一种双向同源比对的非模式物种细胞注释方法与流程
1.本发明属于细胞注释技术领域,具体涉及一种双向同源比对的非模式物种细胞注释方法。
背景技术:
2.单细胞转录组的细胞注释分析是很依赖数据库的分析内容,前者是赋予数据生物学意义的重要基础,单细胞转录组通过对每个细胞的mrna添加识别标签,从而实现将reads归属到其来源细胞,获取单细胞的基因表达丰度。但是,在这个过程中,我们仅能得到细胞的基因表达量,而无法知道这个细胞属于什么细胞类型,为了破除这种隔离,常用的方法是通过细胞表达细胞类型相关标记基因的情况来赋予细胞具有生物学意义的细胞类型标签,以便在后续分析中基于细胞类型讨论样本的分子特性。然而,非模式物种缺乏系统的标记基因研究,标记基因获取难度大,从而缺乏细胞表达细胞类型相关标记基因的情况的研究,因而,成为非模式物种细胞注释的主要阻碍。
3.标记基因作为细胞类型识别的重要指标,在许多文章都被证明了其具有保守性,例如,cell杂志上报道了狨猴与人、小鼠视网膜标记基因的同源性(peng y r,shekhar k,yan w,et al.molecular classification and comparative taxonomics of foveal and peripheral cells in primate retina[j].cell,2019,176(5):1222-1237.e22.),也报道了肝细胞的标记基因在人、小鼠、猕猴、猪、仓鼠、鸡、斑马鱼的同源性(guilliams m,bonnardel j,haest b,et al.spatial proteogenomics reveals distinct and evolutionarily conserved hepatic macrophage niches[j].cell,2022,185(2):379-396.e38.),science杂志上报道了屏状核的标记基因在人、小鼠、海龟、鬃狮蜥的同源性(norimoto h,fenk l a,li h h,et al.a claustrum in reptiles and its role in slow-wave sleep[j].science,2020,578(7795):413-418.)。
[0004]
以上报道提示了通过同源比对,借鉴成熟的模式物种的标记基因数据库,可以摆脱非模式物种目前缺乏系统的标记基因研究,对于细胞类型的标记基因在单细胞数据中的分布情况相关信息的依赖,有利于实现非模式物种单细胞研究从0到1的突破,但是,现有使用的同源比对方法都是单向比对,即将待分析物种的基因比对到模式物种的基因(例如小鼠),这种比对方法容易产生一个基因存在多个同源基因或假阳性同源基因的情况,对数据分析结果产生干扰,从而影响结果的正确性。
技术实现要素:
[0005]
为了克服现有技术中的上述不足,本发明的目的在于提供一种双向同源比对的非模式物种细胞注释方法,可以精确对非模式物种的基因进行同源比对,得到与模式物种同源的基因,利用与模式物种数据库同源的基因来完成相关分析,实现对非模式物种的细胞注释。
[0006]
为了实现上述目的,本发明采用以下技术方案:
[0007]
本发明提供一种双向同源比对的非模式物种细胞注释方法,包括以下步骤:
[0008]
s1、对非模式物种进行单细胞转录组测序,得到单细胞转录组测序的数据;
[0009]
s2、将非模式物种的基因组和模式物种的基因组进行比对,保留唯一比对上的基因对;
[0010]
s3、将模式物种的基因组和非模式物种的基因组进行比对,保留唯一比对上的基因对;
[0011]
s4、将所述s2中保留的唯一比对上的基因对和所述s3中保留的唯一比对上的基因对进行比较,保留相同的基因对,作为双向同源比对的基因对;
[0012]
s5、在模式物种的标记基因数据库中,将所述双向同源比对的基因对中的非模式物种的基因替代所述双向同源比对的基因对中与非模式物种同源的基因,得到非模式物种的表达量矩阵;
[0013]
s6、通过软件调用所述非模式物种的表达量矩阵的基因数据进行注释,得到非模式物种细胞类型的注释结果。
[0014]
进一步地,所述s2和所述s3中,采用blast方法将模式物种的基因组和非模式物种的基因组进行比对。
[0015]
进一步地,所述s5中,所述模式物种的标记基因数据库中包括模式物种的表达量矩阵,在所述模式物种的表达量矩阵中,将双向同源比对的基因对的模式物种的基因id替换为所述双向同源比对的基因对中与模式物种同源的基因,得到所述非模式物种的表达量矩阵。
[0016]
进一步地,采用singler软件调用所述非模式物种的表达量矩阵的基因数据进行注释,得到所述非模式物种细胞类型的注释结果。
[0017]
进一步地,在得到所述非模式物种细胞类型的注释结果后,利用模式物种的配体-受体数据库,根据得到的非模式物种细胞类型的注释结果,可以基于注释的所述非模式物种细胞类型对非模式物种细胞间进行细胞通讯分析。
[0018]
进一步地,在得到所述非模式物种细胞类型的注释结果后,将所述非模式物种细胞类型的注释结果按照细胞类型进行统计,通过表格对非模式物种细胞类型的分布进行可视化。
[0019]
进一步地,所述模式物种的标记基因数据库包括人的标记基因数据库和小鼠的标记基因数据库。
[0020]
与现有技术相比,本发明提供的一种双向同源比对的非模式物种细胞注释方法具有的有益效果如下:
[0021]
可以精确对非模式物种的基因进行同源比对,得到与模式物种同源的基因,保证了后续数据分析结果的准确性,根据得到的与模式物种同源的基因,采用软件调用模式物种的标记基因数据库的基因数据进行注释,实现对非模式物种的细胞类型注释,一方面完善了非模式物种的细胞类型解析,从而建立属于非模式物种细胞本身的标记基因体系,另一方面将模式物种的配体-受体数据库应用到非模式物种的细胞通讯分析中,使非模式物种的细胞通讯分析成为可能,实现从0到1的突破。
附图说明
[0022]
为了更清楚地说明本技术实施例中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本技术的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
[0023]
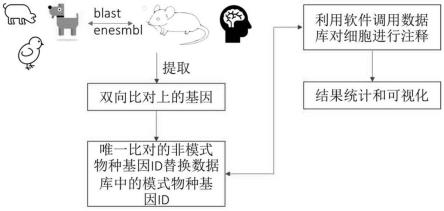
图1是实施例1提供的双向同源比对的非模式物种细胞注释方法的流程示意图。
[0024]
图2是实施例2提供的大鼠的细胞人工注释的t-sne图。
[0025]
图3是实施例2提供的大鼠的基因组blast双向同源比对到小鼠的t-sne图。
[0026]
图4是实施例2提供的大鼠的基因组ensembl双向同源比对到小鼠的t-sne图。
[0027]
图5是实施例3提供的大鼠的基因组blast双向同源比对到人的t-sne图。
[0028]
图6是实施例3提供的大鼠的基因组ensembl双向同源比对到人的t-sne图。
[0029]
图7是根据文章中提供的数据采用本发明提供的双向同源比对的非模式物种细胞注释方法进行细胞类型注释得到的umap图。
[0030]
图8是文章中得到的有关大鼠比对人的原始umap图。
[0031]
图9是实施例5采用双向同源比对的非模式物种细胞注释方法得到的大鼠比对人的umap图。
[0032]
图10是实施例6采用双向同源比对的非模式物种细胞注释方法得到的大鼠比对小鼠的umap图。
[0033]
图11是反映实施例6和文章得到的细胞类型注释结果的关系的桑基图。
具体实施方式
[0034]
为了使本技术要解决的技术问题、技术方案及有益效果更加清楚明白,以下将结合实施例对本技术的技术方案进行清楚、完整的描述。应当理解此处所描述的具体实施例仅仅用以解释本技术,并不用于限定本技术。
[0035]
实施例1
[0036]
请参阅附图1,本实施例提供的一种双向同源比对的非模式物种细胞注释方法包括以下步骤:
[0037]
s1、对非模式物种进行单细胞转录组测序,得到单细胞转录组测序的数据;
[0038]
s2、将非模式物种的基因组和模式物种的基因组进行比对,保留唯一比对上的基因对;
[0039]
s3、将模式物种的基因组和非模式物种的基因组进行比对,保留唯一比对上的基因对;
[0040]
s4、将s2中保留的唯一比对上的基因对和s3中保留的唯一比对上的基因对进行比较,保留相同的基因对,作为双向同源比对的基因对;
[0041]
可理解地,将模式物种的基因组和非模式物种的基因组进行双向同源比对得到的双向同源比对的基因对很准确,不会出现现有使用的同源比对方法采用单向比对容易产生一个基因存在多个同源基因或假阳性同源基因的情况,保证了后续数据分析结果的准确性。
[0042]
具体的,在本发明实施例的一种实施方式中,当非模式物种有ensembl id号时采
用ensembl方法将模式物种的基因组和非模式物种的基因组进行双向同源比对。
[0043]
在本发明实施例的一种实施方式中,直接将采用blast方法将模式物种的基因组和非模式物种的基因组进行双向同源比对。
[0044]
s5、在模式物种的标记基因数据库中,将双向同源比对的基因对中的非模式物种的基因替代双向同源比对的基因对中与非模式物种同源的基因,得到非模式物种的表达量矩阵;
[0045]
具体的,模式物种的标记基因数据库中包括模式物种的表达量矩阵,在模式物种的表达量矩阵中,将双向同源比对的基因对的模式物种的基因id替换为双向同源比对的基因对中与模式物种同源的基因,得到非模式物种的表达量矩阵。
[0046]
s6、通过软件调用非模式物种的表达量矩阵的基因数据进行注释,得到非模式物种细胞类型的注释结果。
[0047]
具体的,采用singler软件调用非模式物种的表达量矩阵的基因数据进行注释,得到非模式物种细胞类型的注释结果。
[0048]
需要说明的是,在得到非模式物种细胞类型的注释结果后,利用模式物种的配体-受体数据库,根据得到的非模式物种细胞类型的注释结果,可以基于注释的非模式物种细胞类型对非模式物种细胞间进行细胞通讯分析。
[0049]
另外,在得到非模式物种细胞类型的注释结果后,也可以将非模式物种细胞类型的注释结果按照细胞类型进行统计,通过表格对非模式物种细胞类型的分布进行可视化。
[0050]
本发明的方法可以精确对非模式物种的基因进行同源比对,得到与模式物种同源的基因,保证了后续数据分析结果的准确性,根据得到的与模式物种同源的基因,采用软件调用模式物种的标记基因数据库的基因数据进行注释,实现对非模式物种的细胞类型注释,一方面完善了非模式物种的细胞类型解析,从而建立属于非模式物种细胞本身的标记基因体系,另一方面将模式物种的配体-受体数据库应用到非模式物种的细胞通讯分析中,使非模式物种的细胞通讯分析成为可能,实现从0到1的突破。
[0051]
实施例2
[0052]
本实施例根据实施例1提供的一种双向同源比对的非模式物种细胞注释方法,将大鼠的基因组双向同源比对到小鼠的基因组后,得到大鼠的细胞注释结果,具体是,图2是大鼠的细胞人工注释的t-sne图;图3是大鼠的基因组blast双向同源比对到小鼠的t-sne图;由于大鼠的基因有ensembl id号,图4是大鼠的基因组ensembl双向同源比对到小鼠的t-sne图,将大鼠的细胞类型的注释结果按照细胞类型进行统计,通过表格对大鼠的细胞类型的分布进行可视化,结果如表1所述,需要说明的是人工注释是通过人主观观察标记基因的分布来判定细胞类型,从而对非模式物种细胞进行注释,是现阶段细胞注释结果可信度的基准线。
[0053]
表1大鼠的基因组双向同源比对到小鼠的大鼠的细胞注释结果统计
[0054][0055]
结合图2、图3和图4,从表1的大鼠的基因组双向同源比对到小鼠的细胞注释结果统计可以看出,由于大鼠和小鼠是近源关系,人工注释和通过双向同源比对得到的细胞类型注释的结果近乎相同,没有区别,双向同源比对得到的细胞类型注释结果仅比人工注释结果多1种细胞类型;采用ensembl的同源比对方式比对的细胞数量要比blast同源比对方式比对的细胞数量多,但是最终通过同源比对方式得到的细胞类型注释的结果近乎相同,没有区别,说明本发明通过双向同源比对对非模式物种细胞进行注释的统计结果也是可信的。
[0056]
实施例3
[0057]
本实施例根据实施例1提供的一种双向同源比对的非模式物种细胞注释方法,将大鼠的基因组双向同源比对到人的基因组后,得到大鼠的单细胞注释结果,具体是,图2是大鼠的细胞人工注释的t-sne图;图5是大鼠的基因组blast双向同源比对到人的t-sne图;由于大鼠的基因有ensembl id号,图6是大鼠的基因组ensembl双向同源比对到人的t-sne图,将大鼠的细胞类型的注释结果按照细胞类型进行统计,通过表格对大鼠的细胞类型的分布进行可视化,结果如表2所示。
[0058]
表2大鼠的基因组双向同源比对到人的大鼠的细胞注释结果统计
[0059]
omnibus,geo)中公开的gse号为gse165153的有关大鼠单细胞转录组原始测序数据,采用本发明提供的双向同源比对的非模式物种细胞注释方法进行细胞类型注释,得到的umap图如附图7所示,而文章中得到的有关大鼠比对人的原始umap图如附图8所示,本实施例得到的umap图虽然和文章的不一样,从细胞类型注释结果来看,根据文章的说明内容,图7中的myeloid1对应图8的macro,图7中的myeloid2对应图8的cam,图7中的myeloid3对应图8的mono,本发明提供的双向同源比对的非模式物种细胞注释方法得到的细胞类型注释结果和文章一致,说明本发明提供的双向同源比对的非模式物种细胞注释方法的注释结果准确。需要说明的是本实施例得到的umap图虽然和文章的不一样,原因是在于降维过程中,将高维数据降维到低维空间时,umap会在低维空间选择随机的若干个点作为起始位点,然后以这些点的坐标为基础计算其它高维空间点对应的低维坐标,正是这种随机性的存在,对于同一份数据,在进行两次降维时,计算得到的坐标值就会有所不同,虽然本实施例在同一台计算机上会设定随机数种子,保证两次降维使用固定的起始随机数,从而保证两次降维图形一样,但是文章的数据在分析时和本实施例使用的是不同的计算机,随机数会发生变化,从而导致本实施例得到的umap图虽然和文章的不一样,但是本实施例得到的细胞类型的注释结果是和文章一致的。
[0064]
实施例5
[0065]
图9是本实施例采用的双向同源比对的非模式物种细胞注释方法得到的大鼠比对人的umap图,结合附图8有关大鼠比对人的umap图,从图中看出本实施例得到的umap图和文章得到的umap图虽然有差异,但实际上这两幅图差异并不大,只是文章和本实施例采用的是不同的标记基因数据库,文章中使用的是gsm号为gsm2967055名为tabula muris的骨髓单细胞转录组测序数据,图8的bc1和bc2合并为图9的b cells,图8的mdc1和pdc合并为图9的dendritic cells,图8的myeloid1/2/3、granulo和micro合并为图9的monocytes,其中,图8的myeloid1/2/3、granulo和micro都是髓系细胞,但是本实施例的人的标记基因数据库中的髓系只有monocytes,图9的cd4 tc更名为cd4+t cells,图9的cd8 tc更名为cd8+t cells,图9的nk被拆分为nk cells和nk t cells,因此本实施例得到的umap图和文章得到的umap图一致。
[0066]
实施例6
[0067]
图10是本实施例采用双向同源比对的非模式物种细胞注释方法得到的大鼠比对小鼠的umap图,图11是桑基图,反映本实施例和文章的注释方法得到的细胞类型注释结果的关系,从图中可以看出,将大鼠的基因组分别比对到人和小鼠的数据库得到的细胞类型注释结果受到标记基因数据库类型的影响,但是在准确性上实际上并没有差异,正是因为数据库之间存在差异,在实际对非模式物种进行细胞类型注释时,需要调用符合非模式物种的标记基因数据库。
[0068]
综上所述,实施例2和实施例3中将非模式物种进行比对注释的可信度还是比较高的,但还是取决于与模式物种的近缘程度;采用ensembl的同源比对方式比对的细胞数量要比blast同源比对方式比对的细胞数量多,但是最终通过同源比对方式得到的细胞类型注释的结果差异很小,具体应用取决于研究物种是否有ensembl id号,如果有,则可以推荐使用ensembl比对方法,比对覆盖率高,运算速度快;如果没有,则使用blast比对方法,适用范围广,灵活度高。实施例4和实施例5中,将大鼠的基因组分别和人和小鼠的基因组双向同源
比对得到的细胞类型注释结果都比较准确,和文章中得到的细胞类型注释结果一致,说明本发明提供的双向同源比对的非模式物种细胞注释方法可以适用于这人和小鼠的标记基因数据库,从而提高了在实际应用中的适用范围。
[0069]
上述实施方式仅为本发明的优选实施方式,不能以此来限定本发明保护的范围,本领域的技术人员在本发明的基础上所做的任何非实质性的变化及替换均属于本发明所要求保护的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1