一种过滤转录组测序数据中rRNA序列的方法和装置与流程
一种过滤转录组测序数据中rrna序列的方法和装置
技术领域
1.本发明属于转录组测序数据处理技术领域,具体地,涉及一种过滤转录组测序数据中rrna序列的方法和装置。
背景技术:
2.转录组是某一物种在特定组织或细胞在某一发育阶段或功能状态下转录出来的所有rna的总和,主要包括mrna和非编码rna(non-coding rna,ncrna)。转录组研究是基因功能及结构研究的基础和出发点,通过高通量测序,能够全面快速地获得几乎所有转录组序列信息,从而能够从整体水平研究基因功能以及基因结构,揭示特定的生物学过程,已广泛应用于植物候选基因发掘、功能鉴定及遗传改良等领域。
3.目前,rna测序(rna sequenclng,rna-seq)技术已成为了转录组学研究的重要手段之一。其成本低、限制少、准确性高。通常会根据测序对象长度的不同,在测序建库的时候选择不同的片段大小,因此测序读长也会有所不同。一般来讲,如果要进行microrna测序的话,通常将microrna分离出来,单独进行测序即可。如果要对mrna测序,通常建库时选择200-300bp大小片段,采用125pe/150pe测序。由于长链非编码rna(lncrna)存在正向转录和反向转录,所以常采用链特异性建库测序。
4.有参转录组数据测序下机后原始数据(raw data)是一些150bp左右的短序列(也叫reads),这些短序列不能直接用于数据分析。为了确保准确可信的分析结果,需要对原始数据进行预处理,包括去除测序接头(建库过程中引入)和低质量的测序数据(由于测序仪本身的误差造成的)。得到的有效数据(valid data)用于和物种的参考基因组进行比对。
5.以上是转录组分析中常用流程,但是在原核生物中,mrna只占全部rna的1~5%,其余绝大部分是核糖体rna(rrna),因此若要测序mrna,首先必须先将mrna纯化出来,然而,原核生物的mrna并不像真核生物mrna一样具有polya的结构,因此,无法直接利用oligot将mrna纯化出来。如果直接用总rna(total rna)进行测序,那么测序的效率一定会非常差,因为测序得到的序列与参考基因组比对时会有非常大比例比对上核糖体、支原体或线粒体,严重影响比对效率。因此,在与参考基因组比对分析之前,需要先去除核糖体、支原体以及线粒体的序列,获得纯净的mrna序列进行下一步分析。
6.目前,本领域通常采取的措施是构建一个所有物种的核糖体序列库,在分析不同物种的有参转录组种都使用该核糖体库进行过滤。然而,现有的数据库存在以下的弊端:
7.1.由于数据库包含非常多的物种,但是仍然不能保证所有核糖体序列都包含在内;
8.2.由于数据库选择了所有物种的核糖体序列,在比对过程中会因序列相似性过滤掉不属于核糖体序列的序列。
9.因此,本领域亟需一种快速准确过滤转录组测序数据中核糖体序列。
技术实现要素:
10.为了解决上述技术问题,本发明采用的技术方案如下:
11.本发明第一方面提供一种建立物种rrna序列数据库的方法,包括以下步骤:
12.s1,获得物种的第一转录组测序数据及参考基因组数据;
13.s2,将转录组数据与参考基因组数据进行比对,获得未比对序列,
14.s3,选取未对比序列的前m条,与ncbi数据库进行比对,其中m=8000~20000;
15.s4,获得比对上的rrna序列,建立rrna序列数据库。
16.在本发明的一些实施方案中,步骤s1中获得物种的转录组数据后进一步包括对所述转录组数据进行预处理的步骤:(1)过滤接头reads;(2)过滤含有n(未知碱基)的比例大于5%的reads;(3)过滤质量值q≤10的碱基数占整个read的20%以上的reads。
17.进一步地,所述第一转录组测序数据转录组建库时采用链特异性建库,转录组测序可以保留转录组测序时转录本的方向信息,因此还可以确定转录本是来源于基因组的正链或是负链。
18.在本发明的一些实施方案中,步骤s2中,利用hisat2比对软件进行比对。
19.在本发明的一些实施方案中,步骤s3中,所述与ncbi数据库进行比对是指将所述未对比序列上传至ncbi进行比对。在本发明的另一些实施方案中,本领域技术人员也可以将ncbi数据库下载到本地进行比对,以提高比对效率。当然,本领域技术人员也可以与其他数据库进行比对,以丰富能够比对上的rrna序列。在本发明的一些具体实施方案中,所述与ncbi数据库中比对是指与ncbi数据库中的nt数据库进行比对。
20.在本发明中,所述m也可以根据实际的序列数目进行合理选择。
21.进一步地,步骤s4中,所述比对上是指比对e value小于预设阈值。在本发明的一些实施方案中,所述预设阈值设置为1e-5,即ncbi比对结果中e value小于1e-5即认为比对上。
22.本发明第二方面提供利用本发明第一方面任一所述方法基于物种的第一转录组测序数据建立的rrna序列数据库。
23.进一步地,本领域技术人员也可以利用该物种的多个转录组测序数据通过上述同样的方法获得更加完善的rrna序列数据库。
24.本发明的第三方面提供一种过滤物种的第二转录组测序数据中rrna序列的方法,包括以下步骤:
25.将所述第二转录组测序数据与本发明第二方面所述的rrna序列数据库进行比对,过滤掉能够比对上的序列。
26.本发明针对一个物种建立rrna序列数据库的目的是为了对该物种的其他转录组数据实现快速过滤或者去除rrna序列。因此,此处的第二转录组测序数据是指待去除rrna序列的任意该物种的转录组数据,当然,也包括用于建立所述rrna序列数据库的第一转录组测序数据本身。由于仅利用未与参考基因组比对上的序列的一部分进行比对,因此,也能够大大提高过滤效率。
27.在本发明的一个实施方案中,利用bowtie2利用比对,比对参数设置如下:
[0028]-i最小fragment长度使用默认参数0;
[0029]-x最大fragment长度使用默认参数500;
[0030]
选择
–
loca模式下的
‑‑
sensitive-local参数即-d 15-r 2-n 0-l 20-i s,1,0.75。具体参数说明:-d 15比对时将一个种子延长后得到比对结果,如果不产生更好的或次好的比对结果,则该次比对失败,当失败次数连续达到15次后,则该条read比对结束;-r 2如果一个read所生成的种子在参考序列上匹配位点过多,当每个种子平均匹配超过300个位置,则通过一个不同的偏移来重新生成种子进行比对;重新生成种子的次数;-n 0进行种子比对时允许的mismatch数为0;-l 20设定种子的长度为20;-i设定两个相邻种子间所间距的碱基数,该参数是公式计算得到,此参数三部分组成:(a)计算方法,包括常数(c)、线性(l)、平方根(s)和自然对数(g);(b)一个常数;(c)一个系数。s,1,0.75即为f(x)=1+0.75*。
[0031]-loca模式是对read进行局部比对,read两端的一些碱基不比对,从而使比对得分满足要求。该模式下得分罚分参数
–
ma默认为2。
[0032]
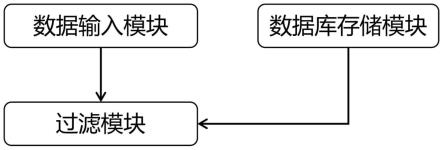
本发明的第四方面提供一种过滤物种的第二转录组测序数据中rrna序列的装置,包括:
[0033]
数据输入模块,用于获得所述第二转录组测序数据;
[0034]
数据库存储模块,用于存储本发明第二方面所述的rrna序列数据库;
[0035]
过滤模块,分别与所述数据输入模块和所述数据库存储模块连接,用于将所述第二转录组测序数据与所述rrna序列数据库进行比对,过滤掉能够比对上的序列并输出。
[0036]
本发明第五方面提供一种计算机设备,包括:
[0037]
存储器,用于存储计算机程序;
[0038]
处理器,用于执行所述计算机程序时实现如本发明第三方面任一所述方法的步骤。
[0039]
本发明第六方面提供一种计算机可读存储介质,所述计算机可读存储介质上存储有计算机程序,所述计算机程序被处理器执行时实现如本发明第三方面任一所述方法的步骤。
[0040]
本发明的有益效果
[0041]
相对于现有技术,本发明具有以下有益效果:
[0042]
本发明首先提供一种可以快速物种名构建该物种的rrna序列数据库,然后转录组测序数据经过预处理后对使用此rrna序列数据库进行针对性地过滤rrna序列。能够快速、准确地获得纯净的mrna数据,进行后续分析。
附图说明
[0043]
图1示出了本发明实施例2小鼠物种中未与参考基因组比对上的部分reads与ncbi数据库比对的结果。
[0044]
图2示出了利用本发明构建的rrna序列数据库进行过滤后的结果。
[0045]
图3示出了本发明实施例3番鸭物种中未与参考基因组比对上的部分reads与ncbi数据库比对的结果。
[0046]
图4示出了利用本发明实施例3构建的rrna序列数据库进行过滤后的结果。
[0047]
图5示出了本发明实施例的过滤转录组测序数据中rrna序列的装置示意图。
具体实施方式
[0048]
除非另有说明、从上下文暗示或属于现有技术的惯例,否则本技术中所有的份数和百分比都基于重量,且所用的测试和表征方法都是与本技术的提交日期同步的。在适用的情况下,本技术中涉及的任何专利、专利申请或公开的内容全部结合于此作为参考,且其等价的同族专利也引入作为参考,特别这些文献所披露的关于本领域中的定义。如果现有技术中披露的具体术语的定义与本技术中提供的任何定义不一致,则以本技术中提供的术语定义为准。
[0049]
本技术中的数字范围是近似值,因此除非另有说明,否则其可包括范围以外的数值。数值范围包括以1个单位增加的从下限值到上限值的所有数值,条件是在任意较低值与任意较高值之间存在至少2个单位的间隔。这些仅仅是想要表达的内容的具体示例,并且所列举的最低值与最高值之间的数值的所有可能的组合都被认为清楚记载在本技术中。
[0050]
为了使本发明所解决的技术问题、技术方案及有益效果更加清楚明白,以下结合实施例,对本发明进行进一步详细说明。
[0051]
实施例
[0052]
以下例子在此用于示范本发明的优选实施方案。本领域内的技术人员会明白,下述例子中披露的技术代表发明人发现的可以用于实施本发明的技术,因此可以视为实施本发明的优选方案。但是本领域内的技术人员根据本说明书应该明白,这里所公开的特定实施例可以做很多修改,仍然能得到相同的或者类似的结果,而非背离本发明的精神或范围。
[0053]
除非另有定义,所有在此使用的技术和科学的术语,和本发明所属领域内的技术人员所通常理解的意思相同,在此公开引用及他们引用的材料都将以引用的方式被并入。
[0054]
那些本领域内的技术人员将意识到或者通过常规试验就能了解许多这里所描述的发明的特定实施方案的许多等同技术。这些等同将被包含在权利要求书中。
[0055]
下述实施例中的他实验方法,如无特殊说明,均为常规方法。下述实施例中所用的仪器设备,如无特殊说明,均为实验室常规仪器设备;下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。
[0056]
实施例1来源物种的参考基因组来源于ensembl的转录组测序数据核糖体序列过滤
[0057]
ensembl是由英国sanger研究所wellcome基金会(wtsi)和欧洲分子生物学实验室所属分部欧洲生物信息学研究所(embi-ebi)共同协作运营的一个项目。ensembl项目得到的数据可以用于支持基因组的比较基因组、进化、序列突变和转录调控方面研究。
[0058]
ensembl数据库中包含了多类物种的rrna数据库,可以直接下载后进行比对,如能对比上,则进行过滤。
[0059]
发明人建立了针对转录组数据来源物种的下载方法,若来源物种是植物,下载ftp://ftp.ensemblgenomes.org/pub/release-版本/plants/fasta/物种/ncrna/*ncrna.fa.gz,若来源物种是真菌,下载ftp://ftp.ensemblgenomes.org/pub/release-版本/fungi/fasta/物种/ncrna/*ncrna.fa.gz,若来源物种是动物,下载ftp://ftp.ensembl.org/pub/release-版本/fasta/物种/ncrna/*ncrna.fa.gz。当然,若网址发生更新,下载地址也可以进行相应更新。
[0060]
若转录组测序数据的来源物种的参考基因组来源于ensembl数据库,则直接利用
下载的rrna数据库进行比对过滤即可。但是,对于来源物种的参考基因组非来源于ensembl数据库的转录组测序数据,则需要建立新的方法进行rrna序列过滤。
[0061]
实施例2来源物种的参考基因组非来源于ensembl的小鼠转录组测序数据核糖体序列过滤
[0062]
本实施例提供来源物种的参考基因组非来源于ensembl的小鼠转录组测序数据过滤rrna序列的方法。
[0063]
具体地,获得小鼠的转录组数据(利用链特异性建库方法得到),该物种参考基因组来源于ncbi数据库。
[0064]
1.转录组测序数据预处理
[0065]
(1)过滤接头(adaptor)的reads;
[0066]
(2)过滤含有n(n表示无法确定碱基信息)的比例大于5%的reads;
[0067]
(3)过滤低质量(质量值q≤10的碱基数占整个read的20%以上)的reads。
[0068]
预处理后得到高质量转录组测序数据(clean data)。
[0069]
2.第一比对
[0070]
用hisat2比对软件将clean data与参考基因组进行比对。得到比对的结果和未对比上序列的文件(unmapped.bam文件)。使用bamtofastqs软件将bam文件转换为fastq文件(unmapped.fastq)。
[0071]
3.提取
[0072]
转换后的fastq文件中有些reads的丰度很高,一些reads的丰度比较低,这些低丰度的reads占总reads比例比较低。为此,发明人在对unmapped.fastq进行一个丰度统计,并进行排序,得到丰度排序后的unmapped.uniq.fa文件。转换后的unmapped.uniq.fa文件中,丰度高的reads排在前面。为了提高比对效率,发明人提取丰度前10000的reads,进行后续比对。
[0073]
4.第二比对
[0074]
将包含10000条reads的fasta文件上传到ncbi进行blast比对,结果如图1所示。获得比对结果后筛选比对上rrna的条目,选择下载全部rrna的fasta文件,建立rrna数据库。
[0075]
5.过滤
[0076]
利用步骤4建立的rrna数据库对cleandata进行双端rrna序列过滤,具体地,使用比对软件bowtie2的2.2.0版本将clean data与rrna数据库进行比对,比对方式选择fr,比对参数设置如下:
[0077]-i最小fragment长度使用默认参数0;
[0078]-x最大fragment长度使用默认参数500;
[0079]
选择
–
loca模式下的
‑‑
sensitive-local参数即-d 15-r 2-n 0-l 20-i s,1,0.75。具体参数说明:-d 15比对时将一个种子延长后得到比对结果,如果不产生更好的或次好的比对结果,则该次比对失败,当失败次数连续达到15次后,则该条read比对结束;-r 2如果一个read所生成的种子在参考序列上匹配位点过多,当每个种子平均匹配超过300个位置,则通过一个不同的偏移来重新生成种子进行比对;重新生成种子的次数;-n 0进行种子比对时允许的mismatch数为0;-l 20设定种子的长度为20;-i设定两个相邻种子间所间距的碱基数,该参数是公式计算得到,此参数三部分组成:(a)计算方法,包括常数(c)、线性
(l)、平方根(s)和自然对数(g);(b)一个常数;(c)一个系数。s,1,0.75即为f(x)=1+0.75*。
[0080]-loca模式是对read进行局部比对,read两端的一些碱基不比对,从而使比对得分满足要求。该模式下得分罚分参数
–
ma默认为2。
[0081]
通过bowtie2比对得到过滤rrna序列后的双端fastq数据,与参考基因组比对结果如图2所示,过滤后即可进行后续的转录组分析。去除rrna前后转录组分析耗时如表1所示:
[0082]
表1 去除rrna前后小鼠转录组分析耗时
[0083]
去除rrna前样本cleandata转录组比对分析耗时5小时去除rrna后样本cleandata转录组比对分析耗时4.5小时
[0084]
由此可见,利用本实施例方法去除rrna后,小鼠转录组比对分析耗时缩短了10%,效果显著。
[0085]
另外,由于转录组建库时采用链特异性建库(fr-firstrand),转录组测序可以保留转录组测序时转录本的方向信息,因此还可以确定转录本是来源于基因组的正链或是负链。
[0086]
实施例3来源物种的参考基因组非来源于ensembl的番鸭转录组测序数据核糖体序列过滤
[0087]
利用实施例2的方法对来源物种的参考基因组非来源于ensembl的番鸭转录组测序数据过滤rrna序列的方法。
[0088]
具体地,获得番鸭的转录组数据(利用链特异性建库方法得到),该物种参考基因组来源于ncbi数据库。
[0089]
用hisat2比对软件将clean data与参考基因组进行比对。unmapped.fastq转换unmapped.fasta按照reads丰度排序为将包含reads丰度前10000条reads的fasta文件上传到ncbi进行blast比对,结果如图3所示。获得比对结果后筛选比对上rrna的条目,选择下载全部rrna的fasta文件,建立rrna数据库。
[0090]
通过bowtie2比对得到过滤rrna序列后的双端fastq数据,与参考基因组比对结果如图4所示。过滤后即可进行后续的转录组分析。去除rrna前后转录组分析耗时如表2所示:
[0091]
表2 去除rrna前后番鸭转录组分析耗时
[0092]
去除rrna前样本cleandata转录组比对分析耗时6小时去除rrna后样本cleandata转录组比对分析耗时5小时
[0093]
由此可见,利用本实施例方法去除rrna后,番鸭转录组比对分析耗时缩短了17%左右,效果十分显著。
[0094]
实施例4过滤转录组测序数据中rrna序列的装置
[0095]
基于实施例2的方法,本实施例提供一种过滤转录组测序数据中rrna序列的装置,如图5,包括:
[0096]
数据输入模块,用于获得该物种的转录组测序数据,此处与建立rrna序列数据库使用的转录组测序数据为同一组数据;
[0097]
数据库存储模块,用于存储实施例2建立的rrna序列数据库;
[0098]
过滤模块,分别与数据输入模块和数据库存储模块连接,能够将转录组测序数据与rrna序列数据库进行比对,过滤掉能够比对上的序列并输出。
[0099]
在本发明提及的所有文献都在本技术中引用作为参考,就如同每一篇文献被单独
引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本技术所附权利要求书所限定的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1