一种肺鳞癌筛查用微小RNA的优选方法
一种肺鳞癌筛查用微小rna的优选方法
技术领域
1.本发明属于生物和分子诊断技术领域,具体涉及一种肺鳞癌筛查用微小rna的优选方法。
背景技术:
2.肺癌是发病率和死亡率最高的癌症种类之一,分为非小细胞肺癌和小细胞肺癌,其中约有85%为非小细胞肺癌(nsclc),又以肺鳞癌(lusc)是最常见的非小细胞肺癌类型之一。肺鳞癌又称肺鳞状上皮细胞癌,常见于具有吸烟史的中老龄男性患者,由支气管黏膜柱状上皮细胞化生后形成的,主要发病于中心气道,具有生长缓慢的特点,对筛查具有一定的难度。
3.目前术前纤支镜、剖胸探查和放射成像已用于肺鳞癌的诊断,但这些侵入性诊断技术对患者的依从性较差,且价格昂贵,难以用于广谱人群的肿瘤筛查。开发非侵入性、准确性高的生物标志物和筛查手段,促进肺鳞癌的检测和诊断显得尤为重要。近年来,免疫治疗在临床实践中取得可喜的成果,如肿瘤标志物ca125、肿瘤标志物scc、肿瘤标志物cea等手段,但这些手段存在阳性率低,特异性不高,敏感度对肺鳞癌较低,如何提高非小细胞肺癌患者在免疫治疗的准确性、特异性和敏感度,如何选择最有可能从免疫治疗获益的患者是当前面临的主要挑战。
4.鉴于此,探索和研究肺鳞癌筛查的微小rna,建立相对准确的检测/预测模型,以期提高筛选免疫治疗中获益的肺鳞癌患者的准确性,有助于促进靶向药物的开发和未来肺鳞癌的临床治疗。
技术实现要素:
5.本发明的目的是为了解决上述技术问题,提供一种肺鳞癌筛查用微小rna的优选方法,采用本发明标志物可为肺鳞癌制定治疗方案和预后判断提供新的依据,以提高患者的临床预后。为实现本发明目的所使用的技术方案为:
6.一种肺鳞癌筛查用微小rna的优选方法,包括以下步骤:
7.(1)基于不同的数据库构建不同的肺鳞癌相关微小rna数据集;
8.(2)分别对不同的数据集进行log2(x+1)对数标准化;
9.(3)对标准化后的数据集计算标准化均数差(smd),并对smd建立效应模型获取;
10.(4)根据smd结果选取差异表达微小rna分子(der);
11.(5)对der进行验证其可靠性;
12.(6)对确认的der运用发现队列,并挑选出最优的生物标志物组合;
13.(7)对最优的生物标志物组合进行auc、灵敏度和特异度对比。
14.优选地,在步骤(1)中,所述数据库包括但不限于geo、tcga、oncomine数据库。
15.优选地,在步骤(1)中,所述微小rna数据集在数据库内采取检索后,纳入标准为:a、研究物种是人,b、微小rna微阵列数据集,c、肺鳞癌亚型明确;排除标准为:a、数据集之间
存在重复样本,b、微小rna表达数据不全或不可获取。
16.优选地,在步骤(3)中,所述smd建立效应模型获取,其中,异质性检验所得的i2值不大于50%时,选用固定效应模型;i2值大于50%时,选用随机效应模型。
17.优选地,在步骤(4)中,der须满足以下任一点:(1)smd》0且95%置信区间不包含0;(2)smd《0且95%置信区间不包含0。
18.优选地,在步骤(5)中,对der进行wilcoxon秩和检验进一步验证其可靠性。
19.优选地,在步骤(6)中,对通过确认的der,运用发现队列,在r语言中运用“my.stepwise.glm”算法和广义线性模型对der进行识别划分,并从中的所有组合中挑选出最优的生物标志物组合。
20.优选地,在步骤(7)中,对单个der绘制roc曲线,并将结果与组合生物标志物的曲线下面积(auc)、灵敏度和特异度对比,从而评估组合标志物的优劣;auc、灵敏度和特异度的阈值范围均为0至1。
21.优选地,在步骤(7)中,采用发现队列和验证队列绘制基于最优的生物标志物组合的roc曲线,通过auc评估生物标志物筛查肺鳞癌的准确性。
22.本发明还提供一种肺鳞癌筛查用微小rna模型,通过如以上所述的方法构建得到。
23.综上所述,由于本发明采用了上述技术方案,本发明具有以下技术效果:
24.本发明使用生物信息学分析,发现了检测肺鳞癌相关基因,通过机器学习算法,建立了预测肺鳞癌的组合微小rna(mir-4425与mir-130b-3p),并在较大规模队列中进行了验证,mir-4425与mir-130b-3p分子组合具有一定的筛查肺鳞癌的准确性。
25.通过筛查组合微小rna组合的表达值筛查患者是否有肺鳞癌,并且该方法具有快速简便,筛查准确度高,相对无创,并且成本低的特点。
附图说明
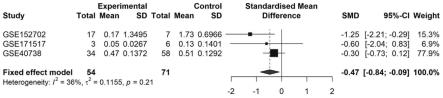
26.图1是对数据集gse171517、数据集gse152702、数据集gse40738的smd计算结果图。
27.图2是在发现队列中计算mir-4425联合mir-130b-3p的auc图。
28.图3是在验证队列a中计算mir-4425联合mir-130b-3p的auc图。
29.图4是在验证队列b中计算mir-4425联合mir-130b-3p的auc图。
具体实施方式
30.为使本发明的目的、技术方案及优点更加清楚明白,以下举出优选实施例,对本发明进一步详细说明。然而,需要说明的是,说明书中列出的许多细节仅仅是为了使读者对本发明的一个或多个方面有一个透彻的理解,即便没有这些特定的细节也可以实现本发明的这些方面。
31.实施例1
32.一种肺鳞癌筛查用微小rna的优选方法,包括以下步骤:
33.步骤1.从公共数据库(包括但不限于geo、tcga、oncomine数据库)中检索肺鳞癌相关的微小rna队列,检索策略:“lungand(squamous or nsclc or[non-small cell])and(mrna or gene)”。
[0034]
步骤2.根据如下标准纳入或排除相关数据集。纳入标准:(1)研究物种是人;(2)微
小rna微阵列数据集;(3)肺鳞癌亚型明确。排除标准如下:(1)数据集之间存在重复样本;(2)微小rna表达数据不全或不可获取。
[0035]
步骤3.下载符合上述标准的数据集,对其进行log2(x+1)对数标准化。在r语言等软件中,运用meta等软件包对标准化后的数据集计算标准化均数差(smd)。smd通过建立效应模型获取,其中,异质性检验所得的i2值不大于50%时,选用固定效应模型;i2值大于50%时,选用随机效应模型。根据smd结果选取差异表达微小rna分子(der)。der须满足以下任一点:(1)smd》0且95%置信区间不包含0;(2)smd《0且95%置信区间不包含0。
[0036]
步骤4.对der进行wilcoxon秩和检验,进一步验证der的可靠性。
[0037]
步骤5.对通过确认的der,运用发现队列,在r语言中运用“my.stepwise.glm”算法和广义线性模型对der进行识别划分,并从中的所有组合中挑选出最优的生物标志物组合。
[0038]
步骤6.生物标志物筛查肺鳞癌的效能对比。对单个der绘制roc曲线,并将结果与组合生物标志物的曲线下面积(auc)、灵敏度和特异度对比,从而评估组合标志物的优劣(横向对比)。auc、灵敏度和特异度的阈值范围均为0至1,其值越大,说明组合生物标志物筛查肺鳞癌患者的准确性越高。
[0039]
步骤7.对发现队列和验证队列绘制基于最优的生物标志物组合的roc曲线,通过auc评估生物标志物筛查肺鳞癌的准确性(纵向对比及验证)。
[0040]
实施例2
[0041]
步骤1:从公共数据库(geo、tcga数据库)中检索肺鳞癌相关的微小rna队列,检索策略:“lung and(squamous or nsclc or[non-small cell])and(mrna or gene)”。
[0042]
步骤2:根据如下标准纳入或排除相关数据集。纳入标准:(1)研究物种是人;(2)微小rna微阵列数据集;(3)肺鳞癌亚型明确。排除标准如下:(1)数据集之间存在重复样本;(2)微小rna表达数据不全或不可获取。
[0043]
步骤3:下载符合纳入和排除标准的共3个数据集(共包含54例肺鳞癌患者和71例正常人),并分别对这些数据集进行log2(x+1)对数标准化。
[0044]
步骤4:汇总各个数据集下人类物种的所有微小rna分子,共获取2062个微小rna。
[0045]
步骤5:对获取的微小rna逐一进行smd计算,以三个数据集的样本量进行划分。先用最少样本的数据集gse171517进行建模,再用稍多样本量的数据集gse152702进行初步验证,最后用最大样本量的数据集gse40738进行最终验证,见图1。图1显示,i2值(36%)小于50%,故采用固定效应模型计算smd。结果显示,固定效应模型提示微小rna let-7b-5p在肺鳞癌病人血液中的表达水平比正常人低(smd《0且95%置信区间不包含0)。
[0046]
步骤6:对2062个微小rna逐一进行smd计算,最终确定16个der,如表1所示。
[0047]
表1肺鳞癌患者和正常人的血液中的差异表达微小rna
[0048][0049]
步骤7:将三个数据集分别划分为发现队列、验证队列a以及验证队列b。基于发现队列对筛选到的16个der进行所有可能的组合,运用“my.stepwise.glm”算法和广义线性模型识别筛选出最优的两分子组合(mir-4425与mir-130b-3p)。
[0050]
步骤8:对发现队列、验证队列a以及验证队列b分别计算上述两分子的auc。结果如表2所示,mir-4425联合mir-130b-3p具有很高的筛查肺鳞癌患者的准确性。两分子联合的生物标志物的准确度高于任一单个der分子。
[0051]
表2肺鳞癌患者和正常人的血液中的差异表达微小rna
[0052][0053]
对发现队列、验证队列a以及验证队列b分别计算mir-4425联合mir-130b-3p的auc,结果如图2-4。图2显示,在发现队列中,两分子组合(mir-4425与mir-130b-3p)具有很高的筛查肺鳞癌的准确性。图3显示,在验证队列a中,两分子组合(mir-4425与mir-130b-3p)具有很高的筛查肺鳞癌的准确性。图4显示,在验证队列b中,两分子组合(mir-4425与mir-130b-3p)具有一定的筛查肺鳞癌的准确性。
[0054]
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以作出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1