一种基于内质网应激特征基因的胃癌预后风险模型和应用
1.本发明属于肿瘤标志物和生物医学检测技术领域,具体涉及一种基于内质网应激特征基因的胃癌预后风险模型和应用。
背景技术:
2.胃癌是全球第三大癌症死亡原因和第五大常见恶性肿瘤,每年约有超过100万例新病例。中国是胃癌高发国家,新增病例和死亡病例分别占全球总数的42.6%和45.0%,年龄标准化5年生存率为27.4%。胃癌样本早期症状隐匿,治疗效果及预后差。多数胃癌样本确诊时已是晚期,导致总体预后不佳,表现为转移、高肿瘤内异质性和化学耐药性。尽管免疫治疗、靶向治疗和转化治疗在胃癌治疗方面迅速发展,但大多数样本的总生存率仍较低。
3.在肿瘤发生发展过程中,肿瘤细胞的高代谢和快速增殖导致肿瘤内部缺血缺氧,使肿瘤细胞进入持续的内质网应激状态。研究表明特定强度的内质网应激可以通过多种机制影响癌症的发生发展,包括促进癌细胞生长和转移、血管生成、免疫逃逸和放化疗耐药性。内质网应激尤其对胃癌的发生、进展和治疗具有重要影响,其可通过与幽门螺杆菌、eb病毒相互作用促进胃癌的进展,还可通过促进胃癌细胞的上皮间质转化导致胃癌细胞迁移和侵袭。
4.生物标志物可以有效的预测癌症样本的预后。近年来,许多研究将基因作为肿瘤发展和预后的生物标志物。目前,组织学诊断和肿瘤-淋巴结-转移(tnm)分期仍是评价胃癌预后的主要方法。由于胃癌的高度异质性和样本的个体差异,即使具有相似的临床和病理特征甚至相同的tnm分期的样本,其预后和治疗效果也具有很大差异。这表明以往的胃癌预后评价指标可能已经扩展到预测样本预后结局和治疗益处的极限。因此,急需鉴定新的生物标志物以辅助完善目前的预后指标,为胃癌的预后评价及个体化治疗提供依据。
技术实现要素:
5.针对胃癌的高度异质性和样本的个体差异缺少准确的预后指标的问题,本发明提供了一种内质网应激的胃癌风险预后评分模型及构建方法和应用。
6.为了达到上述目的,本发明采用了下列技术方案:
7.一种基于内质网应激特征基因的胃癌预后风险模型,内质网应激基因为:nos3、pon1、cxcr4、matn3、anxa5、serpine1;
8.风险评分模型=(0.052
×
nos3表达水平)+(0.137
×
pon1表达水平)+(0.067
×
cxcr4表达水平)+(0.131
×
matn3表达水平)+(0.116
×
anxa5表达水平)+(0.09
×
serpine1表达水平)。
9.一种基于内质网应激特征基因的胃癌预后风险模型的构建方法,包括以下步骤:
10.步骤1,从癌症基因组图谱tcga(http://cancergemome.nih.gov/)中获取rna sequence数据集和临床数据,作为训练集;从geo数据库中(http://www.ncbi.nlm.nih.gov/geo/)获取胃癌样本作为验证集;
11.为了保证数据的可比性,对rna-seqs数据进行每百万转录本(tpm)转化,并通过log2(tpm+1)转化进行数据归一化以便后续分析;
12.步骤2,从genecards数据库(https://www.genecards.org/)中获取内质网应激相关特征基因ers-rgs;使用r软件中的“limma”包进行胃癌组织与癌旁组织之间的差异表达分析,为了纳入更全面的胃癌差异基因,根据p《0.05和|log2(fold change)|》0作为筛选标准;利用r软件中的“survival”包进行单因素cox回归分析,筛选出具有预后价值的内质网应激特征相关的胃癌差异表达基因;
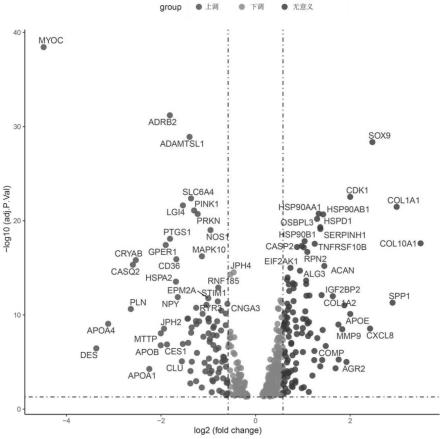
13.步骤4,通过使用“glmnet”r程序包进行最小绝对收缩算子(least absolute shrinkage and selection operator,lasso)回归分析于训练集中构建多基因风险模型;
14.筛选出构建风险评分模型的基因包括:nos3、pon1、cxcr4、matn3、anxa5、serpine1;
15.构建出风险评分模型:
16.风险评分模型=(0.052
×
nos3表达水平)+(0.137
×
pon1表达水平)+(0.067
×
cxcr4表达水平)+(0.131
×
matn3表达水平)+(0.116
×
anxa5表达水平)+(0.09
×
serpine1表达水平);
17.步骤4,通过模型构建基因相应的回归系数,计算每位样本的风险评分通过样本风险评分中位数将胃癌样本分为高-低风险两组;使用geo数据库(http://www.ncbi.nlm.nih.gov/geo/)中的数据(n=387)作为验证集;将成功构建的风险评分模型应用于验证集中进行验证;
18.利用“survminer”程序包对鉴定出的高低风险组进行kaplan-meier(k-m)曲线分析,比较两组样本总生存期(os)差异;使用“timeroc”程序包绘制时间依赖的受试者工作特征曲线(receiver operating characteristic curve,roc),计算胃癌样本在多个时间点的曲线下面积(area under the curve,auc),以评估风险模型预测胃癌样本预后的能力;在验证集中使用相同风险评分公式和临界值验证模型的准确性。
19.根据上述一种基于内质网应激特征基因的胃癌预后风险模型在制备试剂盒中的应用。
20.根据上述试剂盒在胃癌患者总体生存率预测产品中的应用。
21.根据上述试剂盒在胃癌患者总体生存率诊断产品中的应用。
22.根据上述试剂盒在胃癌患者总体生存率辅助诊断产品中的应用。
23.本发明使用内质网应激相关特征基因开发了一种实用性强的胃癌预后风险模型,以预测胃癌患者的总体生存期。本发明分析了来自tcga数据库和geo数据库中胃癌患者的基因表达谱。将从tcga数据库中收集到的数据作为训练集,共纳入了375例胃癌样本和32例癌旁样本。并使用geo数据库中387例胃癌样本进行验证。通过r软件包“limma”筛选出tcga数据库中胃癌组织和癌旁的差异表达基因(deg)。通过genecards数据库,确定出deg中的er应激相关基因。基于训练集中deg数据,我们使用单变量cox回归分析以及lasso回归分析建立出具有6个内质网应激相关特征基因的预后模型,并将胃癌患者分为高、低风险组。通过结合临床特征和风险评分构建列线图,以预测胃癌患者的生存可能性。校准曲线验证了列线图预测与实际观测之间具有良好的一致性。在训练队列中胃癌患者的风险评分与os显著相关(p<0.05)。
24.roc曲线分析显示,在3年、5年和8年随访中,auc分别为0.695、0.786和0.698。同样在验证集中,其3年、5年和8年的auc值分别为0.580、0.625和0.627。预测性能已得到充分验证。由风险模型确定的风险评分经通过独立预后因素分析确定可作为独立于其他临床病理特征的预后因子。基于6个er应激相关基因的风险评分可以很好地将胃癌患者分为高风险、低风险人群,这可能有助于临床治疗方案的选择。
25.与现有技术相比本发明具有以下优点:
26.本发明建立了具有于6个内质网应激特征相关基因的预后模型,并将胃癌样本分为高、低风险组。在训练集与验证集中均显示出良好的预测性能。
附图说明
27.图1为差异表达的内质网应激相关特征基因火山图;
28.图2为通过单变量cox回归分析鉴定与预后显著相关的ers-rgs森林图;
29.图3表示在tcga训练集中基于ers-rgs开发预后模型;图3中(a-b)通过lasso回归分析确定了6个ers-rgs;
30.图4表示在tcga训练集中验证基于ers-rgs开发的预后模型;图4中(a)对特征定义的风险组的生存分析;(b)6个ers-rgs构建预后模型的时间依赖性roc曲线;
31.图5表示在geo验证集中验证基于ers-rgs开发的预后模型;图5中(a)对特征定义的风险组的生存分析;(b)6个ers-rgs构建预后模型的时间依赖性roc曲线;
32.图6为构建生存预测的诺模图;图6中(a)森林图显示对风险评分进行的单因素和多因素独立预后分析结果;(b)结合了风险评分和临床信息特征的列线图;(c)校准曲线显示列线图预测的存活率和实际存活率之间高度一致。
具体实施方式
33.为了能够更清楚地理解本发明的上述目的、特征和优点,下面结合具体实施例对本发明做进一步说明。需要说明的是,在不冲突的情况下,本技术的实施例及实施例中的特征可以相互组合。
34.在下面的描述中阐述了很多具体细节以便于充分理解本发明,但是,本发明还可以采用不同于在此描述的其他方式来实施,因此,本发明并不限于下面公开说明书的具体实施例的限制。
35.实施例1
36.一种基于内质网应激特征基因的胃癌预后风险模型
37.步骤1,数据来源
38.从tcga数据库(http://cancergemome.nih.gov/)中获得375例胃癌和32例癌旁样本的转录组数据及相应临床信息用作训练集样本;从geo数据库中(http://www.ncbi.nlm.nih.gov/geo/)下载387名胃癌样本作为验证集样本(gse84437)。为了保证数据的可比性,对rna-seqs数据进行每百万转录本(tpm)转化,并通过log2(tpm+1)转化进行数据归一化以便后续分析。从genecards数据库(https://www.genecards.org/)中获取ers相关特征基因(ers-rgs)。
39.步骤2,ers-rgs的差异表达分析及功能富集分析
40.使用r软件中的“limma”包进行胃癌组织与癌旁组织之间的差异表达分析。为了纳入更全面的胃癌差异基因,根据p《0.05和|log2(fold change)|》0作为筛选标准。r软件包“clusterprofiler”用来对差异基因进行基因本体功能(gene ontology,go)分析和京都基因与基因组百科全书通路(kyoto encyclopedia of genes and genomes pathway,kegg)分析及可视化,表征胃癌差异基因参与的关键通路并揭示潜在的分子机制。
41.步骤3,预后模型的构建与外部数据集验证
42.利用r软件中的“survival”包进行单因素cox回归分析,筛选出具有预后价值的ers特征相关的胃癌差异表达基因。通过使用“glmnet”程序包进行最小绝对收缩算子(least absolute shrinkage and selection operator,lasso)回归分析构建多基因风险模型。通过模型构建基因相应的回归系数计算每位样本的风险评分(风险评分=∑(回归系数
×
模型构建基因表达量))。将训练集样本风险评分中位数作为cut-off值分为高-低风险两组。成功构建的风险评分模型在外部数据集(geo数据集)中进行验证。如图5所示。
43.步骤4,风险模型的效能评估及独立预后因素分析
44.利用“survminer”程序包对鉴定出的高低风险组进行kaplan-meier(k-m)曲线分析,比较两组样本总生存期(os)差异。通过使用“timeroc”程序包绘制时间依赖的受试者工作特征曲线(receiver operating characteristic curve,roc),计算胃癌样本分别在3年、5年和8年os的曲线下面积(area under the curve,auc),进而评估风险模型预测胃癌样本预后的能力。为了评估该风险模型是否具有评估胃癌样本预后的独立预后价值,对风险评分进行了单因素和多因素独立预后分析。通过“rms”程序包将多因素独立预后分析鉴定出的独立预后因子作为变量,绘制列线图,用以综合判断样本3年、5年和8年的生存率。
45.步骤5,免疫微环境状态及肿瘤突变负荷分析
46.为了评估高低风险组胃癌肿瘤微环境中免疫细胞浸润状况,通过基于反卷积的cibersort算法分析两组间特征免疫细胞浸润丰度。利用颗粒酶a(gzma)和穿孔蛋白1(prf1)的表达量的几何平均值计算细胞溶解活性评分(cytolytic activity,cyt score)。肿瘤突变负荷(tumor mutation burden,tmb)为每百万碱基中被检测出的体细胞基因编码错误、碱基替换、基因插入或缺失错误的总数。通过“maftools”程序包确定胃癌样本的突变状态从而计算tmb。
47.步骤6,统计学方法
48.所有统计分析和可视化均基于r语言(version 4.1.3)及r程序包。p值小于0.05认为有统计学意义。
49.实施例2鉴定胃癌ers相关预后基因并构建cox风险模型
50.基于tcga训练集队列进行胃癌-健康样本差异基因的分析,共获得了5054个显著上调基因与4229个显著下调基因。从genecards数据库共获取了785个ers相关特征基因(ers-rgs)。其中,共444个基因作为胃癌的差异表达基因,包括168个显著下调基因与276个显著上调基因。go和kegg功能富集显示,差异的ers特征基因主要富集在内质网中的蛋白加工、ecm受体相互作用以及未折叠蛋白反应等生物过程(p《0.05)。基于上述444个胃癌差异表达的ers特征基因进行单因素cox回归分析,如图2所示,共筛选出12个与预后显著相关的高风险基因进行lasso回归分析(hr》1),如图3所示,得到最优λ值=6以及每个基因的β回归系数。代入基因得到风险评分模型公式为:risk score=0.052*nos3表达量+0.137*pon1表
达量+0.067*cxcr4表达量+0.131*matn3表达量+0.116*anxa5表达量+0.09*serpine1表达量。根据训练集队列中样本风险评分中位数2.369将样本分为高风险组(n=169)与低风险组(n=168)两组。
51.实施例3风险模型的预后效能评估及验证结果
52.风险曲线可视化结果显示,高风险组胃癌样本死亡比例高于低风险组,提示高风险组样本具有不良预后。nos3、pon1、cxcr4、matn3、anxa5和serpine1均在高风险组中高表达,说明以上六个模型构建基因的高表达与高风险呈正相关。
53.kaplan-meier曲线表明,低风险组在训练集(p《0.0001)与验证集(p=0.0013)中均展现出更高的生存率。时间依赖roc分析结果显示,tcga数据集中3年时间auc值=0.695,5年时间auc值=0.786,8年时间auc值=0.698;验证集中5年时间auc值=0.625,表明该风险模型对胃癌样本预后预测具有良好的敏感性和特异性,尤其对于预测胃癌样本5年总生存率。
54.实施例4风险评分的独立预后因素分析并建立列线图
55.将年龄、性别、肿瘤分级(grade)、肿瘤分期(stage)和风险评分作为变量纳入单因素独立预后因素分析。单因素独立预后分析结果显示风险评分是与胃癌预后显著相关的危险因素(hr=3.601,95%ci:2.300~5.637,p《0.001)。多因素独立预后分析结果显示风险评分是独立于其他临床病理特征的预后因子(hr=3.598,95%ci:2.290~5.655,p《0.001)。为了进一步整合临床信息,实现对胃癌样本多元生存分析,将多因素独立预后分析确定的具有独立预后价值的因子纳入并绘制列线图,用于综合分析胃癌样本3年、5年和8年的生存率。如图6所示。
56.实施例5胃癌高低风险组ers状态与免疫微环境状态评估
57.atf6、hspa5、xbp1和atf4等相关蛋白的细胞内表达水平作为检测细胞或组织中ers强度最常用的指标,通过检测375名胃癌样本中这些标志物的表达水平。特征基因在高风险组中表达水平显著较高(p《0.05),表明高风险组的er应激强度明显高于低风险组(p《0.05)。cibersort结果显示了高低风险组胃癌样本肿瘤微环境中免疫细胞浸润丰度差异,高风险组活化的cd4记忆t细胞丰度显著低于低风险组,而巨噬细胞m0和m2丰度显著高于低风险组(p《0.05)。此外,高风险组常见免疫检查点的表达水平显著高于低风险组(p《0.05)。高风险组样本的细胞溶解活性评分也显著升高(p《0.05)。
58.本发明说明书中未作详细描述的内容属于本领域专业技术人员公知的现有技术。尽管上面对本发明说明性的具体实施方式进行了描述,以便于本技术领域的技术人员理解本发明,但应该清楚,本发明不限于具体实施方式的范围,对本技术领域的普通技术人员来讲,只要各种变化在所附的权利要求限定和确定的本发明的精神和范围内,这些变化是显而易见的,一切利用本发明构思的发明创造均在保护之列。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1