一种基于融合神经网络的LncRNA-疾病关联预测方法
一种基于融合神经网络的lncrna-疾病关联预测方法
技术领域
1.本发明属于lncrna与疾病关联预测技术领域,尤其涉及一种基于融合神经网络的lncrna与疾病关联预测方法。
背景技术:
2.长非编码rna(long non-coding rna,lncrna)是一种非蛋白编码rna,其长度超过200个核苷酸,过去人们普遍认为lncrna对基因表达几乎没有影响。目前,诸多研究表明人类身体各种复杂疾病(如癌症)的形成发展与lncrna的突变和失调的病理机制密切相关。lncrna gman或ephrin a1的调节机制在胃肿瘤转移和发展过程中发挥了重要的调控作用;在癌症进行免疫治疗过程中,命名为limit的癌症免疫原性lncrna可能是治疗靶点;lncrna-hotair在不同的癌细胞中扮演着致癌分子的角色,其表达水平是乳腺癌、胃癌、结直肠癌和宫颈癌等癌症诊断和治疗的潜在生物标志物。因此,识别疾病相关的lncrna将有助于在lncrna水平上了解人类复杂的疾病机制、疾病诊断和治疗。近年来随着生物技术的发展,实验发现的lncrna-疾病关联数据逐渐增加,研究者整理这些数据并建立了lncrnadisease、lnc2cancer、hdmm等lncrna-疾病关联数据库。目前,已知的lncrna-疾病关联仅仅只是小部分,而生物实验验证耗时长且成本昂贵。随着人工智能技术的发展和大数据技术的成熟,研究人员利用计算方法分析和处理已知数据,能够加速发现lncrna和疾病之间的潜在关联,已成为生物实验识别lncrna-疾病关联的有效补充。基于计算的lncrna-疾病关联预测方法分为基于矩阵分解的方法和基于机器学习的方法两类。目前,由于生物数据的完整性、模型选择和实验设计等各个方面仍存在一定的局限性,现有的lncrna-疾病预测方法仍然面临着许多挑战。
技术实现要素:
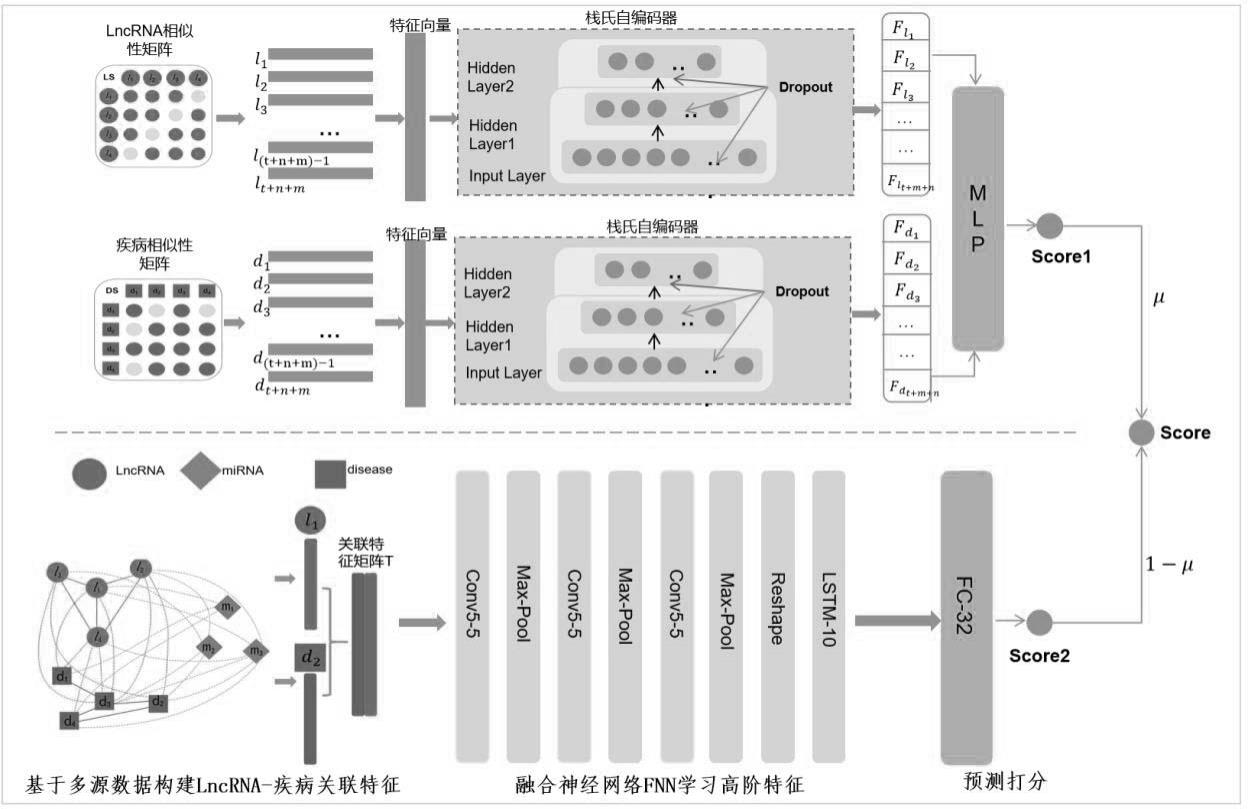
3.本发明目的是提供一种预测精准度更高的关联预测方法,为实现这一目地,本发明基于lncrna与疾病关联数据来源类型多,特征提取不够充分的特点,提出一种基于融合神经网络和栈式自编码器的lncrna与疾病关联预测方法,整体框架见图1所示,本发明技术方案的整体步骤如下:1)整合了lncrna相似性、疾病相似性、lncrna-疾病关联、lncrna-mirna相互作用以及mirna-疾病关联等多源数据构建特征矩阵,并将构建的特征矩阵分别输入到方法的两个模块,其中lncrna 和疾病
ꢀꢀ
关联特征示例见图2所示;2)基于栈氏自编码器sae的特征提取模块学习lncrna-疾病对的低维网络表示,基于融合神经网络fnn的特征提取模块充分利用卷积神经网络(convolutional neural network,cnn)和长短期记忆递归神经网络(long short term memory,lstm)的不同优势,学习lncrna-疾病时间依赖和共享参数的高级特征。两个模块都经过全连接层和softmax层,得到lncrna-疾病的相关可能性即关联得分;3)最后赋予两个模块的预测分值不同的权值,对分值进行加权融合得到预测得
分,从而筛选出疾病相关lncrna。
附图说明
4.图1体系结构图图2 lncrna l
1 和疾病d2关联特征示例图。
具体实施方式
5.本发明的一种基于融合神经网络fnn(fusion neural networks,fnn)和栈式自编码器(stacked autoencoder,sae)的lncrna-疾病关联预测方法,整体框架见图1所示,整体步骤如下:步骤1:整合lncrna功能相似性、疾病语义相似性、lncrna-疾病互作、lncrna-mirna相互作用以及mirna-疾病关联多源数据构建特征矩阵;步骤2:基于sae模块学习lncrna-疾病对的低维网络表示,基于融合神经网络fnn模块进行高阶特征的提取,学习lncrna-疾病时间依赖和共享参数的高级特征;两个模块都经过全连接层和softmax层,得到lncrna-疾病的相关可能性即关联得分;步骤3:赋予两个模块的预测分值不同的权值,对分值进行加权融合得到预测得分,筛选出疾病相关lncrna。
6.根据权利要求1所述lncrna-疾病关联预测方法,从生物学角度构建数据特征,整合来自多个来源的lncrnas、疾病和mirnas的关联、相似性等异质数据,构建lncrna功能相似性特征、疾病语义相似性特征和lncrna-疾病关联特征;lncrna功能相似性特征构建方式如下:在已经验证的生物数据库中发现相似的lncrna可能与相似的疾病有关联,通过计算两种疾病的相似衡量lncrna之间的功能相似度;采用lncrna之间的功能相似度算法计算两种lncrna的功能相似度,构建lncrna功能相似性矩阵ls,ls(i,j)表示lncrna l(i)与lncrna l(j)之间的相似度,其值在0~1之间变化; 越接近于1,表示l(i)和l(j)越相似;ls作为栈氏自编码器特征提取模块的输入;疾病语义相似性特征构建方式如下:根据疾病的dags模型提取疾病之间的语义相似性,完成对疾病的语义相似度的权值定义,构建疾病语义相似度矩阵ds;ds(i,j)表示疾病之间的语义相似度,取值范围为0~1,ds(i,j)越接近于1,表示d(i)和d(j)越相似;ds作为栈氏自编码器特征提取模块的另一个输入;lncrna-疾病关联特征构建方式如下:根据生物信息库中已证实的lncrna-疾病、lncrna-mirna、疾病-mirna之间所存在的相互影响作用,分别构建由lncrna与疾病组成的关系矩阵 、lncrna与mirna组成的关系矩阵 以及疾病与mirna组成的关系矩阵 ,ld、lm和dm取值均为0或1,若lncrna li与mirna mj相互作用,lm
ij
=1,反之 lm
ij
=0;对于lncrna li和疾病 dj,如果与它们相关联的lncrna、疾病以及mirna及其互作关系有较多的相同,那么li和dj的关联性将很大;将lncrna l
i 和所有lncrna的相似关系记作 x1,li与所有疾病的互作关系用向量x
2 表示,li与每个mirna的相互作用用向量x
3 表示;与li类似方法,疾病dj与所有lncrna的互作关系用向量y1表示,疾病dj与各个疾病的语义相似性记为y2,向量y3表示疾病dj与每个mirna的互作关系,lncrna l
1 和疾病d2构建的特征矩阵拼接;属性向量通过公式(1)进行拼接:
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式1从而得到lncrna l
1 和疾病d2的属性关联嵌入矩阵 t,其中,(nl+nd+nm)
ꢀ×
2表示为关联矩阵 t 的维度;该lncrna和疾病的关联嵌入矩阵 t 作为基于融合神经网络fnn的特征提取模块的输入。
7.根据权利要求1所述lncrna-疾病关联预测方法,融合栈式自编码器sae和融合神经网络fnn两个模块构建预测方法,两个模块都经过全连接层和softmax层,得到lncrna-疾病的相关可能性即关联得分;基于栈氏自编码器sae的特征提取模块:栈式自编码器模块由多个自动编码器形成深层神经网络,每层包含一定数量的神经元且每层输出连接到连续层的隐藏单元相同;在该特征提取模块,设置输出维度设为32,迭代次数为100,batchsize参数为128;该模块将lncrna相似性特征矩阵ls和疾病相似性特征矩阵ds作为输入,分别送入由多个连接层组成的两个自编码器进行特征学习,生成高阶特征向量;最后采用多层感知机(multi-layer perceptorn, mlp)网络模型,实现评分预估,将高阶特征向量f馈入到全连接层,表示如公式2所示:
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式2其中,l表示多层感知机的第l层,w
l
、b
l
分别表示l层的权值矩阵和偏置向量;σ
l
为激活函数;用于预测方法收敛的relu函数的如公式3所示:
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式3通过最小化目标函数对模型进行训练,从而达到损失最小化,使用交叉熵代价函数c获得最佳预测,如公式4所示:
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式4其中,x表示训练样本的索引,t表示不同标签的索引,y表示样本的真实标签,a表示样本x模型输出;在最后的多层感知机mlp中获得每种lncrna与疾病之间关联的最终预测,当预测的每个lncrna-疾病样本对超过阈值时,认为该lncrna与潜在的疾病具有相关性;softmax激活函数的功能是将未归一化的输入映射到一组指数化和归一化的概率中,该模块使用softmax函数计算lncrna和疾病的关联概率 score1,关联概率score1计算方法如公式5所示:
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式5其中,w代表参数矩阵,b为偏置向量,z
sae
为输入特征向量;基于融合神经网络fnn的特征提取模块:利用融合神经网络对lncrna和疾病构建的特征矩阵进行特征提取;在特征学习之前,为了充分学习特征边缘的信息,在每次输入前对输入层的周围进行补零操作,执行过程见公式6-12;
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式6
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式7
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式8
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式9
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式10
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式11
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式12其中,f是lncrna和疾病构建的特征矩阵,作为cnn+lstm网络的输入模块,σ(sigmoid)用于模型计算的损失函数,tanh表示激活函数,
⊙
符号用于点乘的计算,w表示权重矩阵,b为偏置向量参数;在cnn卷积神经网络部分,首先使用一个具有5个卷积核的卷积层,然后对数据进行最大池化降维,总共交替使用3个卷积层和池化层,以获得更重要的更深层次的信息;在lstm循环神经网络部分,使用一层10个神经元的双向lstm,最后基于全连接层,用relu激活函数进行馈入,统一维数为32维;构建的特征矩阵经过融合神经网络之后,会经过一个全连接层来获取关联预测的概率分值score2;融合卷积后不同特征图展平合并为一维向量z
fnn
,预测概率见公式13所示:
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式13其中,w参数矩阵,b为偏置向量,z
fnn
为输入特征向量。
8.根据权利要求1所述lncrna-疾病关联预测方法,赋予两侧预测模块不同的权值,对预测分值进行加权融合来预测lncrna-疾病的关系,融合方法见公式14所示:
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
公式14其中,μ表示左侧基于栈式自编码器的关联预测模块对关联预测分值的贡献,1-μ表示右侧基于融合神经网络的关联预测模型对关联预测分值的贡献,score是一个向量,包含lncrna和疾病有关联的预测分值和无关联的预测分值;当预测有关联的分值比预测无关联的分值大时,就可以认为该lncrna-疾病是有关联的,否则,则认为lncrna和疾病就没有关联。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1