一种鲜味肽筛选方法及筛选模型
本发明涉及鲜味肽,尤其涉及一种鲜味肽筛选方法及筛选模型。
背景技术:
1、2002年,鲜味被列为继甜、苦、酸、咸四种基本味之后的第五种基本味。鲜味可以通过减少饮食中钠的摄入量来降低成年人患慢性疾病的风险。除l-谷氨酸钠(msg)外,还发现一些双功能酸、游离l-氨基酸、多肽及其衍生物或反应产物具有鲜味。特别是多肽及其衍生物是调味品的重要组成部分。因此,发现鲜味肽对于制备新的鲜味调味品或食品添加剂具有重要意义。
2、目前已报道的鲜味肽有cm、gcg、edg、tessse和rgeneseeegaivt等100多种。目前主要利用多维色谱和超高效液相色谱-电喷雾电离-四极杆飞行时间质谱(uplc-esi-qtofms/ms)鉴定蛋白水解物中的鲜味肽。然而,传统的鲜味肽筛选方法存在以下不足:(1)传统的鲜味物质挖掘工作首先需要选择一个鲜味基料,然后需要借助超滤、凝胶过滤色谱、反相-高效液相色谱和质谱联用等设备进行潜在鲜味肽筛选;最后筛选结果需要进行人工感官实验鉴定。在人力、时间、经济等方面成本较高,实验周期长。(2)传统鲜味肽挖掘过程需要大量的鲜味来源物质进行实验提炼,料成本较高。(3)传统的鲜味肽判断依赖感官员培训小组,每次人工感官小组需要针对特定的鲜味基料进行3-6个月的系统培训,培训周期长,其需要针对特殊的鲜味来源物进行培训,结果难度迁移,对于新的来源物难以在短时间内形成高效统一的判断。(4)传统的鲜味肽挖掘工作结果准确率不高,且容易缺漏,难以充分挖掘。
3、计算机辅助筛选可应用于鲜味肽的筛选和鉴定,且耗时短,有着统一的衡量标准,成本低。该系列方法包括分子对接、分子指纹建模、分子描述符qsar建模。这三种方法在活性分子挖掘领域有着广泛的应用,但是分子对接是通过受体的特征以及受体和药物分子之间的相互作用方式来进行药物设计的方法。主要研究分子间(如配体和受体)相互作用,并预测其结合模式和亲合力的一种理论模拟方法。分子指纹是通过一组特定位数(128/1024/2048)、0/1填充的字节串来描述分子特征结构的矩阵。常被用作分子特征化的方法,将3维分子结构信息转为2维信息以用于机器学习。分子描述符从多个维度(0-3维)、多个角度(物理化学性质指标)、多个描述方式(定性/定量)描述所需要表征的分子,常用于定量构效关系的研究,如分子组成(如氢键供体数、化学键数)、理化性质(如酯水分布系数)描述符、分子场描述符以及分子形状描述符。
技术实现思路
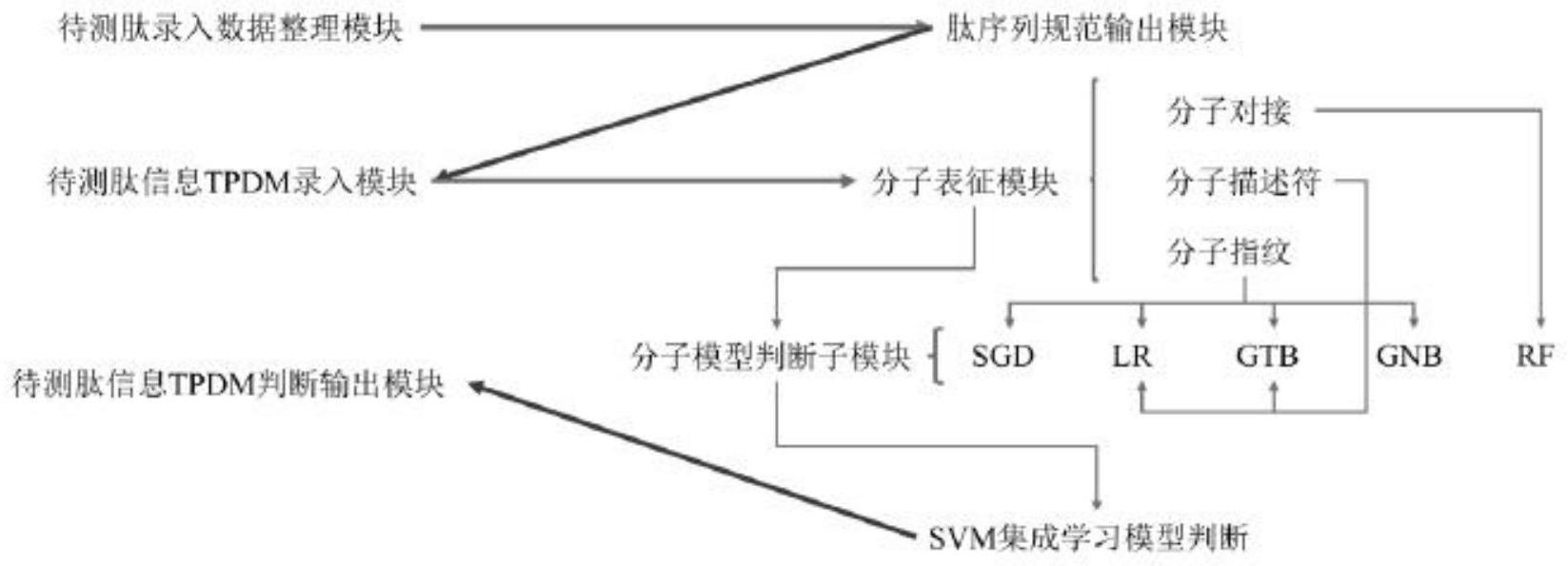
1、针对上述存在的问题,本发明旨在提供一种基于分子对接、分子描述符、分子指纹与集成算法相结合的鲜味肽筛选方法及筛选模型,能够对鲜味肽进行快速,精确的筛选,且该筛选方法可重复使用。
2、为了实现上述目的,本发明所采用的技术方案如下:
3、一种鲜味肽筛选方法,其特征在于,包括以下步骤,
4、s1:整理现有的鲜味肽数据,建立数据库;
5、s2:基于现有的鲜味肽本身的结构碎片构建鲜味肽的分子指纹特征数据;
6、s3:基于分子对接技术解析的鲜味肽与鲜味受体t1r1/t1r3相互作用方式构建分子间互作残基特征数据;
7、s4:基于分子描述符,获得鲜味肽理化性质的分子描述符特征数据;
8、s5:使用机器学习算法对步骤s2-s4中得到的数据分别建立鲜味肽筛选子模型;
9、s6:使用支持向量机算法对鲜味肽筛选子模型进行集成,建立鲜味肽筛选模型;
10、s7:利用步骤s6中建立的鲜味肽筛选模型对鲜味肽进行筛选。
11、进一步的,步骤s2中所述的分子指纹特征数据包括morgan148、322、428、509、598、650、805、952、1150、1409、1573、1687、1706、1907和2017。
12、进一步的,步骤s5中使用机器学习算法对步骤s2中得到的分子指纹特征数据建立的鲜味肽筛选子模型包括随机梯度下降判别模型模型、分子指纹特征数据逻辑回归模型、分子指纹特征数据梯度提升树模型和高斯分布朴素贝叶斯判别模型。
13、进一步的,步骤s3中所述的分子间互作残基特征数据包括:hi_a_2_leu,hi_a_3_leu,hi_a_108_asp,hi_a_154_thr,hi_a_157_ala,hi_a_158_leu,hi_a_161_pro,hi_a_163_leu,hi_a_179_lys,hi_a_181_gln,hi_a_182_tyr,hi_a_183_pro,hi_a_218_asp,hi_a_246_pro,hi_a_419_trp,hi_b_19_thr,hi_b_56_arg,hi_b_57_pro,hi_b_106_pro,hi_b_107_val,hi_b_152_val,hi_b_155_lys,hi_b_156_phe,hi_b_179_thr,hi_b_245_leu,hdb_a_48_ser,hdb_a_50_cys,hdb_a_52_gln,hdb_a_107_ser,hdb_a_109_ser,hdb_a_148_ser,hdb_a_150_asn,hdb_a_151_arg,hdb_a_161_pro,hdb_a_217_ser,hdb_a_218_asp,hdb_a_219_asp,hdb_a_222_gln,hdb_a_247_phe,hdb_a_248_ser,hdb_a_249_ala,hdb_a_276_ser,hdb_a_278_gln,hdb_b_15_leu,hdb_b_17_pro,hdb_b_56_arg,hdb_b_57_pro,hdb_b_58_ser,hdb_b_146_ser,hdb_b_148_glu,hdb_b_155_lys,hdb_b_178_glu,hdb_b_179_thr,hdb_b_215_asp,hdb_b_217_glu,hdb_b_221_gln,psp_a_247_phe,sb_a_151_arg,sb_b_155_lys,sb_b_220_arg,sb_b_247_arg,sb_b_252_arg的次数,若没有发生鲜味肽与鲜味受体t1r1/t1r3的交互,则对应的分子间互作残基特征数据为0;其中,a是t1r1蛋白,b是t1r3蛋白,hdb是氢键相互作用,hi是疏水相互作用,sb是盐桥,psp是∏-堆叠。
14、进一步的,步骤s5中使用机器学习算法对步骤s3中得到的分子间互作残基特征数据建立的鲜味肽筛选子模型为随机森林模型。
15、进一步的,步骤s4中所述的分子描述符特征数据包括bcut2d_mwlow、bcut2d_logphi、smr_vsa1、minestateindex、vsa_estate5、vsa_estate6、vsa_estate7、mollogp、肽序列中d出现的次数、肽序列中e出现的次数、肽序列中d、e出现的次数和、d在肽序列中第一次出现的位置、e在肽序列中第一次出现的位置;若肽序列同时包括d、e,则对应的分子描述符特征数据为1。
16、进一步的,步骤s5中使用机器学习算法对步骤s4中得到的分子描述符特征数据建立的鲜味肽筛选子模型包括分子描述符特征数据逻辑回归模型和分子描述符特征数据梯度提升树模型。
17、进一步的,一种鲜味肽筛选模型,包含上述所述的鲜味肽筛选方法。
18、进一步的,所述筛选模型包括前往网页展示系统和后端计算与分析系统,所述后端计算与分析系统在进行数据计算和分析时采用所述鲜味肽筛选方法。
19、本发明的有益效果是:
20、1、本发明中公开了一种鲜味肽筛选方法,基于现有的鲜味肽本身的结构碎片构建鲜味肽的分子指纹特征数据,基于分子对接技术解析的鲜味肽与鲜味受体t1r1/t1r3相互作用方式构建分子间互作残基特征数据,基于分子描述符,获得鲜味肽理化性质的分子描述符特征数据,然后使用机器学习算法对得到的不同数据分别建立鲜味肽筛选子模型,使用鲜味肽筛选子模型进行集成,建立鲜味肽筛选模型,利用鲜味肽筛选模型对鲜味肽进行筛选和识别,可快速准确的识别出鲜味肽,鲜味肽的筛选准确率显著提升,且筛选时间显著缩短,从而降低了筛选过程的经济成本。
21、2、本发明中公开了一种鲜味肽筛选模型,包括前往网页展示系统、后端计算与分析系统,所述后端计算与分析系统在进行数据计算和分析时采用所述鲜味肽筛选方法,其中,后端计算与分析系统在进行数据计算和分析时采用具体的鲜味肽筛选方法,该模型将所有的流程打造成一个流程化的平台操作界面,能够进行轻度依赖/无依赖人工经验的高效筛选,根据所需要活性的种类,上传对应的数据即可进行特色化的筛选,模型迁移性强。
- 还没有人留言评论。精彩留言会获得点赞!