一种胶质母细胞瘤分类与预测方法、系统、设备及介质
本发明涉及生物学技术和计算机,特别涉及一种基于基因特征的人工智能计算模型得到胶质母细胞瘤分类与预测方法、系统、设备及介质。
背景技术:
1、胶质母细胞瘤(gbm)是一种高级胶质瘤(iv级),是最具侵袭性和致死性的胶质瘤。临床上,gbm预后不良,只有不到5%的患者在确诊后存活时间超过5年。gbm的发生机制尚不清楚,且由于缺乏有效的治疗方法,容易复发。组织形态学的模糊性和肿瘤的异质性给gbm的诊断、预后预测和治疗带来了挑战。组织学诊断的经验在观察者之间产生了相当大的差异,限制了诊断的可重复性,这导致了暂时性的病理诊断和明显的临床混乱。更重要的是,gbm在组织学和遗传学上表现出显著的瘤间和瘤内异质性,具有不同的突变或表现出不同的表型和表观遗传状态,反映出基因组的不稳定性,导致不同的治疗或结果。胶质瘤的发病机制、治疗和预后在分子水平上成为当前研究的重点。基于基因谱和分子生物学特征的gbm分子分类是对传统的基于病理学的gbm分类的必要补充。
2、基于分子的诊断、患者分层和治疗越来越重要。2016年,世卫组织更新了指南,将形态学和遗传变异结合起来,导致对几种脑肿瘤实体的分类进行了重大重组,特别是胶质瘤。2016年who基于gbm的异柠檬酸脱氢酶(idh)基因突变状态分类的两个重要实体是idh野生型gbm和idh突变型gbm,无法对idh进行完全评估的患者归为gbm nos。多组学研究在肿瘤基因组图谱研究网络(tcga)、中国胶质瘤基因组图谱(cgga)等数据库中解释了gbm的概况,揭示了gbm复杂的遗传谱。这些异常分子包括1p和19q编码缺失(少突胶质细胞瘤特异性)、idh基因突变、pten(磷酸酶和紧张素同源物)基因突变、tp53突变、tert(端粒酶逆转录酶)基因启动子突变、atrx(α-地中海贫血/智力低下综合征x相关)基因启动子突变和egfr(上皮生长因子受体)基因扩增,这些都迫使临床病理学重新考虑gbm的治疗。基于异常分子的gbm分类逐渐缩短了从诊断到治疗的时间,显著提高了准确性和靶向性。
3、基因组学作为一门数据驱动的科学而兴起,它通过对基因组尺度数据的探索来发现新的特性,而不是通过测试先入为主的模型和假设。基因组学的应用包括寻找基因型和表型之间的联系,发现患者分层的生物标记物,预测基因的功能。基因组数据太大而且太复杂,不能仅通过对偶相关的目视调查来挖掘,需要分析工具来支持发现意料之外的关系,推导出新的假设和模型,并做出预测。机器学习算法适合数据驱动的科学,它的性能很大程度上取决于每个变量是如何计算的。神经网络是由连续的基本操作组成的机器学习模型,它将前面操作的结果作为输入,计算越来越复杂的特征。深度神经网络能够通过发现高复杂性的相关特征来提高预测的准确性。目前,人工智能广泛应用于gbm预测模型,有监督和无监督的学习方法来训练深度神经网络,但是仍然没有精度较高的预测性较好的分类器对异质性较强的gbm进行分类,提供最佳诊断标准。
技术实现思路
1、本发明针对现有技术的缺陷,提供了一种胶质母细胞瘤分类与预测方法、系统、设备及介质。
2、为了实现以上发明目的,本发明采取的技术方案如下:
3、一种胶质母细胞瘤分类与预测方法,包括以下步骤:
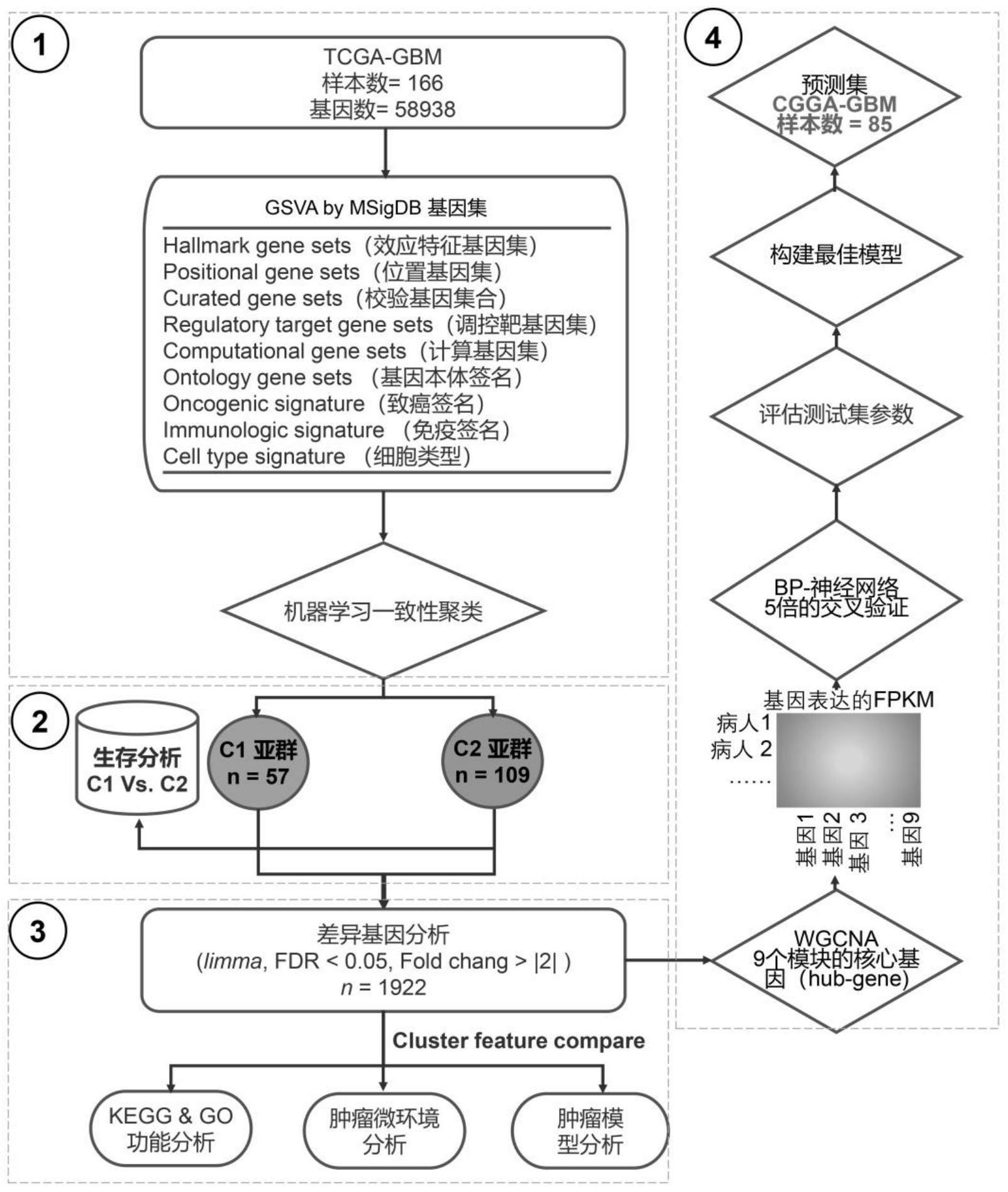
4、s1:下载tcga-gbm和cgga-gbm患者转录组数据作为测试数据集;
5、tcga-gbm为来自于the cancer genome atlas program(tcga)数据库,数据类型是rna-seq和病人临床随访数据,简称为tcga-gbm。
6、cgga-gbm为来自于自中国胶质瘤数据库(the chinese glioma genome atlas,cgga),数据类型是rna-seq和病人临床随访数据,简称为cgga-gbm。
7、s2:使用gsva计算基因特征数据集(msigdb),并合并成初始数据集。
8、s3:对初始数据集进行无监督聚类的一致性聚类,得到与预后相关的新亚型。
9、s4:新亚型差异基因分析,得到新亚型免疫特异性。
10、s5:利用wgcna计算tcga-gbm基因表达谱,划分为基因模块,并得到每个模块的核心基因(hub gene)。
11、s6:使用反向传播(bp)神经网络,构建对新亚型预测的最佳分类器。
12、进一步地,步骤s2具体为:计算基因特征数据集;
13、使用由r语言中的gsva包计算对来自分子特征数据库(msigdb)中的9中类型基因集的评分,合并每个病人对基因集的富集评分,得到多条基因特征,作为以下分类的初始数据集。
14、进一步地,步骤s3具体为:
15、使用r语言中的consensusclusterplus包,进行无监督聚类,对80%的样本进行重采样,重复10次。根据分布函数图确定最佳聚类数,找到新亚型。
16、进一步地,步骤s4具体为:
17、在基因参与的功能分析方面,使用r软件包clusterprofiler进行信号通路和基因本体的富集分析。
18、对于肿瘤微环境分析(tme)分别使用xcell1,cibersort,patientimmunophenoscore和quantiseq进行免疫浸润分析,得到免疫特异性。
19、进一步地,步骤s4中,对新亚型进行差异分析,得到的差异基因(degs),和主要参与的kegg通路和go生物过程。
20、对新亚型进行肿瘤微环境分析(tme)
21、对新亚型进行耐药分析,得到耐药性。
22、对新亚型进行肿瘤驱动基因靶点分析,得到新亚型的肿瘤驱动基因靶点。
23、进一步地,s4中将差异基因阈值设置为fdr<0.05和log2foldchange>|2|,满足此条件的基因为2个亚群之间的明显差异。
24、进一步地,s5具体为:利用wgcna计算s1中下载得到的tcga-gbm转录组数据,计算每个基因的中位绝对偏差(mad),并去掉了前50%的中位绝对偏差最小的基因。使用r软件包的goodsamplesgenes方法去除异常值基因和样本。
25、利用wgcna构建无标度共表达网络。对所有配对基因进行pearson相关矩阵和平均连锁法,为了把基因表达趋势具有一致性的基因合并,将具有相似表达谱的基因划分为基因模块,并筛选每个模块的核心基因。
26、进一步地,s6具体为:通过r包的neuralnet包使用bp神经网络模型,设置了11种不同隐含信息的bp模型,其中隐含层为1,隐含神经元为,分别设置为4,5,6,7,8;隐层为2,隐神经元为5或6;隐层为3,隐神经元为5或6;隐层为4,隐神经元为5或6。使用5次交叉验证,并将所有tcga-gbm转录组数据中的来自基因模块的核心基因(hub genes)基因数据分为训练折叠集和测试折叠集。将整个过程重复100次(k=100),计算平均敏感性(averagesensitivity)、平均特异(average specificity)、平均精密(average precision)和平均准确度(average accuracy)。
27、通过上述计算对基因特征贡献度排序。
28、本发明还公开了一种胶质母细胞瘤分类与预测系统,该系统能够用于实施上述的一种胶质母细胞瘤分类与预测方法,具体的,包括:数据输入模块、基因特征数据集计算模块、聚类模块、新亚型特征分析模块、基因模块划分模块和分类器构建模块;
29、数据输入模块:将tcga-gbm和cgga-gbm患者转录组数据作为测试数据集输入;
30、基因特征数据集计算模块:使用gsva计算基因特征数据集,并合并成初始数据集。
31、聚类模块:对初始数据集进行无监督聚类的一致性聚类,得到与预后相关的新亚型。
32、新亚型特征分析模块:新亚型差异基因分析,得到新亚型免疫特异性。
33、基因模块划分模块:利用wgcna计算tcga-gbm基因表达谱,划分为基因模块,并得到每个模块的核心基因。
34、分类器构建模块:使用反向传播(bp)神经网络,构建对新亚型预测的最佳分类器。
35、本发明还公开了一种计算机设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现上述胶质母细胞瘤分类与预测方法。
36、本发明还公开了一种计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行时实现上述胶质母细胞瘤分类与预测方法。
37、与现有技术相比,本发明的优点在于:
38、本发明根据gbm基因特征集无监督聚类,得到与预后相关的清晰病人亚型分类,清晰呈现不同亚型病人的免疫特异性,是迄今为止免疫差异最明显的分类,并找到现有技术未被关注的靶点基因,能够给出不同人群的治疗指导方案。同时,利用反向传播神经网络构建高精度分类器,在临床可精准诊断、分层和指导治疗。
- 还没有人留言评论。精彩留言会获得点赞!