一种基于异质蛋白特征杂交的抗体亲和力预测方法及系统与流程
本发明属于生物基因,尤其涉及一种基于异质蛋白特征杂交的抗体亲和力预测方法、系统及应用。
背景技术:
1、在抗体设计优化领域中,抗体与目标抗原的亲和力是重要指标。但由于指数增长的氨基酸排列组合的可能,找到高亲和力的抗体序列需要模型从多个方面来表征蛋白,来尽可能的增加对于现有数据样本的利用,减少对训练样本总量的需求。传统的抗体/抗原蛋白表征方法是基于蛋白序列的。但基于序列的方法效率非常低,因为抗原抗体结合情况与他们的结构匹配密切相关,纯蛋白序列的方法,很难发现影响结合亲和力的关键氨基酸。因此当下流行的蛋白表征方法主要是基于蛋白结构的。基于蛋白结构的表征,又细分为基于所有原子,基于碳阿尔法,基于蛋白指纹和基于蛋白表面,这些蛋白表征互有优劣。
2、目前常见的抗体亲和力预测,需要对已知抗体抗原复合物结构进行建模,然后基于传统原子对的力场模型来预测突变导致的亲和力变化。但这类方法需要提前得到复合物的晶体结构,在很多现实场景下并不适用。另外一种基于蛋白序列的亲和力预测,虽然摆脱了复合物结构的限制,但由于蛋白序列与亲和力之间的隐式关系,在现有的数据量下,无法取得很好的预测精度。现有基于蛋白结构的模型,对于蛋白表征处理比较单一。基于蛋白表面或者蛋白全原子的表征,对于没有蛋白结合导致的表面形变的情况,表现较好,但是当抗原抗体结合诱导蛋白表面变化时,基于蛋白表面特征的模型的泛化能力显著降低。基于蛋白碳阿尔法或者蛋白指纹的表征,虽然有更强的泛化能力,但是对于无显著诱导形变的情况,预测能力较低。
3、因此,上述的现有技术确实有待提出更佳解决方案的必要性。
技术实现思路
1、本发明的目的是提供一种基于异质蛋白特征杂交的抗体亲和力预测方法、系统和应用,基于将这些特征融合杂交可以显著提升模型预测能力的理论基础,将多个不同类型的蛋白表征利用基于结构的图神经网络进行深度的特征融合。杂交后的特征,既包括了蛋白表面特征,可以精准匹配抗原抗体的特定区域,也包括了蛋白指纹,在更高维度匹配对应位置和计算亲和力的强弱,从而增强模型泛化能力,提升模型对现有数据的利用度。
2、本发明一方面提供了一种基于异质蛋白特征杂交的抗体亲和力预测方法,包括:
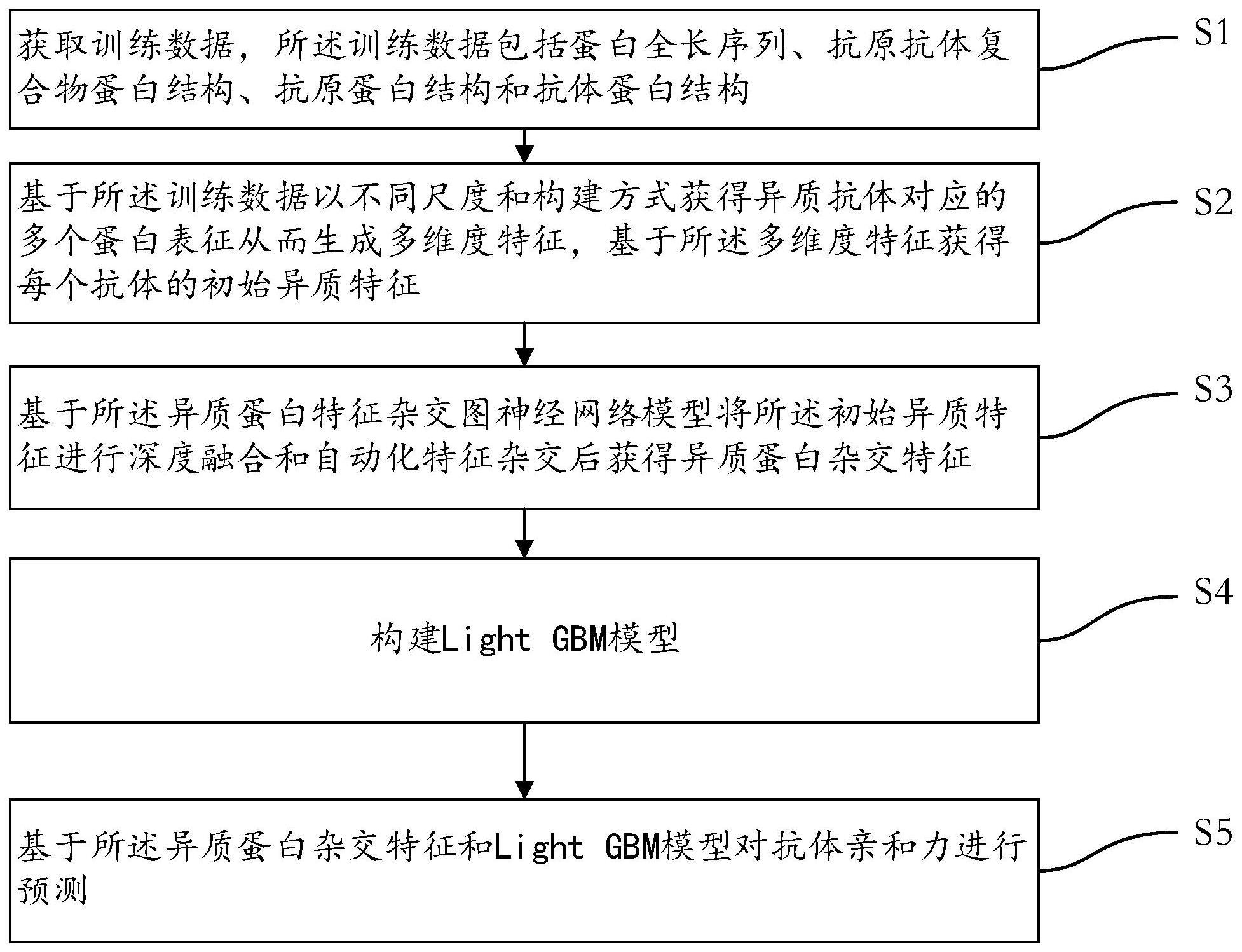
3、s1,获取训练数据,所述训练数据包括蛋白全长序列、抗原抗体复合物蛋白结构、抗原蛋白结构和抗体蛋白结构;
4、s2,基于所述训练数据以不同尺度和构建方式获得异质抗体对应的多个蛋白表征从而生成多维度特征,基于所述多维度特征获得每个抗体的初始异质特征;
5、s3,基于所述异质蛋白特征杂交图神经网络模型将所述初始异质特征进行深度融合和自动化特征杂交后获得异质蛋白杂交特征;
6、s4,构建light gbm模型;
7、s5,基于所述异质蛋白杂交特征和light gbm模型对抗体亲和力进行预测。
8、优选的,所述s2包括:
9、s21,基于蛋白全长序列,分别计算抗原和抗体与训练集中蛋白的代表序列之间的相似度,提取蛋白可变区cdr,计算蛋白可变区cdr对应区域的序列相似度,作为第一特征;
10、s22,基于所述蛋白全长序列和所述蛋白可变区cdr对应区域的蛋白序列,统计所述蛋白全长序列和所述蛋白序列的理化性质作为第二特征,所述理化性质包括亲脂性,电荷分布以及序列kmer;
11、s23,基于抗原抗体复合物蛋白结构,提取其中的原子对信息并计算其中的原子对的能量,作为第三特征;
12、s24,基于抗原蛋白结构和抗体蛋白结构,提取抗原蛋白结构和抗体蛋白结构中的氨基酸层面的化学性质,计算可形成的氢键个数,作为第四特征;
13、s25,基于抗原蛋白结构和抗体蛋白结构获得蛋白指纹,作为第五特征;
14、s26,基于抗原蛋白结构和抗体蛋白结构获得蛋白的表面的几何信息,作为第六特征;
15、s27,将第一特征到第六特征相加得到每个抗体的初始异质特征xv。
16、优选的,所述s21计算抗原和抗体与训练集中蛋白的代表序列之间的相似度基于smith-waterman算法实现。
17、优选的,所述s23计算其中的原子对的能量基于foldx力场实现。
18、优选的,所述s25的所述蛋白指纹基于哈希方法实现。
19、优选的,所述s3包括:
20、s31,构建多层图神经网络作为所述异质蛋白特征杂交图神经网络模型,所述异质蛋白特征杂交图神经网络模型中将蛋白氨基酸作为图神经网络的点,当两个蛋白氨基酸之间的距离小于1纳米时,两点之间形成一条边;
21、s32,多层图神经网络中的浅层网络和深层网络之间进行信息交互,表征不同特征之间的非线性关系,从而实现第一特征到第六特征的自动化特征融合获得异质蛋白杂交特征。
22、优选的,所述异质蛋白特征杂交图神经网络模型为:
23、
24、其中,是节点v在第k层的表示,h0v表示用异质特征xv对抗体氨基酸属性进行初始化;代表聚合函数;是节点u在第k-1层的表示;g(k)u代表聚合函数,n(v)代表所有图上的节点,xuv代表边uv的特征向量,在k次图卷积之后,捕获了它们的k次邻域信息;最后,一个读出函数用于聚合第k次图网络输出的所有节点表示,获得整个分子的杂交特征表示zg:
25、
26、优选的,所述s5包括:
27、将整个分子的杂交特征表示zg作为light gbm模型的初始输入,最终通过所述light gbm模型输出抗体亲和力的预测结果。
28、本发明的第二方面提供一种基于生物活性指纹的分子相似度计算系统,包括:
29、数据获取模块(101),用于获取训练数据,所述训练数据包括蛋白全长序列、抗原抗体复合物蛋白结构、抗原蛋白结构和抗体蛋白结构;
30、特征生成模块(102),用于基于所述训练数据以不同尺度和构建方式获得异质抗体对应的多个蛋白表征从而生成多维度特征,基于所述多维度特征获得每个抗体的初始异质特征;
31、融合杂交模块(103),用于基于所述异质蛋白特征杂交图神经网络模型将所述初始异质特征进行深度融合和自动化特征杂交后获得异质蛋白杂交特征;
32、模型构建模块(104),用于构建light gbm模型;
33、预测模块(105),用于基于所述异质蛋白杂交特征和light gbm模型对抗体亲和力进行预测。
34、本发明的第三方面在于提供一种基于生物活性指纹的分子相似度计算系统在抗体药物开发中的应用。
35、本发明提供的系统、方法以及应用,具有如下有益的技术效果:
36、本发明创新性地提出了基于特征杂交的抗原抗体亲和力预测算法,克服了传统方法泛化能力较弱,数据利用度不足的缺点,通过将不同尺度,不同计算维度的特征,深度融合,使得预测抗原抗体亲和力的准确性和泛化能力大幅提升,为新型抗体药物的发现与优化带来极大的便利,提高了新型药物设计和开发的效率,缩短研发周期,具有较高的实用价值。
- 还没有人留言评论。精彩留言会获得点赞!