一种基于矩阵补全算法的药物-miRNA关联预测方法
本发明涉及矩阵补全算法,尤其涉及一种基于鲁棒主成分分析的矩阵补全算法。
背景技术:
1、micrornas(mirnas)是一类单链非编码rna分子,它们在肿瘤发生和宿主-病原体相互作用中发挥着重要作用,将其作为药物的治疗靶点已经成为了临床治疗的新思路。探索药物-mirna的潜在关联对药物研发和疾病治疗具有重大意义。考虑到传统的生物实验成本高,风险大,亟须提出先进的计算模型来筛选具有高关联概率的药物-mirna样本。目前的计算方法可以分为基于图推理的模型以及基于矩阵补全的模型。其中,前者通过在异构图中执行某些图推理算法来获得关联评分,但是会存在一些限制,比如无法过滤噪声,无法预测与新的药物相关的mirnas等;后者通过对药物-mirna关联矩阵执行补全算法来获得关联评分,然而,由于核范数并不是秩函数的最优凸近似,因此这类模型总是会获得高度偏置的解,预测性能无法得到保证。对此,本发明提出一种新的基于矩阵补全算法的药物-mirna关联预测方法。
技术实现思路
1、本发明的目的是为了解决上述药物-mirna相互作用预测领域所存在的困难,提供一种基于矩阵补全算法的药物-mirna相互作用预测方法,整合药物和mirna的多源数据,帮助科研工作者进行正确的预测。本发明的技术方案如下:
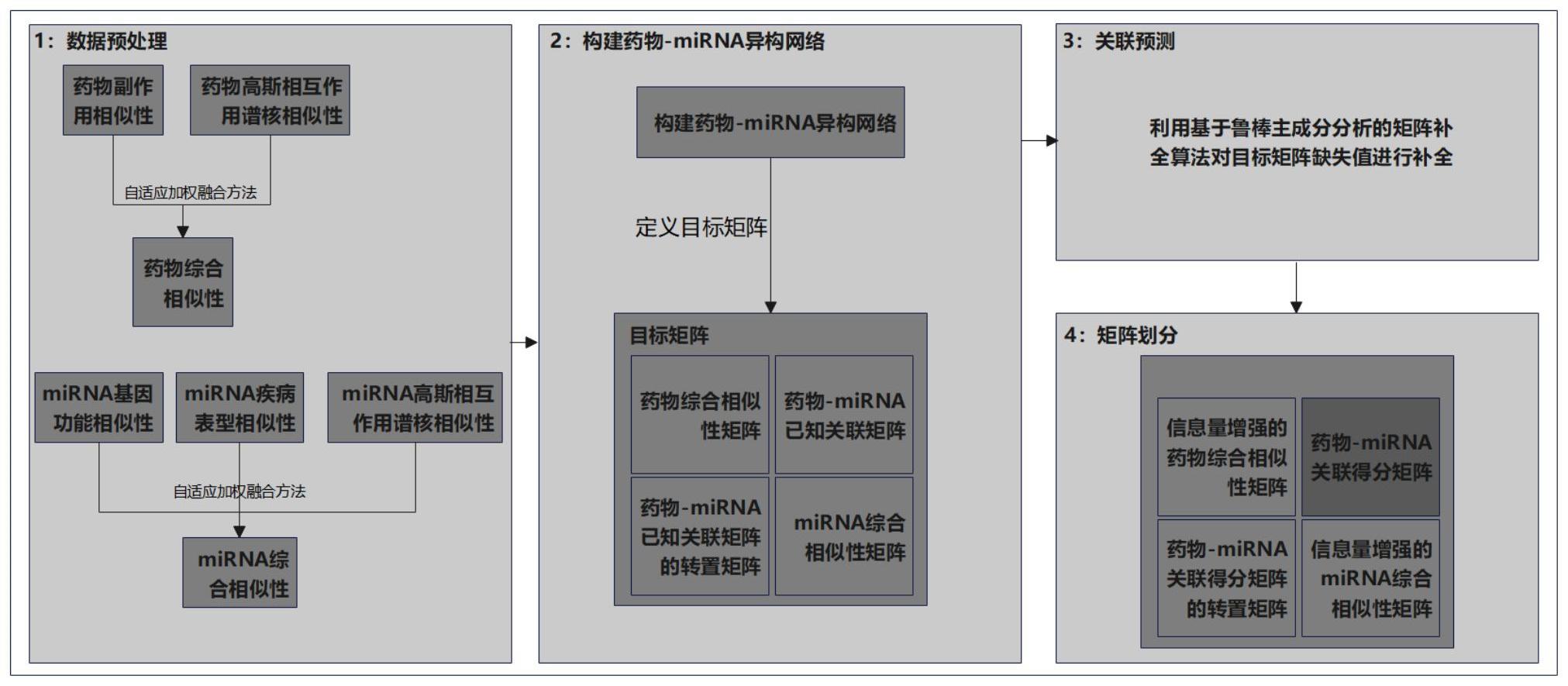
2、a、基于药物的副作用相似性和高斯相互作用谱核相似性构建两个独立的药物相似性网络,基于mirna的基因功能相似性,疾病表型相似性以及高斯相互作用谱核相似性构建三个独立的mirna相似性网络。
3、b、基于自适应加权融合方法,将两个独立的药物相似性网络整合为一个综合的药物相似性网络,将三个独立的mirna相似性网络整合为一个综合的mirna相似性网络。
4、c、基于综合的药物相似性网络,综合的mirna相似性网络以及已知的药物-mirna相互作用信息构建一个药物-mirna异构网络,并定义其邻接矩阵作为目标矩阵,即矩阵补全算法的输入矩阵。
5、d、基于矩阵补全算法实现目标矩阵缺失值的补全。
6、e、基于矩阵划分方法筛选出预测评分矩阵以得到所有药物-mirna对的关联得分。
7、根据权利a所述的基于药物的副作用相似性和高斯相互作用谱核相似性构建两个独立的药物相似性网络,基于mirna的基因功能相似性,疾病表型相似性以及高斯相互作用谱核相似性构建三个独立的mirna相似性网络。本发明使用从sider数据库下载的药物副作用信息计算药物副作用相似性,使用从sm2mirna数据库下载的药物-mirna相互作用数据分别计算药物和mirna的高斯相互作用谱核相似性,基于mirbase和mir2disease数据库下载的mirna-基因关联,mirna-疾病关联信息分别计算mirna的基因功能相似性,疾病表型相似性。
8、根据权利b所述的基于自适应加权融合方法,将两个独立的药物相似性网络整合为一个综合的药物相似性网络,将三个独立的mirna相似性网络整合为一个综合的mirna相似性网络。本发明使用自适应加权融合算法实现各个相似性网络的非线性融合,保证了相似性数值介于0-1之间,增强了数据可解释性。
9、根据权利c所述的基于综合的药物相似性网络,综合的mirna相似性网络以及已知的药物-mirna相互作用信息构建一个药物-mirna异构网络,并定义其邻接矩阵作为目标矩阵,即矩阵补全算法的输入矩阵。本发明依次将综合的药物相似性矩阵,药物-mirna关联矩阵,药物-mirna关联矩阵的转置矩阵,综合的mirna相似性矩阵按从左到右,从上到下的顺序依次拼接即得到药物-mirna异构网络的邻接矩阵,它同时也是矩阵补全算法的目标矩阵。
10、根据权利d所述的基于矩阵补全算法实现目标矩阵缺失值的补全。本发明设计了一种全新的鲁棒主成分分析框架,其假设目标矩阵是一个低秩矩阵和一个噪声矩阵的和,得到低秩矩阵的方式是使其伽马范数最小化,得到噪声矩阵的方式是使其1范数最小化。同时设置了参数来调控噪声矩阵的稀疏性。为了得到最优解,本发明根据增广拉格朗日乘子法框架分别设计了低秩矩阵,噪声矩阵,拉格朗日乘子以及自适应参数的迭代更新规则。
11、根据权利e所述的基于矩阵划分方法筛选出预测评分矩阵以得到所有药物-mirna对的关联得分,本发明根据目标矩阵的拼接方式对得到的输出矩阵进行切分,得到了信息量增强的药物相似性矩阵,信息量增强的mirna相似性矩阵,去除噪声且缺失值已被补全的药物-mirna关联矩阵,补全值即为药物-mirna对的关联分数。
技术特征:
1.一种基于矩阵补全算法的药物-mirna关联预测方法,包括以下部分:
2.根据权利要求1所述的基于药物的副作用相似性和高斯相互作用谱核相似性构建两个独立的药物相似性网络,基于mirna的基因功能相似性,疾病表型相似性以及高斯相互作用谱核相似性构建三个独立的mirna相似性网络。本发明使用从sider数据库下载的药物副作用信息计算药物副作用相似性,使用从sm2mirna数据库下载的药物-mirna相互作用数据分别计算药物和mirna的高斯相互作用谱核相似性,基于mirbase和mir2disease数据库下载的mirna-基因关联,mirna-疾病关联信息分别计算mirna的基因功能相似性,疾病表型相似性。
3.根据权利要求1所述的基于自适应加权融合方法,将两个独立的药物相似性网络整合为一个综合的药物相似性网络,将三个独立的mirna相似性网络整合为一个综合的mirna相似性网络。本发明使用自适应加权融合算法实现各个相似性网络的非线性融合,保证了相似性数值介于0-1之间,增强了数据可解释性。
4.根据权利要求1所述的基于综合的药物相似性网络,综合的mirna相似性网络以及已知的药物-mirna相互作用信息构建一个药物-mirna异构网络,并定义其邻接矩阵作为目标矩阵,即矩阵补全算法的输入矩阵。本发明依次将综合的药物相似性矩阵,药物-mirna关联矩阵,药物-mirna关联矩阵的转置矩阵,综合的mirna相似性矩阵按从左到右,从上到下的顺序依次拼接即得到药物-mirna异构网络的邻接矩阵,它同时也是矩阵补全算法的目标矩阵。
5.根据权利要求1所述的基于矩阵补全算法实现目标矩阵缺失值的补全。本发明设计了一种全新的鲁棒主成分分析框架,其假设目标矩阵是一个低秩矩阵和一个噪声矩阵的和,得到低秩矩阵的方式是使其伽马范数最小化,得到噪声矩阵的方式是使其1范数最小化。同时设置了参数来调控噪声矩阵的稀疏性。为了得到最优解,本发明根据增广拉格朗日乘子法框架分别设计了低秩矩阵,噪声矩阵,拉格朗日乘子以及自适应参数的迭代更新规则。
6.根据权利要求1所述的基于矩阵划分方法筛选出预测评分矩阵以得到所有药物-mirna对的关联得分,本发明根据目标矩阵的拼接方式对得到的输出矩阵进行切分,得到了信息量增强的药物相似性矩阵,信息量增强的mirna相似性矩阵,去除噪声且缺失值已被补全的药物-mirna关联矩阵,补全值即为药物-mirna对的关联分数。
技术总结
本发明提供了一种基于矩阵补全算法的药物‑miRNA关联预测系统,涉及生物信息、机器学习领域。首先,我们整合药物和miRNA的多源数据,构建两个独立的药物相似性网络以及三个独立的miRNA相似性网络。然后,通过自适应加权融合方法,将这些不同的相似性网络分别集成到药物和miRNA的综合相似性网络中。基于药物和miRNA的综合相似性网络以及已知的药物‑miRNA相互作用信息构建药物‑miRNA异构网络,并定义了一个目标矩阵来表示它。随后,将目标矩阵输入到矩阵补全算法中,通过增广拉格朗日乘子法迭代优化以补全目标矩阵中的缺失值。最终,利用矩阵划分方法将输出矩阵划分为信息量增强的药物相似性矩阵,信息量增强的miRNA相似性矩阵以及去除噪声且缺失值被补全的药物‑miRNA关联得分矩阵。
技术研发人员:王淑栋,任传儒,李韵音,庞善臣,王福钰,刘英业
受保护的技术使用者:中国石油大学(华东)
技术研发日:
技术公布日:2024/1/15
- 还没有人留言评论。精彩留言会获得点赞!