基于Cox回归分析的胰腺癌的预后模型构建方法
本发明属于医学领域,尤指一种基于cox回归分析的胰腺癌的预后模型构建方法。
背景技术:
1、胰腺癌(cancer of pancreas,pancreatic cancer)是消化道常见的恶性肿瘤之一,多发生于胰头部。腹痛及无痛性黄疸为胰头癌的常见症状。糖尿病患者长期大量吸烟,高脂肪高动物蛋白饮食者,发病率相对增高,本病多发于中老年人,男性患者远较绝经前的妇女多,绝经后妇女发病率与男性相仿。发病原因尚不清楚,发现些环境因素与胰腺癌的发生有关。已定的首要危险因素为吸烟,糖尿病胆石病饮酒(包括啤酒)以及慢性胰腺炎等进食高脂肪高蛋白饮食和精制的面粉食品,胃切除术也是发生胰腺癌的危险因素,其死亡率极高。
2、截止到目前,手术是唯一可以治愈胰腺癌的方法,但只有不到20%的确诊患者可以从切除手术中受益,而且根治性切除后复发的风险很高。就生存时间而言,一项研究表明,可以手术切除的胰腺癌患者的5年生存率约为20%。在过去的十几年中,胰腺癌的治疗在研究上取得了一些成就,比如,在辅助治疗和转移性疾病治疗方面有越来越多的方法可供选择。在辅助治疗方面,改良folfirinox(mfolfirinox)方案和吉西他滨方案均能够提高胰腺癌患者的无病生存率和总生存率,一项研究的结果显示,mfolfirinox组3年无病生存率为39.7%,总生存率为63.4%,吉西他滨组无病生存率为21.4%,总生存率为48.6%。asco指南推荐术后辅助治疗首选mfolfirinox,而采用单一疗法时首选吉西他滨。然而,尽管有辅助治疗,复发率仍然很高,有69%到75%的患者在2年内复发。转移性胰腺癌治疗方面,folfirinox方案是良好ecog表现状态患者的有效一线治疗选择,较吉西他滨相比,folfirinox中位生存期增加了4.3个月(11.1月vs 6.8月)。
3、尽管治疗上有上述那么多令人欣喜的进展,但胰腺癌的预后仍然很差,需要更多的研究,尤其在个体化治疗方面。
技术实现思路
1、本发明的目的是提出基于cox回归分析的胰腺癌的预后模型构建方法,通过使用和lassocox回归分析方法构建胰腺癌的预后模型,该模型在胰腺癌的预后中是独立的影响因素,预测能力在训练数据集、验证数据集和完整数据集里都得到了验证,并且比现在已经发表的lncrnas模型和tnm分期系统有更高的预测准确性。
2、为了达到上述目的,在本发明的提供基于cox回归分析的胰腺癌的预后模型构建方法,方法包括:
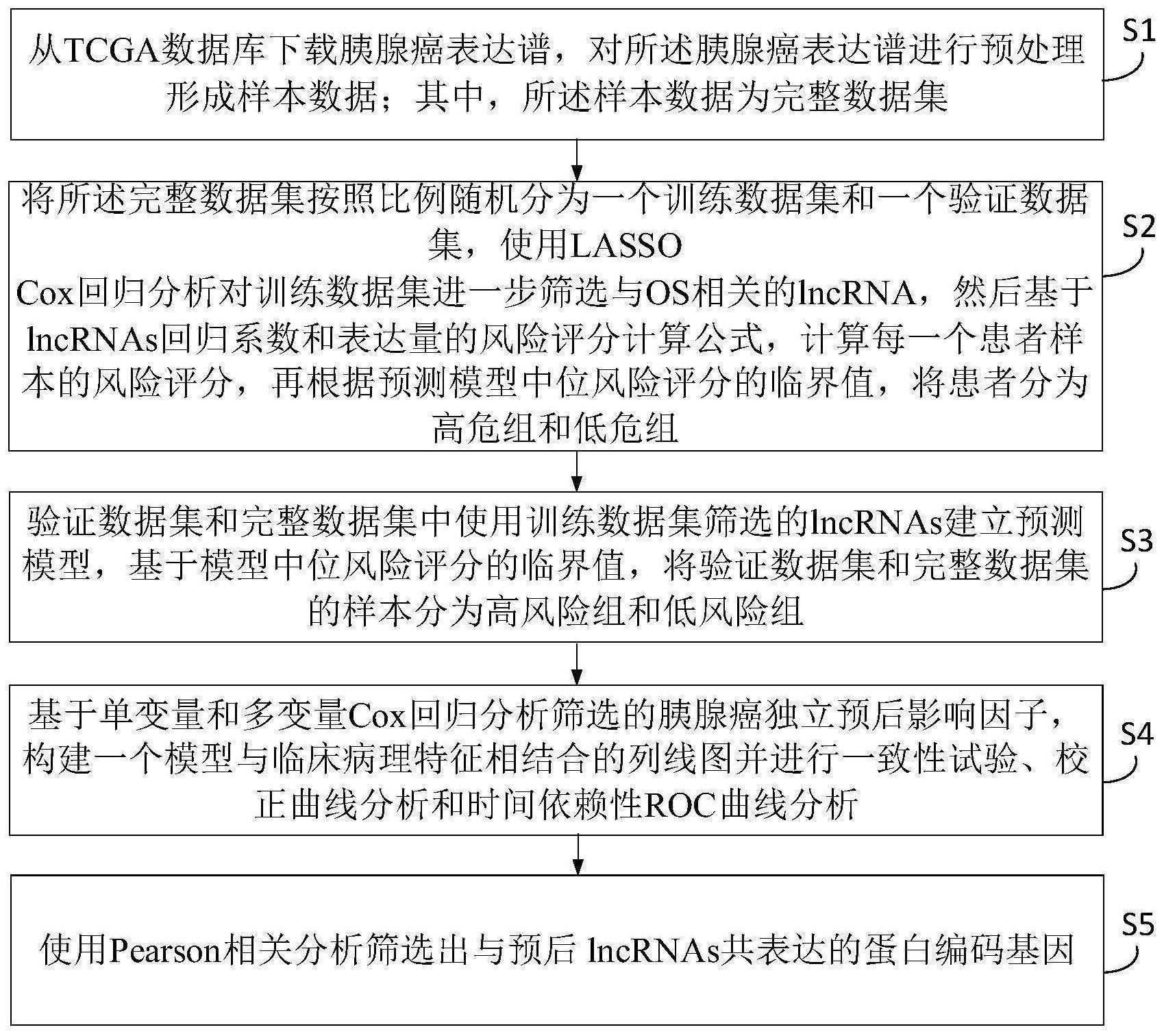
3、步骤一、从tcga数据库下载胰腺癌表达谱,对所述胰腺癌表达谱进行预处理形成样本数据;
4、步骤二、所述样本数据为完整数据集,将所述完整数据集按照比例随机分为一个训练数据集和一个验证数据集,使用lassocox回归分析对训练数据集进一步筛选与os相关的lncrna,然后基于lncrnas回归系数和表达量的风险评分计算公式,计算每一个患者样本的风险评分,再根据预测模型中位风险评分的临界值,将患者分为高危组和低危组;
5、步骤三、验证数据集和完整数据集中使用训练数据集筛选的lncrnas建立预测模型,基于模型中位风险评分的临界值,将验证数据集和完整数据集的样本分为高风险组和低风险组;
6、步骤四、基于单变量和多变量cox回归分析筛选的胰腺癌独立预后影响因子,构建了一个模型与临床病理特征相结合的列线图并进行一致性试验、校正曲线分析和时间依赖性roc曲线分析;
7、步骤五、使用了pearson相关分析筛选出与预后lncrnas共表达的蛋白编码基因;
8、其中,所述步骤一中预处理的步骤包括:对基因进行了整理和注释,然后用r语言的edger软件包对胰腺癌表达谱进行了处理,对比ensembl id后,对基因进行分离筛选出平均表达值大于1的lncrna进行下一步的分析,在胰腺癌表达谱筛选|logfc|>1和p<0.05的差异表达lncrna,并进行单变量cox回归分析,筛选出与os相关的lncrna,再删除临床信息不完整、生存时间为0、重复的样本数据;
9、所述与os相关的lncrna为l031658.1,abca9-as1,dnah17-as1,ap003086.1,ac018755.4;
10、所述风险评分包括:每个样本的风险评分计算公式为:风险评分=-0.23189*al031658.1的表达量+0.20984*abca9-as1的表达量+0.03709*dnah17-as1的表达量+-0.26114*ap003086.1的表达量+0.15556*ac018755.4的表达量,其中所述风险评分公式即为预测模型,以风险评分的中位数为临界值,其中:大于等于临界值的为高危组,小于临界值的为低危组。
11、进一步地,步骤六、使用lassocox回归分析进一步选择并建立基于lncrnas的预测模型,用于预测胰腺癌患者3年和5年的生存率。
12、进一步地,所述步骤三中,根据所述临界值将测试数据集和完整数据集的患者分为高风险组和低风险组,并进行kaplan–meier和对数秩检验。
13、进一步地,所述步骤四中,所述临床病理特征包括lncrnas模型、年龄、性别、饮酒、放疗史、化疗史、家族史、吸烟、肿瘤分化和病理分期。
14、进一步地,所述步骤五中,所诉使用了pearson相关分析筛选出与预后lncrnas共表达的蛋白编码基因的标准为相关系数绝对值>0.4、p<0.001,选出来的蛋白编码基因使用cytoscape的cluego和cluepedia分别进行kegg、go功能富集分析和绘图,用于寻找预后lncrnas作用的通路。
15、进一步地,所述胰腺癌独立预后影响因子为lncrnas模型、化疗史和病理分期。
16、进一步地,基于所述胰腺癌独立预后影响因子构建列线图,每个样本均有胰腺癌患者lncrnas模型风险评分水平、化疗史和病理分期的得分,将该胰腺癌患者获得的三个得分相加计算列线图的总得分,用于预测该胰腺癌患者3年和5年的生存率;
17、绘制列线图预测值和实际观测值进行评估对比该胰腺癌患者3年和5年的生存率的一致性。
18、进一步地,所述选出来的蛋白编码基因使用cytoscape的cluego和cluepedia分别进行kegg、go功能富集分析和绘图的步骤具体包括:
19、从tcga下载的胰腺癌数据中提取所有的mrna,筛选出条件满足|logfc|>2,p<0.05的且在胰腺癌和癌旁正常组织中差异表达的mrna;
20、将筛选后的mrna与之前筛选的5个lncrna进行pearson分析;
21、使用cytoscape的cluego进行了kegg和go功能富集分析,并用cluepedia绘图。本发明的有益技术效果至少在于以下几点:
22、(1)本发明构建的预后模型的能力优于之前发表的lncrnas模型和tnm分期系统,并且,当预后模型和tnm分期系统联合使用时,可以极大地提高胰腺癌预后预测准确性;
23、(2)基于构建的模型和化疗史、病理分期可以构建一个胰腺癌的临床综合预后预测模型,其预测能力良好;
24、(3)基于与这5个lncrna共表达的mrna的kegg和go富集分析发现,p≤0.05的通路有30个,p≤0.00001的有3个,分别为淋巴细胞活化、b细胞活化和白细胞活化,这说明,这5个lncrna对胰腺癌预后的影响与免疫在一定程度上有关,对预测胰腺癌患者的生存率很大地提高了准确性,也可以结合临床进行更针对性的治疗。
- 还没有人留言评论。精彩留言会获得点赞!