一种用于水牛的GBS全基因组关联分析方法与流程
本发明涉及基因组关联分析,更具体地说,它涉及一种用于水牛的gbs全基因组关联分析方法。
背景技术:
1、通过育种实现群体遗传改良是提高水牛业生产水平和效率的关键。我国水牛业生产水平与发达国家有较大差距,根本原因是我国水牛群体的遗传水平低,依靠传统的育种技术难以改变这种状况。以基因组选择为核心的分子育种技术提供了机遇,与传统育种技术相比,该技术可大幅提高群体遗传改良速率和生产效益。
2、产奶、健康、生长和繁殖性状是水牛最为重要的几个经济性状,经济性状的优劣直接影响水牛产业。多年以来,通过传统的育种方法对水牛经济性状进行的遗传改良也取得了一定效果,但由于周期长,经济性状复杂,受诸多基因的控制,传统育种方法已很难对经济性状取得较大的遗传进展。近些年来,随着科技的飞速发展,分子标记辅助育种已成为改良遗传性状的新方法。
3、目前,全基因组snp分型技术主要有基因分型芯片和测序两种方法。基因分型芯片方法尽管技术稳定,结果重复率高,但该技术分型一个实验样本成本高,而且对于群体遗传学研究领域,群体分型的成本更为昂贵,加之受技术所限,存在snp多态位点在不同群体中通用性差,标记密度低,不能精细功能基因定位和全基因组关联分析。
4、目前,一项新的以高通量测序为基础的技术被开发出来,即gbs(genotyping-by-sequencing)技术指通过测序进行基因分型,通过选取合适的限制性内切酶结合高通量群体测序构建snp分子标记,可用于分子标记开发、超高密度遗传图谱构建、群体遗传分析、群体gwas分析等领域。该技术方法与芯片相比,实验步骤简单,成本低;一次测序获得数量庞大的snp位点,密度高;可以检测出基因组上未知变异位点中新的snp;有无参考基因组物种均适合;测序片段完整,snp标记转化成功率高。
5、全基因组关联分析(genome-wide association study,gwas)是一种对全基因组范围内的常见遗传变异(单核苷酸多态性和拷贝数)总体关联分析的方法,该方法以自然群体为研究对象,以长期重组后保留下来的基因(位点)间连锁不平衡(linkagedisequilibrium,ld)为基础,将目标性状表型的多样性与基因(或标记位点)的多态性结合起来分析,可直接鉴定出与表型变异密切相关且具有特定功能的基因位点或标记位点。采用gwas技术在全基因组范围内进行研究,能够一次性对多个性状进行定位,适用于定位性状关联区间、功能基因研究、开发性状选育和功能标记等方面的研究。gwas技术作为一种新的方法在动物育种领域得到广泛应用。全基因组关联分析旨在从全基因组范围内寻找与性状关联的单链核苷酸多态性(single nucleotide polymorphism,snps),其得到的结果更具可靠性。近几年,gwas在牛分子育种方面以辅助选育方式进行应用,在水牛分子育种方面还处于实验研究阶段。目前大部分gwas研究都是基于芯片分型技术,该技术只能检测已知snp多态位点,无法发现新的位点,且操作复杂,成本高。基于上述原因,急需开发一种通用型、经济实惠,便于操作适合水牛的gbs全基因组关联分析方法。为水牛育种的分子选育和遗传改良提供技术支撑。
技术实现思路
1、本发明的目的是提供一种用于水牛的gbs全基因组关联分析方法,对水牛群体的经济性状进行分析。
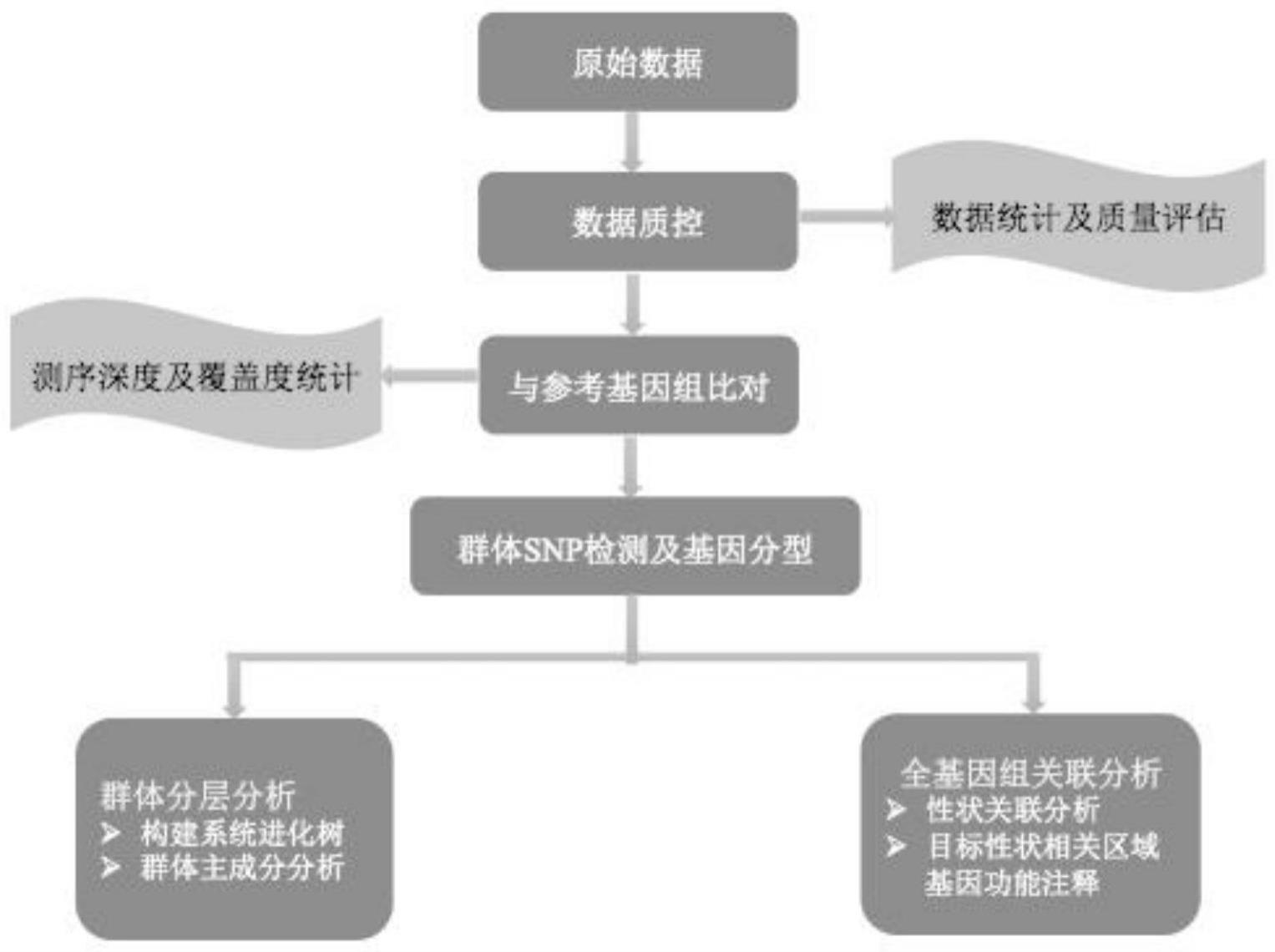
2、本发明的上述技术目的是通过以下技术方案得以实现的:一种用于水牛的gbs全基因组关联分析方法,所述全基因组关联分析方法包括以下步骤:
3、s1.测序数据质控;
4、s2.比对参考基因组;
5、s3.snp检测和注释;
6、s4.群体分层分析;
7、s5.全基因组关联分析。
8、本发明进一步设置为:所述测序数据质控方法如下:
9、1)过滤掉含有接头序列的水牛测序序列;
10、2)当单端测序序列中未测出的碱基含量超过该条序列长度比例的10%时,需要去除此对碱基序列;
11、3)当单端测序序列中含有的低质量(<=5)碱基数超过该条序列长度比例的50%时,需要去除此对碱基序列;
12、4)经过上述对水牛测序数据的严格过滤,得到高质量的有效数据。
13、本发明进一步设置为:所述比对参考基因是将s1中得到的有效数据与参考基因组做比对,得到比对率、平均测序深度以及其他相关数据。
14、本发明进一步设置为:所述snp检测和注释操作如下:
15、(1)检测水牛群体的snp位点,并对获得的多态性位点过滤,得到高质量的snp位点;
16、(2)对得到的高质量的snp位点进行群体snp注释。
17、本发明进一步设置为:所述群体分层分析可采用群体进化树分析和群体主成分分析两种分析方法。
18、本发明进一步设置为:所述全基因关联分析分为性状关联分析和目标性状相关区域基因功能注释两个步骤。
19、综上所述,本发明具有以下有益效果:利用该方法技术可以检测基因组上未知变异位点中新的snp,snp标记转化成功率高;一次测序获得数以百万计snp位点,密度高;获取的每个snp标记位点的成本比传统芯片技术降低一个数量级;该方法数据准确,技术稳定,操作简单,重复性高;利用高通量测序手段获得水牛snp标记与性状关联分析,能够更全面、更精准的定位于目标性状相关的基因或分子模块,为水牛育种的分子选育和遗传改良提供更为精准的候选靶标的效果。
技术特征:
1.一种用于水牛的gbs全基因组关联分析方法,其特征是:所述全基因组关联分析方法包括以下步骤:
2.根据权利要求1所述的一种用于水牛的gbs全基因组关联分析方法,其特征是:所述测序数据质控方法如下:
3.根据权利要求2所述的一种用于水牛的gbs全基因组关联分析方法,其特征是:所述比对参考基因是将s1中得到的有效数据与参考基因组做比对,得到比对率、平均测序深度以及其他相关数据。
4.根据权利要求1所述的一种用于水牛的gbs全基因组关联分析方法,其特征是:所述snp检测和注释操作如下:
5.根据权利要求1所述的一种用于水牛的gbs全基因组关联分析方法,其特征是:所述群体分层分析可采用群体进化树分析和群体主成分分析两种分析方法。
6.根据权利要求1所述的一种用于水牛的gbs全基因组关联分析方法,其特征是:所述全基因关联分析分为性状关联分析和目标性状相关区域基因功能注释两个步骤。
技术总结
本发明公开了一种用于水牛的GBS全基因组关联分析方法,涉及基因组关联分析技术领域,其技术方案要点是:该方法包括测序数据质控、比对参考基因、SNP检测和注释、群体分层分析和全基因组关联分析几个步骤。利用该方法技术可以检测基因组上未知变异位点中新的SNP,SNP标记转化成功率高;一次测序获得数以百万计SNP位点,密度高;获取的每个SNP标记位点的成本比传统芯片技术降低一个数量级;该方法数据准确,技术稳定,操作简单,重复性高;利用高通量测序手段获得水牛SNP标记与性状关联分析,能够更全面、更精准的定位于目标性状相关的基因或分子模块,为水牛育种的分子选育和遗传改良提供更为精准的候选靶标。
技术研发人员:郑海英,尚江华,杨春艳,郑威
受保护的技术使用者:广西壮族自治区水牛研究所
技术研发日:
技术公布日:2024/1/16
- 还没有人留言评论。精彩留言会获得点赞!