基于DNA序列数据的细胞特异性增强子预测方法与系统
本发明属于信息处理,尤其涉及基于dna序列数据的细胞特异性增强子预测方法与系统。
背景技术:
1、本部分的陈述仅仅是提供了与本发明相关的背景技术信息,不必然构成在先技术。
2、增强子是dna序列中的非编码片段,可调节基因表达和转录。作为一类调控元件,增强子控制着特异性基因表达、细胞生长和分化以及细胞癌变等各种细胞活动。增强子的突变或异常表达会破坏基因调控网络,从而影响细胞功能、组织发育和疾病进展。因此,增强子的鉴定对于研究基因表达和调控至关重要。
3、近年来已经提出了几种用于增强子识别的计算方法,但这些方法所基于的数据集存在两个问题:
4、首先,该数据集中的增强子被提取为固定长度(200bp)的短序列,导致这些方法是否可以用于不等长序列并在不等长序列上保持优越性能是未知的。
5、此外,该数据集是一个混合通用数据集,而研究表明增强子具有细胞特异性,在通用数据集上研究的增强子预测方法应用性不强。
6、因此,已有方法enhancer-if认识到这些不足,提取了八个细胞系的增强子,即采用基准数据集,以预测细胞特异性增强子,但该方法仍存在特征提取方案简单、预测细胞特异性增强子的性能不佳、性能验证不全面的缺点。
技术实现思路
1、为克服上述现有技术的不足,本发明提供了基于dna序列数据的细胞特异性增强子预测方法,用以识别细胞特异性增强子。
2、为实现上述目的,本发明的一个或多个实施例提供了如下技术方案:
3、第一方面,公开了基于dna序列数据的细胞特异性增强子预测方法,包括:
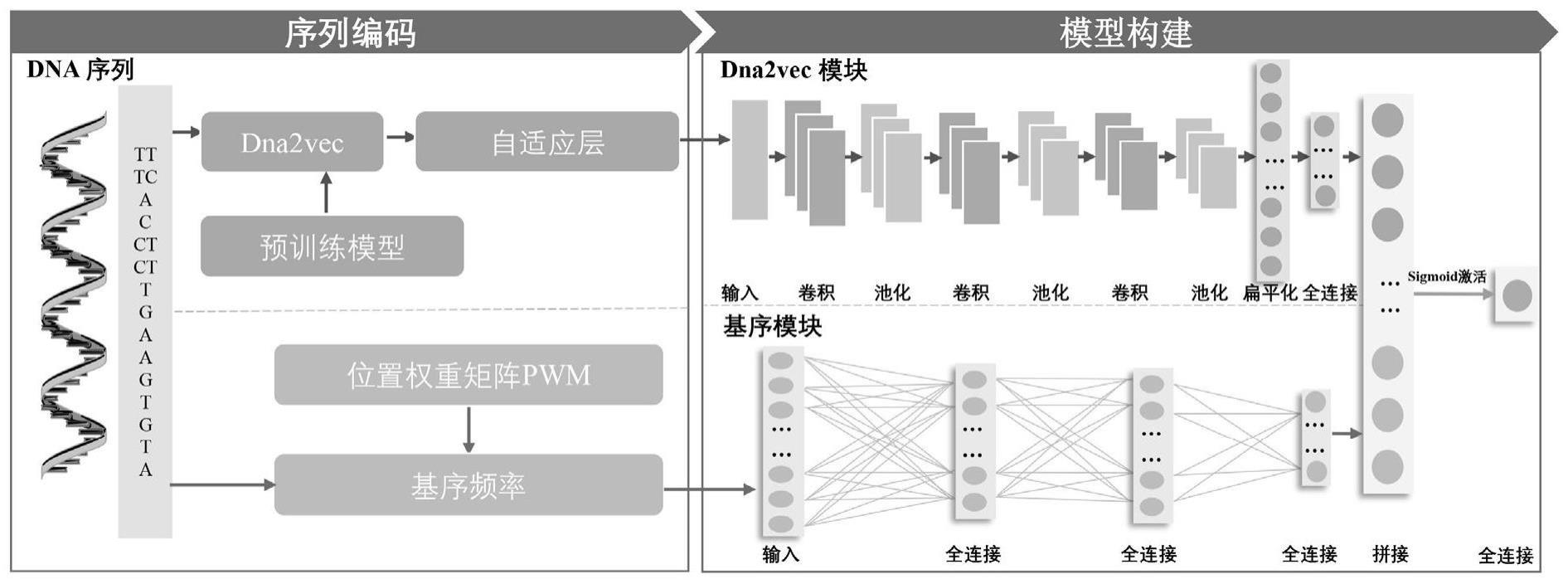
4、针对dna序列采用两种不同特征编码的方式提取序列信息,两种不同特征编码对应获得dna2vec特征和基序频率特征;
5、其中,采用dna2vec特征编码时,利用dna2vec中提供的预训练dna模型来索引序列编码;
6、采用基序频率特征编码时,提取每个dna序列中tfbs基序的数量,并将其转换为频率;
7、融合dna2vec特征和基序频率特征用来构建深度学习模型;
8、基于深度学习模型判断dna序列数据是否属于增强子。
9、作为进一步的技术方案,所述dna2vec用于计算dna序列中可变长度k-mers的分布式表示,其使用人类基因组序列作为学习语料库,在word2vec中使用连续skip-gram模型进行无监督训练,将k-mers嵌入到连续向量空间中。
10、作为进一步的技术方案,在word2vec中使用连续skip-gram模型进行无监督训练时,将3个核苷酸作为一个单词进行预训练性能最优。
11、作为进一步的技术方案,在word2vec中使用连续skip-gram模型进行无监督训练时,具体为:
12、设每条dna序列的长度为l,当k=3时,将其分为(l-2)个单词,使用dna2vec中的预训练模型,获得了每个单词对应的第一特征向量,所有单词的特征向量被连接起来,获得第二特征向量。
13、作为进一步的技术方案,使用自适应池化操作将特征维度归一化,以便将它们输入到深度学习模型中。
14、作为进一步的技术方案,提取每个dna序列中tfbs基序的数量时,从数据库中获得基序的位置权重矩阵,用于与该数据集中的序列数据进行滑动比例匹配,具体匹配规则:
15、假设基序的长度为lm,则pwm是一个行为lm、列为4的矩阵,表示当碱基分别为a、c、g和t时每个碱基的对应分数;
16、假设dna序列的长度为ls,将该序列划分为长度为lm的子序列片段,步长为1,从而得到ls-lm+1个子序列片段;
17、对于每个子序列片段,将每个碱基对应分数的总和作为最终匹配分数并与相应的阈值分数进行比较,以确定该子序列片段是否与基序匹配。
18、作为进一步的技术方案,子序列片段比较的规则如下:
19、其中,j的值取为0,1,2和3,对应于子序列片段中的碱基为a,c,g和t;q表示匹配分数;
20、假设p表示相应基序的p值阈值得分(10-4),则当q>p时,认为子序列片段与此基序匹配;
21、通过遍历每个序列,获得了每个dna序列中每个tfbs基序的数量信息。
22、作为进一步的技术方案,将基序数量除以每条序列长度得到的特征向量作为深度学习模型的输入。
23、作为进一步的技术方案,所述深度学习模型包括:dna2vec模块和一个基序模块;
24、dna2vec模块和基序模块分别使用dna2vec和基序频率这两种序列编码方案作为输入。
25、作为进一步的技术方案,在dna2vec模块中,数据交替通过三个一维卷积层和三个最大池化层以从dna序列中提取特征,其中,卷积层使用卷积计算提取输入的复杂特征,最大池化层采用下采样方法,输出每个子区域的最大值;
26、被提取到的特征通过dropout层,以便在训练阶段暂时从网络中随机删除某些神经元,防止过拟合。
27、作为进一步的技术方案,在基序模块中,数据通过三个全连接层降维;
28、被提取到的特征通过dropout层。
29、dna2vec模块经过特征提取后生成第三特征向量,基序模块生成第四特征向量,将这两个特征向量连接成一个一维向量,并使用具有一个神经元的线性层和“sigmoid”激活函数输出预测值;
30、如果预测值大于设定值,则认为样本为正样本,即认为该序列为增强子;否则,认为样本为负样本,即认为该序列为非增强子。
31、第二方面,公开了基于dna序列数据的细胞特异性增强子预测系统,包括:
32、序列信息提取模块,被配置为:针对dna序列采用两种不同特征编码的方式提取序列信息,两种不同特征编码对应获得dna2vec特征和基序频率特征;
33、其中,采用dna2vec特征编码时,利用dna2vec中提供的预训练dna模型来索引序列编码;
34、采用基序频率特征编码时,提取每个dna序列中tfbs基序的数量,并将其转换为频率;
35、深度学习模型构建模块,被配置为:融合dna2vec特征和基序频率特征用来构建深度学习模型;
36、增强子判断模块,被配置为:基于深度学习模型判断dna序列数据是否属于增强子。
37、以上一个或多个技术方案存在以下有益效果:
38、本发明使用了两个可用于提取序列信息的序列编码方案:dna2vec特征编码和基序频率特征编码,通过融合dna2vec特征和基序频率特征来构建深度学习模型并用于预测细胞特异性增强子。本发明提出的预测模型能够通过dna序列数据准确地判断该序列是否属于增强子,证明了本发明优于现有对特异性增强子进行预测的方法;本发明提出的预测模型在其他多个数据集(通用数据集、增强子-启动子数据集)上都表现出优越的性能,证明了本发明在实际应用中的鲁棒性。
39、本发明附加方面的优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。
- 还没有人留言评论。精彩留言会获得点赞!