一种小分子肽阿胶的多肽谱匹配数据分析方法及系统与流程
本发明涉及蛋白质数据分析,具体涉及一种小分子肽阿胶的多肽谱匹配数据分析方法及系统。
背景技术:
1、多肽谱数据分析是生物信息学和蛋白质组学领域关于质谱数据的处理,它在研究蛋白质的结构、功能、表达以及疾病相关蛋白质标志物的发现等方面发挥着重要的作用。其中质谱仪是用于分析多肽谱数据的关键设备。质谱仪可以通过质量-电荷比来测量样本中多肽离子的质量,并生成质谱图谱。其中,基于质谱数据进行蛋白鉴定和定量是多肽谱数据分析的主要任务之一。蛋白鉴定可以通过比较质谱数据与已知数据库的匹配情况来确定样本中存在的蛋白质。蛋白定量则是通过比较不同样本中特定质谱峰的强度或峰面积来定量不同样本中蛋白质的表达差异。
2、目前常见的,通过多肽谱数据与已知蛋白质数据库进行比对,对多肽谱数据中的肽段和蛋白质进行鉴定,但是由于有些多肽谱的特征峰的峰强度和峰分布相近,导致难以准确对多肽谱数据对应的蛋白质进行识别。
技术实现思路
1、为了解决通过多肽谱数据与已知蛋白质数据库进行比对时,存在难以准确对多肽谱数据对应的蛋白质进行识别的技术问题,本发明的目的在于提供一种小分子肽阿胶的多肽谱匹配数据分析方法及系统,所采用的技术方案具体如下:
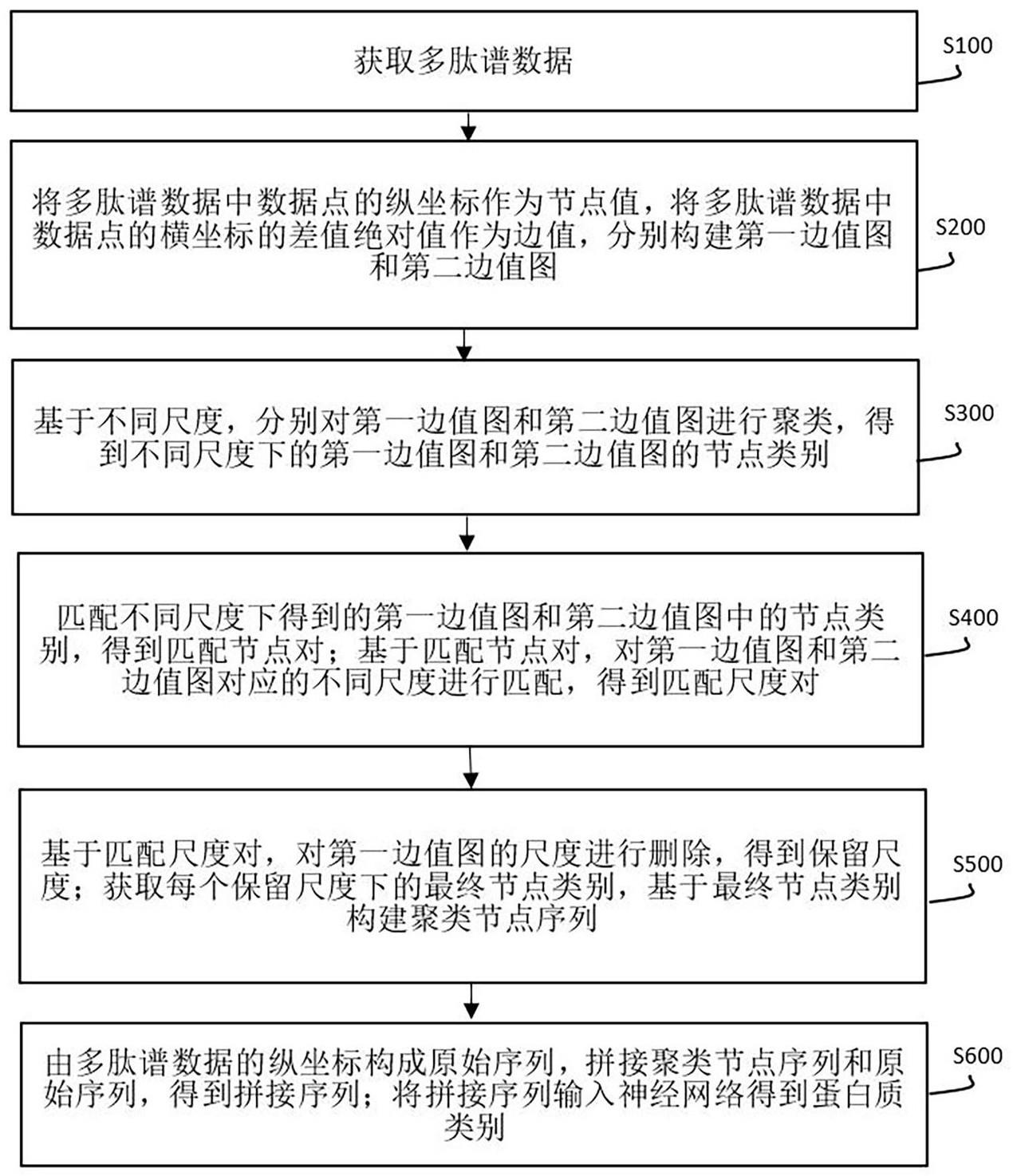
2、第一方面,本发明一个实施例提供了一种小分子肽阿胶的多肽谱匹配数据分析方法,该方法包括以下步骤:
3、获取多肽谱数据;
4、将多肽谱数据中数据点的纵坐标作为节点值,将多肽谱数据中数据点的横坐标的差值绝对值作为边值,分别构建第一边值图和第二边值图;
5、基于不同尺度,分别对第一边值图和第二边值图进行聚类,得到不同尺度下的第一边值图和第二边值图的节点类别;
6、匹配不同尺度下得到的第一边值图和第二边值图中的节点类别,得到匹配节点对;基于匹配节点对,对第一边值图和第二边值图对应的不同尺度进行匹配,得到匹配尺度对;
7、基于匹配尺度对,对第一边值图的尺度进行删除,得到保留尺度;获取每个保留尺度下的最终节点类别,基于最终节点类别构建聚类节点序列;
8、由多肽谱数据的纵坐标构成原始序列,拼接聚类节点序列和原始序列,得到拼接序列;将拼接序列输入神经网络得到蛋白质类别。
9、优选的,所述分别构建第一边值图和第二边值图,包括:
10、对于任意一条多肽谱数据,将每个纵坐标值作为节点值,将两个纵坐标值对应的横坐标的差值绝对值作为边值,构建图结构,作为原始图结构;
11、将原始图结构中的节点值去除,只保留节点与边值,得到第一边值图;
12、将原始图结构中的一个边值对应的两个节点的节点值的最小值与最大值的比值,替代原始的边值,并去除节点值,得到为第二边值图。
13、优选的,基于不同尺度,分别对第一边值图和第二边值图进行聚类,得到不同尺度下的第一边值图的节点类别,包括:
14、对第一边值图通过格里-纽曼算法进行聚类,当该算法中去除最大介数的边产生新类别时,记为一个新尺度,得到第一边值图在该尺度下的多个节点类别。
15、优选的,所述匹配不同尺度下得到的第一边值图和第二边值图中的节点类别,得到匹配节点对,包括:
16、对于第一边值图的任意尺度a下的节点类别与第二边值图的任意尺度b下的节点类别,将第一边值图在尺度a下的多个节点类别作为左侧节点,将第二边值图在尺度b下的多个节点类别作为右侧节点,将节点对应的节点类别的交并比作为边值,利用km匹配算法对第一边值图在尺度a下的多个节点类别和第二边值图在尺度b下的多个节点类别进行匹配,得到匹配节点对。
17、优选的,所述基于匹配节点对,对第一边值图和第二边值图对应的不同尺度进行匹配,得到匹配尺度对,包括:
18、将所有匹配节点对所对应的边值之和作为第一匹配值;
19、将第一边值图对应的每个尺度作为左侧节点,将第二边值图对应的每个尺度作为右侧节点,将两个节点对应的两个尺度下的类别数量中最小数量值与最大数量值的比值与第一匹配值的乘积作为边值,利用km匹配算法对第一边值图和第二边值图对应的不同尺度进行匹配,得到匹配尺度对。
20、优选的,所述基于匹配尺度对,对第一边值图的尺度进行删除,得到保留尺度,包括:
21、对于第一边值图,将尺度数量和预设倍数的乘积作为基准尺度,将基准尺度中的节点类别称之为第一节点类别,当第一节点类别中的节点在最大尺度到基准尺度下,都属于同一类别时,将对应的尺度作为保留尺度。
22、优选的,所述获取每个保留尺度下的最终节点类别,包括:
23、基于匹配节点对,对于每个第一边值图的保留尺度下的节点类别,得到在第二边值图中对应的节点类别;计算每个第一边值图中保留尺度下的节点类别和第二边值图中对应的节点类别的交集,将非交集的节点从第一边值图中保留尺度下的节点类别和第二边值图中对应的节点类别中进行删除,得到最终节点类别。
24、优选的,所述基于最终节点类别构建聚类节点序列,包括:
25、对于每个最终节点类别,将最终节点类别中元素在原始序列中最靠前的次序位置,作为首换位置,将首换位置之后的元素值置换为最终节点类别中的元素值,得到聚类节点序列。
26、优选的,所述神经网络的训练集为拼接序列,神经网络的损失函数为交叉熵损失函数。
27、第二方面,本发明一个实施例提供了一种小分子肽阿胶的多肽谱匹配数据分析系统,包括存储器、处理器以及存储在所述存储器中并可在所述处理器上运行的计算机程序,所述处理器执行所述计算机程序时实现上述一种小分子肽阿胶的多肽谱匹配数据分析方法。
28、本发明实施例至少具有如下有益效果:
29、本发明首先构建第一边值图和第二边值图,对第一边值图和第二边值图进行聚类,得到节点类别,实现先将边值图中的节点进行初分类,便于后续对节点类别进行分析,以体现多级蛋白质结构的目的;对第一边值图和第二边值图对应的不同尺度进行匹配,得到匹配尺度对,对第一边值图的尺度进行删除,得到保留尺度,通过聚类过程中节点所属类别由类别中节点数量多变为类别中节点数量少的过程,即由大尺度变为小尺度,整体向局部转换的过程来体现蛋白质的多级结构;获取每个保留尺度下的最终节点类别,基于最终节点类别构建聚类节点序列,通过把同一个节点类别的节点放到一块,形成聚类节点序列,使得该序列尽可能多的体现多级蛋白质结构;最后拼接聚类节点序列和原始序列得到拼接序列,将拼接序列输入神经网络得到蛋白质类别,其中拼接序列尽可能多的体现了多级蛋白质结构,进而辅助神经网络对多肽所属蛋白质类别的识别,大大提高了对多肽谱数据对应的蛋白质进行识别的准确度和精度。
技术特征:
1.一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,该方法包括以下步骤:
2.根据权利要求1所述的一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,所述分别构建第一边值图和第二边值图,包括:
3.根据权利要求1所述的一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,基于不同尺度,分别对第一边值图和第二边值图进行聚类,得到不同尺度下的第一边值图的节点类别,包括:
4.根据权利要求1所述的一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,所述匹配不同尺度下得到的第一边值图和第二边值图中的节点类别,得到匹配节点对,包括:
5.根据权利要求4所述的一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,所述基于匹配节点对,对第一边值图和第二边值图对应的不同尺度进行匹配,得到匹配尺度对,包括:
6.根据权利要求1所述的一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,所述基于匹配尺度对,对第一边值图的尺度进行删除,得到保留尺度,包括:
7.根据权利要求1所述的一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,所述获取每个保留尺度下的最终节点类别,包括:
8.根据权利要求1所述的一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,所述基于最终节点类别构建聚类节点序列,包括:
9.根据权利要求1所述的一种小分子肽阿胶的多肽谱匹配数据分析方法,其特征在于,所述神经网络的训练集为拼接序列,神经网络的损失函数为交叉熵损失函数。
10.一种小分子肽阿胶的多肽谱匹配数据分析系统,包括存储器、处理器以及存储在所述存储器中并可在所述处理器上运行的计算机程序,其特征在于,所述处理器执行所述计算机程序时实现如权利要求1~9任意一项所述一种小分子肽阿胶的多肽谱匹配数据分析方法的步骤。
技术总结
本发明涉及蛋白质数据分析技术领域,具体涉及一种小分子肽阿胶的多肽谱匹配数据分析方法及系统。该方法适用于对多肽谱数据对应的蛋白质识别鉴定的分析。基于获取的多肽谱数据构建第一边值图和第二边值图;删除部分第一边值图的尺度,得到保留尺度;获取每个保留尺度下的最终节点类别,构建聚类节点序列,通过聚类过程中节点所属类别由类别中节点数量多变为类别中节点数量少的过程,即由整体向局部转换的过程来体现蛋白质的多级结构;拼接聚类节点序列和原始序列,得到拼接序列;将拼接序列输入神经网络得到蛋白质类别,提高了对多肽谱数据对应的蛋白质进行识别的准确度和精度。
技术研发人员:邓来义,袁绪银,赵淑荣,潘少杰
受保护的技术使用者:山东鲁润阿胶药业有限公司
技术研发日:
技术公布日:2024/1/16
- 还没有人留言评论。精彩留言会获得点赞!