合成生物学功能元件的挖掘方法、装置、设备及存储介质
本发明涉及生物学元件挖掘,尤其涉及一种合成生物学元件挖掘方法、装置、电子设备及存储介质。
背景技术:
1、随着生命科学及相关学科的飞速发展,合成生物学应运而生。合成生物学旨在通过理性设计,精准建立或改造生物元件、基因回路与调控系统,以期实现特定的细胞功能。元件(parts)一般被认为是能执行特定生物学功能的最小dna序列单元。元件的类型多种多样,但是元件设计与搭建过程常常受到元件刻画不清楚、元件多样性缺乏等问题的困扰。合成生物学的大部分基础元件都是基于生物学发现发展而来的,其相关研究主要发表在生物化学和分子生物学领域,而这种发现依赖于多年的知识积累和专家的启发。基础元件被人们发现后,往往经过工程化建立可根据具体应用场景选择的元件库。相关技术中,合成生物学元件挖掘主要包括基于同源性元件挖掘和基因组特征或系统发育关系挖掘,其中根据同源性元件挖掘只能覆盖整个序列空间的一小部分,通过基因组特征或系统发育关系挖掘建立在对目标功能具有足够的生物机制或进化过程的理解上。为了用生物元件信息学方法实现元件的工程化挖掘,需要翻阅大量文献才能总结出挖掘所需的特征。同时,人工阅读文献可能遗漏其他领域非热点文献的有效信息,从而失去更多拓展元件多样性的机会。并且,因为自然语言具有上下文依赖和一词多义的特性,而生物学知识本身是丰富而复杂的,当前的实体识别算法无法从文本中识别功能描述,也无法将文献信息的丰富性与数据库的标准化和结构化相结合,导致无法找到更多含有预期功能的蛋白质或基因序列。
技术实现思路
1、本发明提供一种合成生物学元件挖掘方法、装置、电子设备及存储介质,用以解决相关技术中的元件挖掘方法覆盖范围小或挖掘所需的生物学知识建立在人工阅读文献总结的基础之上,效率低、有偏差,影响元件多样性拓展,以及,当前的实体识别工具无法从文本中识别功能描述,也无法将文献信息的丰富性与数据库的标准化和结构化相结合,导致无法找到更多含有预期功能的蛋白质或基因序列的缺陷。
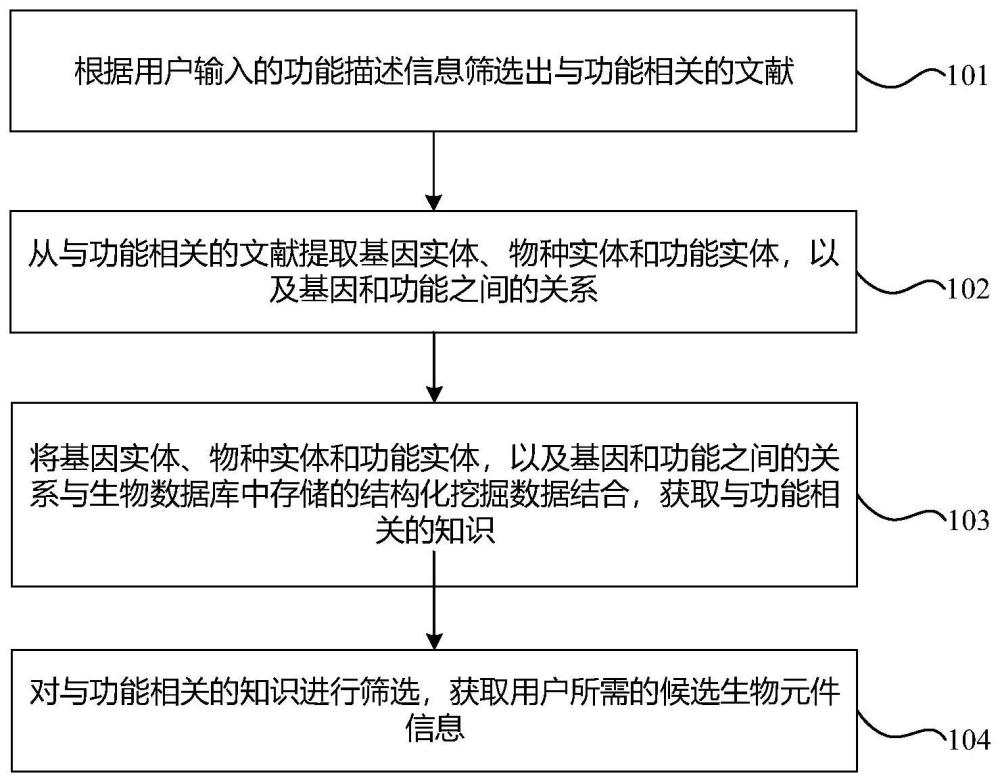
2、本发明提供一种合成生物学元件挖掘方法,包括:
3、根据用户输入的功能描述信息筛选出与所述功能相关的文献;
4、从与所述功能相关的文献提取基因实体、物种实体和功能实体,以及基因和功能之间的关系;
5、将基因实体、物种实体和功能实体,以及基因和功能之间的关系与生物数据库中存储的结构化挖掘数据结合,获取与所述功能相关的知识;
6、对与所述功能相关的知识进行筛选,获取用户所需的候选生物元件信息。
7、根据本发明提供的一种合成生物学元件挖掘方法,所述根据用户输入的功能描述信息与所述功能相关的文献,包括:
8、基于所述用户输入的功能描述信息获取全量文献数据;
9、在引文网络上进行搜索分群,将所述全量文献数据划分为多个论文簇并提取每个论文簇的主要主题;
10、获取用户选择的主题后,使用分类模型获取与选定主题相关的文献作为与所述功能相关的文献;
11、其中,所述分类模型以生物医学语言表示模型作为语义特征,基于文献标题、摘要文本和全文进行训练得到。
12、根据本发明提供的一种合成生物学元件挖掘方法,所述从与所述功能相关的文献提取基因实体、物种实体和功能实体,以及基因和功能之间的关系,包括:
13、基于字典与深度学习结合模型识别出基因实体和物种实体,所述字典与深度学习结合模型对生物医学语言表示模型进行微调的模型,其训练数据基于biose标记法进行标注;
14、基于功能实体识别模型识别出功能实体,所述功能实体识别模型的训练数据基于已标注样本使用主动学习方法在文献池中挑选预测信息熵的词语数量高于预设值的文章样本进行标注得到;
15、将所述基因实体、物种实体和功能实体匹配到标准的数据库上,获取有效基因序列条目和功能条目;
16、基于最近邻匹配方法,将所述功能条目中的每个生物学描述功能实体匹配上对应基因和物种,以获取基因和功能之间的关系。
17、根据本发明提供的一种合成生物学元件挖掘方法,所述将所述基因实体、物种实体和功能实体匹配到标准的数据库上,获取有效的基因序列条目与功能条目,包括:
18、将所述功能实体匹配到标准的数据库上,获取所有功能条目;
19、计算对于任意一个生物学功能实体的词嵌入之和p和功能条目的特征表征o的余弦相似度cos(o,p),其中o与p的长度相同;
20、对所有的功能条目根据余弦相似度从大到小进行排序,保留余排名前k个功能条目;
21、根据功能条目之间的包含关系构建树结构,所述树结构包括多个功能条目子节点和多个功能条目归并父节点;
22、获取包含功能条目子节点数量大于预设值的归并父节点,选取深度最深的预设数量归并父节点对应的功能条目作为有效功能条目。
23、根据本发明提供的一种合成生物学元件挖掘方法,所述将所述基因实体、物种实体和功能实体匹配到标准的数据库上,获取有效的基因序列条目与功能条目,包括:
24、根据关系抽取方法获取所述基因实体、物种实体对应的基因-物种对,将所述基因-物种对中的基因和物种做逻辑关系与运算后输入标准的数据库,以检索出有效的基因序列条目;
25、和/或,
26、将所述功能实体在标准的数据库上进行检索,获取所有功能条目;
27、根据目标数据库功能文本的长度设置滑窗窗口,在每个滑窗窗口内通过预训练的生物医学语言表示模型生成待匹配摘要文本的词嵌入向量;
28、计算每个滑窗内的词嵌入向量的和,以及每个功能条目的特征表征与每个滑窗内的词嵌入向量的和之间的余弦相似度;
29、保留余弦相似度最大值作为该功能条目与生物学描述功能实体的滑窗相似度;
30、设定余弦相似度的第一截断阈值和截断层级阈值,保留滑窗相似度大于第一截断阈值且层级数大于截断层级阈值的功能条目;
31、对保留的功能条目进行父节点归并,以保证所有留下的功能条目都没有父节点保留;
32、设定余弦相似度的第二截断阈值,根据滑窗相似度进行排序并将排序后的功能条目写入功能条目计算队列;
33、构建功能条目输出队列,每次从所述功能条目计算队列中取出一个滑窗相似度最高的功能条目,并与功能条目输出队列中所有元素计算余弦相似度;
34、若所述功能条目输出队列中不存在与之相似度大于第二截断阈值的功能条目则将该功能条目加入所述功能条目输出队列,将所述功能条目输出队列中的功能条目作为有效功能条目。
35、根据本发明提供的一种合成生物学元件挖掘方法,所述将基因实体、物种实体和功能实体,以及基因和功能之间的关系与生物数据库中存储的结构化挖掘数据结合,获取与所述功能相关的知识,包括:
36、对于生物数据库中存储的结构化挖掘数据进行聚簇操作,每个簇内包括不同数据的基因本体数据库注释与基因本体文本注释;
37、对所述基因本体数据库注释与基因本体文本注释进行基因本体富集操作以去除噪音得到清晰生物学概念;
38、对相似生物学概念对应的基因本体术语通过基因本体拓扑相似度聚类,对于每个具有代表性的基因本体术语,使用基因本体数据库注释背景频率计算并归一化具有此注释的数据的数量;
39、构建基因本体富集网络,边权重反映两个基因本体术语共享的数据的数量:
40、保留两个基因本体术语共享的数据的数量超过预设比例,且深度大于预设值的基因本体术语,将保留下来的基因本体术语间的基因本体富集网络作为与所述功能相关的知识。
41、根据本发明提供的一种合成生物学元件挖掘方法,所述对与所述功能相关的知识进行筛选,获取用户所需的候选生物元件信息,包括:
42、将所述与所述功能相关的知识发送至用户,以接收用户的对部分功能条目的评分结果;
43、将每个标记的功能条目分数根据富集网络边权重传递给其他基因本体,以得到所有功能条目的评分结果;
44、根据所述所有功能条目的评分结果获取用户所需的候选生物元件信息。
45、根据本发明提供的一种合成生物学元件挖掘方法,所述对与所述功能相关的知识进行筛选,获取用户所需的候选生物元件信息,还包括:
46、构建功能域网络,并对所述功能域网络中功能域注释进行聚类,得到功能域聚簇结果;
47、结合基因本体富集结果和与基因本体数据库的交叉引用进行分析,对序列进行功能域维度的打分;
48、根据打分结果获取用户所需的候选生物元件信息。
49、本发明还提供一种电子设备,包括存储器、处理器及存储在所述存储器上并可在所述处理器上运行的计算机程序,所述处理器执行所述程序时实现上述任一项所述的合成生物学元件挖掘方法。
50、本发明还提供一种非暂态计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现上述任一项所述的合成生物学元件挖掘方法。
51、本发明提供的合成生物学元件挖掘方法、装置、电子设备及存储介质,通过根据用户输入的功能描述信息与所述功能相关的文献;从与功能相关的文献提取基因实体、物种实体和功能实体,以及基因和功能之间的关系;将基因实体、物种实体和功能实体,以及基因和功能之间的关系与生物数据库中存储的结构化挖掘数据结合,获取与功能相关的知识;对与功能相关的知识进行筛选,获取用户所需的候选生物元件信息;通过将文本挖掘出的非结构化数据与生物医药领域数据库中结构化数据的有机融合,在基因、功能和知识结构三个层面进行交叉比对,实现了元件的通用化、智能化、自动化挖掘,是合成生物学丰富元件库、提高元件多样性的渠道。
- 还没有人留言评论。精彩留言会获得点赞!