基于知识图谱嵌入表示再学习的抗肿瘤药物药效预测方法
本发明涉及肿瘤细胞药物敏感性检测与评估,尤其是一种基于知识图谱嵌入表示再学习的抗肿瘤药物药效预测方法。
背景技术:
1、由于肿瘤的异质性和遗传的多样性,同种癌症的个体患者对即使相同的药物,也会得到不同的疗效反应。盲目用药将引起严重的毒副作用,甚至造成过度治疗。基于网络表示学习方法已被证明有效提取样本的基因调控特征,具有较好的肿瘤细胞药效敏感性预测能力。
2、然而,现有的基于网络表示学习方法在提取基因调控特征的过程中需要将样本融合到先验基因调控网络,进而学习融合网络嵌入表示。这种对所有样本构建融合网络的方式,忽略了新样本的加入需要重新构建融合网络表示学习模型这一缺陷,为现场应用带来不便,不利于预测能力的提升。
技术实现思路
1、为解决新样本的加入需要重新构建融合网络并重新训练融合网络表示学习模型的缺陷,本发明的目的在于提供一种通过对融合基因调控特征再学习,不仅解决了基因高通量数据的高维性,还提高了肿瘤细胞药物敏感性预测性能的基于知识图谱嵌入表示再学习的抗肿瘤药物药效预测方法。
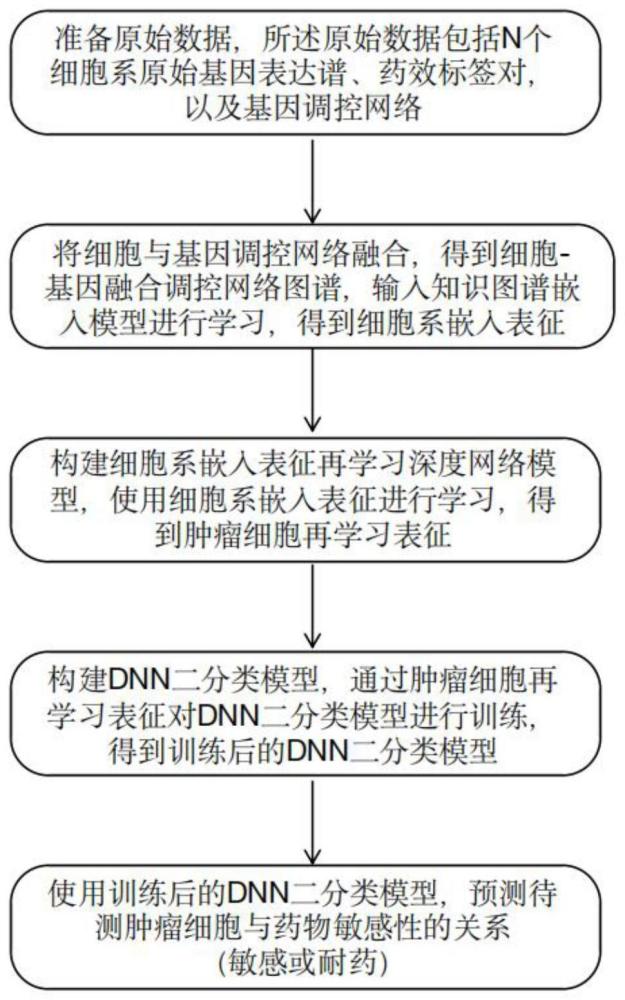
2、为实现上述目的,本发明采用了以下技术方案:一种基于知识图谱嵌入表示再学习的抗肿瘤药物药效预测方法,该方法包括下列顺序的步骤:
3、(1)准备原始数据:所述原始数据包括n个细胞系原始基因表达谱、药效标签对,以及基因调控网络;
4、(2)获得细胞系嵌入表征embed1:即将细胞与基因调控网络融合,得到细胞-基因融合调控网络图谱,将细胞-基因融合调控网络图谱输入知识图谱嵌入模型进行学习,得到细胞系嵌入表征embed1;
5、(3)构建细胞系嵌入表征再学习深度网络模型,使用细胞系嵌入表征embed1对细胞系嵌入表征再学习深度网络模型进行学习,得到肿瘤细胞再学习表征embed;
6、(4)构建dnn二分类模型,通过肿瘤细胞再学习表征embed对dnn二分类模型进行训练,得到训练后的dnn二分类模型;
7、(5)将肿瘤细胞再学习表征embed输入训练后的dnn二分类模型,预测待测肿瘤细胞与药物敏感性的关系。
8、所述步骤(2)具体包括以下步骤:
9、(2a)构建细胞-基因融合调控网络图谱:将所有肿瘤细胞节点与基因调控网络相融合,拟合肿瘤细胞样本基因表达的概率密度分布,将落在分位数z1-α以外的基因作为该细胞的热点基因,链接热点基因与肿瘤细胞节点,获得细胞-基因融合调控网络图谱;
10、(2b)将细胞-基因融合调控网络图谱输入知识图谱嵌入模型,计算所有肿瘤细胞样本的基因调控融合表达特征表示,具体包括以下步骤:
11、(2b1)抽取细胞-基因融合调控网络图谱中的正三元组;
12、(2b2)进行负三元组采样,得到负三元组集合,使用如下公式计算负三元组的重要性:
13、
14、其中,α是一个常数,代表采样率,(h′j,r,o′j)代表第j个负三元组样本,h′表示负三元组样本头部向量表示,o′表示负三元组样本尾部向量表示,r表示负三元组样本关系向量表示,pj=||h′o r-o′||为样本的打分函数,o表示哈德曼乘积;
15、(2b3)对得到的正三元组、负三元组进行打分以计算总损失loss:
16、
17、其中,g(h′i,r,o′i)是负三元组样本i的权重,m为负三元组样本数,б表示sigmoid激活函数,γ表示一个常数,p(h,r,o)为正三元组的打分函数;
18、(2b4)使用adam优化算法更新细胞-基因融合调控网络图谱的所有节点和边的调控融合特征表示;
19、(2b5)重复步骤(2b2)至步骤(2b4),直至步骤(2b3)所示的损失函数收敛,将细胞系节点的调控融合特征表示作为细胞系嵌入表征embed1。
20、所述步骤(3)具体包括以下步骤:
21、(3a)构建细胞系嵌入表征再学习训练集,所述细胞系嵌入表征再学习训练集由细胞系原始基因表达谱与细胞系嵌入表征embed1组成;
22、(3b)构建细胞系嵌入表征再学习深度网络模型,即一维卷积神经网络,一维卷积神经网络设置多个卷积层,通过设施不同的卷积核大小,经过卷积、激活、批标准化、池化操作对细胞系嵌入表征进行处理,在卷积后有一个全连接层作为整个卷积网络的输出,丢弃率dropout设置为0.5;
23、(3c)将细胞系嵌入表征再学习训练集输入构建的细胞系嵌入表征再学习深度网络模型,经过设置的一维卷积神经网络得到输出的肿瘤细胞再学习表征embed2,将肿瘤细胞再学习表征embed2与细胞系嵌入表征embed1进行对比,比较均方误差,将均方误差作为一维卷积神经网络的损失函数;
24、
25、其中,n为细胞系的个数,yi为肿瘤细胞再学习表征embed2中的值,为细胞系嵌入表征embed1中的值;
26、(3d)使用adam优化算法更新肿瘤细胞再学习表征embed2;
27、(3e)重复步骤(3c)至步骤(3d),直至步骤(3c)中的损失函数收敛,获得肿瘤细胞再学习表征embed。
28、所述步骤(4)具体包括以下步骤:
29、(4a)构建药效预测训练集,所述药效预测训练集由肿瘤细胞再学习表征embed与药效标签对组成;
30、(4b)构建dnn二分类模型:
31、(4b1)将药效预测训练集输入构建的dnn二分类模型,得到细胞系对药物敏感的概率,并判断输出为敏感还是耐药,得到敏感关系;dnn二分类模型设置多个隐藏层,并根据细胞系嵌入表征的维度设置层间单元数,dnn二分类模型的隐藏层l≥3,层间使用relu激活函数,输出层单元神经元数量为1,激活函数设置为sigmoid,以做分类任务;sigmoid函数输出的是事件概率,将输出规定在0和1之间,当结果大于某一阈值时,阈值取0.5,将其划分正类,即划分为敏感;
32、(4b2)根据步骤(4b1)得到的敏感关系,以及药效标签的真实敏感关系,计算二元交叉熵损失,以作为dnn二分类模型的损失函数;
33、
34、其中,n为细胞系的个数,yi为二元标签值0或者1,p(yi)为属于yi标签值的概率;
35、(4b3)使用adam算法优化dnn二分类模型输出的敏感关系;
36、(4b4)重复步骤(4b1)至步骤(4b3),直至步骤(4b2)的损失函数收敛,得到训练后的dnn二分类模型。
37、所述步骤(5)具体是指:使用训练后的dnn二分类模型,采用肿瘤细胞再学习表征embed预测待测肿瘤细胞与药物敏感性关系,敏感为1,耐药为0:
38、
39、其中,f表示训练后的dnn二分类模型,zi代表embed中待预测的第i个肿瘤细胞经过sigmoid函数输出药物反应为敏感的概率;如果输出药物反应为敏感的概率大于0.5,则输出1,表示对该药物敏感;如果输出药物反应为敏感的概率小于0.5,则输出0,表示对该药物耐药。
40、由上述技术方案可知,本发明的有益效果为:第一,通过对原始基因表达谱和细胞系嵌入表征构建卷积神经网络模型,实现新样本直接使用训练好的模型进行表征表示,解决了现有方法中新样本的加入需要重新训练模型的缺陷;第二,通过对细胞系嵌入表征再学习,整合了肿瘤细胞系原始表达谱、药效标签对,以及基因调控网络信息,提高了肿瘤细胞药物敏感性预测性能;第三,引入深度学习编码技术解决了基因高通量数据高维性难题。
- 还没有人留言评论。精彩留言会获得点赞!