在甲状腺肿瘤分类中的MIRNA表达特征的制作方法
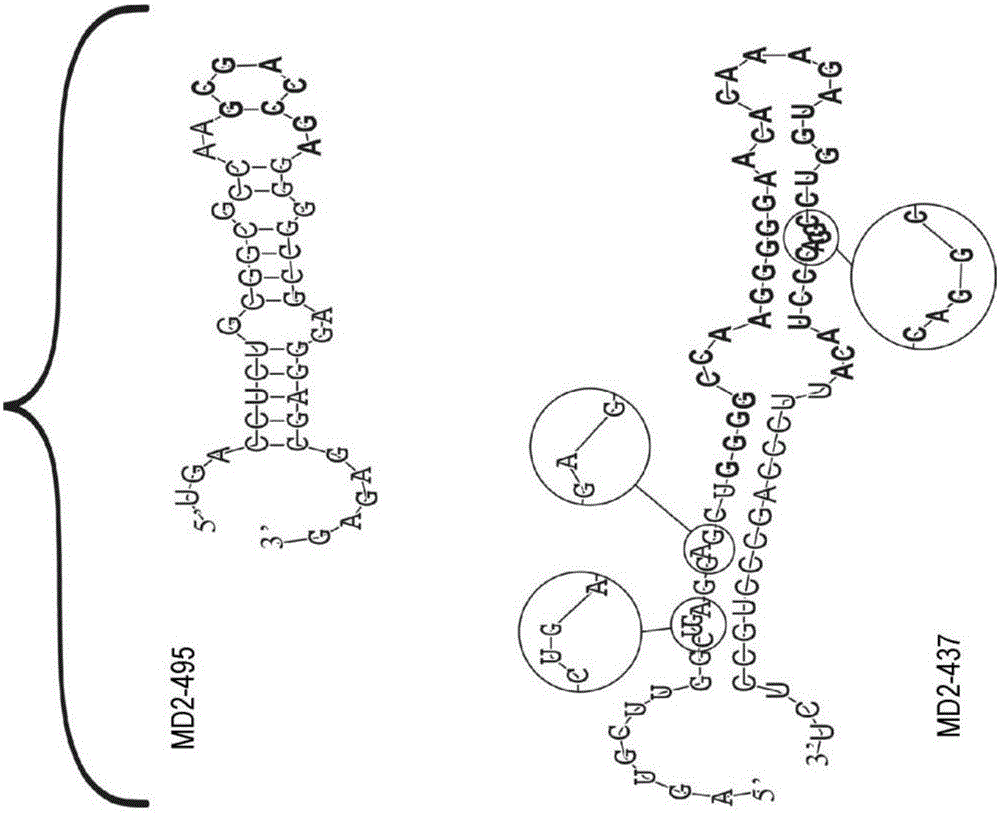
本发明涉及用于甲状腺肿瘤分类的方法。本发明尤其涉及与特定甲状腺肿瘤相关的微RNA分子。发明背景甲状腺结节的准确诊断持续挑战着治疗甲状腺疾病患者的医生。通常对具有细胞学上未定类结节的患者进行诊断性手术,尽管大部分这些结节术后证实为良性。FNA细胞学在术前诊断的这种限制导致临床需要可靠的术前分子标记以区分良性甲状腺结节和恶性甲状腺结节。微RNA(miR)是一类重要的调控RNA,其对于广泛的生物过程具有深远影响。这些小的(通常为18至24个核苷酸长度)非编码RNA分子可以通过促进RNA降解、抑制mRNA翻译并且还通过影响基因转录而调节蛋白质表达模式。miR在多种过程例如发育和分化、控制细胞增殖、应激反应和新陈代谢中起到关键作用。在多种人体癌症中发现许多miR的表达被修改,在一些情况下表明这种修改可以为肿瘤发展的成因。甲状腺由两种主要类型的细胞形成:滤泡细胞和C细胞或滤泡旁细胞。滤泡细胞产生甲状腺激素,其为人体新陈代谢的调节剂。甲状腺激素的过量产生(甲状腺功能亢进)引起快速的或不规则的心跳、睡眠问题、神经质、饥饿、体重减轻和太过温暖的感觉。相反,甲状腺功能减退会引起新陈代谢放缓、疲劳和体重增加。甲状腺激素的释放通过由脑下垂体产生的促甲状腺激素(TSH)调节。C细胞产生降血钙素,其为负责钙的使用的激素。淋巴细胞和基质细胞也存在于甲状腺中。甲状腺癌在美国是第八大最常见的癌症,是在美国最快速增加的癌症,每年有超过60000的新诊断案例,在2014年引起约1800人死亡。甲状腺癌表现为可触及的甲状腺结节。不同类型的甲状腺肿瘤发展自不同类型的细胞,其为严重性和最佳施用疗法的决定因素。甲状腺中的多数赘生物和肿瘤是良性的(非癌性的),然而其他是恶性的(癌性的)。大约95%的甲状腺癌为由甲状腺滤泡细胞产生的分化型甲状腺癌(DTC)。DTC有两个组织学亚型:乳头状甲状腺癌(PTC)型(90%至95%)和滤泡性甲状腺癌(FTC)型(5%至10%)。诊断甲状腺癌最常用的方法为通过细针穿刺(FNA)的活组织检查。将FNA样本进行细胞学的常规检查以确定结节是良性的或是恶性的。取决于机构和医生,FNA样本细胞学检查的敏感性和特异性分别为68%至98%和72%至100%。不幸的是,在采集的FNA标本的情况中至少25%或不足以进行诊断或无法通过细胞学确定。在现有的医疗实践中,未确定结果的多数患者经历手术,并承受手术过程的所有风险和后果。随访结果显示仅有25%的手术患者诊断患有癌症,这意味着75%的患者经受了不必要的手术过程。当检测细胞化学标记或基因标记时,没有能够独自提供可靠结果的独特标记以替代甲状腺病变的形态学诊断。US7319011描述了检测在测试滤泡性甲状腺标本中的基因DDIT3、ARG2、ITM1、C1orf24、TARSH和ACO1中的任一个的表达以区分滤泡性腺瘤(FA)和滤泡性癌(FC)。US7670775描述了CCND2、PCSK2和PLAB的表达分析以鉴别恶性甲状腺组织。US6723506描述了与诊断和治疗甲状腺滤泡性癌有关的PAX8-PPAR1分子的分子表征。US7378233描述了在乳头状甲状腺癌的24例(69%)中出现BRAF基因的T1796A突变。在寻求会克服未定类细胞学的不确定性并最终消除对非癌患者的不必要手术的分子诊断测试方面已经进行了不断努力[Chen,Y.T.等人.(2008)Mod.Pathol.21,1139–1146;He,H.等人.(2005)Proc.NatlAcad.Sci.USA102,19075–19080;Nikiforova,M.N.等人.(2009)Endocr.Pathol.20,85–91;Pallante,P.等人.(2006)Endocr.Relat.Cancer13,497–508;Nikiforova,M.N.等人.(2008)J.Clin.Endocrinol.Metab.93,1600–1608;Visone,R.等人.(2007)Endocr.Relat.Cancer14(3):791-8;US2014/0030714A1;US8541170;US2012/0220474A1;US8465914;US7598052;US8202692;WO2013/066678;WO2012/129378;US2013/0237590;EP2772550A1;Pallante等人.(2010)Endocrine-RelatedCancer17F91-F104;Dettmer等人.(2014)JMolEndocrinol.Mar6;52(2):181-9]。尽管如此,仍存在大量挑战。开发不仅具有高敏感性和特异性、还能够处理无法进行细胞学分析而被列入未定类样品类别的样本的分子试验有很大的必要性。本发明提供了对该挑战的解决方案。发明概述本发明人研发了新的整合技术平台用于分析和表征甲状腺临床样本中的微RNA,其包括一般由手术获得的切除的活组织检查和一般由细针穿刺(FNA)获得的细胞学标本,用于将甲状腺病变分类为良性或恶性及其亚型。公开了作为潜在生物标记的新型微RNA。因此,在第一方面,本发明提供了甲状腺病变样本的分类方法,该方法包括以下步骤:a.从有需要的受试者中获得甲状腺病变样本;b.测量样本中至少四种核酸的表达水平,所述核酸包含SEQIDNO1至SEQIDNO308的序列、其变体或具有与其至少约80%一致性的序列;c.确定核酸表达谱;d.对核酸表达谱应用分类器算法:e.基于应用于所述样本的核酸表达谱的算法结果,将所述甲状腺病变分类为良性肿瘤、恶性肿瘤、或良性肿瘤或恶性肿瘤的亚型。在本发明方法的一个实施方式中,在步骤(b)或(c)之后还包括获得至少一对核酸表达水平的比例德步骤;其中在步骤(d)中所述分类器算法可应用于核酸表达谱、至少一对核酸的所述比例、或其组合中的任一个。在本发明方法的一个实施方式中,所述核酸序列包含SEQIDNO.1至SEQIDNO.37中任一的序列、其变体、或具有与其至少约80%一致性的序列。在本发明方法的另一个实施方式中,所述核酸序列包含SEQIDNO.1至SEQIDNO.25中任一的序列、其变体、或具有与其至少约80%一致性的序列。在本发明方法的另一个实施方式中,所述甲状腺病变样本通过细针穿刺(FNA)活组织检查获得。在一个具体实施方式中,所述样本为来自FNA活组织检查的涂片。在本发明方法的另一个实施方式中,所述甲状腺病变为小于1cm的结节。在本发明方法的另一个实施方式中,算法为机器学习型算法。在本发明所述方法的一个具体实施方式中,所述算法还使核酸表达谱结合来自所述样本的临床数据或基因数据。在本发明方法的另一个实施方式中,在步骤(b)之后当至少一种所述核酸的表达水平低于或高于甲状腺细胞的阈值时,基于所述核酸的表达水平将所述样本舍弃。在本发明方法的另一个实施方式中,所述样本具有少于50个甲状腺细胞。在本发明方法的另一个实施方式中,通过杂交、扩增或者下一代测序方法实施所述测量。在本发明方法的一个具体实施方式中,所述杂交包括使样本接触探针,其中探针包括(i)微RNA的DNA等同物,(ii)其互补物,(iii)与(i)或(ii)至少80%一致性的序列,或(iv)与SEQIDNO.1至SEQIDNO.25中的任一种的至少八个连续核苷酸杂交的核酸序列。在本发明方法的另一个具体实施方式中,所述探针连接于固体载体。在本发明方法的另一个具体实施方式中,扩增为实时聚合酶链反应(RT-PCR),所述RT-PCR扩增方法包括正向引物和反向引物,任选地包括用探针进行杂交。在本发明方法的另一个实施方式中,所述方法还包括根据所述甲状腺病变为良性或恶性向所述受试者施用差异化治疗。在本发明方法的另一个具体实施方式中,所述病变为恶性的,所述治疗为手术、化疗、放疗、激素疗法或任意其他推荐治疗中的任一种。在另一方面,本发明提供用于将甲状腺病变样本分类的方案,该方案包括以下步骤:a.从有需要的受试者中获得甲状腺病变样本;b.测量样本中至少四种核酸的水平,所述核酸包含序列SEQIDNO.1至SEQIDNO.308、其变体或具有至少80%与其一致性的序列;c.确定在所述样本中与特定细胞类型有关的核酸表达;d.其中(i)至少一种非甲状腺细胞标记的核酸的表达水平在阈值以上确定该样本被舍弃;或(ii)非甲状腺细胞标记的表达水平在阈值以下确定该样本继续进行步骤(e)用于进一步分析;e.当样本未在步骤(d)中被舍弃时,确定核酸表达谱;f.对微RNA表达谱应用分类器算法;g.基于应用于所述样本的核酸表达谱的算法结果,将所述甲状腺病变分类为良性肿瘤、恶性肿瘤、或良性肿瘤或恶性肿瘤的亚型。在本发明方案的一个实施方式中,在步骤(b)之后还包括获得至少一对核酸表达水平比例的步骤;其中在步骤(f)中所述分类器算法可以应用于核酸表达谱、至少一对核酸的所述比例或其组合中的任一个。在本发明方案的另一个实施方式中,所述核酸序列包含SEQIDNO.1至SEQIDNO.37中任一的序列、其变体或具有与其至少约80%一致性的序列。在本发明方案的另一个实施方式中,所述核酸序列包含SEQIDNO.1至SEQIDNO.25中任一的序列、其变体或具有与其至少80%一致性的序列。在本发明方案的另一个实施方式中,所述甲状腺病变样本通过细针穿刺(FNA)活组织检查获得。在一个具体实施方式中,所述样本为来自FNA活组织检查的涂片。在本发明方案的另一实施方式中,所述甲状腺病变为小于1cm的结节。在本发明方案的另一实施方式中,所述样本具有少于50个甲状腺细胞。在本发明方案的另一个实施方案中,所述算法为机器学习型算法。在本发明方案的另一个实施方案中,通过杂交、扩增或者下一代测序方法实施测量。在另一方面,本发明提供了用于甲状腺肿瘤分类的工具盒,所述试剂盒包含:a.用于进行甲状腺肿瘤分类的探针,其中所述探针包含以下任一个:(i)包含序列SEQIDNO1至SEQIDNO308中至少一种的微RNA的DNA等同物,(ii)其互补物;(iii)与(i)或(ii)至少80%一致性的序列,(iv)与SEQIDNO1至SEQIDNO182中任一种的至少八个连续核苷酸杂交的核酸序列,或者(v)与RT-PCR产物杂交的核酸序列;任选地b.用于使用所述探针的说明书。在一个实施方式中,所述试剂盒还包含正向PCR引物和反向PCR引物。在另一个实施方式中,本发明的试剂盒可包含正向引物和反向引物。在另一个实施方式中,本发明的试剂盒还可以包含用于进行原位杂交分析的试剂。在本发明的另一方面,用于甲状腺肿瘤分类的试剂盒包含:(a)至少一种正向RT-PCR引物,例如包含序列SEQIDNO.270至SEQIDNO.293的引物中的至少一种;(b)反向引物;(c)与由RT-PCR扩增的分子进行杂交的至少一种探针,例如在实施例中提供的探针;任选地(d)用于实施所述RT-PCR的说明书、或用于甲状腺肿瘤分类的说明书中的任一种。在一个实施方式中,所述探针为普通探针。在另一个实施方式中,所述探针为微RNA序列特异性探针。在另一方面,本发明提供了分离核酸,所述核酸包含与SEQIDNO.27至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139、SEQIDNO.140、SEQIDNO.307和SEQIDNO.308中的任一序列至少80%一致性的至少12个连续核苷酸。在另一方面,本发明提供了药物组合物,其包含本文描述的分离核酸作为活性剂,和任选的辅料、载体、稀释剂和赋形剂。因此,所述核酸分子可作为活性剂包含于药物组合物、制剂或药剂中。在另一方面,本发明提供了包含本文描述的分离核酸的载体。在另一方面,本发明提供了包含本文描述的分离核酸的探针。在另一方面,本发明提供了包含本文描述的分离核酸的生物芯片。在另一方面,本发明提供了本文描述的分离核酸在药剂制备中的用途。附图说明图1:在Giemsa染色的乳头状癌(Pap-carc.)涂片和非乳头状癌(N-Pap-carc.)涂片中的微RNA表达。散点图显示来自Giemsa染色的乳头状癌涂片(y轴,n=1)相对于Giemsa染色的非乳头状癌涂片(x轴,n=1)的微RNA(miR)差异化表达。如通过微阵列所测量的,数据以归一化荧光单位显示。平行线描述在任一方向的样品之间的1.5倍变化。在两个样品中,灰十字表示未测试(NT)的对照探针或中值信号<300。在乳头状癌涂片中上调五种微RNA(hsa-miR-146b-5p、hsa-miR-222-3p、hsa-miR-221-3p、hsa-miR-21-5p和hsa-miR-31-5p)。图2A至2B:通过下一代测序检测的新型微RNA。图2A显示在甲状腺组织中检测的两种新型微RNA、MD2-495(上)和MD2-437(下)预测的二级结构。图2B显示在11个切除的甲状腺样本中的每一个中两种新型微RNA的表达。图3A至3B:恶性样本中的微RNA表达相对于良性样本中的微RNA表达。散点图显示在恶性结节中微RNA(y轴)的中值表达水平相对于良性结节中微RNA(x轴)的中值表达水平,微RNA包括miR-125b-5p、miR-222-3p和miR-146b-5p(高亮)。每个十字表示微RNA,并包括对照的序列、低表达的微RNA和不可靠的探针(NT)。虚线表示1.5倍。图3A显示了在组I中的分析。图3B显示了在组II中的分析。图4:MiR-375在髓样病变中的表达。该图显示miR-375在髓样病变(菱形,Med)中的表达对比于在恶性非髓样病变(方形,Mal-n-med)中的表达以及在良性病变中的表达(圆形,B)。线表示每组的中值表达。p值=1.2e-42。倍数变化=201.4。图5A至5B:可以用不同染料染色样本,可以检测微RNA。该图显示在用不同染料染色的恶性样本(M)或良性样本(B)中miR-146b-5p的中值表达水平。图5A显示用May-GrünwaldGiemsa染色的样本中miR表达对比于用DiffQuik染色的样本中miR表达。p值=0.18(Wilcoxon)。倍数变化中值(med.f-ch)=1.0。图5B显示用DiffQuik染色的样本中miR表达对比于在Papanicolaou染色的样本中miR表达。p值=0.56(Wilcoxon)。倍数变化中值(med.f-ch)=1.1。图6A至6B:Hurthle细胞标记。该图显示了相对于没有Hurthle细胞指示的滤泡性腺瘤,在显示Hurthle细胞的滤泡性腺瘤中的MID-16582表达更高。Sign.=显著的;Diff.=差异的;f-ch=倍数变化;Bl.=血液;NT,未测试。图6A:y轴和x轴显示在未记录为具有Hurthle细胞的FA(滤泡性腺瘤)样本(n=22)中miR的表达水平的中值数组相对于具有Hurthle细胞的FA样本(n=9)中miR的表达水平的中值数组。虚线系数线=×1.5。Bl.=血液。NT,未测试。图6B:y轴和x轴显示在未显示Hurthle细胞的FA样本(n=21)中miR的PCR表达水平的中值相对于具有Hurthle细胞指示的FA样本(n=9)中miR的PCR表达水平的中值。虚线系数线=±0.6。图7:具有微RNA的甲状腺检测组的恶性样本和良性样本的曲线图。x轴和y轴分别显示在良性(B)样本(n=166)中miR的表达水平相对于在恶性(M)样本(n=187)中miR的表达水平。hsa-miR-222-3p(SEQIDNO.1至SEQIDNO.2)、hsa-miR-551b-3p(SEQIDNO.3至SEQIDNO.4)、hsa-miR-31-5p(SEQIDNO.5至SEQIDNO.7)、hsa-miR-125b-5p(SEQIDNO.9)、hsa-miR-146b-5p(SEQIDNO.10至SEQIDNO.11)、hsa-miR-152-3p(SEQIDNO.12至SEQIDNO.13)、hsa-miR-346(SEQIDNO.14)、hsa-miR-181c-5p(SEQIDNO.15)、hsa-miR-424-3p(SEQIDNO.16)、和hsa-miR-375(SEQIDNO.8)的微RNA中值表达水平为高亮的。数量指(50-归一化的Ct值)。菱形(◆)表示正规化子。Sign.=显著的;Diff.=差异的;f-ch=倍数变化。虚线系数线=±0.6。图8A至8C:使用微RNA表达值,将判别式分析分类器用于分类恶性(菱形,M)样本和良性(方形,B)样本。图8A:两种微RNA(hsa-miR-551b-3p和hsa-miR-146b-5p)的归一化值用作分类的特征。该分类器的敏感性为84.8%且特异性为68.9%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图8B:三种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p和hsa-miR-31-5p)的归一化值用作为分类的特征。该分类器的敏感性为82.9%且特异性为72.2%。分类错误的样本(miscl.)由点表示。图8C:8种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-375、hsa-miR-125b-5p、hsa-miR-152-3p、hsa-miR-181c-5p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为83.5%且特异性为81.5%。图9A至9C:使用微RNA表达比例的归一化值,判别式分析分类器用于将恶性(菱形,M)样本和良性(方形,B)样本分类。图9A:两种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p和hsa-miR-31-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为78%且特异性为79.5%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图9B:三种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p)的归一化值用作为分类的特征。该分类器的敏感性为81.1%且特异性为82.1%。分类错误的样本(miscl.)由点表示。图9C:8种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-200c-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为74.4%且特异性为84.1%。图10A至10C:使用微RNA和微RNA比例的组合的归一化值,判别式分析分类器用于将恶性(菱形,M)样本和良性(方形,B)样本分类。图10A:一种微RNA比例和一种微RNA(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为82.9%且特异性为82.8%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图10B:一种微RNA比例和两种微RNA(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p)的归一化值用作为分类的特征。该分类器的敏感性为82.9%且特异性为82.8%。图10C:5种微RNA和3种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-375)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为93.3%且特异性为42.4%。图11A至11C:使用微RNA的归一化值,K最近邻算法(KNN)分类器用于将恶性(M)样本和良性(B)样本分类。图11A:6种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-375、hsa-miR-125b-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为82.3%且特异性为68.2%。图11B:8种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-375、hsa-miR-125b-5p、hsa-miR-152-3p、hsa-miR-181c-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为82.9%且特异性为74.2%。图11C:12种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-375、hsa-miR-125b-5p、hsa-miR-152-3p、hsa-miR-181c-5p、hsa-miR-486-5p、hsa-miR-424-3p、hsa-miR-200c-3p、hsa-miR-346)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为81.1%且特异性为68.9%。图12A至12B:使用微RNA比例的归一化值,KNN分类器用于将恶性(M)样本和良性(B)样本分类。图12A:6种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该图显示了其中x轴表示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为78%且特异性为58.9%。图12B:8种miR比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-200c-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示了其中x轴表示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为80.5%且特异性为65.6%。图13A至13C:使用微RNA和微RNA比例组合的归一化值,KNN分类器用于将恶性(M)样本和良性(B)样本分类。图13A:4种微RNA和2种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-31-5p、hsa-miR-222-3p)的归一化值用作为分类的特征。该图显示了其中x轴表示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为85.4%且特异性为66.9%。图13B:5种微RNA和3种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-375)的归一化值用作为分类的特征。该图显示了其中x轴表示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为83.5%且特异性为70.9%。图13C:7种微RNA和5种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-375、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-152-3p)的归一化值用作为分类的特征。该图显示了其中x轴表示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为83.5%且特异性为66.9%。图14A至14C:使用归一化的微RNA值,支持向量机(SVM)分类器用于将恶性(菱形,M)样本和良性(方形,B)样本分类。图14A:三种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p和hsa-miR-31-5p)的归一化值用作为分类的特征。该分类器的敏感性为82.3%且特异性为68.2%。分类错误的样本(miscl.)由点表示。图14B:6种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-375、hsa-miR-125b-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为83.5%且特异性为75.5%。图14C:8种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-375、hsa-miR-125b-5p、hsa-miR-152-3p、hsa-miR-181c-3p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为86%且特异性为75.5%。图15A至15C:使用微RNA比例的归一化值,SVM分类器用于将恶性(菱形,M)样本和良性(方形,B)样本分类。图15A:三种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p)的归一化值用作为分类的特征。该分类器的敏感性为83.5%且特异性为80.8%。分类错误的样本(miscl.)由点表示。图15B:6种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为83.5%且特异性为80.1%。图15C:8种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-200c-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为82.9%且特异性为80.8%。图16A至16C:使用微RNA值和微RNA比例组合的归一化值,SVM分类器用于将恶性(菱形,M)样本和良性(方形,B)样本分类。图16A:2种微RNA和1种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p)的归一化值用作为分类的特征。该分类器的敏感性为82.9%且特异性为83.4%。分类错误的样本(miscl.)由点表示。图16B:4种微RNA和2种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-31-5p、hsa-miR-222-3p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为86%且特异性为80.1%。图16C:5种微RNA和3种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-375)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为86.6%且特异性为79.5%。图17A至17C:使用微RNA的归一化值,判别式分析集成分类器用于将恶性(菱形,M)样本和良性(方形,B)样本分类。图17A:两种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p)的归一化值用作为用于分类的特征。该分类器的敏感性为84.8%且特异性为64.2%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图17B:三种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p和hsa-miR-31-5p)的归一化值使作为分类的特征。该分类器的敏感性为84.1%且特异性为65.6%。分类错误的样本(miscl.)由点表示。图17C:8种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-375、hsa-miR-125b-5p、hsa-miR-152-3p、hsa-miR-181c-3p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为84.8%且特异性为74.8%。图18A至18C:使用微RNA比例的归一化值,判别式分析集成分类器用于将恶性(菱形,M)样本和良性(方形,B)样本分类。图18A:两种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为83.5%且特异性为73.5%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。Fig.18B:三种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p)的归一化值用作为分类的特征。该分类器的敏感性为86%且特异性为79.5%。分类错误的样本(miscl.)由点表示。图18C:8种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-200c-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴表示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为84.1%且特异性为78.1%。图19A至19C:使用微RNA和微RNA比例的归一化值的组合,判别式分析集成分类器用于将恶性(菱形,M)样本和良性(方形,B)样本分类。图19A:一种微RNA和一种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为85.4%且特异性为78.8%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图19B:2种微RNA和1种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p)的归一化值用作为分类的特征。该分类器的敏感性为85.4%且特异性为78.1%。分类错误的样本(miscl.)由点表示。图19C:5种微RNA和3种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-375)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为86%且特异性为82.8%。图20:髓样(“Med.”)样本、非髓样恶性(“Mal.”)样本和良性(“Ben.”)样本的归一化的hsa-miR-375表达(Exp.)水平以点阵图显示。线表示每组的中值。在每组内,为提高点的可见性,点沿x轴随机分布。图21:非髓样恶性(“Mal.”)样本和良性(“Ben.”)样本的归一化的hsa-miR-146b-5p表达(Exp.)水平以点阵图显示。线表示每组的中值。在每组内,为提高点的可见性,点沿x轴随机分布。图22:非髓样恶性(“Mal.”)样本和良性(“Ben.”)样本的归一化的微RNA比例hsa-miR-146b-5p:hsa-miR-342-3p表达水平以点阵图显示。线表示每组的中值。在每组内,为提高点的可见性,点沿x轴随机分布。图23A至23C:使用微RNA的归一化值,判别式分析分类器用于将不确定的恶性(菱形,M)样本和良性(方形,B)样本分类。图23A:两种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为80%且特异性为56.3%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图23B:三种微RNA(hsa-miR-146b-5;hsa-miR-551b-3p;hsa-miR-222-3p)的归一化值用作为用于分类的特征。该分类器的敏感性为82.6%且特异性为59.5%。分类错误的样本(miscl.)由点表示。图23C:8种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-125b-5p、hsa-miR-31-5p、hsa-miR-375、hsa-miR-152-3p、hsa-miR-181c-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案而y轴显示正确诊断的混淆矩阵。该分类器的敏感性为81.7%且特异性为71.4%。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。图24A至24C:使用微RNA比例的归一化值,判别式分析分类器用于将不确定的恶性(菱形,M)样本和良性(方形,B)样本分类。图24A:两种微RNA的比例(hsa-miR-146b-5p-hsa-miR-342-3p、hsa-miR-31-5p-hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为80%且特异性为72.2%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图24B:三种微RNA的比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p)的归一化值用作为分类的特征。该分类器的敏感性为80%且特异性为69%。分类错误的样本(miscl.)由点表示。图24C:8种微RNA比例的归一化值不确定是恶性样本还是良性样本。8种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案而y轴显示正确诊断的混淆矩阵。该分类器的敏感性为80%且特异性为66.7%。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。图25A至25C:使用微RNA和微RNA比例的归一化值的组合,判别式分析分类器用于将未定类的恶性(菱形,M)样本和良性(方形,B)样本分类。图25A:一种微RNA和一种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p)的归一化值用作为分类的特征。该分类器的敏感性为80%且特异性为73.8%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图25B:2种微RNA和1种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为79.1%且特异性为73%。图25C:5种微RNA和3种微RNA的比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p、hsa-miR-31-5p)的归一化值用作为分类的特征。该分类器的敏感性为87.8%且特异性为67.5%。该图显示其中x轴显示了分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。图26A至26C:使用微RNA的归一化值,KNN分类器用于将未定类的恶性(M)样本和良性(B)样本分类。图26A:6种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-125b-5p、hsa-miR-31-5p、hsa-miR-375)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为78.3%且特异性为65.9%。图26B:8种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-125b-5p、hsa-miR-31-5p、hsa-miR-375、hsa-miR-152-3p、hsa-miR-181c-5p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为82.6%且特异性为73%。图26C:12种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-222-3p、hsa-miR-125b-5p、hsa-miR-31-5p、hsa-miR-375、hsa-miR-152-3p、hsa-miR-181c-5p、hsa-miR-424-3p、hsa-miR-486-5p、hsa-miR-200c-3p、hsa-miR-346)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.),而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为73.9%且特异性为68.3%。图27A至27B:使用微RNA比例的归一化值,KNN分类器用于将未定类的恶性(M)样本和良性(B)样本分类。图27A:6种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为80.9%且特异性为65.9%。图27B:8种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为用于分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为76.5%且特异性为62.7%。图28A至28C:使用微RNA和微RNA比例的归一化值,KNN分类器用于将未定类的恶性(M)样本和良性(B)样本分类。图27C:3种微RNA和3种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.),而y轴显示正确诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为76.5%且特异性为57.9%。图28B:5种微RNA和3种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-222-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p、hsa-miR-31-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为78.3%且特异性为64.3%。图28C:12种微RNA和微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p、hsa-miR-31-5p、hsa-miR-375、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-152-3p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为80.9%且特异性为67.5%。图29A至29C:使用微RNA的归一化值,SVM分类器用于将未定类的恶性(菱形,M)样本和良性(方形,B)样本分类。图29A:三种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p)的归一化值用作为分类的特征。该分类器的敏感性为82.6%,特异性为54.8%。分类错误的样本(miscl.)由点表示。图29B:6种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-125b-5p、hsa-miR-31-5p、hsa-miR-375)的归一化值用作为分类的特征。该分类器的敏感性为82.6%且特异性为59.5%。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。图29C:8种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-125b-5p、hsa-miR-31-5p、hsa-miR-375、hsa-miR-152-3p;hsa-miR-181c-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为90.4%且特异性为60.3%。图30A至30C:使用微RNA比例的归一化值,SVM分类器用于将未定类的恶性(菱形,M)样本和良性(方形,B)样本分类。图30A:三种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p)的归一化值用作为分类的特征。该分类器的敏感性为81.7%且特异性为67.5%。分类错误的样本(miscl.)由点表示。图30B:6种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为88.7%且特异性为63.5%。图30C:8种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.),而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为87.8%且特异性为58.7%。图31A至31C:使用微RNA和微RNA比例的归一化值的组合,SVM分类器用于将未定类的恶性(菱形,M)样本和良性(方形,B)样本分类。图31A:2种微RNA和1种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为80%且特异性为71.4%。图31B:4种微RNA和2种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为89.9%且特异性为51.6%。图31C:5种微RNA和3个微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-222-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p、hsa-miR-31-5p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为84.3%且特异性为68.3%。图32A至32C:使用微RNA的归一化值,判别式分析集成分类器用于将未定类的恶性(菱形,M)样本和良性(方形,B)样本分类。图32A:两种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为85.2%且特异性为45.2%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图32B:三种微RNA(hsa-miR-551b-3p、hsa-miR-146b-5p、hsa-miR-222-3p)的归一化值用作为分类的特征。该分类器的敏感性为84.3%且特异性为45.2%。分类错误的样本(miscl.)由点表示。图32C:8种微RNA(hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-125b-5p、hsa-miR-31-5p、hsa-miR-375、hsa-miR-152-3p;hsa-miR-181c-5p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为88.7%且特异性为64.3%。图33A至33C:使用微RNA比例的归一化值,判别式分析集成分类器用于将未定类的恶性(菱形,M)样本和良性(方形,B)样本分类。图33A:两种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为86.1%且特异性为61.1%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图33B:三种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p)的归一化值用作为分类的特征。该分类器的敏感性为87%且特异性为57.1%。分类错误的样本(miscl.)由点表示。图33C:8种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为89.6%且特异性为65.1%。图34A至34C:使用微RNA和微RNA比例的归一化值的组合,判别式分析集成分类器用于将未定类的恶性(菱形,M)样本和良性(方形,B)样本分类。图34A:一种微RNA和一种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p)的归一化值用作为分类的特征。该分类器的敏感性为83.5%且特异性为58.7%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图34B:2种微RNA和1种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为85.2%且特异性为65.9%。分类错误的样本(miscl.)由点表示。图34C:5种微RNA和3种微RNA比例(hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-146b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-31-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-125b-5p、hsa-miR-31-5p)的归一化值用作为分类的特征。该图显示其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为87.8%且特异性为62.7%。图35:未定类的非髓样恶性(“Mal.”)样本和良性(“Ben.”)样本的hsa-miR-146b-5p的归一化的表达(Exp.)水平以点阵图显示。线表示每组的中值。在每组内,为提高点的可见性,点沿x轴随机分布。图36:未定类的非髓样恶性(“Mal.”)样本和良性(“Ben.”)样本的微RNA比例hsa-miR-146b-5p:hsa-miR-342-3p的归一化的表达水平(Exp.)以点阵图显示。线表示每组的中值。在每组内,为提高点的可见性,点沿x轴随机分布。图37A至37C:使用微RNA的归一化值,判别式分析分类器用于将BethesdaIV恶性(菱形,M)样本和良性(方形,B)样本分类。图37A:两种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为91.5%且特异性为42.9%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图37B:三种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p)的归一化值用作为分类的特征。该分类器的敏感性为91.5%且特异性为39.7%。分类错误的样本(miscl.)由点表示。图37C:8种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-375、hsa-miR-181c-5p、hsa-miR-31-5p、hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为89.4%且特异性为47.6%。图38A至38C:使用微RNA比例的归一化值,判别式分析分类器用于将BethesdaIV恶性(菱形,M)样本和良性(方形,B)样本分类。图38A:两种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为89.4%且特异性为28.6%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图38B:三种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p;hsa-miR-146b-5p:hsa-miR-342-3p;hsa-miR-31-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为91.5%且特异性为30.2%。分类错误的样本(miscl.)由点表示。图38C:8种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为80.9%且特异性为57.1%。图39A至39C:使用微RNA和微RNA比例的归一化值,判别式分析分类器用于将BethesdaIV恶性(菱形,M)样本和良性(方形,B)样本分类。图39A:一种微RNA和一种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p)的归一化值用作为分类的特征。该分类器的敏感性为93.6%且特异性为33.3%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图39B:一种微RNA和两种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为89.4%且特异性为41.3%。分类错误的样本(miscl.)由点表示。图39C:4种微RNA和4种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为87.2%且特异性为46%。图40A至40C:使用微RNA的归一化值,KNN分类器用于将BethesdaIV恶性样本和良性样本分类。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。图40A:6种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-375、hsa-miR-181c-5p)的归一化值用作为分类的特征。该分类器的敏感性为72.3%且特异性为39.7%。图40B:8种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-375、hsa-miR-181c-5p、hsa-miR-31-5p、hsa-miR-138-5p)的归一化值用作为分类的特征。该分类器的敏感性为66%且特异性为61.9%。图40C:12种微RNA((hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-375、hsa-miR-181c-5p、hsa-miR-31-5p、hsa-miR-138-5p、hsa-miR-200c-3p、MID-16582、hsa-miR-346、hsa-miR-152-3p)的归一化值用作为分类的特征。该分类器的敏感性为66%且特异性为61.9%。图41A至41B:使用微RNA比例的归一化值,KNN分类器用于将BethesdaIV恶性样本和良性样本分类。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。图41A:6种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p)的归一化值用作为分类的特征。该分类器的敏感性为78.7%且特异性为61.9%。图41B:8种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该分类器的敏感性为80.9%且特异性为50.8%。图42A至42C:使用微RNA和微RNA比例的归一化值,KNN分类器用于将BethesdaIV恶性样本和良性样本分类。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。图42A:4种微RNA和2种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p)的归一化值用作为分类的特征。该分类器的敏感性为63.8%且特异性为46%。图42B:4种微RNA和4种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p)的归一化值用作为分类的特征。该分类器的敏感性为68.1%且特异性为49.2%。图42C:6种微RNA和6种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p、hsa-miR-375、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-181c-5p、MID-16582:hsa-miR-200c-3p)的归一化值用作为分类的特征。该分类器的敏感性为74.5%且特异性为58.7%。图43A至43C:使用微RNA的归一化值,SVM分类器用于将BethesdaIV恶性样本和良性样本分类。图43A:三种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p)的归一化值用作为分类的特征。该分类器的敏感性为97.9%且特异性为22.2%。恶性=M(菱形);良性=B(方形)。图43B:6种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-375、hsa-miR-181c-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为89.4%且特异性为38.1%。图43C:8种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-375、hsa-miR-181c-5p、hsa-miR-31-5p、hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.),而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为91.5%且特异性为55.6%。图44A至44C:使用微RNA比例的归一化值,SVM分类器用于将BethesdaIV恶性(菱形,M)样本和良性(方形,B)样本分类。图44A:三种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p;hsa-miR-31-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为100%。图44B:6种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为93.6%且特异性为33.3%。图44C:8种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为93.6%且特异性为31.7%。图45A至45C:使用微RNA和微RNA比例的归一化值组合,SVM分类器用于将BethesdaIV恶性(菱形,M)样本和良性(方形,B)样本分类。图45A:一种微RNA和两种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为93.6%且特异性为22.2%。分类错误的样本(miscl.)由点表示。图45B:4种微RNA和2种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为95.7%且特异性为31.7%。图45C:4种微RNA和4种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为91.5%且特异性为36.5%。图46A至46C:使用微RNA的归一化值,判别式分析集成分类器用于将BethesdaIV恶性(菱形,M)样本和良性(方形,B)样本分类。图46A:两种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为91.5%且特异性为39.7%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图46B:三种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p)的归一化值用作为分类的特征。该分类器的敏感性为89.4%且特异性为39.7%。图46C:8种微RNA(hsa-miR-125b-5p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-375、hsa-miR-181c-5p、hsa-miR-31-5p、hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为93.6%且特异性为46%。图47A至47C:使用微RNA比例的归一化值,判别式分析集成分类器用于将BethesdaIV恶性(菱形,M)样本和良性(方形,B)样本分类。图47A:两种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为93.6%且特异性为19%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图47B:三种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为93.6%且特异性为17.5%。分类错误的样本(miscl.)由点表示。图47C:8种微RNA比例(hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p、hsa-miR-222-3p:hsa-miR-486-5p、MID-16582:hsa-miR-200c-3p、hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-200c-3p:hsa-miR-486-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为89.4%且特异性为44.4%。图48A至48C:使用微RNA和微RNA比例的归一化值组合,判别式分析集成分类器用于将BethesdaIV恶性(菱形,M)样本和良性(方形,B)样本分类。图48A:一种微RNA和一种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p)的归一化值用作为分类的特征。该分类器的敏感性为91.5%且特异性为33.3%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图48B:一种微RNA和两种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为89.4%且特异性为36.5%。分类错误的样本(miscl.)由点表示。图48C:4种微RNA和4种微RNA比例(hsa-miR-125b-5p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-551b-3p、hsa-miR-222-3p、hsa-miR-146b-5p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-138-5p)的归一化值用作为分类的特征。该图显示了其中x轴显示分类器答案(Clas.Ans.)而y轴显示真实诊断(真实类别=re.cl.)的混淆矩阵。该分类器的敏感性为91.5%且特异性为34.9%。图49:BethesdaIV非髓样恶性(“Mal.”)样本和良性(“Ben.”)样本的hsa-miR-146b-5p的归一化表达(实验)水平以点阵图显示。线表示每组的中值。在每组中,点沿x轴随机分布。图50:BethesdaIV非髓样恶性(“Mal.”)样本和良性(“Ben.”)样本的微RNA比例hsa-miR-146b-5p:hsa-miR-342-3p的归一化表达(实验)水平以点阵图显示。线表示每组的中值。在每组中,点沿x轴随机分布。图51:使用判别式分析分类器将恶性(菱形,M)样本和良性(方形,B)样本分类,其中恶性组包括髓样肿瘤的样本。两种微RNA(hsa-miR-222-3p、hsa-miR-551b-3p)的归一化值用作为分类的特征。该分类器的敏感性为85.2%且特异性为53.6%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图52:使用判别式分析分类器将恶性(菱形,M)样本和良性(方形,B)样本分类,其中恶性组包括髓样肿瘤的样本。两种微RNA比例(hsa-miR-125b-5p:hsa-miR-138-5p、hsa-miR-146b-5p:hsa-miR-342-3p)的归一化值用作为分类的特征。该分类器的敏感性为84.7%且特异性为80.8%。灰色阴影区域标记其中样本被分类器确定分类为恶性的空白。图53:hsa-miR-486-5p和hsa-miR-200c-3p的表达模式决定了样本的品质。分析四个血液涂片(BS)样本的hsa-miR-486-5p(SEQIDNO.22)和hsa-miR-200c-3p(SEQIDNO.23-24)表达和其在恶性(M)样本和良性(B)样本中的表达的对比。显示了两种miR的归一化值(使用所有正规化子来归一化)。图54:甲状腺良性肿瘤的亚型。建立良性肿瘤的两个亚型,滤泡性腺瘤(FA,y轴,n=81)和Hashimoto病(Hash.,x轴、n=6)的微RNA表达谱(中值)。每个十字表示微RNA或微RNA比例。hsa-miR-125b-5p:hsa-miR-200c-3p比例与FA相关,同时hsa-miR-342-3p和hsa-miR-31-5p的表达与Hashimoto病相关。菱形表示正规化子。显著的微RNA(t-test的p值<0.05)由圆圈表示。图55:恶性甲状腺肿瘤的亚型。建立恶性甲状腺肿瘤的两种亚型,乳头状癌(Pap.;y轴,n=161)和滤泡性癌(FC;x轴,n=16)的微RNA表达谱。每个十字表示微RNA或微RNA比例。菱形为正规化子。显著的微RNA(t-test的p值<0.05)被圈出。只将归一化的微RNA值标记。未标记的圆圈表示显著比例。图56:表示经由FNA获得的用于诊断未定类甲状腺结节样本的方案的流程图。发明详述尽管在甲状腺病变确诊的研究方面积累了大量努力,然而大量的技术问题仍没有解决方案。由于所获得材料的品质,在细针穿刺(FNA)样本中的甲状腺病变诊断仍具有挑战性。样本中的低细胞数量、血液量、甲状腺肿瘤细胞和非肿瘤细胞之间的比例使得提取会提供决定性结果的足够材料具有挑战性。本发明提供用于区分恶性甲状腺肿瘤和良性甲状腺肿瘤、以及甲状腺肿瘤的具体亚型的敏感的、特异性的和准确的方法。区分甲状腺肿瘤的不同亚型对于向患者提供最好和最适当的治疗是重要的。本发明提供甲状腺肿瘤分类和诊断领域中现有技术的显著改进。本发明开发了用于甲状腺病变分类的整合平台,其通过谱化和表征由FNA活组织检查获得的甲状腺临床样本中的微RNA表达,同时还通过微RNA谱化克服了例如样本中低细胞数量和样本中低血液量的障碍而实现。该技术平台作为甲状腺结节术前管理的辅助工具被应用于将甲状腺病变分为良性赘生物或恶性赘生物,以及甲状腺肿瘤的亚型。发明人意外地开发了用于分类良性甲状腺病变和恶性甲状腺病变以及甲状腺癌特定亚型和滤泡性病变的方法,同时通过实施基于微RNA谱的特定算法而整合用于滤除次优样本的步骤。该方法是总体方案的一部分,其中可以使用具有来自FNA样本涂片的现存的或可用的临床细胞学载片,而无需产生或采集来自于患者的其他材料。本方法还包括来自细胞学样本的微量RNA材料中的微RNA分析。FNA样本一经收集,将一代或若干代材料涂于载片上。目前可用的方法通常要求使用若干代以具有用于分析的足够材料。本发明人开发一种方法,其中甚至只有一个FNA载片提供用于微RNA检测的足够材料。此外,本发明人能够测量由小至0.1cm甲状腺结节获得的FNA样本中的微RNA丰度。考虑到约50%的甲状腺病变小于1cm,这是特别有意义的[Jung等人(2014)JClinEndocrinolMetab99:E276–E285]。另外,发明人开发的方法能够分析具有非常少量细胞的样本,例如具有50个细胞,最高为120个细胞和以上的样本。本方法包括排除或淘汰缺少甲状腺细胞和/或其中非甲状腺细胞如血细胞过度表现的样本的步骤。本发明经由谱化记为SEQIDNO.1至SEQIDNO.308的微RNA表达而确认甲状腺病变的独特微RNA表达特征。更具体地,本发明人开发基于微RNA组表达水平而分类甲状腺临床样本的平台,该微RNA组包含至少两种微RNA,选自:hsa-miR-31-5p(SEQIDNO.5至SEQIDNO.7)、hsa-miR-424-3p(SEQIDNO.16)、hsa-miR-222-3p(SEQIDNO.1至SEQIDNO.2)、hsa-miR-146b-5p(SEQIDNO.10至SEQIDNO.11)、hsa-miR-346(SEQIDNO.14)、MID-16582(SEQIDNO.25)、hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)、hsa-miR-181c-5p(SEQIDNO.15)、hsa-miR-125b-5p(SEQIDNO.9)、hsa-miR-375(SEQIDNO.8)、hsa-miR-486-5p(SEQIDNO.22)、hsa-miR-551b-3p(SEQIDNO.3至SEQIDNO.4)、hsa-miR-152-3p(SEQIDNO.12至SEQIDNO.13)、hsa-miR-200c-3p(SEQIDNO.23至SEQIDNO.24)和hsa-miR-138-5p(SEQIDNO.19至SEQIDNO.21)、或与其至少80%、至少85%、或至少90%一致性的序列。该平台是基于使用稳健的组的训练性研究而建立的,其还包含作为正规化子的附加微RNA的测量。本发明对于其中FNA标本表现出细胞病理学中非决定性的结果,通常被称为“未定类”的25%的案例尤其有用,其包括分类为Bethesda类别III、Bethesda类别IV和Bethesda类别V的甲状腺病变样本。在现有的医疗实践中,具有落入该类别的标本的患者经历重复的FNA流程和手术,包括叶切除术或甲状腺切除术。因此,在一个实施方式中,本发明提供用于分类属于“未定类”案例的甲状腺病变样本的方法,“未定类”案例分类为BethesdaSystem的类别III、类别IV和类别V(本文进一步描述)。在一个具体实施例中,本发明提供将分类为BethesdaSystem类别IV的甲状腺病变样本分类的方法,其涉及“滤泡性腺瘤”或“疑似滤泡性腺瘤”,其被认为是最难进行分类的类别。因此,本发明首先提供用于管理无法通过细胞病理学分析归类的甲状腺病变样本的方案。感兴趣的具体样本为通过FNA获得的那些。在一个实施方式中,使用来自FNA样本的常规涂片。在另一个实施方式中,可以使用防腐液中的FNA样本。从FNA样本中提取总RNA,并测量微RNA的表达。在一个实施方式中,测量约2200个微RNA的表达。在另一个实施方式中,测量182个微RNA的表达,其包括序列SEQIDNO.1至SEQIDNO.182。在另一个实施方式中,测量包括序列SEQIDNO.1至SEQIDNO.37的微RNA的表达。在另一个实施方式中,测量选自hsa-miR-31-5p(SEQIDNO.5至SEQIDNO.7)、hsa-miR-424-3p(SEQIDNO.16)、hsa-miR-222-3p(SEQIDNO.1至SEQIDNO.2)、hsa-miR-146b-5p(SEQIDNO.10至SEQIDNO.11)、hsa-miR-346(SEQIDNO.14)、MID-16582(SEQIDNO.25)、hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)、hsa-miR-181c-5p(SEQIDNO.15)、hsa-miR-125b-5p(SEQIDNO.9)、hsa-miR-375(SEQIDNO.8)、hsa-miR-486-5p(SEQIDNO.22)、hsa-miR-551b-3p(SEQIDNO.3至SEQIDNO.4)、hsa-miR-152-3p(SEQIDNO.12至SEQIDNO.13)、hsa-miR-200c-3p(SEQIDNO.23至SEQIDNO.24)和hsa-miR-138-5p(SEQIDNO.19至SEQIDNO.21)、或与其至少80%、至少85%、或至少90%一致性的序列中的至少3个、至少4个、至少5个、至少6个、至少7个、至少8个、至少9个、至少10个、至少11个、至少12个、至少13个、至少14个或全部微RNA,并将其用于分类。在另一个实施方式中,将甲状腺样品分类为恶性或良性包括测量以下序列的表达水平:hsa-miR-222-3p(SEQIDNO.1至SEQIDNO.2)、hsa-miR-551b-3p(SEQIDNO.3至SEQIDNO.4)、hsa-miR-31-5p(SEQIDNO.5至SEQIDNO.7)、hsa-miR-375(SEQIDNO.8)、hsa-miR-125b-5p(SEQIDNO.9)、hsa-miR-146b-5p(SEQIDNO.10至SEQIDNO.11)、hsa-miR-152-3p(SEQIDNO.12至SEQIDNO.13)、hsa-miR-346(SEQIDNO.14)、hsa-miR-181c-5p(SEQIDNO.15)、hsa-miR-424-3p(SEQIDNO.16)、hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)、hsa-miR-138-5p(SEQIDNO.19至SEQIDNO.21)、hsa-miR-486-5p(SEQIDNO.22)、hsa-miR-200c-3p(SEQIDNO.23至SEQIDNO.24)、MID-16582(SEQIDNO.25)或其任意组合、或与其至少80%、至少85%、或至少90%一致性的序列,将表达水平供给分类器,该分类器将样本分析和分类为恶性或良性。因此,本发明提供用于在有需要的受试者中区分恶性甲状腺肿瘤病变和良性甲状腺肿瘤病变的方法,所述方法包括从所述受试者获得甲状腺肿瘤病变样本,或提供从所述受试者获得的生物样本,在一个或更多个所述样本、或包括SEQIDNO:1至SEQIDNO:25中的至少4个微RNA、或者与其至少80%、至少85%、或至少90%一致性的序列、或所述微RNA的组合中通过杂交或扩增确定表达谱,通过使用分类器算法比较所述表达谱和参考阈值;确定甲状腺病变是恶性或者良性。在一个具体实施方式中,本发明的方法用于区分恶性甲状腺肿瘤病变或良性甲状腺肿瘤病变的亚型。在一个实施方式中,本发明的方法包括测量包括SEQIDNO:1至SEQIDNO:25中的至少四种微RNA的表达,获得所述样本的微RNA表达谱值,基于所述值使用分类器确定甲状腺病变是恶性或良性,任选地还将该样本分类为恶性亚型或良性亚型中的一种。在一个具体实施方式中,通过杂交的所述确定表达谱包括使样本和探针接触,该探针与SEQIDNO:1至SEQIDNO:25中的每一个杂交,或者与与其至少80%、至少85%、至少90%一致性的序列杂交。在另一个实施方式中,通过杂交的所述确定表达谱包括使样本和探针接触,该探针与包括序列SEQIDNO:1至SEQIDNO:25的所述微RNA的至少8个、至少10个、至少12个、至少14个、或至少16个连续核苷酸杂交。本发明还提供将样本分类为恶性、或良性、和/或亚型所述样本的方法,借此,进一步测量微RNA在样本中的表达水平,获得表达谱,任选地应用表达数据的多步分析计算微RNA比例。所述多步分析包括向微RNA表达谱、微RNA比例或其组合中的至少一个平行地或连续地应用一种或更多种算法。所述多步分析可还包括分析一种或更多种单一微RNA的表达水平,该表达水平可以指示样本的整体品质。以任意顺序或任意组合,可以包括于多步分析的准则的实例为:非恶性细胞标记的表达、与甲状腺肿瘤特定亚型相关的微RNA表达等。因此例如,一个步骤可以为检测非肿瘤细胞标记的表达是高于还是低于在例如训练数据集的数据集中建立的阈值,在这种情况下,该样本是不合格的。另一个步骤可以为检测与甲状腺肿瘤亚型相关的微RNA表达或微RNA表达比例,例如,如果hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)的表达相比于在如训练数据集的数据集中建立的阈值是非常高的,可以将该样本分类为良性、进一步的亚型例如Hashimoto病。或者,如果hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)表达相比于在如训练数据集的数据集中建立的阈值是非常高的,由于缺少足够甲状腺细胞,该样本是不合格的。另一个任选的步骤可涉及MID-16582(SEQIDNO.25)的表达水平,可以用于确定样本是否可被舍弃,或是否使用分类器具体分析MID-16582(SEQIDNO.25)是高的(相比于在训练集中建立的阈值)样本。在本发明的一个具体实施方式中,所述非甲状腺细胞标记为血细胞标记。在本发明的另一个具体实施方式中,所述细胞标记为上皮细胞标记。在本发明的另一个具体实施方式中,所述细胞标记为血细胞标记、白血球标记或上皮细胞标记。血细胞标记的实例为hsa-miR-486-5p(SEQIDNO.22)、hsa-miR-320a(SEQIDNO.173)、hsa-miR-106a-5p(SEQIDNO.150)、hsa-miR-93-5p(SEQIDNO.182)、hsa-miR-17-3p(SEQIDNO.160)、hsa-let-7d-5p(SEQIDNO.144)、hsa-miR-107(SEQIDNO.152)、hsa-miR-103a-3p(SEQIDNO.149)、hsa-miR-17-5p(SEQIDNO.161)、hsa-miR-191-5p(SEQIDNO.163)、hsa-miR-25-3p(SEQIDNO.167)、hsa-miR-106b-5p(SEQIDNO.151)、hsa-miR-20a-5p(SEQIDNO.166)、hsa-miR-18a-5p(SEQIDNO.40)、hsa-miR-144-3p(SEQIDNO.154)、hsa-miR-140-3p(SEQIDNO.51)、hsa-miR-15b-5p(SEQIDNO.157)、hsa-miR-16-5p(SEQIDNO.159)、hsa-miR-92a-3p(SEQIDNO.181)、hsa-miR-484(SEQIDNO.179)、hsa-miR-151a-5p(SEQIDNO.156)、hsa-let-7f-5p(SEQIDNO.)、hsa-let-7a-5p(SEQIDNO.141)、hsa-let-7c-5p(SEQIDNO.143)、hsa-let-7b-5p(SEQIDNO.142)、hsa-let-7g-5p(SEQIDNO.146)、hsa-let-7i-5p(SEQIDNO.147)、hsa-miR-185-5p(SEQIDNO.162)、hsa-miR-30d-5p(SEQIDNO.172)、hsa-miR-30b-5p(SEQIDNO.170)、hsa-miR-30c-5p(SEQIDNO.171)、hsa-miR-19b-3p、hsa-miR-26a-5p(SEQIDNO.168)、hsa-miR-26b-5p(SEQIDNO.169)、hsa-miR-425-5p(SEQIDNO.176)、MID-19433(SEQIDNO.133)和hsa-miR-4306(SEQIDNO.177)。白血球标记的实例为hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)、hsa-miR-146a-5p和hsa-miR-150-5p(SEQIDNO.59)。上皮细胞标记的实例为hsa-miR-200c-3p(SEQIDNO.23至SEQIDNO.24)、hsa-miR-138-5p(SEQIDNO.19至SEQIDNO.21)、hsa-miR-3648(SEQIDNO.174)、hsa-miR-125b-5p(SEQIDNO.9)、hsa-miR-125a-5p(SEQIDNO.153)、hsa-miR-192-3p(SEQIDNO.164)、hsa-miR-4324(SEQIDNO.178)、hsa-miR-376a-3p(SEQIDNO.175)。如本文所述,所述微RNA比例为一对微RNA的归一化表达水平,其中一个微RNA的归一化表达水平用作分子,另一个微RNA的归一化表达水平为分母。在另一个具体实施方式中,所述确定表达谱包括使样本与包含如本文在实施例中示例的正向引物和反向引物的RT-PCR试剂接触,产生RT-PCR产物。在另一个具体实施方式中,所述方法包括使RT-PCR产物与如本文在实施例中所示例的特异性探针或普通探针或其组合接触,检测并测量PCR产物。在另一个实施方式中,所述确定表达谱包括使用微阵列等通过杂交测定微RNA表达。在另一个实施方式中,所述确定表达谱包括通过下一代测序测量微RNA表达。在另一个实施方式中,所述方法任选地还包括确定作为正规化子的至少一种微RNA的表达谱。在一个实施方式中,表1所述的任意微RNA可以用作正规化子。在一个具体实施方式中,使用包括SEQIDNO.26至SEQIDNO.37、或与其至少80%、85%、90%或95%一致性序列的任意微RNA作为正规化子。本发明人已经出人意料地发现,当使用如本文所定义和示例的不同类别的许多标记时,改进了甲状腺肿瘤样本的分类。所述标记可以为以下任一种:包括SEQIDNOS:1至SEQIDNOS:25、或者与其至少80%、85%、90%或95%一致性的序列的恶性标记、次级标记和细胞型标记或其任意组合。为了实施本发明的方法,可以使用全套的标记。或者,可以使用恶性标记、次级标记和细胞型标记的任意组合。因此,该方法可以包括至少一个恶性标记,其与至少一个次级标记和/或至少一个细胞型标记联合。取决于数据分析,可以以原始特征或归一化特征的形式使用每个细胞型标记。或者,为了确定样本是否具有足够的相关材料以实施分类,或者该样本是否应该被舍弃,可将细胞型标记用作在实施分类之前的初步测试。另一个选择为使用细胞型标记作为最终分类器的一部分,其中由分类器使用细胞类型标记的特征。另一种选择为使用细胞型标记作为MiR比例的分母,其任选地由分类器使用。例如,恶性标记或次级标记的表达水平可被细胞型标记的表达水平除,获得在分类器中使用的miR比例。因此,区分在有需要的受试者中的恶性和良性甲状腺肿瘤病变的方法的另一实施方式中,所述分类器可为以下任一种:单分类器、多步分类器、使用所有恶性标记的分类器、使用恶性标记子集的分类器、使用所有恶性标记和次级标记的分类器、使用恶性标记子集和次级标记子集的分类器、使用所有恶性标记、次级标记和细胞型标记的分类器、使用所有恶性标记、次级标记和细胞型标记的子集的分类器、使用所有恶性标记或其子集以及细胞型标记或其子集的分类器。在本发明方法或方案的其他实施方式中,可通过进一步结合算法分类器结果和所分析甲状腺样本可获得的其他临床数据或分子数据来改进分类的进行。其他可获得的数据可以关于甲状腺病变,例如结节尺寸、结节数量;其可以关于从其获得样本的受试者中可获得的其他临床信息,例如分子测试结果,例如其他分子标记的表达、基因标记、生化测试结果、血液测试结果、尿测试结果、再发率、预后数据、家族史、患者病史等。可结合的其他数据为甲状腺基因数据,例如突变分析、基因融合、染色体重排、基因表达、蛋白质表达等。治疗型适应症可以根据用本发明方法或方案获得的诊断而不同。典型地,存在可以施用于甲状腺癌患者的五种疗法:手术、放疗、化疗、甲状腺激素疗法和靶向疗法。手术是甲状腺癌的最常用疗法。可使用以下流程之一:-叶切除术:去除发现甲状腺癌的叶。进行该区域内的淋巴结活组织检查以确认其是否含有癌。-甲状腺近全切除术:去除除了非常小部分以外的全部甲状腺。-甲状腺全切除术:去除全部甲状腺。-淋巴切除术:去除含有癌的颈部中的淋巴结。甲状腺切除术是具有若干潜在并发症或后遗症的手术流程,潜在并发症或后遗症包括暂时或永久性声音改变、暂时或永久性低钙、终身需要甲状腺激素替代、流血、感染、由于双侧声带麻痹导致的极小可能性的气道阻塞。因此,防止甲状腺不必要去除的准确诊断是非常需要的。放疗使用高能X射线或其他类型射线以消除癌细胞或阻止其增殖。存在两种类型的放疗。外部放疗使用体外仪器向恶性肿瘤发出射线。体内放疗使用直接置于恶性肿瘤内部或附近的密封在针、核、线或导管内的放射性物质。放疗的选择取决于甲状腺癌的类型和阶段。放疗可作为手术的补充以消除未成功去除的癌细胞。滤泡性甲状腺癌和乳头状甲状腺癌可使用放射性碘(RAI)疗法治疗。RAI通过口服施用,并在任何残留的甲状腺组织中聚集,包括已经扩散到体内其他位置的甲状腺癌细胞。由于只有甲状腺组织摄取碘,RAI破坏甲状腺组织和甲状腺癌细胞而不损伤其他组织。在施用全部治疗剂量的RAI之前,施用小的测试剂量以确定肿瘤是否摄取碘。化疗是甲状腺癌治疗的另一种选择。化疗可通过口服、或通过静脉内注射或肌肉内注射来给药。化疗还可以直接给药于恶性肿瘤影响的区域而不是全身。给药的选择取决于甲状腺癌的类型和阶段。已被批准的用于甲状腺癌治疗的药物的一些实例为:AdriamycinPFS(DoxorubicinHydrochloride)、AdriamycinRDF(DoxorubicinHydrochloride)、Cabozantinib-S-Malate,Caprelsa(Vandetanib)、Cometriq(Cabozantinib-S-Malate)、DoxorubicinHydrochloride,Nexavar(SorafenibTosylate)、SorafenibTosylate和Vandetanib。甲状腺激素疗法为去除激素或阻断其活性并抑制癌细胞增殖的癌症治疗。在甲状腺癌的治疗中,可施用药物以抑制促甲状腺激素(TSH)产生,以避免激素会导致甲状腺癌的生长或再发。另外,由于甲状腺癌治疗特异性地靶向于甲状腺细胞,甲状腺无法产生足够的甲状腺激素。对患者施用甲状腺激素替代药物。靶向治疗使用药物或其他物质以识别并攻击特定癌细胞而不伤害正常细胞。酪氨酸激酶抑制剂(TKI)疗法阻断在甲状腺癌细胞内的信号转导,抑制其生长。凡德他尼(Vandetanib)是用于治疗甲状腺癌的TKI。任意疗法的剂量和持续时间取决于患者的个体评估以及医疗服务人员已知的标准实践。治疗的持续时间为在该时间段中药物制剂或药物组合物的剂量施用的时间。通过分析差异化表达的微RNA,首先区分为良性或恶性,然后将其分类为不同亚型,甲状腺肿瘤的识别和区分可提供亚型之间生物学差异、其不同肿瘤形成过程和类型特异性靶向治疗的可能新靶标的进一步线索。本发明提供定量和定性的诊断分析和方法,通过比较如本文描述的特异的微RNA分子的水平,用于检测、诊断、监测、分级和预测甲状腺癌。该水平在患者样本中测量,患者样本可来自于活组织检查、肿瘤样本、细胞、组织和/或体液。因此,本发明的方法对于识别恶性甲状腺肿瘤的不同亚型以及对于确定诊断之后的治疗过程是特别有用的,该亚型为滤泡癌、乳头状癌、乳头状癌的滤泡变体(FVPC或FVPTC)、包膜内型FVPTC(或包膜内型FVPTC)、髓样癌、未分化甲状腺癌、低分化甲状腺癌。在另一个实施方式中,本发明提供用于分类良性甲状腺肿瘤亚型的方法,例如滤泡性癌、Hashimoto甲状腺炎、增生(甲状腺肿大)。本发明还提供治疗甲状腺癌的方法,所述方法包括如本文所述的区分良性甲状腺肿瘤或恶性甲状腺肿瘤的方法,任选地亚型化甲状腺肿瘤类型,并根据本发明方法提供的诊断给药治疗。本发明的所有方法还任选地可以包括测量其他癌标记的水平。除了在本发明中有用的所述微RNA分子,其他癌标记取决于测试的癌,是本领域技术人员已知的。可以用于确定在来自患者的样本中基因表达水平例如本发明的核酸序列的测定技术是本领域技术人员已知的。该分析方法包括但不限于,放射免疫测定、逆转录酶PCR(RT-PCR)测定、免疫组织化学测定、原位杂交测定、竞争结合测定、RNA印迹测定、ELISA测定、核酸微阵列和生物芯片测定。可以设置一种或更多种核酸序列表达水平的任意阈值以将样本或肿瘤样本指定为两组中的一个。或者,在优选的实施方式中,通过取两种核酸序列表达水平的比例和/或通过例如逻辑回归的方法使本发明的一种或更多种核酸序列的表达水平结合以定义度量,然后将其与之前测量的样本或阈值比较。指定的阈值作为参数,其可用于量化将样本指定为各类的置信度。可以测量指定的阈值以得到有利的敏感性或特异性,这取决于临床情况。与参考数据相关的值产生连续的可测量计分,并提供样本属于甲状腺亚型的特定类别的可能性的诊断信息。在多变量分析中,微RNA特征提供高水平的预后信息。本发明还提供新型微RNA分子,包括标记为SEQIDNO.27至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139、SEQIDNO.140、SEQIDNO.307和SEQIDNO.308的核酸。应理解,本发明还包括对应于SEQIDNO.27至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139、SEQIDNO.140、SEQIDNO.307和SEQIDNO.308中的任一种的cDNA、补序列和反miR。另外,本申请提供包含本文所述的微RNA的组合物、制剂和药剂。在一个具体实施方式中,本发明提供包含作为活性物质的微RNA的组合物、制剂和药剂,该微RNA具有SEQIDNO.27至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139、SEQIDNO.140、SEQIDNO.307和SEQIDNO.308中任一种、其变体、或与其至少80%、至少85%、或至少90%一致性的序列。所述组合物、制剂和药剂还可以任选地包括辅料、载体、稀释剂和赋形剂中的任一个。可以将本文描述的微RNA通过与适当的、药学上可接受的载体或稀释剂组合以配制为组合物、制剂和药剂,并可以配制为固体、半固体、液体或气体形式的制剂,例如片剂、胶囊、粉剂、颗粒、软膏剂、溶液、栓剂、注射剂、吸入剂和喷剂。同样地,微RNA或包含其的药物组合物可以以不同途径施用,包括口服、经口腔、经直肠、非肠道、经腹膜内、经皮内、经皮肤、经气管内等。在某些实施方式中,本发明的药物组合物包含本发明的一种或更多种核酸和一种或更多种赋形剂。在某些这种实施方式中,赋形剂选自水、盐溶液、醇、聚乙二醇、明胶、乳糖、淀粉酶、硬脂酸镁、滑石、硅酸、粘性石蜡、羟甲基纤维素和聚乙烯吡咯烷酮。在某些实施方式中,本发明的药物组合物使用已知技术制备,包括但不限于混合、溶解、造粒、糖衣化、研磨、乳化、胶囊化、包埋或压片过程。在文献中,例如在Gennaro,A.R.(2000)Remington:TheScienceandPracticeofPharmacy,第20版中可以找到用于制备药物组合物的方法。在某些实施方式中,本发明的药物组合物为液体(例如,悬浮液、酏剂和/或溶液)。在某些实施方式中,液体药物组合物使用本领域已知的成分制备,包括但不限于,水、二醇、油、醇、调味剂、防腐剂和着色剂。在某些实施方式中,本发明的药物组合物为固体(例如,粉剂、片剂和/或胶囊)。在某些这种实施方式中,包含本发明的一种或更多种核酸的固体药物组合物使用本领域已知的成分制备,包括但不限于,淀粉、糖、稀释剂、成粒剂、润滑剂、粘合剂和崩解剂。此外,本发明提供了包含本文所述的化合物(核酸)的载体和探针。在一个具体实施方式中,本申请提供包含标记为SEQIDNO.27至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139、SEQIDNO.140、SEQIDNO.307和SEQIDNO.308的核酸、其变体、或与其至少80%、至少85%、或至少90%一致性的序列的载体或探针。应理解本文使用的术语是仅出于描述具体实施方式的目的,而不旨在限制。必须注意的是,如说明书和所附权利要求中所使用的,指示物前不加数量词包括复数指示物,除非本文明确指出其他。为记载本文的数值范围,明确预期了介于其间的具有相同精确度的每个中间数字。例如,对于范围6至9,除了6和9外也预期数字7和8,例如对于范围6.0至7.0,明确预期6.0、6.1、6.2、6.3、6.4、6.5、6.6、6.7、6.8、6.9和7.0。如本文所使用的,术语“异常增殖”指偏离正常的、适当的、或预期过程的细胞增殖。异常细胞增殖可以包括特征与由不适当高水平的细胞分裂、不适当低水平的细胞凋亡或二者引起或介导或导致的指征相关的细胞增殖。该指征可以例如由单一或多种局部异常增殖的细胞、细胞群或组织来表征是否为癌性的或非癌性的、良性的或恶性的。异常增殖是癌症的主要特征之一。如本文所使用的,术语“约”指+/-10%。“附着的”或“固定的”,如本文使用的指探针和固体支撑物,指在探针和固体支撑物之间的结合在结合、洗涤、分析和去除条件下足够稳定。结合可为共价的或非共价的。共价键可直接形成于探针和固体支撑物之间、或通过交联剂或通过在或固体支撑物或探针或二者分子上特定活性基团的内含物来形成。非共价结合可以为静电相互作用、亲水相互作用和疏水相互作用中的一种或更多种。包括于非共价结合中的是分子例如链霉亲和素共价附着到支撑物上,以及生物素标记探针和链霉亲和素的非共价结合。固定化还可以涉及共价相互作用和非共价相互作用的的组合。本文使用的“生物样本”或“样本”指尤其包括核酸、微RNA的生物组织或液体的样本。这种样本包括但不限于从受试者分离的组织或液体。生物样本也可以包括组织的切片例如活组织检查样本和尸体解剖样本、细针穿刺(FNA)样本、出于组织学目的取得的冷冻切片、血液、血液的部分、血浆、血清等。通过从受试者移除细胞样本可提供生物学样本,然而也可通过使用先前分离的细胞(例如,由另一个人、在另一时间和/或出于另一目的而分离的),之后可以经培养或不经培养来提供生物学样本。还可以使用归档的组织,例如具有治疗历史或结果历史的归档组织。在本发明的另一实施方式中,制备FNA活组织切片作为涂片。术语“分类”指程序和/或算法,其中将个体项目基于项目中的一个或更多个固有特征的定量信息(被称为特质、变量、特征、特点等),和基于统计模型和/或先前示踪项目的训练集被置于群或类。如本文所述的,术语“将甲状腺肿瘤分类”指识别甲状腺组织样本的一种或更多种特性(例如,包括但不限于,在癌组织中表达的微RNA的存在、在可能变为癌性的癌前组织中表达的微RNA的存在、在可能转移的癌组织中表达的微RNA的存在)。本文使用的术语“分类器”指用于将甲状腺肿瘤(或病变)分类、区分、或识别为良性或恶性的算法,或用于分类、区分、或识别甲状腺肿瘤亚型的算法。一旦取得任意研究组样本的微RNA表达谱,例如来自训练组,就产生数据库,其中存储组中样本的所有微RNA的表达水平。该数据库还被称作“训练数据”,其用于选择用于分类的优化算法。通过用于分类甲状腺样本的算法,还可使用核酸(或微RNA)比例、单独或与核酸(或微RNA)水平组合。在一个实施方式中,本发明的方法或方案中使用的算法为机器学习型算法。机器学习型算法的实例为判别式分析、K最近邻算法分类器(KNN)、支持向量机(SVM)分类器、逻辑回归分类器、神经网络分类器、高斯混合模型(GMM)、最邻近重心分类器、线性回归分类器、决策树分类器、和随机森林分类器、分类器集成、或其任意组合。当使用判别式分析分类器时,判别式可为线性的、二次的、线性协方差矩阵对角线、二次协方差矩阵对角线、线性协方差矩阵伪逆、二次协方差矩阵伪逆中的任一种。当使用KNN分类器时,k可以改变且距离测度可以为Pearson相关、spearman相关、Euclidean或cityblock(Manhattan)距离。当使用SVM分类器时,核心可为线性的、高斯的或多项式的。当使用集成方法分类器时,其通常应用算法例如分类树、KNN或判别式分析分类器。集成可以使用或boosting算法或bagging算法,集成学习周期可为二至最高几千。如本文所使用的,“混淆矩阵”指特定的表格布局,其允许算法性能的可视化,典型的算法为监督式学习算法。“混淆矩阵”还可称为列联表或误差矩阵。如本文所使用的“互补”与核酸有关,可指在核酸分子的核苷酸或核苷酸类似物之间的Watson-Crick(例如,A-T/U和C-G)或Hoogsteen碱基对。完全互补指核酸分子的核苷酸或核苷酸类似物之间100%互补的碱基对。在一些实施方式中,互补序列具有反方向(5’-3’)。本发明还提供标记为SEQIDNO.7至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139和SEQIDNO.140的核酸的互补序列。如本文所使用的,“CT特征”或“CT”表示PCR的第一循环,其中扩增跨越了荧光的阈值(循环阈值)。因此,CT的低值表示微RNA的高丰度或高表达水平。在一些实施方式中,将PCR的CT特征归一化以使归一化的CT为表达水平的倒数。在其他实施方式中,PCR的CT特征可被归一化然后取倒数,以使低的归一化的取倒数的CT表示微RNA的低丰度和低表达水平。如本文中所使用的,“数据处理程序”指可体现在包含在软件中的程序,其确定关于一个或更多个样本所采集数据(例如,测试或分析的最终结果)的生物重要性。例如,数据处理程序可以基于采集的数据确定从其中采集或获得样本的甲状腺病变是良性的或是恶性的,或为具体的亚型。在本文的系统和方法中,基于所确定的结果,数据处理程序还可以控制数据采集程序。数据处理程序和数据采集程序可被整合并提供反馈以运行数据采集,从而提供基于分析的判断方法。“检测”指检测在样本中成分的存在。检测还指检测成分的缺失。检测还指或定量或定性地确定成分的水平。“差异化表达”或“表达水平的差异”指在甲状腺样本中微RNA表达模式的定性差别或定量差别。因此,差异化表达的微RNA可以定性地改变其表达,包括活化或失活,例如正常的甲状腺组织相对于病变的甲状腺组织。定性调节的微RNA可以在甲状腺样本或细胞型中表现出可由标准方法检测的表达模式。一些微RNA可以在一种甲状腺样本或细胞型中表达而不在其他甲状腺样本或细胞型中表达,或者在不同细胞型或不同样本之间以不同水平表达。因此,表达的差异可以是定性的,例如,表达可以是调节的、上调的,这导致微RNA的量增加,或下调的,这导致微RNA的量降低。表达差异的程度只需要足够大以通过标准表征方法来定量,标准表征方法例如表达阵列、下一代测序(NGS)、定量逆转录PCR、Northern印迹分析、实时PCR、原位杂交和核糖核酸酶保护。广义地使用术语“表达谱”以包括例如基因组表达谱和微RNA表达谱。如本文所使用的,表达谱指为核酸(或微RNA)表达所获得的数据集。其可以指原始数据或归一化的表达值。表达谱可以通过确定核酸序列的水平德便利方法产生,例如微RNA的定量杂交、标记的微RNA、扩增的微RNA、扩增的cDNA等、定量PCR等。进一步测量核酸序列水平,可以将获得的数据归一化——数据的归一化已经在本申请的其他部分讨论。表达谱允许在两个或更多个样本之间以及在样本和阈值之间的差异化基因表达的分析。此外,可将分类器应用于表达谱以获得关于样本的信息、例如样本的分类、诊断、亚型等。所研究的核酸序列为发现为预测性的核酸序列,包括在本文表1中提供的核酸序列,其中表达谱可包括5个、10个、20个、25个、50个、100个或更多个包括所有列出的核酸序列的表达数据。根据一些实施方式,术语“表达谱”指测量在所测量样本中的核酸序列丰度。在一个具体实施方式中,在每个甲状腺样本中表征微RNA表达谱。如本文所使用的,“表达比例”指两个或更多个核酸即微RNA的相对表达水平,如通过检测生物样本例如甲状腺样本中对应核酸的相对表达水平而确定。由于微RNA表达水平表示为在对数尺度上获得的CT,实际上表达比例通过CT的差值获得而不是通过除法。如本文所使用的,“FDR”或“错误发现率”为用于多重假设检验中以校正多重比较的统计学方法。当进行多重假设检验时,例如在比较具有多重数据特征中两组之间的特征时,通过组之间的可以达到原本被视为统计学显著的水平的随机差异,存在获得假阳性结果越来越高的可能性。为了限制该错误发现的比例,将统计学显著性的定义为仅针对差异达到阈值以下的p值(通过双侧t检验)的数据特征,这取决于进行测试的数量以及在这些测试中获得的p值分布。如本文所使用的,“FNA”涉及“细针穿刺”。细针穿刺活组织检查(FNAB、FNA或NAB),或细针穿刺(FNAC)细胞学检查,是用于研究表皮(就在皮肤以下)肿块或包块的诊断程序,其对于甲状腺病变活组织检查是尤其有用的。活组织检查通过将细空心针插入包块中以收集细胞样本,该细胞样本在染色后在显微镜下检查。可以进行抽出物的细胞学检查(细胞样本评估,FNAC)或组织学检查(活检组织样本评估,FNAB)。由于实施用针穿刺活组织检查代替可以避免大多数的手术(切除或切开)活组织检查,FNA是用于甲状腺结节的流行活组织检查方法。样本采集和制备的详细描述可见于“AtlasofFineNeedleAspirationCytology”HenrykA.Domanski(2014),其内容通过引用并入本文。穿刺样本的制备在现有技术中已被充分描述。通常,适量的抽出物(通常为约一滴)薄而均匀地铺展于显微镜载片上,其之后被染色和装载。以该方法制备的FNA样本也称为“涂片”。关于样品厚度和均匀度,结果应与切片组织学载片一致。FNA涂片的固定通常通过风干(通常称为“风干流程,FNAB”)或使用95%的乙醇或细胞色素氧化酶喷雾作为固定剂来湿法固定。其他适当的液体固定剂为甲醇、丙酮、异丙醇、丙酮/甲醇等。或者,可将FNA样本添加至试管中的防腐剂或与其混合。如本文所提及的,“滤泡性”病变可为滤泡性腺瘤(FA)、滤泡性癌(FC)乳头状癌的滤泡性变体(FVPCA)中的任一种。本文使用的“片段”指核酸的非全长部分。因此,片段本身也是核酸。本文使用的“槽沟结合剂”和/或“小沟结合剂”(MGB)可交换使用,指一般以序列特异性方式适于双链DNA小沟中的小分子。小沟结合剂可为狭长的、平面的分子,其可以采用新月形,因此紧密地位于双螺旋的小沟中,其通常置换水。小沟结合剂分子可以一般具有通过自由旋转的键连接的若干芳香族环,例如呋喃环、苯环或吡咯环。小沟结合剂可为抗生素,例如纺锤菌素、偏端霉素、贝尼尔、戊烷脒和其他芳香族联脒,Hoechst33258、SN6999、金霉抗肿瘤药物例如色霉素和光神霉素、CC-1065、二氢环化吲哚卟啉三肽(DPI3)、1,2-二氢-(3H)-吡咯并[3,2-e]吲哚-7-羧酸盐(CDPI3),以及相关化合物和类似物,其包括NucleicAcidsinChemistryandBiology,第2版,Blackburn和Gait著,OxfordUniversityPress,1996,以及PCT公开申请WO03/078450中所描述的那些,其内容通过引用并入本文。小沟结合剂可为引物、探针、杂交标签补序列或其组合的成分。小沟结合剂可提高其附着的引物或探针的Tm,使得这些引物或探针在较高温度下能够有效杂交。如在本文两种或更多种核酸序列的情况下所使用的“相同的”或“一致性”指具有在特定区域相同的特定百分比的残基的序列。该百分比可通过以下方式计算:最佳地对齐两个序列,对比特定区域内的两个序列,确定在两个序列中出现相同残基的位置数量以获得匹配位置的数量,用匹配位置的数量除以特定区域位置的总数,将结果乘以100以获得序列一致性的百分比。在两个序列具有不同长度或对齐过程产生一个或更多个交错末端且比较的特定区域仅包含单一序列的情况下,单一序列的残基包含于分母中,而不是计算的分子。当比较DNA和RNA序列时,将胸腺嘧啶(T)和尿嘧啶(U)视为等同的。一致性可手动实施或通过使用计算机序列算法例如BLAST、BLAST2.0等实施。如本文所使用的,“原位检测”指检测在原始位点的表达或表达水平,该原始位点在此指组织样本例如活组织切片。如本文所使用的,“标记”指可被光谱方法、光化学方法、生物化学方法、免疫化学方法、化学方法或其他物理学方法检测到的组合物。标记可为不在蛋白质或核酸中天然存在并允许核酸或蛋白质可检测到的任意实体。例如,可用的标记包括32P、荧光染料、电子致密试剂、酶、生物素、地高辛、或半抗原和可以检测到的其他实体等。标记可在任何位置并入核酸和蛋白质中。“逻辑回归”是一类称作广义线性模型的统计模型类别的一部分。逻辑回归能够从变量集中预测离散结果如群组成员关系,变量集可为连续的、离散的、对分的或其任意混合物。从属变量或响应变量可为对分的,例如,两种可能癌症类型中的一种。逻辑回归模拟让步比的自然对数,即属于第一组的概率(P)相对于属于第二组的概率(1–P)的比例,其作为不同表达水平的线性组合(在对数空间)。通过规定当P大于0.5或50%时情况或样本被分类为第一类型,逻辑回归输出可以用作分类器。或者,在其他情况下,计算的概率P可以用作变量,例如1D或2D阈值分类器。如本文所使用的,术语“先验值”指每种类别的概率,例如分至不同类别并根据样品为恶性或良性的可能性而使用,而不需要在分类中关于样品的表达谱的其他认识。先验值可为不同比例的集合,例如80%至20%的恶性-良性、75%至25%的恶性-良性、70%至30%的恶性-良性、65%至35%的恶性-良性、60%至40%的恶性-良性、50%至50%的恶性-良性(即平均的)。另外,先验值可为经验性的,即基于样本在训练组中的分布。可校正先验值以实现预定的敏感性或特异性。如本文所使用的,“标志”为微RNA或核酸序列,将其存在或缺失在样本中测量。“标志”还提供样本状态的指示。如本文所使用的,“恶性标志”为在恶性样本中比在良性样本中以更高水平存在的微RNA或核酸序列。恶性标志可存在于或不存在于测试样本中。如本文所使用的,“次级标志”为用于区分恶性样本和良性样本的微RNA或核酸序列,所述次级标志在恶性样本和良性样本中的表达水平的差值或比值小于在恶性标志中的表达水平的差值或比值。次级标志可存在于或不存在于测试样本中。如本文所使用的,“细胞型标志”指表达与特定细胞类型有关的微RNA或核酸序列。所述细胞型通常存在于样本中,例如血细胞、白血球、红血球、上皮细胞、Hurthle细胞、线粒体富集细胞、淋巴细胞、滤泡细胞、滤泡旁细胞(C细胞)、转移细胞、免疫细胞、巨噬细胞等。包括为“细胞型标志”的其他标志可为物种特异性标志,例如来自细菌、真菌等的标志。如本文所使用的,“正规化子”指其特征(即表达水平)用于归一化各样品的微RNA或核酸序列。正规化子可单独使用(一种微RNA作为正规化子)或作为正规化子集合(超过一种RNA作为正规化子,例如2种、3种、4种、5种、6种、7种、8种、9种、10种、11种、12种、13种、14种、16种、或17种微RNA可用作该集中的正规化子)的一部分。如本文所提及的,在样本中检测的任何微RNA可用作正规化子。为此,本文定义的作为“标志”的微RNA也可以用作“正规化子”。基本上,任何微RNA可以用作正规化子。为此,表示为SEQIDNO1至SEQIDNO182中任一个的微RNA可用作正规化子。表示为SEQIDNO1至SEQIDNO37中任一个的微RNA可用作正规化子。可用作正规化子的微RNA的具体实例为hsa-miR-23a-3p、MID-20094、MID-50969、hsa-miR-345-5p、hsa-miR-3074-5p、MID-50976、MID-50971、hsa-miR-5701和hsa-miR-574-3p。数据值的“归一化”指将原始数据转换为另一比例。可以通过以下步骤实现归一化:减去正规化子集合的表达平均值,减去正规化子集合的表达中值,拟合正规化子的表达值与值的参考集合(使用多项式拟合)并将该拟合应用于所有特征。可使用所有的正规化子或正规化子亚集合。如本文所使用的,“核酸”或“寡核苷酸”或“多核苷酸”指至少两个核苷酸共价地键结在一起。单链的描述也定义了互补链的序列。因此,核酸也包括所描述的单链的互补链。出于相同目的,核酸的多种变体可以用作给定的核酸。因此,核酸也包括基本相同的核酸和其互补链。单链可以提供在严格杂交条件下与靶序列杂交的探针。因此,核酸还包括在严格杂交条件下杂交的探针。核酸可为单链或双链,或者可含有双链-单链序列和单链-双链序列二者的部分。核酸可以为DNA、基因组和cDNA两者、RNA、或其中核酸可以含有脱氧核糖核苷酸和核糖核苷酸的组合的杂合体,以及包括尿嘧啶、腺嘌呤、胸腺嘧啶、胞嘧啶、鸟嘌呤、肌苷、黄嘌呤、次黄嘌呤、异胞嘧啶和异鸟嘌呤的碱基对组合。通过化学合成法或通过重组法可以获得核酸。尽管可以包括核酸类似物,核酸一般含有磷酸二酯键。类似物可以包括非天然存在的键、主链或核苷酸。类似物可以具有至少一个不同键,例如氨基磷酸酯键、硫代磷酸酯键、二硫代磷酸酯键、O-甲基氨基亚磷酸酯键以及肽核酸主链和键。其他核酸类似物包括具有正向主链、非离子主链和非核糖主链的那些,包括US5235033和US5034506中描述的那些,其通过引用并入本文。含有一种或更多种非天然存在的核苷酸或修饰的核苷酸的核酸也包括于核酸的一种定义中。修饰的核苷酸类似物可以位于例如核酸分子的5'末端和/或3'末端。核酸类似物的代表性实例可以选自糖修饰或主链修饰的核糖核苷酸。然而应注意的是,碱基修饰的核糖核苷酸,即含有非天然存在碱基代替天然存在碱基的核糖核苷酸,例如在5位置修饰的尿苷或胞嘧啶核苷,例如5-(2-氨基)丙基尿苷、5-溴尿苷;在8位置修饰的腺苷和鸟苷,例如8-溴鸟苷;去氮核苷酸,例如7-去氮腺苷;O-烷基化核苷酸和N-烷基化核苷酸,例如N6-甲基腺苷也是合适的。2'-OH-基团可以由选自以下的基团替代:H、OR、R、卤素、SH、SR、NH2、NHR、NR2或CN,其中R为C1至C6的烷基、烯基或炔基,卤素为F、Cl、Br或I。修饰的核苷酸还包括与胆固醇通过例如羟脯氨醇键共轭的核苷酸,其如Krutzfeldt等人(Nature2005;438:685-689),Soutschek等人(Nature2004;432:173-178)和WO2005/079397中所描述的,其通过引用并入本文。可由于不同原因对核糖磷酸主链进行修饰,例如以提高这种分子在生理环境下的稳定性和半衰期,增强穿过细胞膜的扩散,或作为生物芯片的探针。主链修饰还可以增强抗降解性,例如在细胞内吞作用环境中。主链修饰还可减小由肝细胞导致的核酸清除,例如在肝脏和甲状腺中。可以制备天然存在核苷酸和类似物的混合物。或者,可以制备不同核苷酸类似物的混合物、天然存在核苷酸和类似物的混合物。因此,本文提供新的分离核苷酸。本文提供的核酸可以为非天然存在的、合成的核酸。因此,本文提供的核酸可以为合成核酸。合成核酸的方法是本领域技术人员已知的,在例如US7579451中描述,其内容通过引用并入本文。核酸可包括序列SEQIDNO:1至SEQIDNO:308中的至少一个或其变体。在一个实施方式中,核酸包括序列SEQIDNO:1至SEQIDNO:182中的至少一个。变体可以为参照核苷酸序列的互补序列。变体可为70%、75%、80%、85%、90%或95%与参照核苷酸序列或其互补序列一致性的核苷酸序列。变体可以为在严格条件下与参照核苷酸序列、其互补序列、或基本与其相同的核苷酸序列杂交的核苷酸序列。本文描述的核酸可以具有约10个至约250个核苷酸的长度。核苷酸可以具有至少10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、35个、40个、45个、50个、60个、70个、80个、90个、100个、125个、150个、175个、200个或250个核苷酸的长度。可以使用合成基因在细胞中合成或表达(体外或体内)核酸。核酸可作为单链分子合成并与基本上互补的核酸杂交以形成双链体。核酸可以单链或双链形式引入细胞、组织或器官,或能够通过合成基因使用本领域技术人员已知的方法而被表达,本领域技术人员已知的方法包括如在US6506559中所描述的,其内容通过引用并入本文。核酸可以包括如表1所示的微RNA序列或其变体。在某些情况下,相同微RNA的变体也提供于表1中。应注意的是,表1中的SEQIDNO.1至SEQIDNO.180表示与天然存在微RNA的序列对应的cDNA,即该序列以胸腺嘧啶(T)代替尿嘧啶(U)出现。应理解,核酸指脱氧核糖核苷酸、核糖核苷酸、或修饰核苷酸以及其以单链或双链形式的聚合物。该术语包括含已知核苷酸类似物或修饰的主链残基或键的核酸,其为合成的、天然存在的和非天然存在的,其具有与参照核酸相似的结合性能,并以与参照核酸类似方式进行新陈代谢。该类似物的实例包括但不限于,硫代磷酸酯、氨基磷酸酯、甲基膦酸酯、手性甲基膦酸酯、2-O-甲基核糖核苷酸、肽-核酸(PNA)和解锁核酸(UNA;参见例如Jensen等人,NucleicAcidsSymposiumSeries52:133-4)、及其衍生物。核甘酸如本领域公知地使用,包括本领域所熟知的具有天然碱基(标准)和修饰的碱基的那些。这些碱基一般位于核苷酸糖部分的1'位置。核苷酸一般包含碱基、糖和磷酸酯基团。核苷酸可以为在糖部分、磷酸酯部分和/或碱基部分不经修饰或经修饰,也可交换地称为核苷酸类似物、修饰核苷酸、非天然核苷酸、非标准核苷酸以及其他(参见例如WO92/07065;WO93/15187,其内容通过引用并入本文)。本领域已知的修饰核酸碱基的若干实例如Limbach等人NucleicAcidsRes.22:2183,1994总结的。可以引入核酸分子中的碱基修饰的一些非限制性实例包括次黄嘌呤、嘌呤、吡啶-4-酮、吡啶-2-酮、苯基、假尿嘧啶、2,4,6-三甲氧基苯、3-甲基尿嘧啶、二氢尿苷、萘基、氨基苯基、5-烷基胞苷(例如5-甲基胞苷)、5-烷基尿苷(例如核糖胸核苷)、5-卤尿苷(例如5-溴尿苷)或6-氮嘧啶或6-烷基嘧啶(例如6-甲基尿苷)、丙炔以及其他(Burgin等人,Biochemistry35:14090,1996)。该方面中的“修饰碱基”指在1'位置不同于腺嘌呤、鸟嘌呤、胞嘧啶和尿嘧啶或其等同物的核苷酸碱基。修饰核苷酸指具有一个或更多个对核苷、碱基、戊糖环或磷酸酯基团修饰的核苷酸。修饰包括通过修饰核苷酸的酶修饰而获得的天然存在的那些,所述酶例如甲基转移酶。修饰核苷酸还包括合成的或非天然存在的核苷酸。核苷酸中合成的或非天然存在的核苷酸修饰包括具有2’位修饰的那些,例如2'-甲氧基乙氧基、2'-氟、2'-烯丙基,2'-O-[2-(甲基氨基)-2-氧代乙基]、4'-巯基、4'-CH2-O-2'-桥,4'-(CH2)2-O-2'-桥、2'-LNA或其他二环或“桥的”核苷类似物,和2'-O-(N-氨基甲酸甲酯),或包含碱基类似物的那些。如本公开所描述的,与2’位修饰核苷酸有关的“氨基”指2'-NH2或2'-O-NH2,其可为修饰的或未修饰的。这种修饰基团例如在US5672695和US6248878中描述。本发明“修饰核苷酸”也可以包括如上所述的核苷酸类似物。如本文使用的,“碱基类似物”指位于修饰核酸中的核酸糖部分1’位置的杂环部分,其能够并入核酸双链体中(或在核酸糖部分取代物的等同位置,其能够并入核酸双链体中)。碱基类似物一般为除常见碱基鸟嘌呤(G)、胞嘧啶(C)、腺嘌呤(A)、胸腺嘧啶(T)和尿嘧啶(U)以外的嘌呤碱基或嘧啶碱基。碱基类似物可与dsRNA中的其他碱基或碱基类似物成为双链体。碱基类似物包括在发明的化合物和方法中使用的那些,例如,US5432272、US6001983和US7579451中公开的那些,其通过引用并入本文。碱基的非限制性实例包括次黄嘌呤(I)、黄嘌呤(X)、313-D-呋喃核糖基-(2,6-二氨基嘧啶)(K)、3-γ-D-呋喃核糖基-(1-甲基-吡唑并[4,3-d]嘧啶-5,7(4H,6H)-二酮)(P)、异胞嘧啶(iso-C)、异鸟嘌呤(iso-G)、1-γ-D-呋喃核糖基-(5-硝基吲哚)、1-γ-D-呋喃核糖基-(3-硝基吡咯)、5-溴尿嘧啶、2-氨基嘌呤、4-硫代-dT、7-(2-噻吩基)-咪唑并[4,5-b]吡啶(Ds)和吡咯-2-甲醛(Pa)、2-氨基-6-(2-噻吩基)嘌呤(S)、2-氧代吡啶(Y)、二氟甲苯基、4-氟-6-甲基苯并咪唑、4-甲基苯并咪唑、3-甲基异喹诺基、5-甲基异喹诺基和3-甲基-7-丙炔基异喹诺基、7-氮杂吲哚基、6-甲基-7-氮杂吲哚基、咪唑并吡啶基、9-甲基-咪唑并吡啶基、pyrrolopyrizinyl、异喹诺基、7-丙炔基异喹诺基、丙炔基-7-氮杂吲哚基、2,4,5-三甲基苯基、4-甲基吲哚基、4,6-二甲基吲哚基、苯基、萘基、蒽基、菲基、芘基、茋基、并四苯基、并五苯基及其结构衍生物(Schweitzer等人,J.Org.Chem.,59:7238-7242(1994);Berger等人,NucleicAcidsResearch,28(15):2911-2914(2000);Moran等人,J.Am.Chem.Soc.,119:2056-2057(1997);Morales等人,J.Am.Chem.Soc.,121:2323-2324(1999);Guckian等人,J.Am.Chem.Soc.,118:8182-8183(1996);Morales等人,J.Am.Chem.Soc.,122(6):1001-1007(2000);McMinn等人,J.Am.Chem.Soc.,121:11585-11586(1999);Guckian等人,J.Org.Chem.,63:9652-9656(1998);Moran等人,Proc.Natl.Acad.Sci.,94:10506-10511(1997);Das等人,J.Chem.Soc.,PerkinTrans.,1:197-206(2002);Shibata等人,J.Chem.Soc.,PerkinTrans.,1:1605-1611(2001);Wu等人,J.Am.Chem.Soc.,122(32):7621-7632(2000);O'Neill等人,J.Org.Chem.,67:5869-5875(2002);Chaudhuri等人,J.Am.Chem.Soc.,117:10434-10442(1995);U.S.专利第6218108号)。碱基类似物还可为通用碱基。“通用碱基”指在修饰核苷酸中位于核苷酸糖部分1'位置的杂环部分,或者在核酸糖部分取代物的等同位置,当存在于核酸双链体中时,其可位于超过一种碱基相反而不改变双螺旋结构(例如磷脂主链的结构)。另外,通用碱基不破坏其所存在于双链体的单链核酸形成靶向核酸的能力。表1:本发明的微RNA1“N”可为G、C、A、T/U中的任一个;miR名称为miRBase注册名称(20期),除了由MID-[数字]或MD2-[数字]表示的miR名称;MID-00078、MID-00321、MID-00387、MID-00671、MID-00672、MID-00690、MID-15965、MID-16318、MID-17144、MID-17866、MID-18468、MID-19433、MID-19434、MID-23168、MID-23794、MID-24496、MID-24705、MD2-495和MD2-437为假定的微RNA,其在RosettaGenomics被预测和/或克隆。核酸也包括表2中所示的微RNA发夹序列或其变体。表2:本发明的微RNA发夹应注意的是,表2中的SEQIDNO.183至SEQIDNO.306表示与天然存在pre-miR的序列对应的cDNA,即该序列以胸腺嘧啶(T)代替尿嘧啶(U)出现。核酸可以以核酸复合物的形式,可以还包括肽、蛋白质、RNA-DNA杂合体、抗体、抗体片段、Fab片段或适配子的一种或更多种。核酸可以包括pri-miRNA序列或其变体。pri-微RNA序列可包括45个至30000个、50个至25000个、100个至20000个、1000个至1500个或80个至100个核苷酸。pri-miRNA序列可包含如本文所述的pre-miRNA、miRNA和miRNA*及其变体。pri-miRNA序列可以包括SEQIDNO.183至SEQIDNO.308或其变体中的任意序列。pri-miRNA可以包括发夹结构。发夹可以包含基本互补的第一核酸序列和第二核酸序列。第一核酸序列和第二核酸序列可以来自37个至50个核苷酸。第一核酸序列和第二核酸序列可以被来自8个至12个核苷酸的第三序列分离。如Hofacker等人所描述的,该发夹结构可以具有小于-25千卡/摩尔的自由能,其由含默认参数的Vienna算法计算(Monatsheftef.Chemie1994;125:167-188),其内容通过引用并入本文。该发夹可以具有4个至20个、8个至12个或10个核苷酸的末端循环。pri-miRNA可以包括至少19%的腺苷核苷酸、至少16%的胞嘧啶核苷酸、至少23%的胸腺嘧啶核苷酸和至少19%的鸟嘌呤核苷酸。核酸也可以包括pre-miRNA序列或其变体。pre-miRNA序列可以包括45个至90个、60个至80个或60个至70个核苷酸。pre-miRNA序列可以包含如本文所述的miRNA和miRNA*。pre-miRNA序列还可以为不包含从pri-miRNA的5’和3’末端算起的0至160个核苷酸的pre-miRNA序列。pre-miRNA序列可以包括序列SEQIDNO.183至SEQIDNO.308或其变体。如本文所描述的,核酸可为在8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、30个、35个、40个、45个、50个或更多个核苷酸区域中,与表1或表2中的核酸序列至少70%、75%、80%、85%、90%、95%、97%、98%或99%一致性(80%至99%具有1%增量)。核酸还可以包括微RNA序列(包括miRNA*)或其变体,包括那些由MID-[数字]表示的假定微RNA。如本文所提及的,微RNA包括已经列于miRBase注册名称(发行20)中的那些miR,以及已经由RosettaGenomics预测和/或克隆并由MID-[数字]表示的假定微RNA。微RNA序列可以包括13个至33个、18个至24个或21至23个核苷酸。微RNA可以具有总计至少5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、31个、32个、33个、34个、35个、36个、37个、38个、39个或40个核苷酸的长度。微RNA序列可为pre-miRNA中最初的13个至33个核苷酸。微RNA序列也可以为pre-miRNA中最后的13个至33个核苷酸。微RNA序列可以包括SEQIDNO.1至SEQIDNO.182中任一个的序列或其变体。本发明使用微RNA用于甲状腺结节的鉴别、分类和诊断。如本文所使用的,“变体”指核酸,其意为(i)参照核苷酸序列的部分;(ii)参照核酸苷序列或其部分的互补序列;(iii)通过点突变而与参照核苷酸序列或其互补序列不同的核酸;(iv)存在普通群组中的参照核苷酸序列的天然存在变体或其互补序列;或(iv)在严格条件下与参照核酸或其互补序列杂交的核酸。如本文所使用的,“探针”指能够通过一种或更多种化学键与靶核酸互补序列结合的寡核苷酸,通常通过互补碱基对,通常经过氢键形成。取决于杂交条件的严格性,探针可以结合缺少与探针序列完整互补的靶序列。例如,为进行杂交试验,探针可以与被检测的微RNA序列中至少8个、至少9个、至少10个、至少11个、至少12个、至少13个、至少14个、至少15个、至少16个、至少17个、至少18个、至少19个、至少20个连续核苷酸互补。或者,为进行PCR试验,探针可与被检测的PCR产物中至少8个、至少9个、至少10个、至少11个、至少12个、至少13个、至少14个、至少15个、至少16个、至少17个、至少18个、至少19个、至少20个连续核苷酸互补。因此,探针可与其靶核酸的至少60%、65%、70%、75%、80%、85%、90%、95%、97%、98%或99%互补或杂交。探针可以为单链或部分单链和部分双链的。探针的成链是由结构、组成和靶序列特性所控制的。探针可以包括标记、附着物、或者本文描述的非天然存在于核酸中的核苷酸序列。例如使用可结合链霉亲和素复合物的生物素,探针可被直接标记或间接标记。“探针”可以为检测本文描述的核酸序列的试剂。探针可以为标记的核酸探针,其能够与本发明核酸序列的部分或由其派生的扩增产物杂交。在一些实施方式中,核酸探针为本文公开的核酸序列的反向互补的核酸分子。探针可以为在严格条件下与本文公开的核酸充分特异性杂交的核酸序列。探针任选地使用荧光分子标记,荧光分子例如荧光素,如6-羧基荧光素(FAM)、吲哚羰花青如QUASAR-670(QUA)、六荧光素(hexafluorocine)如6-羧基六荧光素(HEX)、或其他荧光团分子和任选的猝灭剂。猝灭剂优选与荧光团匹配。猝灭剂的示例性实施例为黑洞猝灭剂BHQ1和黑洞猝灭剂BHQ2,或小沟结合剂(MGB),例如二氢环吡咯并吲哚三肽。其他荧光团和猝灭剂是本领域已知的,并在本文中是类似可用的。因此,本发明还提供探针,所述探针具有本文描述的新核酸序列,其定义为SEQIDNO.27至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139、SEQIDNO.140、SEQIDNO.307和SEQIDNO.308中的任一种或其变体。探针可以用于筛查方法和诊断方法。探针可以附着于或固定于固体载体例如生物芯片上。探针可以具有8个至500个、10个至100个或20个至60个核苷酸的长度。探针可以具有至少8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、35个、40个、45个、50个、60个、70个、80个、90个、100个、120个、140个、160个、180个、200个、220个、240个、260个、280个或300个核苷酸的长度。探针还可以具有10个至60个核苷酸的接头序列。探针还可以包含接头。该接头可以包含本文描述的非天然存在于核酸序列的序列。接头可具有10个至60个核苷酸的长度。接头可以具有20个至27个核苷酸的长度。接头可以为足以使探针总长度为45个至60个核苷酸的长度。接头可以无法形成稳定的二级结构,或无法自身折叠,或无法在含于探针中的核苷酸的非接头部分上折叠。接头序列为异源的,其可以不出现在动物基因组中,探针非接头核酸来自该动物。如本文所使用的,术语“参考值”指相比于试验结果,在统计学上和具体结果相关的值。在一个实施方式中,参考值由将微RNA表达和已知临床结果比较的研究的统计学分析确定。在另一个实施方式中,参考值可以根据所使用的分类器(即算法)而不同。因此,参考值可以为训练数据中的所有微RNA的表达水平(或值)。参考值可以为由分类器建立的一个或更多个阈值。参考值还可以为系数或系数集合。基本上参考值指算法需要的或使用的任何参数。如本文所使用的,“敏感性”可以指二元分类测试在多大程度上正确识别条件的统计学测量,例如其在多大频率上将癌正确分类为两种可能类型中的正确类型。如通过一些绝对标准或金标准所确定的,A类的敏感性为由测试确定的属于A类的情况在A类情况中所占的比例。如本文所使用的,“敏感性”可以指分类测试在多大程度上正确识别条件的统计学测量,例如其在多大频率上将癌正确分类为两种或更多种可能类型中的正确类型。如通过一些绝对标准或金标准所确定的,A类的敏感性为由测试确定的属于A类的情况在A类情况中所占的比例。如本文所使用的,“涂片”指用于检测、一般用于医疗诊断的薄铺于显微镜载片的甲状腺组织样本。FNA涂片通常具有非常少量的细胞,这导致少量的RNA,相应地RNA的量可以为1ng至1000ng、1ng至100ng、1ng至50ng、1ng至40ng。涂片可使用领域技术人员已知的细胞学、组织学或病理学上任意着色剂染色,例如在病理学样本中用于区分细胞的任意着色剂。着色剂的实例为多色着色剂,例如Papanicolaou,其为细胞核着色剂和细胞质着色剂的组合;细胞结构着色剂例如Wright、Giemsa、Romanowsky等;细胞核着色剂例如Hoescht着色剂等;细胞生存力着色剂例如Trypanblue等,酶活性,例如对于HRP的联苯胺以形成可见的沉淀等。如本文所使用的,“特异性”可指二元分类测试在多大程度上正确识别不具有特定条件的情况的统计学测量,例如,当样本为非癌性样本时,二元分类测试在多大频率上将样本正确归类为非癌性。如通过一些绝对标准或金标准所确定的,A类的特异性为由测试确定的属于“非A”类的情况在“非A”类情况中所占的比例。如本文所使用的,“特异性”可指分类测试在多大程度上正确识别不具有特定条件的情况的统计学测量。如通过一些绝对标准或金标准所确定的,A类的特异性为由测试确定的不属于A类的情况在A类情况中所占的比例。如本文所使用的,“癌症的阶段”指癌症进展水平的数据化测量。用于确定癌症阶段的标准包括但不限于,肿瘤尺寸、肿瘤是否扩散到身体其他部位和癌症已经扩散的位置(例如在身体的相同器官或区域以内还是扩散到其他器官)。如本文所使用的,“严格杂交条件”指在该条件下第一核酸序列(例如探针)与第二核酸序列(例如靶)杂交,例如在核酸的复合混合物中。严格条件为序列依赖性的,在不同环境下是不同的。严格条件可以选择为在定义的离子强度pH下,低于特定序列的热熔点(Tm)约5℃至10℃。Tm可以为在平衡(靶序列在Tm过量存在,50%的探针在平衡时被占据)时50%的与目标互补探针与靶序列杂交的温度(在定义的离子强度、pH和核酸浓度下)。严格条件可为盐浓度小于约1.0M钠离子,例如在pH7.0至8.3下约0.01至1.0M钠离子浓度(或其他盐),对于短探针(例如约10个至50个核苷酸),温度为至少约30℃,对于长探针(例如大于约50个核苷酸),温度为至少约60℃。严格条件还可以通过添加去稳定剂例如甲酰胺实现。对于选择性或特异性杂交,阳性特征可以为背景杂交的至少2至10倍。示例性严格杂交条件包括以下:50%的甲酰胺、5xSSC和1%的SDS,在42℃下保温,或5xSSC,1%的SDS,在65℃下保温,在0.2xSSC中洗涤,以及在65℃下0.1%的SDS、DMSO、6XSSPE+0.005%的N-月桂酰肌氨酸+0.005%的TritonX-102、0.06X的SSPE+0.005%的N-月桂酰肌氨酸+0.005%的TritonX-102。如本文所使用的,术语“受试者”指哺乳动物,包括人类和其他哺乳动物。本发明的方法优选应用于人类受试者。如本文所使用的,“癌症的亚型”指影响相同器官的癌症不同类型(例如,甲状腺乳头状癌、甲状腺滤泡性癌和甲状腺乳头状癌的滤泡性变体)。如本文所使用的,“甲状腺病变”可指甲状腺肿瘤,包括甲状腺肿瘤的亚型,例如Hashimoto病、滤泡型癌、乳头状癌、乳头状癌的滤泡性病变(FVPC或FVPTC)、包膜内型FVPC(或包膜内型FVPTC)、非包膜内型(渗透的/扩散的)FVPC或FVPTC、髓样癌、未分化甲状腺癌、或低分化甲状腺癌。如本文所使用的,短语“阈值表达谱”指测量值与之比较以将肿瘤分类的标准表达谱。如本文所使用的,组织样本为使用相关医疗领域的技术人员所熟知的方法由组织活检获得的组织。如本文所使用的,短语“疑似癌性的”指医疗领域技术人员认为含有癌细胞的癌组织样品。由活组织检查获得样本的方法包括包块的粗分配、显微解剖、激光显微解剖或其他本领域已知的细胞分离方法。如本文所使用的,“肿瘤”指所有赘生性细胞生长和增殖,无论恶性或良性,以及所有癌前和癌性细胞和组织。本文所使用的甲状腺病变或肿瘤样本的细胞学分类是基于“TheBethesdaSystemforReportingThyroidCytopathology”,“BSRTC”(Syed,Z.Ali和EdmundS.Cibas著;DOI10.1007/978-0-387-87666-5_1;SpringerScience+BusinessMedia,LLC2010)。BSRTC推荐带有一般诊断分类的各种甲状腺FNA报告,其中每种分类具有隐含的患癌风险。Bethesda分类推荐的命名法如下所示:I.非诊断性的或不符合要求的仅囊肿液几乎为非细胞样本其他(浑浊的血液、凝血制品等)。II.良性与良性滤泡性结节(包括类腺瘤结节、胞质结节等)一致在适当的临床条件下,与淋巴细胞性(Hashimoto)甲状腺炎一致与肉芽肿(亚急性)甲状腺炎一致其他III.未定类显著性的异型性或未定类显著性的滤泡性病变IV.滤泡性肿瘤或疑似滤泡性肿瘤特别在Hurthle细胞(嗜酸瘤细胞)型V.疑似恶性疑似乳头状癌疑似髓样癌疑似转移性癌疑似淋巴瘤其他VI.恶性乳头状甲状腺癌低分化癌甲状腺髓样癌不分化(未分化)癌鳞状细胞癌具有混合特性的癌转移性癌非霍奇金淋巴瘤其他如本文所使用的,“未定类”指进行细胞学检查并根据Bethesda分类而分类为III、IV和V类的甲状腺病变或肿瘤样本。本发明还提供用于鉴别受试者中甲状腺病变亚型的方法,所述甲状腺病变亚型为甲状腺肿瘤的所述恶性亚型或良性亚型。亚型为以下任一种:滤泡性癌、乳头状癌、乳头状癌的滤泡性变体(FVPC或FVPTC)、包膜内型FVPC(或包膜内型FVPTC)、非包膜内型FVPC(或非包膜内FVPTC)、髓样癌、未分化甲状腺癌或低分化甲状腺癌。在另一个实施方式中,所述亚型为Hashimoto甲状腺炎、滤泡性腺瘤或增生中的任一种。在另一个实施方式中,所述亚型为Hashimoto细胞癌。在另一方面,本发明提供用于区分滤泡性腺瘤和滤泡性癌的方法。在另一方面,本发明提供用于区分滤泡性腺瘤和乳头状癌的方法。在另一方面,本发明提供用于区分滤泡性腺瘤和乳头状癌的滤泡性变体的方法。在另一方面,本发明提供用于区分乳头状癌的非包膜内型滤泡性变体和良性病变的方法。在另一方面,本发明提供用于区分乳头状癌和Hashimoto甲状腺炎的方法。“载体”指任意已知载体例如质粒载体、噬菌体载体、噬菌粒载体、粘粒载体或病毒载体。本文所述的核酸可包含于载体中。载体可用于输送核酸。载体优选至少含有提高所负载核酸表达的启动子,在该情况下,核酸优选与该启动子切实连接。载体在宿主细胞中是或不是可复制的,基因转录可以在宿主细胞的细胞核外或细胞核内进行。在后述情况下,核酸可以包含于宿主细胞的基因组。载体可以为DNA载体或RNA载体。载体可以为可自我复制型染色体外载体或融入宿主基因组的载体。在本发明方法或方案的一个实施方式中,通过逆转录聚合酶链反应(RT-PCR)测量微RNA水平。cDNA的靶序列通过靶RNA的逆转录产生,其可以为本文描述的核酸(包含表1和表2中提供的序列)。产生cDNA的已知方法涉及逆转录聚腺苷酸或逆转录具有连接的衔接子序列的RNA。在逆转录之前,RNA可以连接于衔接子序列。通过T4RNA连接酶可进行连接反应以在RNA的3’末端连接衔接子序列。然后可以使用引物进行逆转录(RT)反应,该引物具有与衔接子序列的3’末端互补的序列。或者,使用包含5’衔接子序列poly(T)引物,聚腺苷酸RNA可以用于逆转录(RT)反应中。poly(T)序列可以包含8个、9个、10个、11个、12个、13个或14个连续胸腺嘧啶。然后使用包含与靶核酸互补的至少15个核苷酸和5’末端序列的特异性正向引物,与衔接子序列的3’末端互补的反向引物和包含与靶核酸互补的至少8个核酸的探针,通过实时PCR可以扩增RNA的逆转录。该探针可部分与衔接子序列的5’末端互补。可通过PCR等进行靶核酸(微RNA,包括本文描述的假定性微RNA)逆转录的扩增。PCR反应的第一循环可具有56℃、57℃、58℃、59℃或60℃的退火温度。第一循环可包含1个至10个循环。PCR反应的剩余循环可为60℃。剩余循环可具有2个至40个循环。PCR反应具有正向引物。在一个实施方式中,正向引物可具有与靶核酸相同的15个、16个、17个、18个、19个、20个或21个核苷酸。正向引物的3’末端可对靶核酸和高度相似序列之间的序列差异性敏感。正向引物还可以具有5’突出末端。5’末端可以提高正向引物的熔融温度。5’末端的序列可以包含与靶核酸不相同的序列。5’末端的序列也可以是合成的。5’末端可以包含8个、9个、10个、11个、12个、13个、14个、15个或16个核苷酸。本发明使用的正向引物实例提供于表8中。PCR反应包含反向引物。反向引物可以与靶核酸互补。反向引物也可以包含与衔接子序列互补的序列。本发明使用的反向引物实例提供于实施例8中。用于检测RT-PCR扩增产物的探针可为通用探针或序列特异性探针。通用探针设计为以非序列特异性方式检测RT-PCR扩增产物(或与其杂交)。所述探针为16个至20个核苷酸长,优选18个核苷酸长,并包含与RT引物反向互补的序列,在5’末端包含4个腺嘌呤(A)。序列特异性探针设计为基于在探针序列和RT-PCR产物序列之间全部或部分的互补性检测RT-PCR扩增产物(或与其杂交)。所述探针为20个至28个核苷酸长,优选24个核苷酸长,并在5’末端包含3个核苷酸,其中至少两个为与RT引物互补,然后是10至14个、优选12个胸腺嘧啶(T),然后是6个至10个、优选8个连续核苷酸,其对应于特异性对应的微RNA的反向互补序列。提供包含本文描述的新核苷酸的生物芯片。在一个实施方式中,该生物芯片可以包含识别本文所述的新核苷酸的探针。所述核酸为分离的核酸,其包含至少12个连续核苷酸,其与SEQIDNO.27至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139、SEQIDNO.140、SEQIDNO.307和SEQIDNO.308中任一的序列至少80%一致性。在一个实施方式中,所述分离的核酸包含至少13个、至少14个、至少15个、至少16个、至少17个、至少18个、至少19个或至少20个连续核苷酸,其与SEQIDNO.27至SEQIDNO.29、SEQIDNO.33、SEQIDNO.34、SEQIDNO.139、SEQIDNO.140、SEQIDNO.307和SEQIDNO.308中任一的序列具有一致性。该生物芯片可以包括含有附着的核酸的固体载体、本文描述的探针或多个探针。该探针在严格杂交条件下能够与靶序列杂交。探针可以附着于载体上空间限定的位置。每个靶序列可以使用超过一个探针,可以为重叠探针或为针对具体靶序列不同部分的探针。该探针能够与本领域技术人员所理解的单一病症相关的靶序列杂交。可以将探针首先合成然后附着到生物芯片上,或者可以直接在生物芯片上合成。固体载体可以为经修饰以含有离散单个位点以有利于探针的附着或连接的材料,并适用于至少一种检测方法。载体的代表性实例包括玻璃以及改性或功能化的玻璃、塑料(包括丙烯酸树脂、聚苯乙烯和苯乙烯与其它材料的共聚物、聚丙烯、聚乙烯、聚丁烯、聚氨酯、TeflonJ等)、多糖、尼龙或硝酸纤维素、树脂、二氧化硅或基于二氧化硅的材料包括硅胶和改性硅胶、碳、金属、无机玻璃和塑料。载体可以允许光学检测而没有明显地荧光。载体可为平面的,但也可以使用其他形状的载体。例如,探针可置于试管的内表面以进行流入式样品分析以最小化样品体积。类似地,载体可为柔性的,例如柔性泡沫,包括特定塑料制造的闭孔泡沫。生物芯片和探针可以衍生自化学官能团以进行二者的随后连接。例如,生物芯片可以衍生自的化学官能团包括但不限于:氨基基团、羧基基团、氧代基团或巯基基团。使用这些官能团,可以使用探针上的官能团直接或间接地使用接头使探针附着。探针可以通过5’末端、3’末端或经由内部核苷酸附着于固体支撑物。探针还可以非共价地附着于固体支撑物。例如,可以制备生物素化的寡核苷酸,其可以共价地连接于涂覆有链霉亲和素的表面以实现附着。或者,可以使用例如光聚合和光刻蚀的技术在表面上合成探针。在本发明的另一个实施方式中,可以通过高通量测序法实现测定用于甲状腺病变分类的微RNA。高通量测序法可以涉及合成测序法、连接测序法和超深测序法。合成测序法可以通过使用与核酸标签上的排序元素互补的测序引物启动。在聚合酶反应中,该方法涉及在标记核酸或核酸类似物进入到互补核酸序列增长链之后立刻(基本实时)检测每个核酸的同一性,或者标记核酸或核酸类似物一经(实时)进入到互补核酸序列增长链就检测每个核酸的同一性。在成功结合标记核酸之后,使用本领域已知方法测量特征然后清零。合成序列法的实例是本领域已知的,并在例如US7056676、US8802368和US7169560中描述,其内容通过引用并入本文。合成序列法中可以用于标记核苷酸或核苷酸类似物的标记实例包括但不限于发色团、荧光部分、酶、抗原、重金属、磁探针、染料、磷光基团、放射性材料、化学发光部分、散射纳米颗粒或荧光纳米颗粒、产生拉曼信号的部分和电化学检测部分。合成序列法每小时可以产生至少1000个、至少5000个、至少10000个、至少20000个、30000个、至少40000个、至少50000个、至少100000个或者至少500000个测序片段。该测序片段的每个测序片段可以具有至少40个、至少45个、至少50个、至少60个、至少70个、至少80个、至少90个、至少100个、至少120个或者至少150个碱基。可以通过使用折回PCR和锚定引物在固体载体(或芯片)上进行合成测序法。由于微RNA作为小核酸片段出现——衔接子被添加到片段的5’末端和3’末端。附着于流动细胞通道表面的核酸片段被延长并被桥扩增。片段变为双链的,并将该双链分子变性。变性之后的固相扩增多循环可以在每个流动细胞通道中生成几百万簇的约1000个拷贝的相同模板单链核酸分子。使用引物、聚合酶和四个荧光标记的可逆终止核苷酸进行连续测序。在核酸结合之后,使用激光刺激发色团,捕获图像并记录第一碱基的同一性。去除每个结合碱基的3'终止子和发色团,并重复合并、检测和鉴别步骤。在例如测序平台上使用该技术。另一种测序方法涉及使扩增区域与在LST中和与序列元素互补的引物杂交(列出fasta文件名称的文件)。使用聚合酶、ATP硫酸化酶、荧光素酶、三磷酸腺苷双磷酸酶和载体荧光素和腺苷5’磷酰硫酸保温该杂交复合物。然后,相继加入对应于A、C、G和T(U)的三磷酸脱氧核苷酸。每个碱基的合并伴随着焦磷酸的释放,通过硫酸化酶转变为ATP,这驱动氧化荧光素的合成和可见光的释放。由于焦磷酸释放与合并的碱基是等摩尔的,发出的光与在任一步骤添加的核苷酸数量是成比例的。重复该过程直至确定整个序列。另一种测序方法涉及通过连接方案(简并连接)的四色测序,其涉及使锚定引物与四个位置中的一个杂交。然后进行锚定引物和用荧光染料标记的简并九聚体群组的酶促连接反应。在任意给定的循环,构建使用的九聚体群组以使其位置之一的同一性与附着于该九聚体的发色团的同一性相关。对于在所查询的位置处连接酶区别于互补性的程度,荧光信号允许碱基同一性的推断。实施连接和四色成像之后,剥除锚定引物:九聚物复合物并开始新循环。在实施连接之后的序列信息成像方法是本领域已知的。在一些情况下,高通量测序涉及使用超深测序,例如在Marguiles等人,Nature437(7057):376-80(2005)中所描述的。微RNA测序(miRNA-seq)是一种RNA测序(RNA-Seq)类型,其使用下一代测序或大规模并行高通量DNA测序以对微RNA测序。miRNA-seq与其他形式RNA-seq的不同之处在于输入原料通常富集小RNA。miRNA-seq提供组织特异性表达模式,其可导致疾病关联性和微RNA亚型。miRNA-seq也可以用于发现先前未表征的微RNA,例如表示为SEQIDNO.139至SEQIDNO.140和SEQIDNO.307至SEQIDNO.308的核酸序列。如本文所使用的,术语“诊断”指将病理或症状分类,确定病理严重程度(等级或阶段),监测病理进展,预测病理结果和/或康复预期。如本文所使用的,短语“需要其的受试者”指已知患有癌症的人类受试者、具有患癌风险的人类受试者(例如,有遗传倾向的受试者、有癌症医疗史和/或家族史的受试者、已暴露于致癌物质、职业危害、环境危害的受试者)和/或表现出疑似癌症临床症状的受试者(甲状腺中结节)。另外或者,需要其的受试者可以为经历常规健康体检的健康人类受试者。分析恶性细胞或恶变前期细胞的存在可以在体内或体外实现,借此回收生物样本(例如活组织检查)。该活组织样本包含细胞并可以为切开的或切除的活组织。样本可以从受试者的甲状腺回收,可以使用FNA回收。或者细胞可以从完整切除物中回收。当采用本发明的教导时,可以收集与确定治疗方案、疗程和/或疾病严重程度测量相关的其他信息。如本文所使用的,“治疗方案”指指定提供给需要其的受试者(例如,诊断为具有病症的受试者)的治疗类型、剂量、治疗时间表和/或治疗持续时长的治疗计划。选定的治疗方案可以为预期获得最好临床效果(例如,病状完全治愈)的积极治疗,或可以缓解病理学症状的更稳健治疗,其导致病症的不完全治愈。应理解的是,在某些情况下,治疗方案可以与受试者的某些不适或不利副作用(例如破坏健康细胞或组织)有关。治疗的类型可以包括手术治疗(例如去除病变、患病细胞、组织或器官)、细胞替代疗法、以局部或全身方式施用治疗药物(例如受体激动剂、拮抗剂、激素、化疗药物)、使用外源(例如,外粒子束)和/或内源(例如,近距离放射疗法)暴露于放射线的疗法和/或其任意组合。取决于病症的严重程度和治疗的选择类型,剂量、治疗时间表和治疗持续时长可以不同,本领域技术人员能够通过治疗剂量、治疗时间表和病症持续时长来调整治疗类型。还提供诊断方法。该方法包括检测生物样本中与具体癌症相关的核酸表达水平。患者具体癌症状态的诊断可以考虑到预后和治疗策略的选择。此外,通过确定临时表达的与具体癌症相关的核酸,可分类细胞发育阶段。可以实施标记探针与组织切片或FNA涂片的原位杂交。通过对比单个样本之间的指纹,本领域技术人员可以基于该发现作出诊断、预后、或预测。还应理解的是,指示诊断的核酸序列可以不同于指示预后的核酸序列,细胞状况的分子谱可以导致应答条件或受阻条件之间的差别,或可以预测结果。还提供试剂盒,该试剂盒可以包括本文描述的核酸以及任何或全部的以下物质:分析试剂、缓冲剂、探针和/或引物、和灭菌盐水或另一个药学上可接受的乳化基料和悬浮基料。另外,试剂盒可以包括含有用于本文所述方法实践的指示(例如方案)的指导材料。试剂盒可还以包含用于表达谱数据分析的软件包。例如,试剂盒可以为用于靶核酸序列的扩增、检测、鉴别或定量的试剂盒。该试剂盒可以包含poly(T)引物、正向引物、反向引物和探针。任意本文所述的组合物可以包含于试剂盒中。在一个非限制性实例中,用于分离微RNA的试剂、标记微RNA的试剂、和/或使用阵列评估微RNA群组的试剂包含于试剂盒中。试剂盒还可以包括用于制备或合成微RNA探针的试剂。因此在适当容器中,试剂盒可以包括通过结合标记核酸或未标记核酸用于标记微RNA的酶,该未标记核酸随后会被标记。试剂盒还可以包括一种或更多种缓冲剂,例如反应缓冲剂、标记缓冲剂、洗涤缓冲剂或杂交缓冲剂、用于制备微RNA探针的化合物、用于原位杂交的成分和用于分离微RNA的成分。本发明的其他试剂盒可以包括制备包含微RNA的核酸阵列的成分,从而可以包括例如固体支撑物。提供以下实施例以更充分地说明本发明的某些实施方式。然而,其不应以任何方式解释为限制本发明的广义范围。实施例材料和方法1.微RNA分析可以使用本领域已知方法来评估甲状腺肿瘤样本中微RNA的存在和/或水平,该方法例如Northern印迹法、RNA表达分析如微阵列分析、RT-PCR、高通量测序(下一代测序)、克隆和定量实时聚合酶链反应(qRT-PCR)。确定RNA表达的分析技术是本领域已知的,参见例如Sambrook等人,MolecularCloning:ALaboratoryManual,第三版,ColdSpringHarborPress,ColdSpringHarbor,N.Y.(2001)。以下更详细地描述本文所使用具体方法的实施例。2.RNA提取物FNA细胞阻断样本从7个至10个10μm厚的组织切片中分离总RNA。在二甲苯中、在57℃下将切片保温几次(1至3次)持续5分钟以去除多余的石蜡,然后在环境温度下以10000g离心2分钟。然后用1ml100%的乙醇洗涤样本若干次(约3次)以洗去组织中的二甲苯,然后在环境温度下以10000g离心10分钟。舍弃上清液,在65℃下干燥组织5分钟。通过500μl的缓冲剂B(10mM的NaCl、pH7.5的500mMTris、pH8的20mMEDTA、1%的SDS)中的蛋白酶K溶液(5至12μl的蛋白酶K(例如Sigma或ABI),在45℃下使蛋白质降解几小时(约16小时)。通过在95℃下保温7分钟使蛋白酶K失活。在试管冷却之后,添加10μl的RNA合成内标物(例如,0.15飞摩尔/μl的2个内标物)。使用等体积的酸化酚-氯仿提取RNA,涡旋,然后在4℃下以12000g离心15分钟。然后使用8μl的线性丙烯酰胺、0.1体积的pH5.2的3MNaOAc、和3体积的100%无水乙醇沉淀RNA30分钟至16小时,然后在4℃下以20000g(14000rpm)离心至少40分钟。将小球通过添加1ml的85%冷乙醇洗涤。在37℃下引入DNA酶60分钟以酶解DNA(例如,10μl的TurboTMDNA酶),然后如上所述地使用酸化酚-氯仿提取并用乙醇沉淀。FNA涂片样本(示例)通过在环境温度下在二甲苯中浸渍载玻片若干小时(约2至20小时,通常约16小时)以去除过量石蜡或胶,去除盖玻片(当存在时)之后,从非染色的或染色的(例如,通过Papanicolaou、Giemsa或Diff-Quick)载玻片上的FNA涂片样本分离总RNA。然后用100%的乙醇洗涤载玻片若干次(约3次)以洗掉二甲苯。在双蒸水(DDW)中浸渍载玻片1分钟。使用解剖刀将细胞从载玻片刮除。然后将载玻片用500μl的缓冲液B(10mM的NaCl、pH7.5的500mMTris、pH8的20mM的EDTA、1%的SDS)洗涤,转移至1.7ml试管。在45℃下,通过蛋白酶K(例如,5μl至12μl的Sigma或ABI)降解蛋白质几小时(约16小时)。通过在95℃下保温试管7分钟使蛋白酶K失活。在试管冷却之后,添加10μl的RNA合成内标物(例如,0.15飞摩尔/微升的2个内标物)。使用等体积的酸化酚-氯仿提取RNA,涡旋,然后在4℃下以12000g离心15分钟。然后使用8μl的线性丙烯酰胺、0.1体积的pH5.2的3MNaOAc、和3体积的无水乙醇沉淀RNA30分钟至16小时。然后将试管在4℃下以20000g(14000rpm)离心至少40分钟。将小球用约1ml的85%冷乙醇洗涤。在37℃下引入DNA酶60分钟以酶解DNA(例如,10μl的TurboTMDNA酶,Ambion,LifeTechnologies),然后如上所述地使用酸化酚-氯仿提取并用乙醇沉淀。3.总RNA定量在NanoDrop3300(ND3300)荧光光谱仪中使用染料(ThermoFisherWilmington,DE),通过荧光光谱进行总RNA定量。当使用高浓度的染料(1:200稀释)时,ND3300RNA检测范围为25ng/ml至1000ng/ml,当使用1:2000稀释的染料时,ND3300RNA检测范围为5ng/ml至50ng/ml。ND3300确定的RNA量与检测表达的微RNA高度相关。4.微阵列中的微RNA谱化通过将DNA寡核苷酸探针打印至以下物质以生成常规微阵列(AgilentTechnologies,SantaClara,CA):2172个miR序列、17个阴性对照、23个内标物和10个阳性对照(总共2222个探针)。将每个微RNA探针打印一式三份,运送至位于微RNA补充序列的3’末端的28个核苷酸(nt)接头。将阴性内标物和阳性探针打印3次至200次。使用不匹配基因组的序列设计17个阴性对照探针。设计两组阳性对照探针以与微RNA阵列杂交:(i)在标记之前将合成小RNA内标到RNA以确认标记有效性;(ii)在阵列上印迹用于丰富的小RNA的探针以确认RNA品质,该小RNA如小细胞核RNA(U43、U24、Z30、U6、U48、U44)、5.8s和5s的核糖体RNA中。5.用于微阵列的微RNA的Cy染料标记用Cy3或Cy5,通过将RNA接头、p-rCrU-Cy/染料或若干连续Cy(BioSpringGmbH,IBAGmbH或等同物)连接至3’末端来标记总RNA(20ng至1000ng)(Thomson等人,NatureMethods2004;1:47-53)。标记反应包含总RNA、内标物(0.1至100飞摩尔)、250ng至400ng的RNA-接头-染料、15%的DMSO、1x连接酶缓冲剂和20单位的T4RNA连接酶(NEB或等同物),在4℃下进行1小时,然后在37℃下进行1小时,然后在4℃最高进行40分钟。标记的RNA与30μl的杂交混合物(45μL的10XGEAgilent阻断剂和246μL的2XHi-RPM杂合体的混合物)混合。将标记混合物在100℃下保温5分钟,然后在水浴中冰孵5分钟。在54℃至55℃将载玻片杂交16小时至20小时,然后洗涤两次。第一次洗涤用AgilentGE洗涤缓冲液1(例如,6XSSPE+0.005%的N-月桂酰肌氨酸+0.005%的TritonX-102)在室温下进行5分钟,然后在37℃下用AgilentGE洗涤缓冲液2(例如,0.06XSSPE+0.005%N-月桂酰肌氨酸+0.005%的TritonX-102)进行第二洗涤5分钟。使用微阵列扫描器(Agilent微arrayScannerBundleG2565BA,在100%的XDRHi、10%的XDRLo下分辨率为5μm)扫描阵列。使用适当的软件(FeatureExtraction10.7软件,Agilent)分析阵列图像。6.RT-PCR在1ng至500ng的总RNA上实施聚腺苷酸化和逆转录。在37℃下,在poly(A)聚合酶(poly(A)聚合酶NEB-M0276L)、ATP、具有一致序列的寡T引物和逆转录酶(IIRT,Invitrogen,Carlsbad,CA)的存在下将RNA保温1小时。然后,将cDNA通过RT-PCR扩增。扩增反应包括微RNA特异性正向引物,以及与寡T尾的一致3’序列互补的通用反向引物,正向引物为与特异的微RNA序列的3’互补的(MGB)探针或为与poly(A)衔接子序列部分互补的探针。RT-PCR方法的详细说明可见于出版物WO2008/029295中,其内容通过引用并入本文。确定每个微RNA的循环阈值(CT,在其探针信号达到阈值的PCR循环)。为了能够比较来自RT-PCR的微RNA表达结果和来自微阵列的微RNA表达结果,用50中减去通过RT-PCR获得的每个值(50-CT)。每个患者的每个微RNA的50-CT表达与由微阵列方法获得的信号进行比较。7.阵列数据归一化原始数据集由每个样品的多探针测量信号组成。为进行分析,仅使用设计为测量已知表达水平或经确认的人类微RNA的探针信号。通过对可靠点取对数的方式将一式三份的点组合到一个信号中。所有数据为对数变换的,分析在对数空间进行。通过在两个代表性样本中对每个探针取平均表达水平,计算用于归一化的参照数据向量R,两个代表性样本中每个肿瘤类型中取一个。对于具有数据向量Sk的每个样本k,发现二次多项式Fk以提供在样本数据和参照数据之间的最佳拟合,以使R≈Fk(Sk)。不使用远程数据点(“离群值”)拟合多项式F。对于样本中的每个探针(向量Sk中的元素),由初始值通过用多项式函数Fk将其转换以计算归一化值(在对数空间)以使在对数空间进行统计学分析。为表现和计算倍数变化,通过取指数将数据转换回线性空间。8.miRNA-seq序列库构建取决于所使用的高通量测序平台,可使用多种不同试剂盒进行序列库构建。然而,存在用于准备小RNA测序的若干常规步骤。连接步骤将DNA衔接子添加到小RNA的两端,其在逆转录和PCR扩增期间充当引物结合位点。使用连接酶如T4RNA连接酶,或使用5’RACE反应2添加5’衔接子,将5’衔接子之后的转换成腺苷酸的单链DNA3’衔接子与小RNA连接。衔接子还被设计为捕获具有5’磷酸基团的小RNA,典型的微RNA,而不捕获具有5’羟基的RNA降解产物。逆转录和PCR扩增步骤将连接RNA的小衔接子转换为在测序反应中使用的cDNA克隆产物。然后进行PCR以扩增cDNA序列池。在该步骤中还可以使用设计为具有独特核苷酸标签的引物以生成在合并库多重测序中的ID标签。9.下一代测序(NGS)来自每个FFPE样本的500ngRNA用于小RNA深度测序(miRSeq)。将库负载于序列分析仪(HiSeqTM2000DNA)的两条路径上。每个库平均获得约63000000个测序片段。为发现新微RNA,对原始测序数据(整理引物-衔接子序列)应用序列分析软件(miRDeep2,FriedlanderMR等人,NucleicAcidsRes.2012Jan;40(1):37-52)。10.统计分析对对数转换的归一化荧光信号使用双侧(不成对的)Student’st测试以计算P值。通过设置0.05至0.1的错误发现率(FDR)确定显著差别的阈值,从而校正多重假设测试的结果,获得在0.01至0.06的p值截止值。对于每个差异化表达的微RNA,计算应答运行特性(ROC)曲线的倍数差异(归一化荧光中值的比例)和曲线下面积(AUC)。从统计学分析中排除三组miR:(a)先前发现在血液样本中高表达的miR(由于在FNA样本中血液的高百分比),(b)表达水平与RNA的下降量无关的miR,即这些miR没有显示与测量的RNA量下降有关信号线性下降,(c)表达水平与(b)组中的miR相关的miR。实施例1:预操作的样本中微RNA的检测在几个来自体外FNA活检样本的Papanicolaou、Giemsa和Diff-Quick染色的涂片中,进行微RNA谱的初步研究以确保方法的可行性。由于FNA涂片通常具有非常少的细胞,提供极小量的RNA用于研究,例如1ng至1000ng,首先需要评价在如此低的RNA量下,微RNA是否可检测。因此,在Giemsa染色的乳头状癌涂片和非乳头状癌涂片中,测量约2200个单独微RNA的微RNA表达水平。在乳头状癌涂片中,发现先前显示与乳头状癌相关的五种微RNA(hsa-miR-146b-5p、hsa-miR-31-5p、hsa-miR-222-3p、hsa-miR-221-3p和hsa-miR-21-5p)过度表达。图1显示Giemsa染色的乳头状癌样本和非乳头状癌样本之间微RNA表达的对比,揭示在乳头状癌中高度上调的微RNA标志。这些结果强烈表明了在FNA涂片中可以成功确定微RNA谱。实施例2:恶性甲状腺病变和良性甲状腺病变之间的差异化微RNA表达在本实验分析中使用的样本群组由73个预操作的甲状腺FNA细胞阻断物组成,该甲状腺FNA细胞阻断物选自DepartmentofPathologyTempleUniversityHospital(Philadelphia,USA)的归档材料。该73个样本包括35个良性甲状腺病变样本和38个恶性甲状腺病变样本。该35个良性肿瘤由18个滤泡性腺瘤样本、8个Hashimoto甲状腺炎样本和9个增生(甲状腺肿大)样本组成。该38个恶性肿瘤由10个滤泡性癌和28个乳头状癌组成。28个乳头状癌样本中,9个为乳头状癌,13个为包膜内型的乳头状癌滤泡性变体,6个为非包膜内型的乳头状癌滤泡性变体。组织学诊断最终评估了甲状腺病变的恶性或良性。细胞学分类是基于“TheBethesdaSystemforReportingThyroidCytopathology”(Syed,Z.Ali和EdmundS.Cibas著;DOI10.1007/978-0-387-87666-5_1;SpringerScience+BusinessMedia,LLC2010)。通过InstitutionalReviewBoard(IRB,等同于EthicalReviewBoard)的参与机构批准该研究方案。肿瘤分类是基于WorldHealthOrganization(WHO)指南。其他的群组由在甲状腺切除术后制备的13个甲状腺体外FNA涂片组成,从米兰-比可卡大学(米兰,意大利)获得。从这些样本中提取总RNA(至少10ng),使用含有约2200个miR的常规微阵列谱化微RNA的表达。结果显示若干miR的良性和恶性病变之间表达模式的显著差异(恶性病变对比良性病变的上调或下调),在表3中列出。表3:恶性甲状腺肿瘤中的上调或下调miRNA对比良性甲状腺肿瘤中的上调或下调miRNA使用双侧(不成对的)Student’st测试计算p值。倍数变化表示每组中值的比例。AUC:当使用miRNA分类两组时曲线以下面积。中值:表达值的中值(大约)。基于在35个良性FNA样本和38个恶性样本中的miRNA表达,开发用于区分恶性甲状腺肿瘤和良性甲状腺肿瘤的分类算法。基于发现在这些情况中差异化表达的8个miR(hsa-miR-125b-5p、hsa-miR-21-5p、hsa-miR-222-3p、hsa-miR-221-3p、hsa-miR-146b-5p、hsa-miR-181a-5p、hsa-miR-138-5p和MID-23794),将逻辑回归分类器训练为区分恶性甲状腺病变和良性甲状腺病变,该情况为或在良性或恶性之间或在具体甲状腺肿瘤亚型之间(数据未显示)。该分类器对于鉴别恶性肿瘤达到89%的准确率,87%的敏感性,91%的特异性。hsa-miR-125b-5p、hsa-miR-21-5p、hsa-miR-222-3p、hsa-miR-221-3p、hsa-miR-146b-5p和hsa-miR-181a-5p在恶性病变中表现出更高表达,hsa-miR-138-5p和MID-23794在良性病变中表现出更高表达(数据未显示)。实施例3:区分恶性甲状腺病变和良性甲状腺病变的不同亚型在18个滤泡性腺瘤样本和10个滤泡性癌样本之间比较miR的表达水平。表4中示出滤泡性腺瘤对比滤泡性癌中的上调或下调微RNA。表4:滤泡性腺瘤中上调或下调miRNA对比滤泡性癌中上调或下调miRNA使用双侧(不成对的)Student’st测试计算p值。倍数变化表示每组中值的比例。AUC:当使用miRNA分类两组时曲线以下的面积。中值:表达值的中值(大约)。比较18个滤泡性腺瘤样本和9个乳头状癌(非滤泡性变体)样本的miR表达水平,使用hsa-miR-146b-5p和hsa-miR-21-5p的表达水平产生具有100%准确率(数据未显示)的分类器用于区分滤泡性腺瘤样本和乳头状癌样本。比较18个滤泡性腺瘤样本相对于19个乳头状癌的滤泡性变体样本的miR表达水平。表5中示出在乳头状癌的滤泡性变体中对比滤泡性腺瘤的上调或下调的微RNA。表5:乳头状癌滤泡性变体(FVPC)对比滤泡性腺瘤(FA)的上调或下调miRNA。使用双侧(不成对的)Student’st测试计算p值。倍数变化表示每组中值的比例。AUC:当使用miRNA分类两组时曲线以下面积。中值:表达值的中值(大约)比较6个非包膜内型的乳头状癌的滤泡性变体样本和35个良性样本的miR表达水平,使用hsa-miR-221-3p和hsa-miR-200b-3p的表达水平产生具有98%准确率、83%敏感性和100%特异性的分类器(数据未显示)。在8个Hashimoto甲状腺炎样本和9个(非滤泡性)乳头状癌样本之间比较miR的表达水平。表6中示出在乳头状癌中相对于Hashimoto甲状腺炎的上调或下调的微RNA。用于比较这两种甲状腺病变的谱特征的miR的最佳候选为hsa-miR-146b-5p、hsa-miR-200a-3p和MID-23794。表6:乳头状癌(PC)对比Hashimoto甲状腺炎(Ht)的上调或下调miRNA。使用双侧(不成对的)Student’st测试计算p值。倍数变化表示每组中值的比例。AUC:当使用miRNA分类两组时曲线以下面积。中值:表达值的中值(大约)实施例4:通过深度测序鉴别新型微RNA生物标志从拉宾医疗中心病理部(DepartmentofPathologyatRabinMedicalCenter)获得来自滤泡性病变的11个FFPE(福尔马林固定,嵌入到石蜡)甲状腺切除样本(从手术活检中获得,在福尔马林中固定并在石蜡中保藏)。该样本包括6个滤泡性腺瘤和5个滤泡性癌。在所有样本中肿瘤细胞含量高于50%。使用序列分析软件发现总共386个新型候选微RNA,其中27个通过qPCR选择为有效的。本文公开两种新型微RNA,MD2-495和MD2-437,其序列提供于表1中,其各自的发夹结构显示于表2中。图2A显示两种新型微RNA的二级结构,其由序列分析软件预测。图2B显示在11个样本中的每个中两种新型微RNA的表达(测序片段的归一化个数)。右侧的颜色编码栏表示表达尺度。实施例5:特异性微RNA在良性甲状腺病变和恶性甲状腺病变中差异化表达从以色列的医疗中心获得染色的甲状腺FNA涂片(群组I);从美国的医疗中心获得甲状腺FNA细胞阻断物(群组II)。对于两个群组,基于被切除肿瘤的组织学诊断,将甲状腺病变最终分类为恶性或良性。表7中显示两个群组中样本的分类总结。表7:FNA样本–群组I和群组IIFNA样本描述群组I群组II病变数(#患者)181(65)73(73)结节性增生(结节性甲状腺肿)139滤泡性腺瘤2718格雷夫斯病30Hashimoto甲状腺炎38良性结节总数4635乳头状癌109乳头状癌的滤泡性变体1319滤泡性癌410髓样癌60甲状腺癌(混合组织学)20恶性结节总数3538Bethesda2类别II、类别VI330Bethesda2类别III、类别IV、类别V48731某些患者患有超过一种病变。2BethesdaSystemforReportingThyroidCytopathology(BSRTC)来自2007年在国立卫生研究院举办的会议(CibasES,AliSZ.TheBethesdaSystemforReportingThyroidCytopathology.AmJClinPathol2009;132:658-65)。该系统基于六种诊断类别获得标准化FNA报告:DCI=非诊断的,DCII=良性,DCIII=非典型/未定类显著性的滤泡性病变(AUS/FLUS),DCIV=滤泡性肿瘤/疑似滤泡性肿瘤(FN/SFN),DCV=疑似恶性和DCVI=恶性。使用上述的内部开发方案从样本中提取含有微RNA片段的高度纯化的RNA。通过常规打印微阵列测量超过2000个微RNA谱化FFPE样本和细胞学(FNA)样本以鉴别差异化表达的微RNA并开发分类器。通过qPCR用常规打印微阵列测量超过2000个微RNA和96个微RNA谱化超过150个甲状腺FNA样本(表7)。图3A(群组I)和3B(群组II)显示在患有恶性结节(y轴)的患者中和在患有良性结节(x轴)的患者中微阵列上微RNA表达水平的中值。对于每个微RNA,两组中的该值通过FDR=0.1的Mann-Whitney测试比较。发现在良性赘生物和恶性赘生物之间微RNA的差异化表达。基于两种微RNA:hsa-miR-146b-5p和hsa-miR-375,恶性涂片相对于良性涂片的分类获得超过85%的准确率(基于10个10倍交叉验证运行的中值,数据未显示)。实施例6:hsa-miR-375为FNA样本中髓样甲状腺癌的显著性标志比较髓样甲状腺癌样本(n=6)和来自其他甲状腺结节的样本(n=75)之间的FNA群组I中hsa-miR-375(SEQIDNO.8)的表达水平。结果示于图4中。因此,hsa-miR-375为FNA样本中髓样甲状腺癌的显著性标志。实施例7:染色甲状腺涂片可以用于微RNA谱化在用不同染料染色的样本中比较微RNA的表达水平以评估微RNA的稳定性和一经染色的微RNA检测水平的再现性。将来自FNA群组I中的总共143个涂片进行如下染色:60个用May-GrünwaldGiemsa染色,64个用DiffQuik染色和19个用Papanicolaou染色。用不同染料染色的相同样本副本中的微RNA表达水平显示出显著的相关性(超出预期)。在图5A-5B中进一步证明了不同染料之间miR-146b-5p表达水平的相似性,其显示hsa-miR-146b-5p(SEQIDNO.10至SEQIDNO.11)的归一化表达水平在用不同染料染色相同样本时是相似的,可以参见52个May-GrünwaldGiemsa-DiffQuik对(图5A)和15个DiffQuik-Papanicolaou对(图5B)。因此,在临床设置中使用的不同细胞学染料(Papanicolaou、May-GrünwaldGiemsa和DiffQuik)不影响微RNA表达的检测和定量。实施例8:甲状腺分类——测试开发总共24个微RNA全部被选为用于确立甲状腺样本为恶性还是良性的状态(表12)。用以上描述的RT-PCR测量微RNA表达。表8提供miR和其各自正向引物的列表。使用以下提供的polyT衔接子实现第一链产生。正向引物是序列特异性的,但是反向引物是通用的。使用针对hsa-miR-31-5p(SEQIDNO.5至SEQIDNO.7)、hsa-miR-5701(SEQIDNO.35)、hsa-miR-424-3p(SEQIDNO.16)、MID-50971(SEQIDNO.34)、MID-20094(SEQIDNO.27至SEQIDNO.28)、MID-50976(SEQIDNO.33)、hsa-miR-3074-5p(SEQIDNO.32)、hsa-miR-222-3p(SEQIDNO.1至SEQIDNO.2)、MID-50969(SEQIDNO.29)、hsa-miR-146b-5p(SEQIDNO.10至SEQIDNO.11)、hsa-miR-346(SEQIDNO.14)、MID-16582(SEQIDNO.25)的miR的通用MGB探针实现RT-PCR产物的检测,或使用表9中提供的miR特异性探针。以下提供了反向引物、polyT衔接子和MGB探针的序列:-反向引物GCGAGCACAGAATTAATACGAC(SEQIDNO.309);-polyT衔接子GCGAGCACAGAATTAATACGACTCACTATCGGTTTTTTTTTTTTVN(SEQIDNO.310),其中“V”可以是A、G或C中的任一个;“N”可以是G、C、A或U/T的任一个;-通用MGB探针AAAACCGATAGTGAGTCG(SEQIDNO.311).表8:测试开发——微RNA和正向引物表9:测试开发——微RNA特异性探针微RNA特异性探针序列SEQIDNO.hsa-miR-342-3pCCGTTTTTTTTTTTTACGGGTGC336hsa-miR-181c-5pCCGTTTTTTTTTTTTACTCACCG337hsa-miR-125b-5pCCGTTTTTTTTTTTTCACAAGTT338hsa-miR-375CCGTTTTTTTTTTTTCACGCGAG339hsa-miR-486-5pCCGTTTTTTTTTTTTCTCGGGGC340hsa-miR-551b-3pCCGTTTTTTTTTTTTCTGAAACC341hsa-miR-23a-3pCCGTTTTTTTTTTTTGGAAATCC342hsa-miR-574-3pCCGTTTTTTTTTTTTGTGGGTGT343hsa-miR-152-3pCGTTTTTTTTTTTTCCAAGTTC344hsa-miR-200c-3pCGTTTTTTTTTTTTCCATCATT345hsa-miR-138-5pCGTTTTTTTTTTTTCGGCCTGA346hsa-miR-345-5pCGTTTTTTTTTTTTGAGCCCTG347表11:在甲状腺测试中微RNA标志基于其在发明人实施的若干初步研究中的表达模式(数据未显示)选择标志微RNA,并提供将其分类为“恶性”、“细胞类型”或用作正规化子的理由。当与这些微RNA在良性样本中的表达的对比时,基于其在恶性样本中的表达水平,确立“恶性标志”hsa-miR-222-3p、hsa-miR-551b-3p、hsa-miR-31-5p、hsa-miR-375、hsa-miR-125b-5p、hsa-miR-152-3p、hsa-miR-346、hsa-miR-181c-5p、hsa-miR-424-3p和hsa-miR-146b-5p。根据以下示范的其模式或表达,发明人选择“细胞类型”标志hsa-miR-486-5p、hsa-miR-342-3p、hsa-miR-138-5p、hsa-miR-200c-3p和MID-16582。在与甲状腺上皮细胞相关的全部血液中发现hsa-miR-486-5p(SEQIDNO.22)。与其他微RNA一起(数据未显示),发现其与甲状腺FNA样本中的血液量有关。因此,hsa-miR-486-5p(SEQIDNO.22)为全部血液标志的一个实例。检测到若干微RNA与miR-486-5p高度相关(>0.85),也可以视为血液标志,包括hsa-miR-320a、hsa-miR-106a-5p、hsa-miR-93-5p、hsa-miR-17-3p、hsa-let-7d-5p、hsa-miR-107、hsa-miR-103a-3p、hsa-miR-17-5p、hsa-miR-191-5p、hsa-miR-25-3p、hsa-miR-106b-5p、hsa-miR-20a-5p、hsa-miR-18a-5p、hsa-miR-144-3p、hsa-miR-140-3p、hsa-miR-15b-5p、hsa-miR-16-5p、hsa-miR-92a-3p、hsa-miR-484、hsa-miR-151a-5p、hsa-let-7f-5p、hsa-let-7a-5p、hsa-let-7c-5p、hsa-let-7b-5p、hsa-let-7g-5p、hsa-let-7i-5p、hsa-miR-185-5p、hsa-miR-30d-5p、hsa-miR-30b-5p、hsa-miR-30c-5p、hsa-miR-19b-3p、hsa-miR-26a-5p、hsa-miR-26b-5p、hsa-miR-425-5p、MID-19433和hsa-miR-4306。一旦测量血液腔中的微RNA表达谱,发明人就观察到在不同的血细胞类型中发现大量微RNA提高(数据未显示)。因此,hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)为微RNA之一,尤其是,其在白血球中富集,因此可视为白血球标志的实例。令人关注的是,显示hsa-miR-342-3p的表达与hsa-miR-150-5p相关,这说明hsa-miR-150-5p也是白血球标志。另外,在白血球中还显示了hsa-miR-146a-5p的表达(数据未显示)。发现hsa-miR-200c-3p(SEQIDNO.23至SEQIDNO.24)和hsa-miR-138-5p(SEQIDNO.19至SEQIDNO.21)在上皮细胞中富集。在初步试验中,用无甲状腺组织材料的血液制备涂片,并与来自甲状腺组织的涂片比较。发现hsa-miR-200c-3p(SEQIDNO.23至SEQIDNO.24)和hsa-miR-138-5p(SEQIDNO.19至SEQIDNO.21)二者在甲状腺涂片(良性和恶性二者)中相比血液涂片以高得多的水平表达(数据未显示)。还发现其他微RNA在上皮细胞中富集(数据未显示)。因此,发现hsa-miR-200c-3p(SEQIDNO.23至SEQIDNO.24)和hsa-miR-138-5p(SEQIDNO.19至SEQIDNO.21)为上皮细胞标志的实例。令人关注的是,发明人发现hsa-miR-138-5p的表达与上皮细胞的存在相关,在数据的某些子集中发现hsa-miR-138-5p在良性样本中是上调的(数据未显示)。发现MID-16582(SEQIDNO.25)在Hurthle细胞中以更高水平表达。在初步研究中,发明人出人意料地发现了该微RNA在存在Hurthle细胞的滤泡性腺瘤中相对在不显示具有Hurthle细胞的滤泡性腺瘤中是上调的(图6A至6B)。该结果可以有助于在Hurthle细胞中发现的线粒体富集。本发明人发现了在Hurthle细胞中发现的MID-16582的序列(SEQIDNO.25)以及其他核酸序列可图谱化为线粒体DNA(数据未显示)。因此,MID-16582为Hurthle细胞标志的实例。测试开发训练集包括约360个不同样品。样品中的大多数为染色的FNA涂片(Papanicolaou、May-GrünwaldGiemsa或Diff-Quik)。45个FNA样本在细胞块中。从以色列、欧洲和美国的医疗中心收集样本。样本中的大多数为“未定类”FNA(根据Bethesda分类法,71个类别III、113个类别IV和74个类别V),其余为“定类”(38个类别II、60个类别VI)。训练集由恶性(n=197)和良性(n=155)甲状腺结节组成,并含有甲状腺结节的八个主要组织学亚型代表。来自甲状腺结节的33个样本尺寸小于1cm。最小的结节尺寸为0.1cm。从大多数测试中排除髓样癌样本,除非有说明之处。表10提供每类样本的分布。表10:训练研究群组组成和Bethesda分布组织学类型No.乳头状癌84乳头状癌,滤泡性变体77滤泡性癌16非特异性癌6髓样的14结节性增生65滤泡性腺瘤81Hashimoto6Graves3恶性总数197良性总数155无结果4BethesdaNo.I0II38III71IV113V74VI60未知98定类总数258未定类总数84将来自常规制备FNA涂片的样本和细胞块用于总RNA提取和RT-PCR扩增。将15个标志微RNA群体和作为正规化子的9个微RNA测试所有样本(表11)。图7中显示在样本子集(n=353)中的训练结果。发现在阈值以上的微RNA表达与恶性样本相关,该微RNA为hsa-miR-222-3p(SEQIDNO.1至SEQIDNO.2)、hsa-miR-551b-3p(SEQIDNO.3至SEQIDNO.4)、hsa-miR-31-5p(SEQIDNO.5至SEQIDNO.7)、hsa-miR-125b-5p(SEQIDNO.9)、hsa-miR-146b-5p(SEQIDNO.10至SEQIDNO.11)、hsa-miR-346(SEQIDNO.14)、hsa-miR-181c-5p(SEQIDNO.15)和hsa-miR-375(SEQIDNO.8)。通过以下公式获得图7中显示的表达水平:[50–每个标志的归一化Ct]。通过减去正规化子的平均信号实现归一化。将全部样本使用的正规化子的平均信号值加上所有检测的表达值,以使值进入更容易计算的范围中。令人关注的是,hsa-miR-125a-5p的表达水平和hsa-miR-125b-5p的表达水平相关。实施例9:甲状腺测试分类器的建立使用四个算法以建立甲状腺测试、判别式分析、K最近邻算法(KNN)、支持向量机(SUV)、判别分析分类器集合(判别式分析集合)中使用的最佳分类器。用以下参数确立先验值:-先验值:对于所有使用的算法,对于恶性样本设置先验值为70%,对于良性样本设置先验值为30%。-样本集合:在该实施例中,分析三个样本设置。一个样本设置包括恶性样本(n=183)和良性样本(n=155),排除恶性髓样样本;在下文和附图中称为“恶性+良性”。另一样品设置包括所有“未定类”样本,其包括所有分类为BethesdaIII、IV和V的样本,在下文和附图中称为“未定类”。第三样本设置包括仅分类为BethesdaIV的样本,在下文和附图中称为“Bethesda”。分类为BethesdaIV的甲状腺病变样本通常难以通过细胞学参数分类。因此,建立基于该样本子集的分类器是重要的。另外,存在技术问题的特定样本(例如,BethesdaII的恶性样本;取自淋巴结的样本)由于各种原因被排除。-将髓样样本从分类中排除。因此,在该实施例中,当提及恶性样本其指非髓样恶性。-微RNA表达水平归一化:使用所谓的正规化子微RNA[hsa-miR-23a-3p、MID-20094、MID-50969、hsa-miR-345-5p、hsa-miR-3074-5p、MID-50976、MID-50971、hsa-miR-5701和hsa-miR-574-3p]将微RNA表达水平归一化,用50减去该值,以使得较低CT与较高表达值相关。-微RNA比例:从微RNA对获得比例以试图从分类器中除去某些因素。因此,例如hsa-miR-31-5p:hsa-miR-342-3p的比例使得白血球在hsa-miR-31-5p(分子)表达中的贡献减少(经由hsa-miR-342-3p的表达,分母)。由于CT为对数尺度,通过从其他中减去一个miR表达产生比例。通过加上常数使每个比例进一步归一化,以使得比例与微RNA归一化值在相同范围内。实施例9.1:判别式分析分类器当使用判别式分析作为算法时,在上述三个样品集合中应用判别式分析的线性判别式类型(使用微RNA表达水平(图8A至8C、图23A至23C和图37A至37C)、微RNA比例(图9A至9C、图24A至24C和图38A至38C)、或微RNA表达水平和微RNA比例的组合(图10A至10C、图25A至25C和图39A至39C)的不同组合作为特征)。如上所述,使用该算法运行三个样品集合。图8A至8C、图9A至9C和图10A至10C提供了该算法对于恶性样本+良性样本的结果。图23A至23C、图24A至24C和图25A至25C提供了该算法对于未定类样本的结果。图37A至37C、图38A至38C和图39A至39C提供了该算法对于BethesdaIV样本的结果。实施例9.2:KNN分类器使用KNN(K最邻近算法)作为算法进行一种分析,其中使用k=5的Pearson相关系数的距离量度。使用微RNA表达水平(图11A至11C、图26A至26C和图40A至40C)、微RNA比例(图12A至12B、图27A至27B和图41A至41B)、或者微RNA表达水平和微RNA比例的组合(图13A至13C、图28A至28C和图42A至42C)的不同组合作为特征,将使用KNN算法的分类器应用于如上所述的三个样品集合(恶性+良性、未定类和BethesdaIV)。如上所述,使用该算法运行三个样品集合。图11A至11C、图12A至12B和图13A至13C提供该算法对于恶性样本+良性样本的结果。图26A至26C、图27A至27B和图28A至28C提供该算法对于未定类样本的结果。图40A至40C、图41A至41C和图42A至42C提供该算法对于BethesdaIV样本的结果。实施例9.3:SVM分类器应用SVM(支持向量机)作为算法进行第三分析,其中使用线性内核。分别使用微RNA表达水平(图14A至14C、图29A至29C和图43A至43C)、微RNA比例(图15A至15C、图30A至30C和图44A至44C)、或者微RNA表达水平和微RNA比例的组合(图16A至16C、图31A至31C和图45A至45C)的不同组合作为特征,将使用SVM算法的分类器应用于如上所述的三个样品集合(恶性+良性、未定类和BethesdaIV)。如上所述,使用该算法运行三个样品集。图14A至14C、图15A至15C和图16A至16C提供了该算法对于恶性样本+良性样本的结果。图29A至29C、图30A至30C和图31A至31C提供了该算法对于未定类样本的结果。图43A至43C、图44A至44C和图45A至45C提供该算法对于BethesdaIV样本的结果。实施例9.4:集成方法分类器使用集成方法作为算法进行第四分析。使用AdaBoost创建最高为100个判别式分析分类器的集合,并将其应用于数据。使用微RNA表达水平(图17A至17C、图32A至32C和图46A至46C)、微RNA比例(图18A至18C、图33A至33C和图47A至47C)、或者微RNA表达水平和微RNA比例的组合(图19A至19C、图34A至34C和图48A至48C)的不同组合作为特征,将使用集成算法的分析应用于如上所述的三个样品集(恶性+良性、未定类和BethesdaIV)。如上所述,使用该算法运行三个样品集。图17A至17C、图18A至18C和图19A至19C提供了该算法对于恶性样本+良性样本的结果。图32A至32C、图33A至33C和图34A至34C提供了该算法对于未定类样本的结果。图46A至46C、图47A至47C和图48A至48C提供了该算法对于BethesdaIV样本的结果。实施例10.用于含有髓质的恶性样本的分类器将实施例9使用的、但包括髓样恶性样本的相同样本集合用于建立分类器。在该样本集合中应用所有分类器,在图51和52中提供了从应用于样本集的判别式分析算法获得的代表集。当使用两个微RNA比例(hsa-miR-125b-5p:hsa-miR-138-5p和hsa-miR-146b-5p:hsa-miR-342-3p)的归一化值作为分类特征时,分类器的敏感性为84.7%,特异性为80.8%。当使用两个微RNA(hsa-miR-222-3p和hsa-miR-551b-3p)的归一化值作为分类特征时,敏感性为85.2%,特异性为53.6%。实施例11:通过细胞特异性标志的表达淘汰样品贯穿本研究的一个重要考虑为结果的准确率,其被提供于已采集FNA样本的患者。实验室倾向于误差以避免提供假阴性结果。另一方面,在FNA样本的分析中,疑似诊断会使患者经历手术,其中25%的案例被发现是不必要的。例如,参考文献中的至少一个报告描述了在9个案例中的7个,具有大量血液或甚至为纯血的甲状腺肿瘤样本被误诊为疑似(Walsh等人.(2012)JClinEndocrinMetab.doi:10.1210/jc.2012-1923)。考虑到该目标,本发明人寻找可以用作细胞类型标志并帮助筛查检测样本品质的微RNA。因此,在训练群组中评价hsa-miR-486-5p(SEQIDNO.22)和hsa-miR-200c-3p(SEQIDNO.23至24)的表达,训练群组包括具有来自良性和恶性(非髓样)甲状腺病变的样本的细胞块,以及仅血液的四个样本(出于该目的制备血液涂片载玻片,如本文所描述的提取RNA)。图53显示了该实验的结果。相比于在训练集中建立的阈值,血液微RNA标志hsa-miR-486-5p非常高,上皮细胞标志hsa-miR-200c-3p非常低。因此使用这些标志滤除血液涂片样本。这种表达模式说明这些样本不具有足够的上皮细胞(由于缺少上皮细胞标志)以继续测试。在测试条件下,该四个血液涂片样本将被淘汰和舍弃。在血液涂片中,相比于阈值,也显示出低的hsa-miR-138-5p(SEQIDNO.19至SEQIDNO.21)的表达(数据未显示)。具有该表达谱的样本适合从甲状腺病变样本分类的方案中被淘汰和/或舍弃。发明人先前建立了与白细胞相关的hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)的表达(数据未显示)。因此,hsa-miR-342-3p相比于阈值的高表达显示缺乏足够的甲状腺细胞,具有该表达谱的样本适合从甲状腺病变样本分类的方案中被淘汰和/或舍弃。同时,hsa-miR-200c-3p的高表达一般为上皮细胞、特别地为甲状腺细胞存在的象征(数据未显示,图53)。因此,hsa-miR-200c-3p高于阈值的表达可以作为样本中甲状腺细胞充足的指标。实施例12:甲状腺肿瘤亚型的分类使用来自Hashimoto(n=6)的样本和来自训练群组的滤泡性腺瘤(FA;n=81)实现良性甲状腺肿瘤亚型的分类。结果示于图54中。hsa-miR-342-3p(SEQIDNO.17至SEQIDNO.18)和hsa-miR-31-5p在Hashimoto样本中的表达比在训练集中建立的阈值高。因此,hsa-miR-342-3p单独或与hsa-miR-31-5p组合的高表达可以用于将样本分类为良性并进一步归类为亚型例如Hashimoto。进一步地,发明人还检测用于亚型良性甲状腺肿瘤的微RNA比例。在该情况下,hsa-miR-125b-5p:hsa-miR-200c-3p的miR比例对于区分滤泡性腺瘤(FA)和Hashimoto样本是重要的(数据未显示)。使用训练群组的样本子集(n=177)实现恶性甲状腺肿瘤亚型的分类。图55提供了分析的一个实例,其中发现在乳头状癌中146b-5p、222-3p、31-5p、125b-5p、551-3p和375高度表达,同时发现MID-16582在滤泡性癌中被高度表达。以下miR对的比例对于分类乳头状癌(PC)样本与滤泡性癌样本是重要的:hsa-miR-146b-5p:hsa-miR-342-3p、hsa-miR-125b-5p:hsa-miR-200c-3p、hsa-miR-222-3p:hsa-miR-486-5p、hsa-miR-31-5p:hsa-miR-342-3p、MID-16582:hsa-miR-200c-3p、MID-16582:hsa-miR-138-5p(数据未显示)。因此,发明人证明了可以使用miR比例特别是分母为细胞标志微RNA如hsa-miR-486-5p、hsa-miR-200c-3p、hsa-miR-138-5p和hsa-miR-342-3p的miR比例,进行恶性甲状腺肿瘤亚型化。实施例13:用于将甲状腺结节分类为恶性或良性的方案图56提供了用于甲状腺结节样本分析的方案的流程图,从FNA样本采集到实验室分析和诊断。从患有甲状腺结节的患者采集FNA样本,并将其进行常规处理。由FNA样本制备涂片。作为第一步,细胞病理学专科医生检查FNA样本并提供分析。在分析为非结论性的情况下,特别是在分类为BethesdaIII、IV或V,即所谓的“未定类”的样本中,将该样品送往RosettaGenomics实验室以进行微RNA谱化和结论性诊断。从经历微RNA谱化的样本中提取总RNA。如以上实施例中所示的,可通过扩增(RT-PCR或NGS)或杂交(微阵列)实施微RNA谱化。该方案可包括以下任意一项:-在分类期间可使用一种或更多种算法,其可以应用于包含单一微RNA表达、微RNA比例或其组合的数据。-其中hsa-miR-375的表达水平高于特定阈值的样本可以被确定为恶性(例如至少10的阈值,或至少18的阈值),例如在图4(由阵列分析的表达)中和图20(由PCR分析的表达)中所证明的。该阈值取决于样本的归一化,以及用于测量微RNA的方法。该阈值也可以为靶敏感性和特异性的函数。-其中hsa-miR-146b-5p的表达水平高于特定阈值的样本会被确定为恶性(例如至少16的阈值),例如在图21、35和49中所证明的。该阈值取决于样本的归一化,以及用于测量微RNA的方法。该阈值也可以为靶敏感性和特异性的函数。-其中进一步归一化的hsa-miR-146b-5p:hsa-miR-342-3p比例高于特定阈值的样本会被确定为恶性(例如至少16的阈值),例如在图22、36和50中所证明的。该阈值取决于样本的归一化,以及用于测量微RNA的方法。-由于不充足的来自肿瘤的材料,正规化子的表达水平可以用作舍弃样本的指标。因此,可以舍弃表现出任意正规化子的低水平、或正规化子表达的最小值、中值或最大值的样本。例如,hsa-miR-23a-3p的低水平(相比于在群组中hsa-miR-23a-3p表达的整体水平)是可能被错误分类的。对应地,hsa-miR-23a-3p的高水平通过提高敏感性和特异性而改善分类(数据未显示)。微RNA表达谱数据的分析使得甲状腺结节诊断为良性或恶性。如图54和图55中所示,包括微RNA表达可以与甲状腺肿瘤亚型相关的结果使得,例如该样本根据其甲状腺肿瘤亚型被进一步分类。前述具体实施例的描述充分揭示了本发明的一般特性,其他人可通过应用现有知识轻而易举地针对不同应用修改和/或调整这些特定实施例,而无需过度实验也无需偏离一般构思,因此,这种调整和修改应该且旨在理解为在实施方式公开的等同方案的含义和范围之内。尽管本发明已经结合其具体实施方式进行描述,许多替代方案、修改方案和变体对本领域技术人员来说是明显的。因此,其旨在包括所有落入随附权利要求的宽范围和精神中的这些替代方案、修改方案和变体。应该理解尽管表明本发明的优选实施方式,详细的说明和具体实施例仅通过举例说明方式给出,因为在本发明的精神和范围内的各种变化和修改通过该详细说明对本领域技术人员会变得显而易见。序列表<110>罗塞塔金诺米克斯有限公司<120>在甲状腺肿瘤分类中的MIRNA表达特征<130>184PCT<150>61/992,756<151>2014-05-13<150>61/992,531<151>2014-05-13<150>62/069,353<151>2014-10-28<150>62/139,066<151>2015-03-27<160>347<170>PatentIn版本3.5<210>1<211>21<212>DNA<213>人<400>1agctacatctggctactgggt21<210>2<211>24<212>DNA<213>人<400>2agctacatctggctactgggtctc24<210>3<211>22<212>DNA<213>人<400>3gacccatacttggtttcagagg22<210>4<211>21<212>DNA<213>人<400>4gcgacccatacttggtttcag21<210>5<211>21<212>DNA<213>人<400>5aggcaagatgctggcatagct21<210>6<211>23<212>DNA<213>人<400>6aggcaagatgctggcatagctgt23<210>7<211>21<212>DNA<213>人<400>7ggcaagatgctggcatagctg21<210>8<211>22<212>DNA<213>人<400>8tttgttcgttcggctcgcgtga22<210>9<211>22<212>DNA<213>人<400>9tccctgagaccctaacttgtga22<210>10<211>22<212>DNA<213>人<400>10tgagaactgaattccataggct22<210>11<211>24<212>DNA<213>人<400>11tgagaactgaattccataggctgt24<210>12<211>21<212>DNA<213>人<400>12tcagtgcatgacagaacttgg21<210>13<211>22<212>DNA<213>人<400>13tcagtgcatgacagaacttggg22<210>14<211>23<212>DNA<213>人<400>14tgtctgcccgcatgcctgcctct23<210>15<211>22<212>DNA<213>人<400>15aacattcaacctgtcggtgagt22<210>16<211>21<212>DNA<213>人<400>16caaaacgtgaggcgctgctat21<210>17<211>23<212>DNA<213>人<400>17tctcacacagaaatcgcacccgt23<210>18<211>24<212>DNA<213>人<400>18tctcacacagaaatcgcacccgtc24<210>19<211>17<212>DNA<213>人<400>19agctggtgttgtgaatc17<210>20<211>23<212>DNA<213>人<400>20agctggtgttgtgaatcaggccg23<210>21<211>24<212>DNA<213>人<400>21agctggtgttgtgaatcaggccgt24<210>22<211>22<212>DNA<213>人<400>22tcctgtactgagctgccccgag22<210>23<211>22<212>DNA<213>人<400>23taatactgccgggtaatgatgg22<210>24<211>23<212>DNA<213>人<400>24taatactgccgggtaatgatgga23<210>25<211>19<212>DNA<213>人<400>25agtgaagcattggactgta19<210>26<211>21<212>DNA<213>人<400>26atcacattgccagggatttcc21<210>27<211>21<212>DNA<213>人<400>27taagccagtttctgtctgata21<210>28<211>25<212>DNA<213>人<400>28tttctaagccagtttctgtctgata25<210>29<211>23<212>DNA<213>人<400>29atgacagattgacatggacaatt23<210>30<211>22<212>DNA<213>人<400>30gctgactcctagtccagggctc22<210>31<211>21<212>DNA<213>人<400>31tgctgactcctagtccagggc21<210>32<211>21<212>DNA<213>人<400>32gttcctgctgaactgagccag21<210>33<211>17<212>DNA<213>人<400>33ctgtctgagcgccgctc17<210>34<211>18<212>DNA<213>人<400>34atactctggtttcttttc18<210>35<211>19<212>DNA<213>人<400>35ttattgtcacgttctgatt19<210>36<211>21<212>DNA<213>人<400>36cacgctcatgcacacacccac21<210>37<211>22<212>DNA<213>人<400>37cacgctcatgcacacacccaca22<210>38<211>23<212>DNA<213>人<400>38tggaagactagtgattttgttgt23<210>39<211>23<212>DNA<213>人<400>39taccctgtagatccgaatttgtg23<210>40<211>23<212>DNA<213>人<400>40taaggtgcatctagtgcagatag23<210>41<211>21<212>DNA<213>人<400>41caacaccagtcgatgggctgt21<210>42<211>22<212>DNA<213>人<400>42tagcttatcagactgatgttga22<210>43<211>22<212>DNA<213>人<400>43tgtaaacatccttgactggaag22<210>44<211>22<212>DNA<213>人<400>44tgctatgccaacatattgccat22<210>45<211>23<212>DNA<213>人<400>45tggcagtgtcttagctggttgtt23<210>46<211>22<212>DNA<213>人<400>46agggacgggacgcggtgcagtg22<210>47<211>23<212>DNA<213>人<400>47tttggcactagcacatttttgct23<210>48<211>22<212>DNA<213>人<400>48aacccgtagatccgaacttgtg22<210>49<211>22<212>DNA<213>人<400>49tcgtaccgtgagtaataatgcg22<210>50<211>22<212>DNA<213>人<400>50gctacttcacaacaccagggcc22<210>51<211>21<212>DNA<213>人<400>51taccacagggtagaaccacgg21<210>52<211>22<212>DNA<213>人<400>52taacactgtctggtaaagatgg22<210>53<211>23<212>DNA<213>人<400>53tgtagtgtttcctactttatgga23<210>54<211>21<212>DNA<213>人<400>54cataaagtagaaagcactact21<210>55<211>22<212>DNA<213>人<400>55tgccctgtggactcagttctgg22<210>56<211>22<212>DNA<213>人<400>56tgagaactgaattccatgggtt22<210>57<211>22<212>DNA<213>人<400>57tcagtgcactacagaactttgt22<210>58<211>22<212>DNA<213>人<400>58ctggtacaggcctgggggacag22<210>59<211>22<212>DNA<213>人<400>59tctcccaacccttgtaccagtg22<210>60<211>23<212>DNA<213>人<400>60ttaatgctaatcgtgataggggt23<210>61<211>23<212>DNA<213>人<400>61aacattcaacgctgtcggtgagt23<210>62<211>23<212>DNA<213>人<400>62aacattcattgctgtcggtgggt23<210>63<211>24<212>DNA<213>人<400>63tttggcaatggtagaactcacact24<210>64<211>22<212>DNA<213>人<400>64tcgtgtcttgtgttgcagccgg22<210>65<211>22<212>DNA<213>人<400>65aactggcctacaaagtcccagt22<210>66<211>21<212>DNA<213>人<400>66tagcagcacagaaatattggc21<210>67<211>23<212>DNA<213>人<400>67cgggtagagagggcagtgggagg23<210>68<211>22<212>DNA<213>人<400>68acagtagtctgcacattggtta22<210>69<211>23<212>DNA<213>人<400>69taacactgtctggtaacgatgtt23<210>70<211>22<212>DNA<213>人<400>70taatactgcctggtaatgatga22<210>71<211>23<212>DNA<213>人<400>71cccagtgttcagactacctgttc23<210>72<211>23<212>DNA<213>人<400>72cccagtgtttagactatctgttc23<210>73<211>22<212>DNA<213>人<400>73tccttcattccaccggagtctg22<210>74<211>22<212>DNA<213>人<400>74ctgtgcgtgtgacagcggctga22<210>75<211>22<212>DNA<213>人<400>75acagcaggcacagacaggcagt22<210>76<211>23<212>DNA<213>人<400>76agctacattgtctgctgggtttc23<210>77<211>22<212>DNA<213>人<400>77acctggcatacaatgtagattt22<210>78<211>22<212>DNA<213>人<400>78tgtcagtttgtcaaatacccca22<210>79<211>22<212>DNA<213>人<400>79ctcagtagccagtgtagatcct22<210>80<211>24<212>DNA<213>人<400>80caagtcactagtggttccgtttag24<210>81<211>21<212>DNA<213>人<400>81aggggtgctatctgtgattga21<210>82<211>22<212>DNA<213>人<400>82taatactgtctggtaaaaccgt22<210>83<211>21<212>DNA<213>人<400>83gcagtccatgggcatatacac21<210>84<211>22<212>DNA<213>人<400>84aagacgggaggaaagaagggag22<210>85<211>22<212>DNA<213>人<400>85aatcgtacagggtcatccactt22<210>86<211>21<212>DNA<213>人<400>86cagcagcacactgtggtttgt21<210>87<211>22<212>DNA<213>人<400>87ttcacagggaggtgtcatttat22<210>88<211>23<212>DNA<213>人<400>88tcggggatcatcatgtcacgaga23<210>89<211>21<212>DNA<213>人<400>89agggggaaagttctatagtcc21<210>90<211>21<212>DNA<213>人<400>90aggaggcagcgctctcaggac21<210>91<211>25<212>DNA<213>人<400>91ggcggagggaagtaggtccgttggt25<210>92<211>24<212>DNA<213>人<400>92tgggctaagggagatgattgggta24<210>93<211>23<212>DNA<213>人<400>93aaggagcttacaatctagctggg23<210>94<211>21<212>DNA<213>人<400>94tggaggagaaggaaggtgatg21<210>95<211>24<212>DNA<213>人<400>95gtgggtagggtttgggggagagcg24<210>96<211>20<212>DNA<213>人<400>96taggatgggggtgagaggtg20<210>97<211>19<212>DNA<213>人<400>97gagggcgggtggaggagga19<210>98<211>23<212>DNA<213>人<400>98ttagggagtagaagggtggggag23<210>99<211>23<212>DNA<213>人<400>99tgaggatatggcagggaagggga23<210>100<211>24<212>DNA<213>人<400>100cccggacaggcgttcgtgcgacgt24<210>101<211>20<212>DNA<213>人<400>101gtgggttggggcgggctctg20<210>102<211>20<212>DNA<213>人<400>102tcagggagtcaggggagggc20<210>103<211>18<212>DNA<213>人<400>103gggctcacatcaccccat18<210>104<211>17<212>DNA<213>人<400>104ttggaggcgtgggtttt17<210>105<211>17<212>DNA<213>人<400>105ggtgggggctgttgttt17<210>106<211>20<212>DNA<213>人<400>106ggctccttggtctaggggta20<210>107<211>20<212>DNA<213>人<400>107ggatccgagtcacggcacca20<210>108<211>17<212>DNA<213>人<400>108ggatggaggaggggtct17<210>109<211>22<212>DNA<213>人<400>109gagcttggatgagctgggctga22<210>110<211>22<212>DNA<213>人<400>110gctgaactgggctgagctgggc22<210>111<211>22<212>DNA<213>人<400>111ttgaggagacatggtgggggcc22<210>112<211>22<212>DNA<213>人<400>112gagcaggcgaggctgggctgaa22<210>113<211>25<212>DNA<213>人<400>113aagggaggaggagcggaggggccct25<210>114<211>24<212>DNA<213>人<400>114agggctggactcagcggcggagct24<210>115<211>22<212>DNA<213>人<400>115ttcagatcccagcggtgcctct22<210>116<211>20<212>DNA<213>人<400>116aactctagcctgagcaacag20<210>117<211>22<212>DNA<213>人<400>117tgggggagtgcagtgattgtgg22<210>118<211>20<212>DNA<213>人<400>118gcggagagagaatggggagc20<210>119<211>21<212>DNA<213>人<400>119agcatgacagaggagaggtgg21<210>120<211>20<212>DNA<213>人<400>120ggaggttgggaagggcagag20<210>121<211>19<212>DNA<213>人<400>121tgagggagtgggtgggagg19<210>122<211>22<212>DNA<213>人<400>122aagtgattggaggtgggtgggg22<210>123<211>17<212>DNA<213>人<400>123cctgtctgagcgacgct17<210>124<211>17<212>DNA<213>人<400>124gagactctcctgtgcag17<210>125<211>22<212>DNA<213>人<400>125tgcagattgtgggtgggaggac22<210>126<211>22<212>DNA<213>人<400>126tgcagctggtggagtctggggg22<210>127<211>22<212>DNA<213>人<400>127tggagaagactggagagggtat22<210>128<211>25<212>DNA<213>人<400>128actaccccaggatgccagcatagtt25<210>129<211>22<212>DNA<213>人<400>129agctggtttgatggggagccat22<210>130<211>22<212>DNA<213>人<400>130cactgattatcgaggcgattct22<210>131<211>22<212>DNA<213>人<400>131cgcctgtgaatagtcactgcac22<210>132<211>22<212>DNA<213>人<400>132gacgtgagggggtgctacatac22<210>133<211>23<212>DNA<213>人<400>133ggctggtccgaaggtagtgagtt23<210>134<211>25<212>DNA<213>人<400>134ggctggtccgagtgcagtggtgttt25<210>135<211>25<212>DNA<213>人<400>135tgtccaaagtaaacgccctgacgca25<210>136<211>18<212>DNA<213>人<400>136ttcccggccaatgcatta18<210>137<211>22<212>DNA<213>人<400>137tttggaggggccgtgacagatg22<210>138<211>22<212>DNA<213>人<400>138ctcccactgcttcacttgacta22<210>139<211>19<212>RNA<213>人<220><221>n<222>(1)..(1)<223>n=a,g,c或u<220><221>m为c_特征<222>(1)..(1)<223>n为a,c,g,或u<400>139ngggccgagggagcgagag19<210>140<211>19<212>RNA<213>人<400>140agugcuuggcugaggagcu19<210>141<211>22<212>DNA<213>人<400>141tgaggtagtaggttgtatagtt22<210>142<211>22<212>DNA<213>人<400>142tgaggtagtaggttgtgtggtt22<210>143<211>22<212>DNA<213>人<400>143tgaggtagtaggttgtatggtt22<210>144<211>22<212>DNA<213>人<400>144agaggtagtaggttgcatagtt22<210>145<211>22<212>DNA<213>人<400>145tgaggtagtagattgtatagtt22<210>146<211>22<212>DNA<213>人<400>146tgaggtagtagtttgtacagtt22<210>147<211>22<212>DNA<213>人<400>147tgaggtagtagtttgtgctgtt22<210>148<211>23<212>DNA<213>人<400>148agcttctttacagtgctgccttg23<210>149<211>23<212>DNA<213>人<400>149agcagcattgtacagggctatga23<210>150<211>24<212>DNA<213>人<400>150aaaagtgcttacagtgcaggtagc24<210>151<211>21<212>DNA<213>人<400>151taaagtgctgacagtgcagat21<210>152<211>23<212>DNA<213>人<400>152agcagcattgtacagggctatca23<210>153<211>24<212>DNA<213>人<400>153tccctgagaccctttaacctgtga24<210>154<211>20<212>DNA<213>人<400>154tacagtatagatgatgtact20<210>155<211>23<212>DNA<213>人<400>155tctggctccgtgtcttcactccc23<210>156<211>22<212>DNA<213>人<400>156tcgaggagctcacagtctagta22<210>157<211>22<212>DNA<213>人<400>157tagcagcacatcatggtttaca22<210>158<211>22<212>DNA<213>人<400>158ccagtattaactgtgctgctga22<210>159<211>22<212>DNA<213>人<400>159tagcagcacgtaaatattggcg22<210>160<211>22<212>DNA<213>人<400>160actgcagtgaaggcacttgtag22<210>161<211>24<212>DNA<213>人<400>161caaagtgcttacagtgcaggtagt24<210>162<211>22<212>DNA<213>人<400>162tggagagaaaggcagttcctga22<210>163<211>23<212>DNA<213>人<400>163caacggaatcccaaaagcagctg23<210>164<211>22<212>DNA<213>人<400>164ctgccaattccataggtcacag22<210>165<211>23<212>DNA<213>人<400>165tgtgcaaatccatgcaaaactga23<210>166<211>23<212>DNA<213>人<400>166taaagtgcttatagtgcaggtag23<210>167<211>22<212>DNA<213>人<400>167cattgcacttgtctcggtctga22<210>168<211>22<212>DNA<213>人<400>168ttcaagtaatccaggataggct22<210>169<211>21<212>DNA<213>人<400>169ttcaagtaattcaggataggt21<210>170<211>22<212>DNA<213>人<400>170tgtaaacatcctacactcagct22<210>171<211>23<212>DNA<213>人<400>171tgtaaacatcctacactctcagc23<210>172<211>22<212>DNA<213>人<400>172tgtaaacatccccgactggaag22<210>173<211>23<212>DNA<213>人<400>173aaaagctgggttgagagggcgaa23<210>174<211>21<212>DNA<213>人<400>174agccgcggggatcgccgaggg21<210>175<211>21<212>DNA<213>人<400>175atcatagaggaaaatccacgt21<210>176<211>23<212>DNA<213>人<400>176aatgacacgatcactcccgttga23<210>177<211>17<212>DNA<213>人<400>177tggagagaaaggcagta17<210>178<211>20<212>DNA<213>人<400>178ccctgagaccctaaccttaa20<210>179<211>22<212>DNA<213>人<400>179tcaggctcagtcccctcccgat22<210>180<211>22<212>DNA<213>人<400>180tagtaccagtaccttgtgttca22<210>181<211>22<212>DNA<213>人<400>181tattgcacttgtcccggcctgt22<210>182<211>23<212>DNA<213>人<400>182caaagtgctgttcgtgcaggtag23<210>183<211>98<212>DNA<213>人<400>183gtggaccggctggccccatctggaagactagtgattttgttgttgtcttactgcgctcaa60caacaaatcccagtctacctaatggtgccagccatcgc98<210>184<211>85<212>DNA<213>人<400>184gtcttctgtatataccctgtagatccgaatttgtgtaaggaattttgtggtcacaaattc60gtatctaggggaatatgtagttgac85<210>185<211>69<212>DNA<213>人<400>185gttctaaggtgcatctagtgcagatagtgaagtagattagcatctactgccctaagtgct60ccttctggc69<210>186<211>90<212>DNA<213>人<400>186gtaccaccttgtcgggtagcttatcagactgatgttgactgttgaatctcatggcaacac60cagtcgatgggctgtctgacattttggtat90<210>187<211>73<212>DNA<213>人<400>187ggccggctggggttcctggggatgggatttgcttcctgtcacaaatcacattgccaggga60tttccaaccgacc73<210>188<211>90<212>DNA<213>人<400>188ggcagtctttgctactgtaaacatccttgactggaagctgtaaggtgttcagaggagctt60tcagtcggatgtttacagcggcaggctgcc90<210>189<211>71<212>DNA<213>人<400>189ggagaggaggcaagatgctggcatagctgttgaactgggaacctgctatgccaacatatt60gccatctttcc71<210>190<211>90<212>DNA<213>人<400>190gtgagtgtttctttggcagtgtcttagctggttgttgtgagcaatagtaaggaagcaatc60agcaagtatactgccctagaagtgctgcac90<210>191<211>130<212>DNA<213>人<400>191ggggagcgggatcccgggccccgggcgggcgggagggacgggacgcggtgcagtgttgtt60ttttcccccgccaatattgcactcgtcccggcctccggcccccccggccccccggcctcc120ccgctacccc130<210>192<211>90<212>DNA<213>人<400>192tctgcttggccgattttggcactagcacatttttgcttgtgtctctccgctctgagcaat60catgtgcagtgccaatatgggaaaagcagg90<210>193<211>82<212>DNA<213>人<400>193gcctgttgccacaaacccgtagatccgaacttgtggtattagtccgcacaagcttgtatc60tataggtatgtgtctgttaggc82<210>194<211>83<212>DNA<213>人<400>194gctggcgacgggacattattacttttggtacgcgctgtgacacttcaaactcgtaccgtg60agtaataatgcgccgtccacggc83<210>195<211>88<212>DNA<213>人<400>195tgcgctcctctcagtccctgagaccctaacttgtgatgtttaccgtttaaatccacgggt60taggctcttgggagctgcgagtcgtgct88<210>196<211>89<212>DNA<213>人<400>196accagacttttcctagtccctgagaccctaacttgtgaggtattttagtaacatcacaag60tcaggctcttgggacctaggcggagggga89<210>197<211>87<212>DNA<213>人<400>197tggtgtggtggggcagctggtgttgtgaatcaggccgttgccaatcagagaacggctact60tcacaacaccagggccacaccacacta87<210>198<211>99<212>DNA<213>人<400>198ccctggcatggtgtggtggggcagctggtgttgtgaatcaggccgttgccaatcagagaa60cggctacttcacaacaccagggccacaccacactacagg99<210>199<211>84<212>DNA<213>人<400>199cgttgctgcagctggtgttgtgaatcaggccgacgagcagcgcatcctcttacccggcta60tttcacgacaccagggttgcatca84<210>200<211>134<212>DNA<213>人<400>200gaggaagccggcggagttctggtatcgttgctgcagctggtgttgtgaatcaggccgacg60agcagcgcatcctcttacccggctatttcacgacaccagggttgcatcatacccatcctc120tccaggcgagcctc134<210>201<211>118<212>DNA<213>人<400>201gcgccctgtgtgtgtctctctctgtgtcctgccagtggttttaccctatggtaggttacg60tcatgctgttctaccacagggtagaaccacggacaggataccggggcaccctctgcgt118<210>202<211>104<212>DNA<213>人<400>202gtcggccggccctgggtccatcttccagtacagtgttggatggtctaattgtgaagctcc60taacactgtctggtaaagatggctcccgggtgggttctctcggc104<210>203<211>85<212>DNA<213>人<400>203acagtgcagtcacccataaagtagaaagcactactaacagcactggagggtgtagtgttt60cctactttatggatgagtgtactgt85<210>204<211>73<212>DNA<213>人<400>204cctggcactgagaactgaattccataggctgtgagctctagcaatgccctgtggactcag60ttctggtgcccgg73<210>205<211>80<212>DNA<213>人<400>205gtatcctcagctttgagaactgaattccatgggttgtgtcagtgtcagacctctgaaatt60cagttcttcagctgggatat80<210>206<211>84<212>DNA<213>人<400>206ggtcttttgaggcaaagttctgagacactccgactctgagtatgatagaagtcagtgcac60tacagaactttgtctctagaggct84<210>207<211>82<212>DNA<213>人<400>207tccccatggccctgtctcccaacccttgtaccagtgctgggctcagaccctggtacaggc60ctgggggacagggacctgggga82<210>208<211>85<212>DNA<213>人<400>208gtcccccccggcccaggttctgtgatacactccgactcgggctctggagcagtcagtgca60tgacagaacttgggcccggaaggac85<210>209<211>87<212>DNA<213>人<400>209tgtcccccccggcccaggttctgtgatacactccgactcgggctctggagcagtcagtgc60atgacagaacttgggcccggaaggacc87<210>210<211>89<212>DNA<213>人<400>210taggctgtatgctgttaatgctaatcgtgataggggtttttgcctccaactgactcctac60atattagcattaacagtgtatgatgcctg89<210>211<211>87<212>DNA<213>人<400>211ggttgcttcagtgaacattcaacgctgtcggtgagtttggaattaaaatcaaaaccatcg60accgttgattgtaccctatggctaacc87<210>212<211>79<212>DNA<213>人<400>212ggtcacaatcaacattcattgctgtcggtgggttgaactgtgtggacaagctcactgaac60aatgaatgcaactgtggcc79<210>213<211>110<212>DNA<213>人<400>213cggaaaatttgccaagggtttgggggaacattcaacctgtcggtgagtttgggcagctca60ggcaaaccatcgaccgttgagtggaccctgaggcctggaattgccatcct110<210>214<211>87<212>DNA<213>人<400>214cctccccccgtttttggcaatggtagaactcacactggtgaggtaacaggatccggtggt60tctagacttgccaactatggggcgagg87<210>215<211>66<212>DNA<213>人<400>215cctcgggctacaacacaggacccgggcgctgctctgacccctcgtgtcttgtgttgcagc60cggagg66<210>216<211>79<212>DNA<213>人<400>216gggagctgagggctgggtctttgcgggcgagatgagggtgtcggatcaactggcctacaa60agtcccagttctcggcccc79<210>217<211>71<212>DNA<213>人<400>217cctggctctagcagcacagaaatattggcacagggaagcgagtctgccaatattggctgt60gctgctccagg71<210>218<211>92<212>DNA<213>人<400>218tgtgctctgggggctgtgccgggtagagagggcagtgggaggtaagagctcttcaccctt60caccaccttctccacccagcatggccggcaca92<210>219<211>83<212>DNA<213>人<400>219ggccccgccaacccagtgttcagactacctgttcaggaggctctcaatgtgtacagtagt60ctgcacattggttaggctgggct83<210>220<211>65<212>DNA<213>人<400>220gagcatcttaccggacagtgctggatttcccagcttgactctaacactgtctggtaacga60tgttc65<210>221<211>89<212>DNA<213>人<400>221gctcgggcagccgtggccatcttactgggcagcattggatggagtcaggtctctaatact60gcctggtaatgatgacggcggagccctgc89<210>222<211>86<212>DNA<213>人<400>222gggcgggggccctcgtcttacccagcagtgtttgggtgcggttgggagtctctaatactg60ccgggtaatgatggaggcccctgtcc86<210>223<211>68<212>DNA<213>人<400>223ccctcgtcttacccagcagtgtttgggtgcggttgggagtctctaatactgccgggtaat60gatggagg68<210>224<211>83<212>DNA<213>人<400>224ggccccgccaacccagtgttcagactacctgttcaggaggctctcaatgtgtacagtagt60ctgcacattggttaggctgggct83<210>225<211>69<212>DNA<213>人<400>225gtctacccagtgtttagactatctgttcaggactcccaaattgtacagtagtctgcacat60tggttaggc69<210>226<211>87<212>DNA<213>人<400>226tccatgtgcttctcttgtccttcattccaccggagtctgtctcatacccaaccagatttc60agtggagtgaagttcaggaggcatgga87<210>227<211>84<212>DNA<213>人<400>227ccaggcgcagggcagcccctgcccaccgcacactgcgctgccccagacccactgtgcgtg60tgacagcggctgatctgtgcctgg84<210>228<211>104<212>DNA<213>人<400>228ggctggacagagttgtcatgtgtctgcctgtctacacttgctgtgcagaacatccgctca60cctgtacagcaggcacagacaggcagtcacatgacaacccagcc104<210>229<211>107<212>DNA<213>人<400>229gaacatccaggtctggggcatgaacctggcatacaatgtagatttctgtgttcgttaggc60aacagctacattgtctgctgggtttcaggctacctggaaacatgttc107<210>230<211>116<212>DNA<213>人<400>230cagctgctggaaggtgtaggtaccctcaatggctcagtagccagtgtagatcctgtcttt60cgtaatcagcagctacatctggctactgggtctctgatggcatcttctagcttctg116<210>231<211>110<212>DNA<213>人<400>231gctgctggaaggtgtaggtaccctcaatggctcagtagccagtgtagatcctgtctttcg60taatcagcagctacatctggctactgggtctctgatggcatcttctagct110<210>232<211>132<212>DNA<213>人<400>232gctcttggcctggcctcctgcagtgccacgctccgtgtatttgacaagctgagttggaca60ctccatgtggtagagtgtcagtttgtcaaataccccaagtgcggcacatgcttaccagct120ctaggccagggc132<210>233<211>83<212>DNA<213>人<400>233ggggctttcaagtcactagtggttccgtttagtagatgattgtgcattgtttcaaaatgg60tgccctagtgactacaaagcccc83<210>234<211>105<212>DNA<213>人<400>234gtgaaactgggctcaaggtgaggggtgctatctgtgattgagggacatggttaatggaat60tgtctcacacagaaatcgcacccgtcaccttggcctacttatcac105<210>235<211>99<212>DNA<213>人<400>235gaaactgggctcaaggtgaggggtgctatctgtgattgagggacatggttaatggaattg60tctcacacagaaatcgcacccgtcaccttggcctactta99<210>236<211>98<212>DNA<213>人<400>236acccaaaccctaggtctgctgactcctagtccagggctcgtgatggctggtgggccctga60acgaggggtctggaggcctgggtttgaatatcgacagc98<210>237<211>95<212>DNA<213>人<400>237ggtctctgtgttgggcgtctgtctgcccgcatgcctgcctctctgttgctctgaaggagg60caggggctgggcctgcagctgcctgggcagagcgg95<210>238<211>79<212>DNA<213>人<400>238cgctcccgccccgcgacgagcccctcgcacaaaccggacctgagcgttttgttcgttcgg60ctcgcgtgaggcaggggcg79<210>239<211>64<212>DNA<213>人<400>239ccccgcgacgagcccctcgcacaaaccggacctgagcgttttgttcgttcggctcgcgtg60aggc64<210>240<211>98<212>DNA<213>人<400>240cgaggggatacagcagcaattcatgttttgaagtgttctaaatggttcaaaacgtgaggc60gctgctataccccctcgtggggaaggtagaaggtgggg98<210>241<211>69<212>DNA<213>人<400>241gatgggcgtcttaccagacatggttagacctggccctctgtctaatactgtctggtaaaa60ccgtccatc69<210>242<211>82<212>DNA<213>人<400>242ggcgtgagggtatgtgcctttggactacatcgtggaagccagcaccatgcagtccatggg60catatacacttgcctcaaggcc82<210>243<211>200<212>DNA<213>人<400>243accccaaggtggagcccccagcgaccttccccttccagctgagcattgctgtgggggaga60gggggaagacgggaggaaagaagggagtggttccatcacgcctcctcactcctctcctcc120cgtcttctcctctcctgcccttgtctccctgtctcagcagctccaggggtggtgtgggcc180cctccagcctcctaggtggt200<210>244<211>100<212>DNA<213>人<400>244gtgctaacctttggtacttggagagtggttatccctgtcctgttcgttttgctcatgtcg60aatcgtacagggtcatccactttttcagtatcaagagcgc100<210>245<211>116<212>DNA<213>人<400>245ctgatctccatcctccctggggcatcctgtactgagctgccccgaggcccttcatgctgc60ccagctcggggcagctcagtacaggatactcggggtgggagtcagcaggaggtgag116<210>246<211>68<212>DNA<213>人<400>246gcatcctgtactgagctgccccgaggcccttcatgctgcccagctcggggcagctcagta60caggatac68<210>247<211>64<212>DNA<213>人<400>247tcctgtactgagctgccccgagctgggcagcatgaagggcctcggggcagctcagtacag60gatg64<210>248<211>100<212>DNA<213>人<400>248cggtcctgctcccgccccagcagcacactgtggtttgtacggcactgtggccacgtccaa60accacactgtggtgttagagcgagggtgggggaggcaccg100<210>249<211>129<212>DNA<213>人<400>249gggatgccacattcagccattcagcgtacagtgcctttcacagggaggtgtcatttatgt60gaactaaaatataaatttcacctttctgagaagggtaatgtacagcatgcactgcatatg120tggtgtccc129<210>250<211>101<212>DNA<213>人<400>250ggatgcacagatctcagacatctcggggatcatcatgtcacgagataccagtgtgcactt60gtgacagattgataactgaaaggtctgggagccactcatct101<210>251<211>105<212>DNA<213>人<400>251tgccagatgtgctctcctggcccatgaaatcaagcgtgggtgagacctggtgcagaacgg60gaaggcgacccatacttggtttcagaggctgtgagaataactgca105<210>252<211>96<212>DNA<213>人<400>252agatgtgctctcctggcccatgaaatcaagcgtgggtgagacctggtgcagaacgggaag60gcgacccatacttggtttcagaggctgtgagaataa96<210>253<211>96<212>DNA<213>人<400>253gggacctgcgtgggtgcgggcgtgtgagtgtgtgtgtgtgagtgtgtgtcgctccgggtc60cacgctcatgcacacacccacacgcccacactcagg96<210>254<211>96<212>DNA<213>人<400>254tggtaagggtagagggatgagggggaaagttctatagtcctgtaattagatctcaggact60atagaactttccccctcatccctctgccctctacca96<210>255<211>59<212>DNA<213>人<400>255tctcaggaggcagcgctctcaggacgtcaccaccatggcctgggctctgctcctcctca59<210>256<211>98<212>DNA<213>人<400>256ctcggttgccgtggttgcgggccctgcccgcccgccagctcgctgacagcacgactcagg60gcggagggaagtaggtccgttggtcggtcgggaacgag98<210>257<211>170<212>DNA<213>人<400>257gttcagtccagggcagcttccctgttctgttaattaaactttgggacattaaaatgggct60aagggagatgattgggtagaaagtattattctattcatttgcctcccagcctacaaaaat120gcctgcttggggtctaatacttcaacggttaaagatgcctggaagagggc170<210>258<211>93<212>DNA<213>人<400>258ggtaactgccctcaaggagcttacaatctagctgggggtaaatgacttgcacatgaacac60aactagactgtgagcttctagagggcagggacc93<210>259<211>112<212>DNA<213>人<400>259ttaggcgctgatgaaagtggagttcagtagacagcccttttcaagccctacgagaaactg60gggtttctggaggagaaggaaggtgatgaaggatctgttctcgtgagcctga112<210>260<211>69<212>DNA<213>人<400>260gtgggtagggtttgggggagagcgtgggctggggttcagggacaccctctcaccactgcc60ctcccacag69<210>261<211>82<212>DNA<213>人<400>261tggtccctcccaatccagccattcctcagaccaggtggctcccgagccaccccaggctgt60aggatgggggtgagaggtgcta82<210>262<211>81<212>DNA<213>人<400>262gctcgactcctgttcctgctgaactgagccagtgtgtaaaatgagaactgatatcagctc60agtaggcaccggagggcgggt81<210>263<211>57<212>DNA<213>人<400>263cccggtgagggcgggtggaggaggagggtccccaccatcagccttcactgggacggg57<210>264<211>113<212>DNA<213>人<400>264aagttaattttgaagctgacttttttagggagtagaagggtggggagcatgaacaatgtt60tctcactccctacccctccactccccaaaaaagtcagcttctcttgttaactt113<210>265<211>82<212>DNA<213>人<400>265ggccccacgtggtgaggatatggcagggaaggggagtttccctctattcccttcccccca60gtaatcttcatcatgcggtgtc82<210>266<211>59<212>DNA<213>人<400>266gcgcgtgcgcccgagcgcggcccggtggtccctcccggacaggcgttcgtgcgacgtgt59<210>267<211>89<212>DNA<213>人<400>267gaggaaaagatcgaggtgggttggggcgggctctggggatttggtctcacagcccggatc60ccagcccacttaccttggttactctcctt89<210>268<211>65<212>DNA<213>人<400>268caaatagcttcagggagtcaggggagggcagaaatagatggccttcccctgctgggaaga60aagtg65<210>269<211>76<212>DNA<213>人<400>269ttctgtgaggggctcacatcaccccatcaaagtggggactcatggggagagggggtagtt60aggagctttgatagag76<210>270<211>53<212>DNA<213>人<400>270ggtgggggttggaggcgtgggttttagaacctatccctttctagccctgagca53<210>271<211>91<212>DNA<213>人<400>271gttctagagcatggtttctcatcatttgcactactgatacttggggtcagataattgttt60gtggtgggggctgttgtttgcattgtaggat91<210>272<211>84<212>DNA<213>人<400>272ggagtgaccaaaagacaagagtgcgagccttctattatgcccagacagggccaccagagg60gctccttggtctaggggtaatgcc84<210>273<211>55<212>DNA<213>人<400>273ccggatccgagtcacggcaccaaatttcatgcgtgtccgtgtgaagagaccacca55<210>274<211>58<212>DNA<213>人<400>274gtgaatgacccccttccagagccaaaatcaccagggatggaggaggggtcttgggtac58<210>275<211>139<212>DNA<213>人<400>275aactgggctgggctgaactgggctgggctgagctgagcttggatgagctgggctgaactg60ggctgggttgagctgggctgggctgagttgagccaggctgatctgggctgagccgagctg120ggttaagccgagctgggtt139<210>276<211>194<212>DNA<213>人<400>276ggctgggctgggctgggctctgctgtgctgtgctgaacagggctgagctgaactgagctg60agctgggctgagctgggctctgctgtgctgtgctgagcagggctgagctgaactgggctg120agctgggctgagctgggctgagttgagcagagctgggttgagcagagctgggctgggctg180ggctgagttgagcc194<210>277<211>72<212>DNA<213>人<400>277cggtttctccttgaggagacatggtgggggccggtcaggcagcccatgccatgtgtcctc60atggagaggccg72<210>278<211>80<212>DNA<213>人<400>278ggcaggtgagcaggcgaggctgggctgaacccgtgggtgaggagtgcagcccagctgagg60cctctgctgtcttatctgtc80<210>279<211>91<212>DNA<213>人<400>279gtgggcaggggaggaagaagggaggaggagcggaggggcccttgtcttcccagagcctct60cccttcctcccctccccctccctctgctcat91<210>280<211>86<212>DNA<213>人<400>280gggcggctgcgcagagggctggactcagcggcggagctggctgctggcctcagttctgcc60tctgtccaggtccttgtgacccgccc86<210>281<211>68<212>DNA<213>人<400>281ctgggggtaggagcgtggcttctggagctagaccacatgggttcagatcccagcggtgcc60tctaactg68<210>282<211>97<212>DNA<213>人<400>282gagctatgattgtgtagctgaactctagcctgagcaacagagtgagatggtcttgttttg60ttgcccaggctggagtccagtgtcaagatcatggctc97<210>283<211>93<212>DNA<213>人<400>283gagctccaaatctgtgcacctgggggagtgcagtgattgtggaatgcaaagtcccacaat60cactgtactccccaggtgcacagattctctctc93<210>284<211>82<212>DNA<213>人<400>284gattggactttattgtcacgttctgattggttagcctaagacttgttctgatccaatcag60aacatgaaaataacgtccaatc82<210>285<211>82<212>DNA<213>人<400>285gattggactttattgtcacgttctgattggttagcctaagacttgttctgatccaatcag60aacatgaaaataacgtccaatc82<210>286<211>77<212>DNA<213>人<400>286ttggctataactatcatttccaaggttgtgcttttaggaaatgttggctgtcctgcggag60agagaatggggagccag77<210>287<211>113<212>DNA<213>人<400>287agcatgacagaggagaggtggaggtaggcgagagtaatataatttctccaggagaacatc60tgagaggggaagttgctttcctgccctggccctttcaccctcctgagtttggg113<210>288<211>55<212>DNA<213>人<400>288aggaggttgggaagggcagagatgagcataaagtttttgccttgtttttcttttt55<210>289<211>91<212>DNA<213>人<400>289aagatgagggagtgggtgggaggtgggaaggctgccccaaatggcctctaacatcccttc60cagtctcctcctcctcctcctccttcttctt91<210>290<211>117<212>DNA<213>人<400>290tatgtacccggagccaaaagtgattggaggtgggtggggttaatgaatagacaagtgtta60aaactaaaagtcacgtctctctctccttcctcctcagttttggcttgatttttcatg117<210>291<211>76<212>DNA<213>人<400>291cttacctagaaattgttgcctgtctgagcgacgcttcaaactcagcttcagcaggtctgc60agggacatcaggtagg76<210>292<211>130<212>DNA<213>人<400>292gtgtctctgtgtttgcaggtgtccagtgtgaggtgcagctggtggagtctgggggaggct60tggtacagcctgggggatccctgagactctcctgtgcagcctctggattcaccttcagta120acagtgacat130<210>293<211>63<212>DNA<213>人<400>293gtcagcctgcaattagtgaaatggaggcacacatgctggtttgcagattgtgggtgggag60gac63<210>294<211>130<212>DNA<213>人<400>294gtgtctctgtgtttgcaggtgtccagtgtgaggtgcagctggtggagtctgggggaggct60tggtacagcctgggggatccctgagactctcctgtgcagcctctggattcaccttcagta120acagtgacat130<210>295<211>88<212>DNA<213>人<400>295ggccttggatggagaagactggagagggtatggaagtgcttggacgtaggacatctgcct60ctctggtctttgtccatcccacagggcc88<210>296<211>71<212>DNA<213>人<400>296agctggttggcattctggccctggttcatgccaactcttgtgttgactaccccaggatgc60cagcatagttg71<210>297<211>103<212>DNA<213>人<400>297ctgccaaagagcagcaagatgagctggtttgatggggagccatcccttgatgaggagaac60ccttcccactctcactcagcctcacccagctgccctgaggcag103<210>298<211>82<212>DNA<213>人<400>298gctcagaagtgatgaattgatcagatagacgaggccgggcttgtccccggccactgatta60tcgaggcgattctgatctgggc82<210>299<211>130<212>DNA<213>人<400>299gctgggtgcagtagcttatgtctgtagtcccagctacttgggaggctgaggtgggaggat60cacctgaggtcaggagtttgggtctgccgtgagctgtgattgcgcctgtgaatagtcact120gcactccagc130<210>300<211>55<212>DNA<213>人<400>300gacgtgagggggtgctacatacagcagctgtgtgtagtatgtgcctttctctgtt55<210>301<211>130<212>DNA<213>人<400>301taggaattctggaccaggcttaaaagactgggatgaggctggtccgaaggtagtgagtta60tctccattgatagttcagtctgtaacagatcaaactccttgttctactcttttttttttt120tttagacaga130<210>302<211>103<212>DNA<213>人<400>302tgggctggtccgagtgcagtggtgtttacaagtatttgattataactagttacagatttc60tttgtttccttctccactcccactgcctcacttgactggccta103<210>303<211>56<212>DNA<213>人<400>303gctctgtccaaagtaaacgccctgacgcactgtgggaagggtgagatgggcaccgc56<210>304<211>63<212>DNA<213>人<400>304gtgagtgggaggggggctgcagcccaaagaggcaacaaaggcccttcccggccaatgcat60tac63<210>305<211>169<212>DNA<213>人<400>305tgtcctcaggcctgctactgatcctgcagccagaagttccagaaagtgaagggatttgga60ggggccgtgacagatgcaggtgccctcaacatccttgccctgtcaccccctgcccagaat120ttgctacttaaatggtacttctctgaagaagatgaggaggaaggggaca169<210>306<211>106<212>DNA<213>人<400>306acagaattcctcttctcccttctcctataacctgttttatttaattaattaattttttag60gctagtcaagtgaagcagtgggagtggaaggaacaaagaaatctgt106<210>307<211>45<212>RNA<213>人<400>307ugagcucugcggcgccaagggaccgaggggccgagggagcgagag45<210>308<211>75<212>RNA<213>人<400>308agugcuuggcugaggagcuggggccaagggggaacacaaauaugguccugacccuacauu60cccagcccugccucu75<210>309<211>22<212>DNA<213>人工序列<220><223>通用反向引物<400>309gcgagcacagaattaatacgac22<210>310<211>46<212>DNA<213>人工序列<220><223>RT反应引物<220><221>m为c_特征<222>(45)..(45)<223>v=a,g或c<220><221>m为c_特征<222>(46)..(46)<223>v=a,g或cn=a,g,c或t<400>310gcgagcacagaattaatacgactcactatcggttttttttttttvn46<210>311<211>18<212>DNA<213>人工序列<220><223>通用探针<400>311aaaaccgatagtgagtcg18<210>312<211>23<212>DNA<213>人工序列<220><223>特异性正向引物<400>312gcagctacatctggctactgggt23<210>313<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>313cagtcatttggcgcgacccatacttggt28<210>314<211>21<212>DNA<213>人工序列<220><223>特异性正向引物<400>314aggcaagatgctggcatagct21<210>315<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>315cagtcatttgggtttgttcgttcggctc28<210>316<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>316cagtcatttgggtccctgagaccctaac28<210>317<211>26<212>DNA<213>人工序列<220><223>特异性正向引物<400>317tggctgagaactgaattccataggct26<210>318<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>318cagtcatttggctcagtgcatgacagaa28<210>319<211>23<212>DNA<213>人工序列<220><223>特异性正向引物<400>319tgtctgcccgcatgcctgcctct23<210>320<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>320cagtcatttggcaacattcaacctgtcg28<210>321<211>21<212>DNA<213>人工序列<220><223>特异性正向引物<400>321caaaacgtgaggcgctgctat21<210>322<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>322cagtcatttgggtctcacacagaaatcg28<210>323<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>323cagtcatttggcagctggtgttgtgaat28<210>324<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>324cagtcatttggctcctgtactgagctgc28<210>325<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>325cagtcatttgggtaatactgccgggtaa28<210>326<211>24<212>DNA<213>人工序列<220><223>特异性正向引物<400>326ttggcagtgaagcattggactgta24<210>327<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>327cagtcatttggcatcacattgccaggga28<210>328<211>29<212>DNA<213>人工序列<220><223>特异性正向引物<400>328catttggctaagccagtttctgtctgata29<210>329<211>27<212>DNA<213>人工序列<220><223>特异性正向引物<400>329tggcatgacagattgacatggacaatt27<210>330<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>330cagtcatttggcgctgactcctagtcca28<210>331<211>22<212>DNA<213>人工序列<220><223>特异性正向引物<400>331cgttcctgctgaactgagccag22<210>332<211>18<212>DNA<213>人工序列<220><223>特异性正向引物<400>332cctgtctgagcgccgctc18<210>333<211>30<212>DNA<213>人工序列<220><223>特异性正向引物<400>333cagtcatttggcatactctggtttcttttc30<210>334<211>30<212>DNA<213>人工序列<220><223>特异性正向引物<400>334agtcatttggcttattgtcacgttctgatt30<210>335<211>28<212>DNA<213>人工序列<220><223>特异性正向引物<400>335cagtcatttggccacgctcatgcacaca28<210>336<211>23<212>DNA<213>人工序列<220><223>特异性探针<400>336ccgttttttttttttacgggtgc23<210>337<211>23<212>DNA<213>人工序列<220><223>特异性探针<400>337ccgttttttttttttactcaccg23<210>338<211>23<212>DNA<213>人工序列<220><223>特异性探针<400>338ccgttttttttttttcacaagtt23<210>339<211>23<212>DNA<213>人工序列<220><223>特异性探针<400>339ccgttttttttttttcacgcgag23<210>340<211>23<212>DNA<213>人工序列<220><223>特异性探针<400>340ccgttttttttttttctcggggc23<210>341<211>23<212>DNA<213>人工序列<220><223>特异性探针<400>341ccgttttttttttttctgaaacc23<210>342<211>23<212>DNA<213>人工序列<220><223>特异性探针<400>342ccgttttttttttttggaaatcc23<210>343<211>23<212>DNA<213>人工序列<220><223>特异性探针<400>343ccgttttttttttttgtgggtgt23<210>344<211>22<212>DNA<213>人工序列<220><223>特异性探针<400>344cgttttttttttttccaagttc22<210>345<211>22<212>DNA<213>人工序列<220><223>特异性探针<400>345cgttttttttttttccatcatt22<210>346<211>22<212>DNA<213>人工序列<220><223>特异性探针<400>346cgttttttttttttcggcctga22<210>347<211>22<212>DNA<213>人工序列<220><223>特异性探针<400>347cgttttttttttttgagccctg22当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1