测定样品中PIK3CA突变状态的方法与流程
本发明涉及一种用于确定以下各项的DNA样品中PIK3CA突变状态的高灵敏度方法:例如,循环肿瘤细胞(CTC)、血浆/血清无细胞DNA(cfDNA)和福尔马林固定石蜡包埋(FFPE)组织。
背景技术:
循环肿瘤细胞(CTC)检测和计数可以充当“液体活组织检查”及响应于全身治疗的早期标志物,而它们的分子表征具有被翻译为个体化靶向治疗并使癌症患者免遭不必要的和无效的治疗的强大潜力。
已经显示在辅佐化疗之前在7.5mL血液中对一个或多个CTC的检测可以准确地预测总体存活期(OS)。在随访的最初5年期间对CTC的持续检测与晚期疾病复发和死亡的风险增加有关,并指出化疗和激素治疗抵抗性残余疾病的存在。最近一项前瞻性临床研究证实一个或多个CTC的存在预测早期复发和减少的总体存活期(OS)。
在转移性乳腺癌(MBC)中,CTC代表无进展存活期(PFS)和OS的独立预后因素,而CTC计数测定法(enumeration assay)(CellSearchTM系统,Veridex)由FDA批准用于转移性乳腺癌、前列腺癌、和结肠直肠癌。疗法的第二个周期之前增加的CTC数量是不良的PFS和OS的早期预测性标志物,并且可以用于监测治疗益处,然而接受治疗的CTC减少随着靶向疗法而更强。在一线治疗前患有MBC的患者中的CTC检测可以定义具有惨淡临床结果的患者的亚组。
现在确立的是,无细胞DNA(cfDNA)从经历细胞凋亡或其他由微环境胁迫诱导的生理事件的细胞释放到循环中并且可以在患有癌症的患者的血液样品中被鉴别。然而,与产生的cfDNA片段的长度(DNA完整性)相关,cfDNA的来源可以区别于凋亡或坏死原点。术语循环肿瘤DNA(ctDNA)基本上包括衍生自肿瘤的总cfDNA的一个亚型。许多研究表明,ctDNA和CTC两者不仅存在于晚期而且甚至存在于早期阶段的癌症患者的血浆/血清和外周血中。
虽然有许多可商购的cfDNA提取试剂盒,但是由于起始材料在提取过程中的损失,效率和产量仍然很低,并且因为缺乏标准化,其定量是可变的。尽管如此,cfDNA提取效率可以直接影响突变检测即测定灵敏度的结果。
磷酸肌醇3-激酶(PI3K)包括在20世纪80年代发现的脂质激酶家族,其负责调解重要的生物功能,例如细胞存活、分化和增殖。磷脂酰肌醇3-激酶(PI3K)/AKT信号传导途径牵涉于包括癌症的人类疾病,并且理解此途径的复杂性可以为治疗性干预提供新途径。在PI3K的p110α催化亚基中的体细胞突变,在许多类型的实体癌如乳腺癌、结肠直肠癌、前列腺癌、卵巢癌、宫颈癌、头颈癌、食道癌、肺癌、脑癌、皮肤癌、肝癌、胰腺癌、胃癌或甲状腺癌中是非常频繁的,并且在响应于分子靶向疗法中起关键作用并与乳腺癌中的HER-2扩增共同发生。PIK3CA的突变已经在18%-40%的乳腺癌患者中进行了报道,而绝大多数(包括大约90%的病例)聚集在外显子9与外显子20中的两个热点区域中。
在CTC、cfDNA或FFPE组织的DNA样品中检测PIK3CA热点突变的临床相关性是很重要的,因为PIK3CA突变的存在与靶向疗法中的药物抗性相关。问题是,突变以非常低的数量存在于临床肿瘤样品中,并且现有方法学的检测限是非常低的,因此导致假阴性,其可以影响临床诊断和患者管理。
ctDNA的分析已被证明为一个有用的工具以评价肿瘤进展及评价预后、诊断和治疗反应。许多研究已经证实ctDNA的临床效用和许多技术已经被开发以增加用于此目的的方法的分析灵敏度。扬库(Janku)等人,使用分批整经法(beaming method),已经表明,在这两个外显子中PIK3CA突变在组织与血浆之间的一致性比例为91%[扬库F(Janku F),等人.参与实验靶向疗法的患晚期癌症患者的血浆无细胞DNA中的可行突变(Actionable mutations in plasma cell-free DNA in patients with advanced cancers referred for experimental targeted therapies).癌靶标(Oncotarget).2015;6:12809-21]。在另一项研究中,使用数字PCR(dPCR)测定,ctDNA中的PIK3CA突变的百分比发现于22.7%的患有乳腺癌的患者中[大城C(Oshiro C),等人.血清DNA中的PIK3CA突变预示着原发性乳腺癌患者中的复发(PIK3CA mutations in serum DNA are predictive of recurrence in primary breast cancer patients).乳腺癌研究与治疗(Breast Cancer Res Treat).2015;150:299-307)。
出于这个原因,用于PIK3CA热点突变的新颖方法已被开发,其特征在于极端灵敏度(0.05%)和高特异性(100%)[马尔苟A(MARKOU A),等人.循环肿瘤细胞中的PIK3CA突变状态可以在患有乳腺癌患者的疾病复发或进展期间改变(PIK3CA mutational status in circulating tumor cells can change during disease recurrence or progression in patients with breast cancer).临床癌症研究(Clin Cancer Res).2014年11月15日;20(22):5823-34]。此测定提供许多优点:它可以在过量的野生型等位基因存在下检测极少量的具有PIK3CA突变的突变等位基因。另外,通过使用所开发的方法,分离自CTC的DNA中的PIK3CA突变在早期乳腺癌患者(20.3%)和患有临床确诊转移的患者(35.1%)两者中均能以比以前报道高得多的百分比检测出。
出于这个原因,用于检测CTC中的PIK3CA热点突变(外显子9和20)的超灵敏和高度特异的方法,基于等位基因特异引发、野生型扩增的竞争性阻断探针、不对称PCR、以及探针解链分析[马尔苟A,等人.循环肿瘤细胞中的PIK3CA突变状态可以在患有乳腺癌患者中的疾病复发或进展期间改变.临床癌症研究.2014年11月15日;20(22):5823-34]的组合而开发和验证。数据还表明,PIK3CA突变状态可以在患有乳腺癌患者中的疾病复发或进展期间改变,并且在CTC中PIK3CA突变的存在与患有临床确诊转移的患者中的较差存活相关联[马尔苟A,等人.CTC中的PIK3CA突变状态可在乳腺癌患者中的疾病复发或进展期间发生改变.临床癌症研究.2014年11月15;20(22):5823-34]。
技术实现要素:
目的是提供一种用于确定例如CTC的样品中的PIK3CA突变状态的改进方法。
这个目的通过根据权利要求1所述的方法完全或部分实现。本发明的实施例和进一步细节在所附的从属权利要求、附图和序列表中阐述。
因此,该方法涉及对样品中PIK3CA等位基因的存在进行确定,即对样品中含有一个或多个突变(如一个或多个热点突变)的PIK3CA等位基因的存在进行确定。该样品可以,例如,属于CTC,但该方法也可用于任何其他类型的生物样品中,例如用于实体瘤血浆/血清或FFPE组织中的无细胞DNA中。一个或多个PIK3CA突变的检测可被用来确定多种类型的癌症,包括结肠癌、乳腺癌、脑癌、甲状腺癌、胰腺癌、前列腺癌、头颈癌、卵巢癌、宫颈癌、肝癌、胃癌、食道癌、皮肤癌和肺癌。当确定罹患癌症的风险时,该方法特别有利,其中PIK3CA突变可能如例如在乳腺癌的早期诊断期间存在。
本发明的方法是基于由L.周(Zhou)等人针对BRAF突变而首次描述的途径[周L等人.通过等位基因特异性PCR、竞争性探针阻断及解链分析的罕见的等位基因富集与检测.生物技术(BioTechniques)2011;50:311-8]。使用这种方法的基本途径,设计从头引物和探针并检查所有的实验条件以便检测CTC样品中的PIK3CA热点突变。解链分析和未标记探针方面自犹他州爱达荷州技术大学取得授权。
根据本发明的方法可以增强在均相体系中的罕见等位基因检测。该方法包括使用竞争性阻断探针和解链分析的非对称的及等位基因特异性聚合酶链反应(PCR)。该方法需要一种与有待扩增的突变等位基因DNA靶标的第一链的3'(三撇(prime))端互补的突变等位基因特异性引物以及一种未标记的阻断探针(竞争性探针),该阻断探针是一种寡核苷酸,其与野生型DNA的相应第一链的野生型序列互补并且有待检测的突变恰好存在于该位置处。此外,该方法包括一个通用引物,其与有待通过PCR扩增的DNA靶标的第二链的3'端互补。
根据本发明的用于确定PIK3CA突变等位基因在例如CTC的样品中存在的方法则包括以下步骤:
·进行非对称的和等位基因特异性聚合酶链反应(PCR),和
·进行在PCR中产生的DNA的解链分析,
该PCR通过使用以下项来进行:
·突变等位基因特异性引物,其与有待扩增的突变等位基因DNA靶标的第一链的3'(三撇)端互补,
·未标记阻断探针,其是与对应于突变等位基因第一链的野生型DNA第一链的野生型序列互补的寡核苷酸,并且在该对应位置中存在有待检测的突变,并且在PCR反应中所述探针被阻断而不作为用于DNA合成的引物;和
·一个通用引物,其与有待通过PCR扩增的DNA靶标的第二链的3'端互补。
解链分析通过利用以下项进行
·为未标记探针的解链探针,该解链探针是一种寡核苷酸,该寡核苷酸包含与野生型等位基因序列互补的序列并且与突变等位基因的序列重叠;和
·用于测量双链DNA组分的解链温度的可检测组分,该双链DNA组分至少包括结合到扩增的突变等位基因链或野生等位基因链的解链探针的双链组分,其中该解链温度在结合到扩增的突变等位基因链的解链探针的双链组分与结合到扩增的野生等位基因链的解链探针之间不同。
然后使用等位基因特异性PCR来富集罕见的等位基因。该等位基因特异性PCR需要一个为突变等位基因特异性引物(反向或正向)的第一引物,将该第一引物设计为对所希望突变等位基因完全特异并且将其3'端设计为恰好在应检测的突变位点处。还使用为通用引物(正向或反向)的第二引物,该第二引物结合到第一引物所结合到的互补链,并且可以用于扩增突变体与野生型链等位基因两者。以这种方式,存在于例如野生型的样品中的其他等位基因是错配的并且非特异性扩增受到限制。然而,通过仅使用一个突变等位基因特异性引物,这种抑制在目前为止所描述的所有情况下不是100%完成的,由于野生型通常以过量的浓度存在。
因此,该方法包括竞争性探针阻断的使用,其中使用一种未标记的阻断探针。该未标记的阻断探针(竞争性探针)是一种恰好在有待检测的突变存在的位置处与野生型序列互补的寡核苷酸。这个未标记的阻断探针在其3'端被阻断用作该PCR中的引物,例如与用于PCR的正常引物相比,通过在其3'端具有额外的磷酸基团而阻断。将这个未标记的阻断探针用于野生型等位基因的竞争性阻断并且以比突变等位基因特异性引物更高的浓度添加,例如该等位基因特异性引物的5至20倍或10倍更高的浓度。突变等位基因特异性引物和未标记的阻断探针的序列存在着重叠,并且当突变等位基因特异性引物和此未标记的阻断引物两者均存在(野生型和突变体)时;该未标记的阻断探针杂交到野生型,并且该突变等位基因特异性引物杂交到突变等位基因。因此,该未标记的阻断探针与等位基因特异性引物竞争增加的灵敏度,因为它被设计成与野生型相匹配,从而准确地结合在野生型等位基因上。该未标记的阻断探针可被设计,使得相应的热点突变尽可能靠近未标记的阻断探针的中心来放置。以这种方式,野生型的非特异性扩增可以降低到最小程度。罕见的等位基因富集是最佳的,其中与等位基因特异性引物相比,阻断探针和反向引物是过量的。
该不对称PCR包括等位基因特异性PCR,其中该突变等位基因特异性引物相对于通用引物是以较低的浓度(例如低10倍)添加的。以这种方式,此突变等位基因特异性引物在PCR中仅由以极低浓度存在的突变等位基因来充分使用。在一个突变等位基因存在下以及在一些PCR循环之后,该突变等位基因特异性引物被充分使用,并且包括该突变信息的链然后被过量生产,因为它被用作用于另一个引物的恒定模板,所述另一个引物对于这两个等位基因是通用的,并且野生型等位基因的扩增通过使用突变等位基因特异性引物和未标记的阻断探针而受限。所产生的单链PCR产物包含该突变信息。在PCR之后,这些是过量的并且被过量的、不完全互补的探针识别,所以解链曲线是在一个较低的温度下。
解链分析跟随PCR反应并且包括以下步骤:将温度从低于所感兴趣的解链温度的温度增加至高于所感兴趣的解链温度的温度,并且检测双链DNA的解链温度。
解链分析包括为一种未标记探针的解链探针的使用,该探针是一个寡核苷酸,其包含与野生型等位基因序列互补并优选地在突变位置周围与突变等位基因的序列重叠的序列。解链探针(未标记的阻断探针)然后可以包括与突变等位基因特异性引物的序列重叠的序列。解链探针提供了与其结合到野生型等位基因相比不同的解链温度用于其结合到突变等位基因。如在此还例举,解链探针可以是未标记的阻断探针。未标记的阻断探针至突变等位基因的解链温度低于野生型等位基因的未标记阻断探针的解链温度。将未标记的阻断探针以非常高的浓度加入,并且这主要用于该反应以阻断野生型序列,其中该突变等位基因特异性引物将不能够非特异性结合到野生型并给予非特异性PCR产物。此外,该相同的未标记的阻断探针识别包含突变信息的单链以及如上所述的野生型单链。其结果是,得到的解链曲线如同等位基因在该探针下的特定签名。
解链探针与不对称PCR产物的互补性第一链之间的解链温度的测量可以通过使用荧光检测技术来进行,其中使用了荧光染料。在一个实施例中,该荧光染料是仅在所测量的样品中的双链DNA存在下发出荧光的染料。该染料可以是LC-绿加(LC-Green Plus)。通过测量双链DNA和荧光染料LC-绿加的荧光的发射,得到解链曲线,这是突变等位基因的特征,因为该突变DNA序列的解链温度低于野生型序列的解链温度。该方法可以包括在PCR反应结束后增加温度;那时所有的产品都是双链并以100%发射荧光。然后将温度逐渐升高,并且当温度达到DNA序列的特征温度,即Tm,荧光开始减少。Tm是如下温度,在该温度下DNA的50%是双链并且50%是单链。
使用染料的解链分析可以,例如,包括以下步骤:55℃至60℃退火持续10s以及95℃持续1min,其中将该温度在55℃至60℃的温度下开始以0.2℃/s的增量(升温速率)逐渐增加,并且通过检测染料(数据采集步骤)来测量解链温度。
在PCR反应中有待扩增的突变等位基因DNA靶标可以包括PIK3CA的外显子9(SEQ NO ID:1)和/或外显子20(SEQ ID NO:2)或由其组成,并且该突变等位基因特异性引物序列与外显子9(SEQ NO ID:1)或外显子20(SEQ NO ID:2)的DNA链互补。
解链探针可以是未标记的阻断探针。该未标记的阻断探针可以具有一个3'-端,与用于扩增的PCR引物相比,该3'-端通过添加磷酸基团修饰。这将阻断未标记的阻断探针作为PCR引物用于DNA链的合成的用途。任选地,未标记的阻断探针可被一个或多个非荧光部分修饰,如但不限于非荧光小沟结合剂、生物素、间隔段、接头、磷酸、碱基类似物、非天然碱、以及类似物。
如上所讨论的,可检测的组分可以包括荧光组分,并且其中解链分析则可以包括检测该荧光组分。荧光组分可以是一种荧光染料,如下组的荧光染料,该组由LC-绿加或SYBR绿I组成,该荧光染料仅在样品中的双链DNA存在下发出荧光。
在该反应中,将未标记的阻断探针以比突变等位基因特异性引物高的浓度进行添加,以阻断野生型等位基因序列的扩增。该未标记的探针优先结合到野生型DNA并且与引物结合竞争。同时,突变等位基因特异性引物的较低浓度导致仅延伸突变等位基因序列。因此,阻断探针的浓度应更于突变体引物,因为有待结合到过量的野生型等位基因。此外,突变等位基因特异性引物与通用引物之间的不同浓度导致过量产生包括突变信息的链以便增加该方法的灵敏度。
该突变可以存在于PIK3CA的外显子9(SEQ NO ID:1)中,其中该突变等位基因特异性引物包含序列5’-TTTCTCCTGATT-3’(SEQ ID NO:3)或由其组成,其中T指示突变位点并且A指示一个额外的错配,该错配抑制野生型等位基因序列的扩增,以便仅增加突变罕见等位基因的扩增并导致加强方法的特异性。该突变等位基因特异性引物的序列可优选地包括或为5’-ACTCCATAGAAAATCTTTCTCCTGATT-3’(SEQ ID NO:4)。
该未标记的阻断探针可以包含以下项或由其组成:序列5’-CTGATCAGTGA-3’(SEQ ID NO:5),其中C指示序列与野生型位点互补的精确位置;以及一个PCR阻断组分,其在PCR反应中阻断未标记的阻断探针作为DNA合成的引物。该未标记的阻断探针的序列可以优选地包括或为5’-CTTTCTCCTGATCAGTGATTTCAGAG-P-3’(SEQ ID NO:6),其中P是充当PCR阻断组分的磷酸。
通用引物可与序列5’-GCTCAAAGCAATTTCTACACGAGA-3’(SEQ ID NO:7)具有75%至100%一致性。这意味着,一个通用引物可以与核酸序列5’-GCTCAAAGCAATTTCTACACGAGA-3’(SEQ ID NO:7)具有至少75%、或至少80%、或至少85%或至少90%一致性,如75%-100%、76%-100%、77%-100%、78%-100%、79%-100%、80%-100%、81%-100%、82%-100%、83%-100%、84%-100%、85%-100%、86%-100%、87%-100%、89%-100%、90%-100%、91%-100%、92%-100%、93%-100%、94%-100%、95%-100%、96%-100%、97%-100%、98%-100%、99%-100%或约100%一致性。
该突变还可以存在于PIK3CA的外显子20(SEQ ID NO:2)中,其中该突变等位基因特异性引物包括序列5’-AATGATGCACG-3’(SEQ ID NO:8)或由其组成,其中G指示突变位点。该突变等位基因特异性引物的序列可以优选地包括或为5’-ATGAAACAAATGAATGATGCACG-3’(SEQ ID NO:9)。
该未标记的阻断探针可以包含以下项或由其组成:序列5’-TGCACATCATG-3’(SEQ ID NO:10),其中A指示序列与野生型位点互补的精确位置;以及一个PCR阻断组分,其在PCR反应中阻断未标记的阻断探针作为DNA合成的引物。该未标记的阻断探针的序列可以优选地包括或为5’-GAATGATGCACATCATGGTGG-P-3’(SEQ ID NO:11),其中P是充当PCR阻断组分的磷酸。
然后通用引物可与序列5’-TCTCAGTTATCTTTTCAGTTCAATGC-3’(SEQ ID NO:12)具有75%至100%一致性。这意味着,一个通用引物可以与核酸序列5’-TCTCAGTTATCTTTTCAGTTCAATGC-3’(SEQ ID NO:12)具有至少75%、或至少80%、或至少85%或至少90%一致性,如75%-100%、76%-100%、77%-100%、78%-100%、79%-100%、80%-100%、81%-100%、82%-100%、83%-100%、84%-100%、85%-100%、86%-100%、87%-100%、89%-100%、90%-100%、91%-100%、92%-100%、93%-100%、94%-100%、95%-100%、96%-100%、97%-100%、98%-100%、99%-100%或约100%一致性。
本文件还涉及一种用于以下项的方法
i)诊断受试者中的恶性赘生性疾病,和/或
ii)预测治疗受试者中的恶性赘生性疾病的效力,和/或
iii)评价治疗受试者中的恶性赘生性疾病的结果,和/或
iv)评价受试者中的恶性赘生性疾病的复发
其中该受试者是患有或怀疑患有恶性赘生性疾病的哺乳动物,
其中所述方法包括根据如在此所述的步骤分析样品中PIK3CA突变等位基因DNA的存在。
如在此所述的方法可以有利地用于检测样品中的PIK3CA突变等位基因DNA的存在,并且
i)诊断受试者中的恶性赘生性疾病,和/或
ii)预测治疗受试者中的恶性赘生性疾病的效力,和/或
iii)评价治疗受试者中的恶性赘生性疾病的结果,和/或
iv)评价受试者中的恶性赘生性疾病的复发
其中该受试者是患有或怀疑患有恶性赘生性疾病的哺乳动物。
所分析的样品可以是一个生物样品,并且所述生物样品可获自受试者。有利的是,该受试者是人。
恶性赘生性疾病可以选自下组,该组由以下各项组成:乳腺癌、结肠癌、子宫内膜癌、食道癌、胃癌(gastric cancer)、头颈癌、肝癌、卵巢癌、甲状腺癌、皮肤癌、胰腺癌、前列腺癌和胃部癌(stomach cancer)。
有利的是,该恶性赘生性疾病是乳腺癌。
当在受试者中诊断和/或预测恶性赘生性疾病时,该方法包括以下步骤
a)从给定受试者获得生物样品
b)如在此所述,进行用于在从所述生物样品中获得的DNA样品中分析PIK3CA突变等位基因DNA的存在的方法
c)在所述DNA样品中检测PIK3CA突变等位基因DNA的存在;和
d)将在所述DNA样品中检测的PIK3CA突变等位基因DNA的量与阳性对照和/或阴性对照进行比较,从而诊断和/或预测受试者中的恶性赘生性疾病。
另外的实施例是其中阳性对照包括来自携带突变的细胞系的细胞。甚至另外的实施例是其中阴性对照包括来自未罹患恶性赘生性疾病的健康受试者的细胞。
当在罹患恶性赘生性疾病的受试者中预测治疗结果或预测对治疗的反应时,该方法包括以下步骤
a)从给定受试者获得生物样品
b)如在此所述,进行用于在从所述生物样品中获得的DNA样品中分析PIK3CA突变等位基因DNA的存在的方法;和
c)在所述DNA样品中检测PIK3CA突变等位基因DNA的存在;和
d)将在所述DNA样品中检测的PIK3CA突变等位基因DNA的量与阳性和/或阴性对照进行比较,从而预测所述受试者中的恶性赘生性疾病的治疗结果,这基于在所述DNA样本中检测到的PIK3CA突变等位基因DNA的存在。
当在正在因恶性赘生性疾病接受治疗的受试者中评价恶性赘生性疾病的治疗效力时,该方法包括以下步骤
a)从正因恶性赘生性疾病接受治疗的受试者的中获得生物样品
b)如在此所述,进行用于在从所述生物样品中获得的DNA样品中分析PIK3CA突变等位基因DNA的存在的方法;
c)在所述DNA样品中检测PIK3CA突变等位基因DNA的存在;和
d)在因恶性赘生性疾病治疗所述受试者期间,在一个或多个时间点重复步骤a)至c),并且其中在所述DNA样品中PIK3CA突变等位基因DNA的相对存在随时间的变化指示治疗效力。
因此,有效治疗的指示是PIK3CA突变等位基因DNA在所述DNA样品中的渐降存在相对于重复该方法的步骤中分析的先前样品的相对变化。
任选地,可以根据本领域已知或在此所述的标准评分系统完成所检测的PIK3CA突变等位基因DNA于所述DNA样品中的评分。
该样品可以是可能包含恶性赘生性疾病的任何样品,优选来自具有恶性赘生性疾病的受试者的生物样品,并且该受试者将在两者之间或目前正在治疗。
当评价恶性赘生性疾病的复发时,该方法包括以下步骤
a)从先前已经具有恶性赘生性疾病的受试者获得生物样品,
b)检测PIK3CA突变等位基因DNA在获自所述生物样品的DNA样品中的存在,
c)在因恶性赘生性疾病治疗所述受试者后的一个或多个时间点重复步骤a)和b),并且其中在所述DNA样品中PIK3CA突变等位基因DNA的相对存在随时间的变化可指示恶性赘生性疾病的复发。
因此,复发的指示是PIK3CA突变等位基因DNA在鉴定恶性赘生性疾病的所述DNA样品中的渐增量(即,PIK3CA突变等位基因DNA在所述DNA样品中的存在的随时间增加)相对于重复该方法的步骤中分析的先前样品的相对变化。
本发明还涉及一种用于检测样品中PIK3CA的外显子9中的突变的存在的多核苷酸,其至少包括序列5’-TTTCTCCTGATT-3’(SEQ ID NO:3)或由其组成,优选地是包括5’-ACTCCATAGAAAATCTTTCTCCTGATT-3’(SEQ ID NO:4)或由其组成的序列,其中T指示突变位点并且A指示额外的错配。该多核苷酸可以有利地在如上所述的方法中用作突变等位基因特异性引物,用于检测PIK3CA的外显子9中的突变的存在。该多核苷酸可以用作乳腺癌的预后标志物。
本发明还涉及一种用于检测样品中PIK3CA的外显子20中的突变的存在的多核苷酸,该多核苷酸至少包括序列5’-AATGATGCACG-3’(SEQ ID NO:8)或由其组成,优选地是包括5’-ATGAAACAAATGAATGATGCACG-3’(SEQ ID NO:9)或由其组成的序列,其中G指示突变位点。该多核苷酸可以在如在此所述的方法中用作突变等位基因特异性引物,用于检测PIK3CA的外显子20中的突变的存在。该多核苷酸可以用作乳腺癌的预后标志物。
本发明还涉及一种用于检测样品如CTC、ctDNA或FFPE中的PIK3CA突变等位基因DNA的试剂盒。用于在样品中检测PIK3CA中的突变的试剂盒也可以用于
i)检测生物样品中的PIK3CA突变等位基因DNA,和/或
i)检测受试者中的恶性赘生性疾病;或
iii)诊断或预测受试者中的恶性赘生性疾病;或
iv)预测治疗受试者中的恶性赘生性疾病的结果;或
v)评价治疗受试者中的恶性赘生性疾病的效力;或
vi)评价受试者中的恶性赘生性疾病的复发;
该试剂盒可以包括
-用于检测样品中的PIK3CA的外显子9中的突变的第一多核苷酸,所述第一多核苷酸至少包括序列5’-TTTCTCCTGATT-3’(SEQ ID NO:3)或由其组成,优选地所述第一多核苷酸包括5’-ACTCCATAGAAAATCTTTCTCCTGATT-3’(SEQ ID NO:4)或由其组成,和/或
-用于检测样品中的PIK3CA的外显子20中的突变的第二多核苷酸,所述第二多核苷酸至少包括序列5’-AATGATGCACG-3’(SEQ ID NO:8)或由其组成,优选地所述第二多核苷酸包括5’-ATGAAACAAATGAATGATGCACG-3’(SEQ ID NO:9)或由其组成。第一和/或第二多核苷酸可以在如以上所述的方法中用作突变等位基因特异性引物。
该试剂盒可以进一步包括至少包括以下项或由其组成的第三多核苷酸:序列5’-CTGATCAGTGA-3’(SEQ ID NO:5)和一个PCR阻断组分,优选地所述第三多核苷酸包括或是5’-CTTTCTCCTGATCAGTGATTTCAGAG-P-3’(SEQ ID NO:6),其中P是磷酸并且A是额外的错配;和/或至少包括以下项或由其组成的第四多核苷酸:序列5’-TGCACATCATG-3’(SEQ ID NO:10)和一个PCR阻断组分,优选地其中所述第四多核苷酸包括或是5’-GAATGATGCACATCATGGTGG-P-3’(SEQ ID NO:11),其中P是磷酸。该第三和/或第四多核苷酸可以在以上所述的方法中用作未标记的阻断探针。
该试剂盒可以进一步包括具有与序列5’-GCTCAAAGCAATTTCTACACGAGA-3’(SEQ ID NO:7)具有75%至100%一致性的序列的第五多核苷酸,和/或与序列5’-TCTCAGTTATCTTTTCAGTTCAATGC-3’(SEQ ID NO:12)具有75%至100%一致性的第六多核苷酸。该第五和/或第六多核苷酸可以在以上所述的方法中用作通用引物。
附图说明
图1示出了当前研究的实验流程图。
图2A-D描绘了针对外显子9 1633G>A(A)和外显子20 3140A>G(B)所开发的PIK3CA突变测定的特异性以及针对外显子9 1633G>A(C)和外显子20 3140A>G(D)所开发的PIK3CA突变测定的灵敏度。
图3A-D显示在患有可动手术的乳腺癌的患者中针对外显子9 1633G>A(A)和外显子20 3140A>G(B)对CTC中的PIK3CA突变的检测。在临床确诊转移的患者中针对外显子9 1633G>A(C)和外显子20 3140A>G(D)对CTC中的PIK3CA突变的检测。
图4描绘了卡普兰-迈耶(Kaplan-Meier)曲线,其估计患有乳腺癌与临床确诊转移的患者以月数计的OS,这相对于CTC中的PIK3CA突变状态。
图5显示在临床确诊为转移性乳腺癌的患者中的无细胞DNA中针对外显子9 1633G>A(A,B)和外显子20 3140A>G(C)对PIK3CA突变的检测。
图6呈现了该方法的原理
图7呈现了针对外显子9和20的核苷酸序列
定义
通常预期本发明中使用的术语符合分子生物学领域的普通技术人员公认的标准定义。如下所列的少数例外已经进一步定义于本发明的范围内。
如在此所使用的“至少一个/种”意指一个/种或多个/种,即1、2、3、4、5、6、7、8、9、10个/种等。
如在此所使用的“检测(detection或detect或detecting)”包括在参考或不参考对照的情况下的定性和/或定量检测(测量水平),并且进一步是指鉴定给定PIK3CA突变等位基因DNA分子的存在、不存在或量。
如在此所使用,术语“核酸序列”、“核酸分子”、“核酸”等是指一种多核苷酸分子(DNA-脱氧核糖核酸,或RNA-核糖核酸),其包含一串核酸碱基。这些核酸碱基是“A”(腺嘌呤)、“T”(胸腺嘧啶)/“U”(尿嘧啶),“C”(胞嘧啶)和“G”(鸟嘌呤)。在RNA中,“T”被替换为“U”。DNA或RNA可以是单链或双链的。通过“对应于”一种核酸序列(在此表示为DNA序列)的RNA序列,旨在指相同的核酸序列,但其中“T”被“U”替换以得到相应的RNA序列。术语“核酸”可以包括DNA和/或RNA序列两者,除非具体提及一者或另一者。
如在此与核酸分子(DNA和RNA分子)结合使用,术语“分离的”意指该分子已经从其原始环境中移出。这意指这样一种核酸分子,当其存在于活的有机体时不是“分离的”。化学键断开和/或通过其他方式将序列从其天然环境分离意指该核酸分子是“分离的”。
如在此所使用的,术语“引物”是指经合成而产生的寡核苷酸,当置于以下条件下时其能作为核酸合成的起始点:其中诱导与核酸链互补的引物延伸产物的合成,即在核苷酸和聚合作用剂如DNA聚合酶、反转录酶或类似物存在下并在合适的温度和pH下。引物优选地是用于最大效率的单链,但可替代地可以是双链。如果是双链,引物首先经过处理以分开其双链,然后再用于制备延伸产物。引物必须足够长以在聚合作用剂存在下引发延伸产物的合成。引物的确切长度将取决于许多因素,包括温度和引物来源。例如,取决于靶序列的复杂性,引物典型地包含15至25个或更多个核苷酸,尽管它可以包含更少的核苷酸。短的引物分子通常需要较冷的温度以形成与模板足够稳定的杂交复合物。
术语“突变等位基因特异性引物”是指以下引物:其与突变等位基因序列杂交并能够在靶序列的变体之间鉴别,因为只有具有这些突变,该引物才在合适的条件下通过核酸聚合酶有效地延伸。具有靶序列的其他变体,延伸是低效或无效的。
术语“正向引物”是指通过以5'到3'方向与变性DNA分析物的一条链的3'端结合形成一个延伸产物的引物。
术语“反向引物”是指通过以3'到5'方向与变性DNA分析物的一条链的5'端结合形成一个延伸产物的引物。
术语“扩增子”是指一种核酸延伸测定如PCR的扩增产物。
术语“未标记的探针”是指未共价连接于染料并被配置成完全或部分地杂交至靶序列的寡核苷酸。该混合物中存在的染料不能结合未标记探针或与未标记探针解离,尤其是当探针与靶序列杂交和解链时。
术语阻断探针或竞争探针是指以下一种寡核苷酸,其与野生型等位基因序列互补并且为了避免非特异性野生型产物扩增而与突变等位基因特异性引物竞争。
如在此所使用,关于寡核苷酸的术语“熔融温度”(Tm)被定义为以下温度,在该温度下DNA的50%形成稳定的双螺旋并且其他50%已被分离成单链分子。如本领域技术人员已知的,PCR退火温度典型地比Tm小几度,其中后者基于反应中的寡核苷酸和盐浓度来计算。
术语“互补的”或“互补性”参考沃森-克里克(Watson-Crick)碱基配对法则涉及的多核苷酸的反向平行链使用。术语“完美互补的”或“100%互补的”是指在反向平行链之间所有碱基都具有沃森-克里克配对的互补序列,即,在多核苷酸双链体中任何两个碱基之间不存在错配。然而,即使在完美互补不存在下,反向平行链之间也形成双链体。术语“部分互补的”或“不完全互补的”是指在反向平行多核苷酸链之间少于100%完美的碱基的任何比对(例如,在多核苷酸双链体中存在至少一个错配或未配对的碱基)。部分互补链之间的双链体通常比完美互补链之间的双链体更不稳定。
术语“多核苷酸”和“寡核苷酸”可以互换使用。
“寡核苷酸”是有时用于描述更短的多核苷酸的术语。
术语“杂交的”和“杂交”是指在两个核酸之间导致形成双链体的碱基配对相互作用。不要求两个核酸在其全长上具有100%的互补性以实现杂交。
如本文件中所使用的,通过“其变体”或“其变体和类似物”,旨在指一个或多个核酸序列,与指定的核酸序列具有的一致性为至少85%或至少90%,如85%-100%、86%-100%、87%-100%、89%-100%、90%-100%、91%-100%、92%-100%、93%-100%、94%-100%、95%-100%、96%-100%、97%-100%、98%-100%、99%-100%或约100%。
PCR(聚合酶链式反应)是用于扩增核酸分子的方法。PCR反应是本领域技术人员公知的技术,并涉及在以下条件下使样品与一对所谓的寡核苷酸引物接触(一个正向引物和一个反向引物),所述条件允许在引物与靶(模板)序列之间的杂交,该靶(模板)序列与这些引物具有互补性并且其靶向可能存在于样品中的序列以便扩增靶序列。
如在此所使用的“诊断”包括疾病性质的鉴定。
如在此所使用的“预后”包括关于疾病的可能结果的预测、关于如由疾病的性质和症状所指示的从疾病恢复的希望。
“真阳性”是指在局部或转移的恶性赘生物中PIK3CA特异性突变的存在。
“假阴性”是指在局部或转移的恶性赘生物中PIK3CA特异性突变的存在并且本身没有通过诊断测定法归类。
“真阴性”是指没有局部或转移的恶性赘生物并且本身通过诊断测定法归类的那些受试者。
“假阳性”是指不具有局部或转移的恶性赘生物而通过常规诊断测定法归类为具有局部或转移的恶性赘生物的那些受试者。
根据上下文,术语“假阳性”也可以是指不具有恶性赘生物而通过诊断测定法归类为具有恶性赘生物或非恶性疾病的那些受试者。
在此所使用的“灵敏度”(如其应用于诊断测定法的上下文中)是指本身被正确鉴定的所有具有局部或转移的恶性赘生物的受试者的比例(即,真阳性的数目除以真阳性和假阴性的数目的总和)。
在此所使用的诊断测定法的“特异性”(如其应用于诊断测定法的上下文中)是指本身被正确鉴定的没有局部和转移的恶性赘生物的所有受试者的比例(即,真阴性的数目除以真阴性和假阳性的数目的总和)。
术语“赘生物(neoplasm)”或“肿瘤(tumor)”可互换使用,并且是指组织的异常肿块,其中肿块的生长超过正常组织的生长,且不与正常组织的生长协调。赘生物或肿瘤可被定义为“良性的”或“恶性的”,其取决于以下特征:细胞分化的程度(包括形态学和功能性),生长速率,局部侵入以及转移。“良性”赘生物通常是分化良好的,特征是比恶性赘生物生长缓慢,并且依然位于原初位点。另外,良性赘生物不具有浸润、侵入或转移至远位点的能力。
“恶性”赘生物通常是低分化的(间变),具有特异性快速生长连同进行性的浸润、侵入以及外围组织的破坏。另外,恶性赘生物具有转移至远的位点的能力。术语“转移”是指癌性细胞从原发性(初始)肿瘤扩散或迁移至另一器官或组织,且典型地是通过以下项可识别的:原发性(初始)肿瘤的组织类型的“继发性肿瘤”或“次级细胞块”的存在,而不是定位次级(转移性)肿瘤的器官或组织的“继发性肿瘤”或“次级细胞块”的存在。例如已经迁移至骨的肺癌被认为是转移的肺癌,并且由源自骨组织中生长的上皮肺细胞的癌细胞组成。
“健康”是指具有良好健康的受试者。这样的受试者表明不存在任何恶性或非恶性的疾病。在本申请的上下文中,“健康个体”只有当他们不存在任何恶性或非恶性疾病才是健康的;“健康个体”可以具有通常不被视为“健康”的其他疾病或病况。
如在此所使用的“受试者”包括人;非人灵长类如黑猩猩和其他猿类和猴类;农场动物如牛、绵羊、猪、山羊和马;家养哺乳动物如狗和猫;实验动物,包括啮齿动物如小鼠、大鼠和豚鼠等。该术语不指示具体的年龄或性别。因此,预期涵盖成人和新生的受试者以及胎儿,无论雄性或雌性。在优选的实施例中,该受试者是哺乳动物,包括人类和非人类哺乳动物。在最优选的实施例中,该受试者是人。
“血浆(Blood plasma)或(plasma)”是血液的稻草色/淡黄色液体成分,其通常保持全血中的血细胞在悬浮液中。它按体积计构成总血的约55%。它是细胞外液(细胞外的所有体液)的血管内液部分。它主要是水(按体积计93%),并包含溶解的蛋白质(包括白蛋白、免疫球蛋白)和纤维蛋白原、葡萄糖、凝血因子、电解质(Na+、Ca2+、Me2+、HCO3-“、CI-”等)、激素以及二氧化碳。
如在此所使用的“生物样品”包括多种从患有或不患有恶性赘生物的任何受试者获得的样品类型。一个典型的受试者是人。例如,生物样品包括从怀疑具有恶性赘生物的个体采集的组织或血液流体获得的样品。
如在此所用的术语“治疗”被定义为通过内科或外科的方式管理患者。该治疗改善或减轻医学病症或疾病的至少一种症状并被要求提供治愈。如在此所使用的术语“治疗结果”或“治疗的结果”是该治疗对患者的物理效应。
如在此所用的术语循环肿瘤细胞(CTC)是已经从原发性肿瘤流出进入脉管并且在血流中循环的细胞。因此CTC构成重要远处器官中的额外肿瘤(转移)的后续生长的种子,从而触发负责绝大多数的癌症相关死亡的机制。
术语无细胞DNA(cfDNA)是指以下DNA,其从经历细胞凋亡或其他由微环境胁迫诱导的生理事件的细胞释放到循环。该cfDNA可以在患有癌症的患者血液中被检测到。
术语循环肿瘤DNA(ctDNA)是指以下DNA,其从原发性肿瘤的细胞释放到循环并提供有关原发性肿瘤或转移性肿瘤的状态的信息。
如在此中所使用的,术语PIK3CA是指所谓的“磷脂酰肌醇-4,5-二磷酸肌醇3-激酶、催化亚基α”基因的法定名称。磷脂酰肌醇3-激酶(PI3K)/AKT信号传导途径牵涉于包括癌症的人类疾病,并且理解此途径的复杂性可以为治疗性干预提供新途径。在PI3K的p110α催化亚基中的体细胞突变是非常频繁的,并且在响应于分子靶向疗法中起关键作用并常与乳腺癌中的HER-2扩增共同发生。PIK3CA的突变已经在18%-40%的乳腺癌患者中进行了报道,而绝大多数(包括大约90%的病例)聚集在外显子9与外显子20中的两个热点区域中,外显子9和外显子20分别编码螺旋结构域和激酶结构域。PI3K途径的异常活化与对HER2定向疗法的反应减弱相关,因为与野生型肿瘤相比,用曲妥珠单抗治疗的HER2-阳性患者的结果在患有PIK3CA-突变的患者中是显著更差的。
术语突变等位基因DNA是指根据参照序列在PIK3CA基因的两个外显子之间包含至少一个突变的DNA序列。该突变等位基因DNA可能在外显子9中具有1633G>A的热点突变或在外显子20中具有3140A>G的热点突变。
该突变是构成基因的DNA序列中的永久改变,这样使得该序列不同于在参照序列中所发现的。突变以大小计变动;它们可以从单一的DNA结构单元(碱基对)影响到一大段包含多个基因的染色体的任何位置。
如在此中所使用的,术语“参照基因组”或“参照序列”是指任何生物体或病毒的任何特定的已知基因组序列(无论是部分的还或完整的),其可以用于给从受试者识别出的序列作参照。例如,用于人类受试者以及许多其他生物体的参照基因组见于美国国家生物技术信息中心(National Center for Biotechnology Information)www.ncbi.nlm.nih.gov。“基因组”是指以核酸序列形式表示的生物体或病毒的完整遗传信息。
术语“野生等位基因链”是指编码特定自然群体中最常见的表型的DNA序列。最初,该野生型被概念化为在一个基因座的标准的“正常”等位基因的产物,与此相反的是通过非标准的“突变体”等位基因产生的产物。
术语“热点突变”是指在一个染色体区域存在的突变,其对遗传损伤/变化比平均值序列更敏感。
具体实施方式
本发明提供了用于在例如循环肿瘤细胞或循环肿瘤DNA的一个样品中进行PIK3CA热点突变的高通量突变检测的超灵敏和高度特异的分子方法。PIK3CA突变的重要性与乳腺癌中响应于分子靶向疗法相关联。
材料与方法
患者
作为训练组,分析了总共78个样品:ⅰ)63个外周血样品;37个来自临床确诊转移的患者并且26个来自健康女性志愿者,用来定义该测定的特异性,以及ii)15个原发性乳腺肿瘤组织(FFPE)。作为独立组,总共175个外周血样本获自118位患有可动手术的乳腺癌的患者以及57位患有临床确诊转移的患者;此外,针对这些乳腺癌患者中的76位(32位患有转移并且44位患有可动手术的乳腺癌)对来自原发性肿瘤的FFPE进行了分析。对于这些样品中的157个,关于CK-19在EpCAM阳性CTC部分中的表达的信息通过先前的研究也是可获得的。在患有可动手术的乳腺癌的118位患者的独立组中,9位患者复发和死于疾病进展(中位随访:42个月)。另外,患有HER2+肿瘤的患者接收曲妥珠单抗持续12个月,然而患有HR+肿瘤的患者接受内分泌治疗(LH/RH类似物加他莫昔芬或芳香酶抑制剂)。根据指引还执行辅助性放射疗法。所有的研究参与者签署了知情同意书以参与该研究,该同意书经协会的伦理和科学委员会(the ethics and scientific committees of the institutions)批准。
CTC的阳性免疫磁性选择
如先前所述将CTC分离自20mL的外周血[斯特拉蒂A(Strati A)等人.通过RT-qPCR的乳腺癌中的循环肿瘤细胞的基因表达谱.BMC癌症(BMC Cancer)2011,11:422]。更具体地,在将外周血用20mL PBS(pH=7.3)稀释之后,通过使用菲可帕克(Ficoll-Paque)TM PLUS(GE医疗集团(GE Healthcare),生物科学公司(Bio-Science AB))以670g在室温下的梯度密度离心持续30min获得外周血单核细胞(PBMC)。将接触面细胞(interface cell)除去,用40mL的无菌PBS(pH=7.3,4℃)以530g持续10min洗涤两次,并再悬浮于10mL的PBS中。将细胞用台盼蓝染色并在血细胞计数器中计数。免疫磁性Ber-EP4[抗上皮细胞粘附分子(EpCAM)]-包被的捕获珠粒(Dynabeads Epithelial Enrich,英杰公司(Invitrogen))被用来富集上皮细胞。
自CTC提取DNA
如先前所述的从CTC提取基因组DNA(gDNA)[西莫尼多M(Chimonidou M)等人.循环肿瘤细胞中的肿瘤抑制和转移抑制基因的DNA甲基化(DNA methylation of tumor suppressor and metastasis suppressor genes in circulating tumor cells).临床化学(Clin Chem)2011;57:1169-77]。除去Trizol的水相后,通过加入150μL的100%乙醇来沉淀(从中间相)DNA。将样品通过颠倒进行混合并在室温下保持2-3min,并且然后将DNA通过离心(2000g,5min,4℃)沉积并在包含0.1mol/L的柠檬酸钠于10%乙醇(500μL)中的溶液中洗涤两次。每次洗涤后,将DNA沉淀在室温下伴随周期性混合储存于洗涤液中持续30min并离心(2000g,5min,4℃)。在这些2次洗涤之后,将DNA沉淀悬浮于1mL的75%乙醇中,在室温下伴随周期性混合保持10-20min,并离心(2000g,5min,4℃)。然后将分离的gDNA进行空气干燥持续15min并溶解于50μL的8mmol/L NaOH中。将DNA浓度在Nanodrop ND-1000分光光度计中进行测定。将来自26位雌性健康志愿者的经采集用于特异性研究的外周血通过使用与针对患者的样品所用的完全相同的方法进行处理。
自血浆提取DNA
根据制造商的说明使用QIAamp循环核酸试剂盒(QIAGEN)从血浆样品分离无细胞DNA。首先,将EDTA中的外周血样品在1小时内用于血浆分离,并且将血浆样品储存在-70℃直到cfDNA分离。恰好在cfDNA分离之前,将血浆样品在室温下解冻,并在4℃以13,400g离心10min以除去残留的沉淀的细胞组分。总共,将2.00mL的血浆与工作液的1.6mL缓冲液ACL(含有1.0μg载体RNA)和200μl蛋白酶K(18mg ml-1)混合,并在60℃孵育30min。然后,如在制造商的方案中所述的处理DNA分离。
引物和探针设计
通过使用PrimerPremier 5软件(第一生物软件国际公司(Premier Biosoft International)),将所有的寡核苷酸均从头经由电脑模拟(in-silico)针对PIK3CA外显子9(SEQ ID NO:1)和外显子20(SEQ ID NO:2)中的每一个进行设计,并且通过IDT(综合DNA技术公司(Intergraded DNA Technologies))合成。外显子9(SEQ ID NO:1)和外显子20(SEQ ID NO:2)的序列参见图7。对于每一个外显子,将一个等位基因特异性引物(与外显子9的1633G>A突变和外显子20的3140A>G突变匹配)、一个未标记的竞争性阻断探针、以及一个用于不对称扩增的引物根据周和其同事的研究进行设计[周L(Zhou L)等人.通过等位基因特异性PCR、竞争性探针阻断和解链分析的罕见等位基因富集与检测(Rare allele enrichment and detection by allele-specific PCR,competitive probe blocking,and melting analysis).生物技术(BioTechniques)2011;50:311-8]。对于外显子9,引物组S1被设计成扩增包含外显子9的热点突变的区域(70bp)。反向引物(等位基因特异性引物)被设计成通过匹配派生等位基因的30端来扩增该突变等位基因。未标记的探针和正向引物被设计成与野生型相匹配。阻断探针与等位基因特异性引物竞争增加的灵敏度。对于外显子20,引物组S2被设计成扩增包含外显子20的热点突变的区域(104bp)。正向引物(等位基因特异性引物)被设计成通过匹配派生等位基因的30端来扩增该突变等位基因。未标记的探针和反向引物被设计成与野生型相匹配。将热点突变尽可能靠近未标记的阻断探针的中心来放置。所有引物和探针被留心地设计成避免染色体22上的假基因的扩增,该假基因与PIK3CA的外显子9具有>95%同源性。所有引物和探针序列在表1中详细给出。
表1:在此研究中设计并使用的引物和探针序列
PCR和解链分析
使用LightScanner 32(爱达荷州技术公司(Idaho Technology),美国)、采用玻璃毛细管(罗氏应用科学公司(Roche Applied Science),德国)获得实时PCR和解链曲线。然而,相同结果也可以通过使用LightCycler 2.0(IVD)仪器和LightCycler 480(罗氏诊断(Roche Diagnostics))来获得。LC-绿加(LC-Green Plus)(爱达荷州技术公司,美国)被用于荧光测量。将从MCF-7(c.1633G>A:E545K;杂合子)和T47D(c.3140A>G:H1047R;杂合子)乳腺癌细胞系中分离的两个gDNA样本用作PIK3CA突变对照。每个外显子的PCR条件和解链分析方案详细描述于表2中。每个外显子的PCR反应混合物详细描述于表3中。
表2:每个PIK3CA外显子的PCR条件和解链分析方案
表3:用于检测外显子9和外显子20中的PIK3CA热点突变的PCR反应混合物
统计分析
CTC中的PIK3CA突变状态与原发性肿瘤之间的相关性是通过使用卡方检验评价的。将科恩(Cohen)氏κ系数(一种用于定性项目的评判间一致性或注释者间一致性的统计量度)用于评价在CTC中的PIK3CA突变与原发性肿瘤之间以及在CTC中的PIK3CA突变与CK-19mRNA表达之间的一致性,因为其通常被认为是比简单的百分之一致性计算更准确的量度(由于k考虑到偶然发生的一致性)。通过使用卡普兰-迈耶(Kaplan-Meier)方法来计算无病间隔(DFI)和总体存活期(OS)曲线并用时序检验进行比较。P值<0.05被认为是统计学上显著的。使用SPSS Windows版本19.0(SPSS,芝加哥,伊利诺州)进行统计分析。
实例1
用于PIK3CA热点突变的超灵敏和高度特异性方法的开发和验证
该研究的实验流程图概述于图1中。
最初,用于CTC中的PIK3CA热点突变(外显子9和20)的超灵敏和高度特异性方法被开发和验证。将该测定在封闭管形式中进行并基于等位基因特异性引发、野生型扩增的竞争性探针阻断、不对称PCR、以及探针解链分析[马尔苟A(Markou A),等人.循环肿瘤细胞中的PIK3CA突变状态可以在患有乳腺癌患者中的疾病复发或进展期间改变(PIK3CA mutational status in circulating tumor cells can change during disease recurrence or progression in patients with breast cancer).临床癌症研究(Clin Cancer Res).2014年11月15日;20(22):5823-34]。在该测定设计中,将等位基因特异性PCR的灵敏度和特异性通过不对称扩增和探针解链分析用未标记的竞争性野生型特异性阻断探针进行增强。该未标记的竞争性探针在60℃的解链分析峰能够指示在这两个外显子中的PIK3CA突变的存在。用WT等位基因特异性引物扩增的WT PIK3CA外显子9的未标记的阻断探针和DNA模板的导数解链曲线的峰,与用突变等位基因特异性引物扩增的未标记的阻断探针和突变体PIK3CA外显子9的DNA模板的解链曲线的峰在4℃左右在这两种情况下不同(结果未显示)。在所有实验中,靶向突变等位基因,该突变是通过用突变等位基因特异性引物扩增的该未标记的阻断探针和突变PIK3CA序列的导数解链进行检测的。因此,如果该峰在该较低的温度60℃下存在的话,则仅检测到突变。对于该突变体和WT两者,在通过使用突变等位基因特异性引物存在该WT的非特异性扩增的情况下,均可看见由于PCR产物且可以在较高温度下被检测的另一个峰。针对PIK3CA外显子9和20中的每者,将所有引物和探针从头经由电脑模拟被留心地设计成避免染色体22上的假基因的扩增,该假基因与PIK3CA的外显子9具有>95%同源性。
方案优化。在若干实验中针对两个外显子将该PIK3CA突变测定进行了如下广泛优化,使用作为来自癌症细胞系(MCF-7和T47D)的阳性和阴性对照gDNA样品及分离自健康供体的野生型(WT)gDNA,相对于:PC退火温度,Mg+2浓度,引物和未标记的探针浓度,PCR循环数,每个不对称PCR步骤的持续时间,不对称PCR与靶DNA量的引物比,以及解链分析条件(数据未示出)。
为了开发具有低检测极限的高度特异性方法以用于检测CTC中的PIK3CA突变,将该方案和条件最初根据最佳结果进行优化。通过使用不对称PCR、野生型阻断探针和探针解链分析增强等位基因特异性PCR扩增和检测。罕见的等位基因富集是最佳的,其中与等位基因特异性引物相比,阻断探针和通用引物是过量的。观察到随着突变等位基因特异性引物的浓度降低,特异性增加而PCR效率降低。不断增加的特异性通过野生型与PIK3CA DNA之间的ΔCq来体现。不断降低的PCR效率通过PIK3CA DNA的Cq的增加来证明。虽然,PCR效率受任一引物的浓度的降低的影响,但仅低浓度的突变等位基因特异性引物增加特异性。为了补偿较低PCR效率,典型地进行80个循环。
具有阻断探针的等位基因特异性富集受退火/延伸温度的影响。当退火/延伸温度在针对匹配的野生型等位基因的野生型阻断探针的解链温度(Tm)与错配的突变等位基因之间时,特异性是最佳的。如果退火温度低于或等于突变等位基因的Tm,则探针抑制野生型和突变等位基因两者的扩增,从而限制了灵敏度。如果退火温度高于或等于野生型等位基因的Tm,则优先阻断和PCR效率降低限制了灵敏度。
实例2
特异性研究。所开发的方法的测定特异性是以与针对患有乳腺癌的患者所遵循的完全相同的方式,首先通过分析分离自26位健康女性志愿者的gDNA来评价。该开发的方法是高度特异的,因为在任何这些样品中不存在以下情况:健康女性供体在PIK3CA的两个外显子中均具有任何突变。图2A和B描绘了所开发的PIK3CA突变测定的特异性,并且示出了在未标记阻断探针存在下在PCR后获得的特有导数解链曲线,所述未标记的阻断探针检测外显子9 1633G>A热点突变(A)和外显子20 3140A>G热点突变(B)。基线是PCR阴性对照。PIK3CA突变是通过用突变等位基因特异性引物扩增的该未标记的阻断探针和突变PIK3CA序列的导数解链进行检测的。仅当此峰在60℃下存在时检测到突变。在存在WT的非特异性扩增的情况下,在较高温度下的另一个峰是由于通过使用突变等位基因特异性引物的PCR产物并且对于该突变体和WT两者均可见。
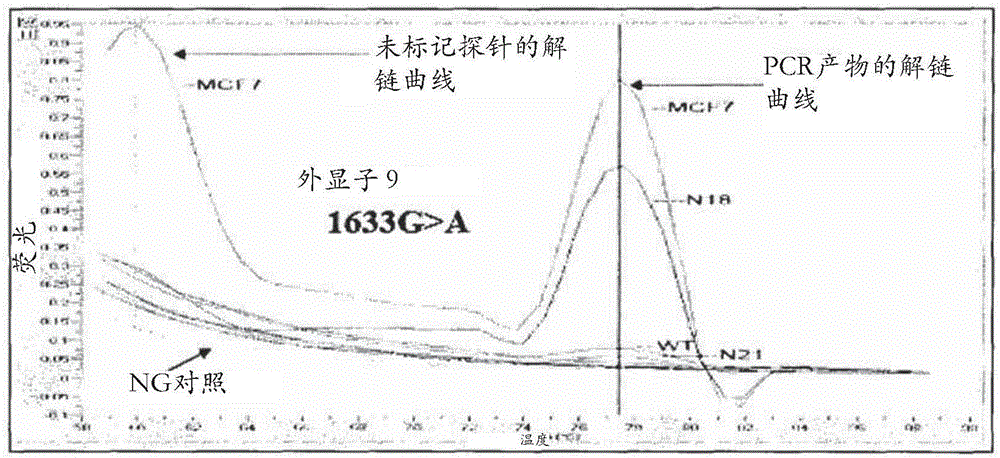
如在图2a中可以看出的,在外显子9中,将健康供体的gDNA(N18)之一通过扩增-受阻突变系统(ARMS)-PCR特异性引物进行扩增,并在77.5℃得到峰,而不是在60.0℃。这可以由以下事实解释:即使通过使用PIK3CA热点突变特异性引物,极低量的以极高浓度存在的野生型序列可以被非特异性地扩增。为了避免这种情况,使用了作为阻滞剂起关键作用的未标记探针,因为它是野生型特异的并结合在与突变-特异性引物相同的序列处。在N18的情况下,该WT序列被非特异性地扩增,这就是为什么在77.5℃检测到解链曲线的峰。然而,在针对未标记探针的解链曲线上在60.0℃没有检测到任何峰,这确切地指示我们正在寻找的特异性突变的存在。这些结果证实了该方法的100%特异性。
实例3
灵敏度研究。将所开发的方法的灵敏度进一步通过将来自细胞系的突变的gDNA与WT的gDNA以50%、25%、12.5%、2.5%、1.25%、0.5%、0.25%、0.125%和0.05%的比例进行混合来评价(参见图2C,针对外显子9 1633G>A,热点突变;以及图2D,针对外显子20 3140A>G,热点突变)。选择用于稀释的WT gDNA样品来匹配突变的gDNA数量、质量和定量循环(Cq),以使PCR偏差最小化。产生解链曲线并且评价区别细胞系稀释物的解链转变与WT样品的解链转变的能力。对于外显子9,有可能清晰地区别对应于0.05%的MCF-7细胞系的稀释物(图2C),而对于外显子20,它也可以区别0.05%的T47D细胞系稀释物的比率(图2D)。解链曲线是高度可再现的。
对于这两个外显子,PIK3CA突变是通过用突变等位基因特异性引物扩增的该未标记的阻断探针和突变PIK3CA序列的导数解链进行检测的。仅当此峰在60℃下存在时检测到突变。在存在WT的非特异性扩增的情况下,在较高温度下的另一个峰是由于通过使用突变等位基因特异性引物的PCR产物并且对于该突变体和WT两者均可见。
特别地,为了得到CTC的分子表征的可靠信息,所使用的突变检测系统的灵敏度、特异性、及稳健性是极其重要的。用于PIK3CA突变的高灵敏度方法已基于HRMA被开发[霍克斯PA(Vorkas PA),等人.通过新颖和高灵敏度的高分辨率小扩增子解链分析方法的PIK3CA热点突变扫描.分子诊断学杂志(J Mol Diagn)2010;12:697-704]。尽管事实上这种方法比传统的桑格测序更灵敏(1%),但当该测定应用于CTC样品中时,CTC中的突变的检测失败了。
实例4
患有临床确诊转移的乳腺癌患者的EpCAM阳性CTC和原发性组织(FFPE)中的PIK3CA突变的检测
作为训练组,将来自患有临床确诊转移的患者的37份外周血样品和15份原发性乳腺肿瘤组织(FFPE)针对PIK3CA突变进行了分析。在EpCAM-阳性CTC部分中,针对外显子9在6/37患者(16.2%)并且针对外显子20在4/37患者(10.8%)中检测到PIK3CA突变。总计,在10/37(27.0%)患有转移性乳腺癌的患者中检测到PIK3CA突变。在原发性乳腺肿瘤组织中,针对外显子9在9/15(60.0%)并且针对外显子20在7/15(46.7%)中检测到PIK3CA突变。存在六种以下情况,其中在相同的FFPE样品中均检测到两者的热点突变;总计,在10/15(66.7%)FFPE样品中检测到PIK3CA突变。
实例5
独立组。随后,在118位患有可动手术的乳腺癌的患者和57位患有临床确诊转移的患者的独立组中评价该测定。对这些患有乳腺癌的患者中的76位,来自原发性肿瘤的FFPE也是可获得的。
可动手术的乳腺癌患者的CTC中的PIK3CA突变的检测。
在该独立组中,来自118位患有可动手术的乳腺癌的患者的EpCAM-阳性CTC部分在原发性癌症之后已被除去,并且在辅助化疗之前已经开始进行了分析。针对外显子9 1633G>A(A)、热点突变(图3A),在3/118(2.5%)中并且针对外显子20 3140A>G、热点突变(图3b),在21/118(17.8%)中检测到PIK3CA突变。存在以下一种情况,其中两者热点突变均在CTC样本中被检测到。总计,在24/118(20.3%)的可动手术的乳腺癌患者中检测到PIK3CA突变。基线是PCR阴性对照。
患有临床确诊转移的乳腺癌患者的CTC中的PIK3CA突变的检测。
在该独立组中,将来自57位患有转移性乳腺癌的患者的EpCAM-阳性CTC部分进行了分析。自这些57位患者中,24位具有骨转移,3位在肝中,2位在脑中,9位在肺中,3位在骨和肝两者中,并且6位在肺和骨中,以及2位在多于两个不同位点中。在EpCAM-阳性CTC部分中,在外显子9 1633G>A热点突变的8/57(14.0%)中(图3C)以及在外显子20 3140A>G热点突变的12/57(21.1%)中(图3D)检测到PIK3CA突变。总计,在该组患者中的20/57(35.1%)中检测到PIK3CA突变。在图3C和3D中的解链曲线中在60℃的峰分别指示外显子9和外显子20突变的存在。在此背景下,如果在60℃未检测到峰,那么该样品被认为是所检测突变的野生型。基线是PCR阴性对照。
相应的原发性肿瘤中的PIK3CA突变的检测。
在该独立组中,将在CTC和相应的原发性肿瘤中的PIK3CA突变状态在76位患有乳腺癌的患者(32位患有临床确诊转移且44位患有可动手术的乳腺癌)中进行比较,关于这些患者相应的FFPE也是可获得的(表4)。
表4.独立组:CTC和相应的原发性肿瘤(FFPE)中的PIK3CA突变状态
外显子9,1633G>A。关于所有患者,在38/76(50%)的原发性肿瘤样品中以及在76位中的6位(7.9%)相应的EpCAM-阳性CTC部分样品中观察到外显子1633G>A热点突变。对于在其原发性肿瘤中携带这种PIK3CA热点突变的4位患者,相同的突变也在CTC中被检测。在34位患者中,此突变在原发性肿瘤中被鉴定,而在EpCAM-阳性CTC部分中未被鉴定,然而发现36位患者在原发性肿瘤和CTC两者中针对此突变均是阴性的。然而,在两种情况下,此热点突变在EpCAM-阳性CTC部分中被鉴定,而在相应的原发性肿瘤中未被鉴定。在患有可动手术的乳腺癌的患者中,1633G>A在21/44(47.7%)的FFPE中以及在1/44(2.3%)相应的CTC样本中被观察到;在其原发性肿瘤中携带这种PIK3CA热点突变的患者中没有一位在CTC中具有相同的突变。在21位患者中,此突变在原发性肿瘤中被鉴定,而在CTC中未被鉴定,然而发现22位患者在原发性肿瘤和CTC两者中针对此突变均是阴性的。然而,在一种情况下,此热点突变在CTC部分中被鉴定,而在相应的原发性肿瘤中未被鉴定。在具有转移的患者中,1633G>A在17/32(53.1%)的FFPE中以及在5/32(15.6%)相应的CTC中被观察到。对于在其原发性肿瘤中携带这种PIK3CA热点突变的4位患者,相同的突变也在CTC中被检测。在13位患者中,此突变在原发性肿瘤中被鉴定,而在CTC中未被鉴定,然而发现14位患者在原发性肿瘤和CTC两者中针对此突变均是阴性的。然而,在一种情况下,此热点突变在CTC部分中被鉴定,而在相应的原发性肿瘤中未被鉴定。
外显子20,3140A>G。关于所有患者,在13/76(17.1%)的FFPE中以及在14/76(18.4%)相应的CTC样品中观察到外显子20 3140A>G热点突变。对于在其原发性肿瘤中携带这种PIK3CA热点突变的4/13患者,相同的突变也在EpCAM-阳性CTC部分中被检测。在9位患者中,此突变在原发性肿瘤中被鉴定,而在CTC中未被鉴定,然而发现53位患者在原发性肿瘤和CTC两者中针对此突变均是阴性的。值得注意的是,在10位患者中,此热点突变仅在EpCAM-阳性CTC部分中被检测,而在相应的原发性肿瘤中未检测到。在患有可动手术的乳腺癌的患者的组中,3140A>G在5/44(11.4%)的FFPE中以及在10/44(22.7%)相应的CTC中被观察到。对于在其原发性肿瘤中携带这种PIK3CA热点突变的2/5患者,相同的突变也在CTC中被检测。在3位患者中,此突变在原发性肿瘤中被鉴定,而在CTC中未被鉴定,然而发现31位患者在原发性肿瘤和CTC两者中针对此突变均是阴性的。然而,在8位患者中,此热点突变仅在CTC中被检测,而在相应的FFPE中未检测到。在具有转移的患者的组中,3140A>G在8/32(25.0%)的原发性肿瘤样品中以及在4/32(12.5%)相应的CTC中被观察到。对于在其原发性肿瘤中携带这种PIK3CA热点突变的2/8患者,相同的突变也在CTC中被检测。在6位患者中,此突变在原发性肿瘤中被鉴定,而在CTC中未被鉴定,然而发现22位患者在原发性肿瘤和CTC两者中针对此突变均是阴性的。然而,在2位患者中,此热点突变仅在CTC中被检测,而在相应的原发性肿瘤中未检测到。
在原发性肿瘤和相应的CTC两者中的这些热点PIK3CA突变的存在之间没有一致性,因为这是在统计学上针对可动手术的和经验证转移的乳腺癌患者或针对所有患者一起来分开评价的(见表4)。
这些发现表明,CTC中的PIK3CA突变状态可以在患有乳腺癌的患者的疾病复发或进展期间变化。当在76位患者的亚组中将CTC和相应的原发性肿瘤中的PIK3CA突变状态进行比较时,可以观察到相同的突变均在少数样品中的原发性肿瘤和CTC两者中存在。在大多数患者中,该突变在原发性肿瘤中被鉴定,而在CTC中未被鉴定,然而许多患者在原发性肿瘤和CTC两者中针对此突变均是阴性的。然而,有以下12个病例,其中热点突变在CTC中被鉴定,而在相应的原发性肿瘤中未被鉴定;在两位患者中,1633G>A在CTC中被观察,但在相应的FFPE中未观察到,然而在10位患者中,3140A>G在CTC中被观察,但在相应的原发性肿瘤中未观察到。已经报道了反映CTC的异质性的类似结果。
实例6
CTC中的PIK3CA突变状态,相对于CK-19mRNA表达。
在该独立组中,针对这些患者中的157位(57位具有临床转移且100位患有早期乳腺癌;表5)进一步评价了CTC中的PIK3CA突变状态是否与CK-19mRNA表达相关。在可动手术的和转移性乳腺癌中,外周血中的CK-19mRNA-阳性细胞的检测是针对隐藏的肿瘤细胞的最灵敏的生物标志物,
表5.独立组:CTC中的PIK3CA突变与CK-19表达之间的比较
在可动手术的乳腺癌中,仅3/100的样品针对PIK3CA突变和CK-19mRNA表达两者均是阳性的(都在外显子20中)。有33/100样品针对CK-19为阳性的(在PIK3CA中没有携带突变),然而值得高度注意的是将PIK3CA热点突变在17位患者的CTC中进行鉴定,所述患者针对CK-19mRNA表达是阴性的。在患有临床确诊转移的患者中,11/57(19.3%)的样品针对PIK3CA突变和CK-19表达两者均是阳性的,6位在外显子9中并且5位在外显子20中。有14/57(24.6%)的样品针对CK-19是阳性的(在PIK3CA中没有携带这些热点突变)。值得高度注意的是将PIK3CA热点突变在9位患者的CTC中进行鉴定,所述患者针对CK-19mRNA表达是阴性的。如果未检测到PIK3CA突变,则这26个样品(17个来自患有可动手术的乳腺癌的患者并且9个来自患有临床确诊转移的患者)将会被表征为CTC阴性的,所述样品被发现针对CTC中的PIK3CA突变为阳性的,但针对CK-19mRNA表达均为阴性的。关于这些热点PIK3CA突变和CK-19mRNA表达在CTC中的存在,没有一致性(见表2)。
本研究的重要观察是针对CK-19mRNA表达为阴性的患者(携带CTC中的PIK3CA热点突变)的显著数目(17/118于可动手术的乳腺癌组中和9/57于转移验证组中)。如果未检测到PIK3CA突变,则这些患者将会被表征为CTC阴性的。CTC的分子表征已表明CTC是高度异质的。这可能,至少部分归因于上皮-间质转化以及间质-上皮转化。在此背景下,未预期所有CK-19阳性的CTC将是PIK3CA突变阳性的,或所有我们的样本(其为CK-19阴性的)将不会于CTC中携带PIK3CA突变。CK-19实时PCR测定是高特异性和灵敏的,并且它已经在先前的研究中展示了其临床意义[斯塔托波罗A(Stathopoulou A),瓦拉传科利斯I(Vlachonikolis I),马弗蒂斯D(Mavroudis D),佩拉基M(Perraki M),古劳斯西丝C(Kouroussis C),阿波斯拖拉基S(Apostolaki S),等人.患有可动手术的乳腺癌的患者的外周血中的细胞角蛋白-19-阳性细胞的分子检测:其预后意义的评价.临床肿瘤学杂志(J Clin Oncol)2002;20:3404-12.]。然而,总是有数个患者不复发(即使他们是CK-19阳性的),或会复发(即使他们是CK-19阴性的),并且甚至当使用FDA批准的CellSearchTM系统时已显示相同的情况,清楚表明一种标志物不是万能的并且不足以验证我们样本中的恶性CTC群体的存在。
实例7
CTC中的PIK3CA突变状态在患有经验证转移的患者中的临床意义。
进一步评价CTC中的PIK3CA突变状态与相对较小组的患有临床确诊转移的患者的临床结果之间的关系。通过使用患者的术后存活进行的卡普兰-迈耶(Kaplan-Meier)存活分析证明了于CTC上携带PIK3CA热点突变的患者(n=20)比那些未携带者(n=37)具有显著较短的OS(P=0.047,时序检验;参见图4)。
一个惊人的发现也是CTC中的PIK3CA突变的存在与转移性患者的较差存活相关联。在任何类型的癌症中,这是首次提出CTC中的基因突变的存在与患者存活相关。使具有PI3K信号传导途径中的激活突变的癌症患者与检测PI3K抑制剂的第一期方案匹配具有改善的应答率和存活[啼四魔薄利多AM(Tsimberidou AM),等人.在I期临床试验方案中的个体化用药:MD安德森癌症中心的倡议(Personalized medicine in a phase I clinical trials program:the MD Anderson Cancer Center initiative).临床癌症研究(Clin Cancer Res)2012;18:6373-83]。关于这一点,我们的发现(即PIK3CA突变可以排他地在CTC中存在,而不存在于原发性肿瘤中)可对患者有利,如最近在一试点前瞻性研究中所示的,在所述前瞻性研究中赫赛汀的给予基于CK-19mRNA阳性的CTC的存在[乔治奥利亚斯V(Georgoulias V),等人.曲妥珠单抗降低患有呈现耐受化疗的CK-19mRNA阳性循环肿瘤细胞的早期乳腺癌患者的临床复发发生率:一项随机II期研究的结果(Trastuzumab decreases the incidence of clinical relapses in patients with early breast cancer presenting chemotherapy-resistant CK-19mRNA-positive circulating tumor cells:results of a randomized phase II study).肿瘤学年鉴((Ann Oncol)2012;23:1744-50]。在PI3K/AKT信号传导途径中的突变(其经常解除对肿瘤细胞的管制)最近已经在乳腺癌干细胞中被确定,认为所述突变在乳腺癌的起始、进展和临床反应中具有中心作用[多诺万CA(Donovan CA),等人.乳腺癌腋淋巴结转移与干细胞突变的相关性(Correlation of breast cancer axillary lymph node metastases with stem cell mutations).美国医学会杂志外科学(JAMA Surg)2013;148:873-8]。
实例8
在具有经验证转移的乳腺癌患者的ctDNA中的PIK3CA突变的检测。
使用所开发的方法来检测分离自具有经验证转移的乳腺癌患者血浆的ctDNA中的热点PIK3CA突变。首先,从血浆中提取的所有ctDNA样品均针对其DNA质量被检查以在用来评价热点突变的外显子9的完全相同的PIK3CA基因区域中验证野生型的DNA质量、引物特异性。将ctDNA样品中的PIK3CA基因的突变状态精确地如先前所述的开发的方法进行检测(图5)。在ctDNA样品中,将PIK3CA突变于4/24(16%)针对外显子9(图5A,5B)和6/24(25%)针对外显子20(图5C)中进行检测。在图5A、5B和5C中的解链曲线中在60℃的峰分别指示外显子9和外显子20突变的存在。在此背景下,如果在60℃未检测到峰,那么该样品被认为是所检测突变的野生型。
在本研究中证明该测定可以检测分离自癌症患者的血浆的ctDNA中的PIK3CA突变。该测定灵敏度是非常低的(0.05%)并且特异性是非常高的;该测定可以在高量的野生型等位基因的存在下检测极罕见的PIK3CA突变等位基因。总的来说,在分离自血浆样品的ctDNA中,24个样品中有4个在外显子9中对1633G>A突变是阳性的并且24个样品中有6个对3140A>G突变是阳性的。循环肿瘤DNA似乎是非常有效的并且是生物标记物的有利来源以用于实时确定肿瘤的突变状态;然而,在这种情况下,需要高度灵敏的、稳健的和特异性的方法。本文证据证明该测定提供一个有用的工具,用于检测分离自癌症患者的血浆的ctDNA中的PIK3CA突变。
序列表
序列表
<110> 制药辅助有限公司(Pharmassist Ltd)
<120> 测定样品中的PIK3CA突变状态的方法
<150> US62/034,231
<151> 2014-08-07
<160> 12
<170> PatentIn版本3.5
<210> 1
<211> 125
<212> DNA
<213> 智人
<400> 1
agtaacagac tagctagaga caatgaatta agggaaaatg acaaagaaca gctcaaagca 60
atttctacac gagatcctct ctctgaaatc actaagcagg agaaagattt tctatggagt 120
cacag 125
<210> 2
<211> 619
<212> DNA
<213> 智人
<400> 2
gtttcaggag atgtgttaca aggcttatct agctattcga cagcatgcca atctcttcat 60
aaatcttttc tcaatgatgc ttggctctgg aatgccagaa ctacaatctt ttgatgacat 120
tgcatacatt cgaaagaccc tagccttaga taaaactgag caagaggctt tggagtattt 180
catgaaacaa atgaatgatg cacgtcatgg tggctggaca acaaaaatgg attggatctt 240
ccacacaatt aaacagcatg cattgaactg aaaagataac tgagaaaatg aaagctcact 300
ctggattcca cactgcactg ttaataactc tcagcaggca aagaccgatt gcataggaat 360
tgcacaatcc atgaacagca ttagaattta cagcaagaac agaaataaaa tactatataa 420
tttaaataat gtaaacgcaa acagggtttg atagcactta aactagttca tttcaaaatt 480
aagctttaga ataatgcgca atttcatgtt atgccttaag tccaaaaagg taaactttga 540
agattgtttg tatctttttt taaaaaacaa aacaaaacaa aaatccccaa aatatataga 600
aatgatggag aaggaaaaa 619
<210> 3
<211> 12
<212> DNA
<213> 人工序列
<220>
<223> 用于PCR的引物
<400> 3
tttctcctga tt 12
<210> 4
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> (引物)
<400> 4
actccataga aaatctttct cctgatt 27
<210> 5
<211> 11
<212> DNA
<213> 人工序列
<220>
<223> (探针)
<400> 5
ctgatcagtg a 11
<210> 6
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> (探针)
<400> 6
ctttctcctg atcagtgatt tcagag 26
<210> 7
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> (引物)
<400> 7
gctcaaagca atttctacac gaga 24
<210> 8
<211> 11
<212> DNA
<213> 人工序列
<220>
<223> (引物)
<400> 8
aatgatgcac g 11
<210> 9
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> (引物)
<400> 9
atgaaacaaa tgaatgatgc acg 23
<210> 10
<211> 11
<212> DNA
<213> 人工序列
<220>
<223> (探针)
<400> 10
tgcacatcat g 11
<210> 11
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> (探针)
<400> 11
gaatgatgca catcatggtg g 21
<210> 12
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> (引物)
<400> 12
tctcagttat cttttcagtt caatgc 26
- 还没有人留言评论。精彩留言会获得点赞!