用于预测体重减轻和体重维持的生物标志物的制作方法
本发明提供了生物标志物和生物标志物组合,它们可用于预测可通过向受试者应用一项或多项膳食干预而获得的体重减轻程度和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性。
背景技术:
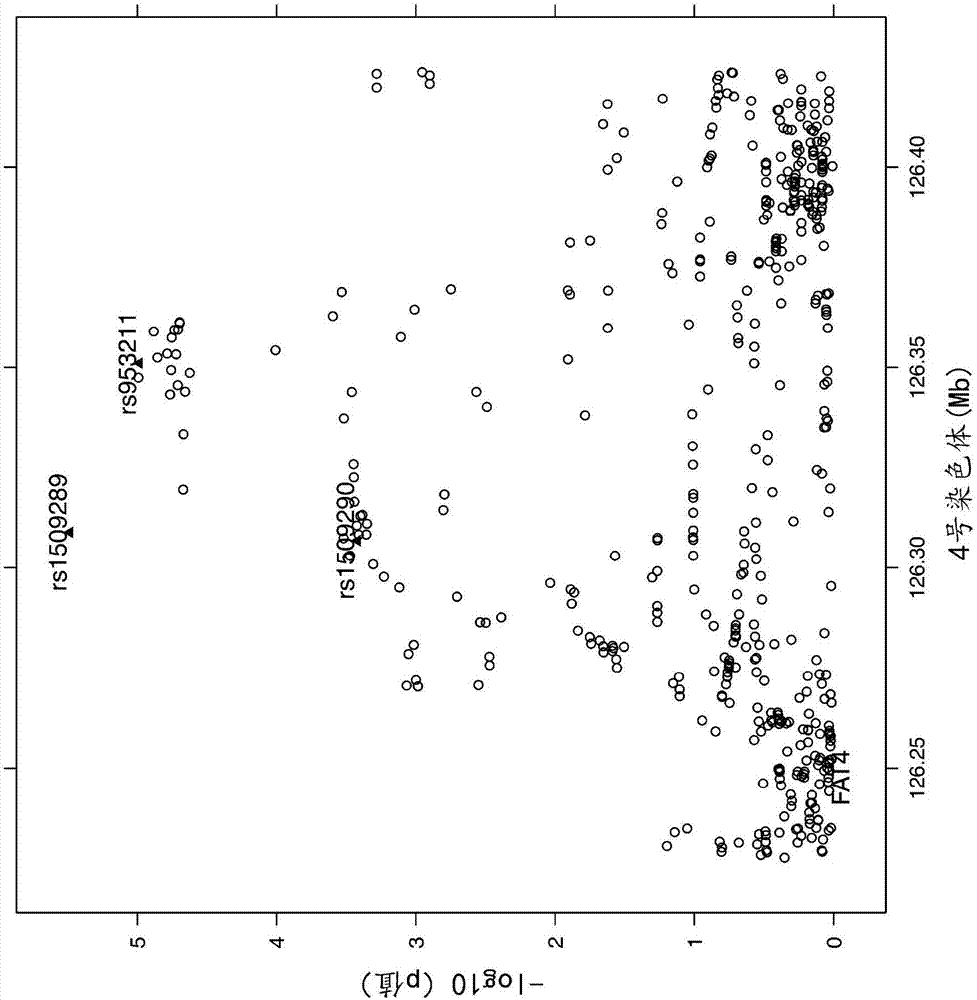
:肥胖是一种慢性代谢疾病,在世界上的许多地区已达到了流行病程度,并且是诸如2型糖尿病、心血管疾病、血脂异常和某些类型的癌症等严重并存病的主要风险因素(worldhealthorgantechrepser.2000;894:i-xii,1-253(世界卫生组织技术报告系列2000,894:i-xii,1-253))。人们早已认识到,低热量膳食干预可以非常有效地降低体重,并且这种体重减轻通常伴随肥胖相关并存病尤其是2型糖尿病的风险的改善。经验数据表明,初始体重的至少10%的体重减轻导致肥胖相关并存病的风险的显著降低(worldhealthorgantechrepser.2000;894:i-xii,1-253(世界卫生组织技术报告系列2000,894:i-xii,1-253))。然而,体重减轻能力显示出较大的受试者间波动性。已证实一定百分比的人群不能通过低热量膳食成功减轻体重(ghosh,s.etal.,obesity(silverspring),(2011)19(2):457-463(ghosh,s.等人,《肥胖》(银泉),2011年,第19卷第2期,第457-463页))。这导致不切实际的体重减轻期望,继而导致不依从、退出(drop-out)和通常不成功的膳食干预。一些研究已证实,本领域中存在用于监测体重减轻的方法,这些方法包括监测血浆中特定生物标志物的水平(例如lijnenetal.,thrombres.2012jan,129(1):74-9(lijnen等人,《血栓形成研究》,2012年1月,第129卷第1期,第74-79页);cugnoetal.,internemergmed.2012jun,7(3):237-42(cugno等人,《内科和急诊医学》,2012年6月,第7卷第3期,第237-242页);以及bladbjergetal.,brjnutr.2010dec,104(12):1824-30)(bladbjerg等人,《英国营养期刊》,2010年12月,第104卷第12期,第1824-1830页))。然而,这些方法不能提供特定受试者可获得的体重减轻程度的预测或指示。在研究生物标志物水平与体重减轻的相关性时无预测价值。使减轻的体重保持稳定对于患者而言也是一个重大挑战。已知的是,体重减轻干预之后仅一年,即会恢复减轻的体重的约三分之一(hensrud,obes.res.9suppl4,348s–353s,2001(hensrud,《肥胖研究》,第9卷增刊4,第348s–353s页,2001年))。此外,膳食引起的体重减轻会引起多种生理变化,这些生理变化会促进体重恢复(sumithranandproietto.,clin.sci.lond.engl.1979124,231–241(2013)(sumithran和proietto,《临床科学》(英国伦敦,1979年创刊),第124卷,第231-241页,2013年))。这些变化包括能量消耗、底物代谢以及参与食欲调节的激素通路方面的改变。迄今为止,我们对这些生理和分子变化的了解仍然非常有限。用于成功计划和设计膳食干预(例如,低热量膳食)的解决方案在于预测体重减轻轨迹的方法的可用性。另外,用于预测患者在体重维持计划期间成功(或未能)使其体重减轻保持稳定的测试的可用性,将有助于成功计划和设计体重管理干预。美国专利申请us2011/0124121公开了一种用于预测体重减轻是否成功的方法。所公开的方法包括:选择正在接受或考虑接受体重减轻疗法诸如胃囊带术的患者,测量患者对热量摄入的一种或多种激素响应,以及基于激素响应来预测体重减轻疗法是否成功。所测量的激素为胃肠激素,诸如胰腺激素。欧洲专利申请ep2420843公开了一种通过确定膳食周期前后血管紧张素i转化酶(ace)的水平来确定某人在有意减轻体重后将维持体重减轻的概率的方法。然而,仍然需要一种能够准确预测受试者体重减轻和体重维持程度的方法。因此,本发明的目标是提供可被容易检测且可便于预测受试者体重减轻和体重维持的生物标志物。此类生物标志物可用于预测体重减轻期间和体重维持期间的体重轨迹,并且可有助于根据患者的生物学体重减轻和体重维持能力来将患者分成适合的治疗组。原钙粘蛋白fat4也被称为钙粘蛋白家族成员14(cdhf14)或fat肿瘤抑制因子同源物4(fat4),它是一种在人类中由fat4基因编码的蛋白质。fat4与hippo信号通路相关联。hippo通路以保守信号通路的形式出现,该通路对于在果蝇和脊椎动物中适当调节器官生长是必不可少的(halder,2011,development,jan;138(1):9-22(halder,2011年1月,《发育》,第138卷第1期,第9-22页))。最近已证实,果蝇fat(ft)钙粘蛋白对调节线粒体形态和代谢具有直接作用(singetal.,2014,cell,158,1293-1308(sing等人,2014年,《细胞》,第158卷,第1293-1308页))。研究表明,ft经蛋白水解裂解所释放的可溶性片段(ftmito)会被转运到线粒体中,并且这种裂解充当协调细胞周期和代谢的开关机制。研究指出,改变ftmito的水平可允许生物体按照变化的能量要求来直接调节代谢速率。技术实现要素:本发明鉴定了这样的生物标志物,它们可用于预测通过向受试者应用一项或多项膳食干预而获得的受试者改变体重相关表型的倾向性。本发明还鉴定了这样的生物标志物,它们可用于预测在一项或多项膳食干预后受试者维持体重相关表型的倾向性。具体地讲,本发明公开了与体重相关表型变化相关的fat4基因的特异性多态性/等位基因,以及基于这些易感性改变的诊断工具和试剂盒。因此,本发明可用于预测体重管理计划(包括体重减轻计划或体重维持计划)的结果。因此,本发明在一个方面提供了一种用于评估可通过向受试者应用一项或多项膳食干预而获得的受试者体重减轻的倾向性和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性的方法,所述方法包括确定fat4基因或其调节元件中一种或多种多态性标志物的存在。本发明在一个方面提供了一种用于评估可通过向受试者应用一项或多项膳食干预而获得的受试者体重减轻的倾向性和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性的方法,所述方法包括确定受试者在选自以下的一个或多个多态性位置处的核苷酸:(i)seqidno:1(rs953211)的第101位(ii)seqidno:2(rs1509290)的第101位(iii)seqidno:3(rs1509289)的第101位以及/或者检测与所述多态性位置遗传连锁的一种或多种生物标志物。在一个实施方案中,所述一种或多种生物标志物(例如,snp)位于fat4基因或其调节元件内。在一个实施方案中,所述生物标志物距离以上所定义的多态性位置不到20、15、10、5、4、3、2、1千碱基(kb)。优选地,所述方法包括确定受试者在seqidno:1的第101位和seqidno:2的第101位处的核苷酸。在一个实施方案中,所述方法包括确定受试者在seqidno:1的第101位、seqidno:2的第101位和seqidno:3的第101位处的核苷酸。在一个实施方案中,所述方法包括确定seqidno:1的第101位处a或g、和/或seqidno:2的第101位处a或g、和/或seqidno:3的第101位处a或g的存在。在一个实施方案中,用于评估通过应用一项或多项膳食干预而获得的受试者维持体重减轻的倾向性的方法包括确定seqidno:1的第101位处a或g的存在,其中体重减轻由身体质量指数(bmi)、脂肪质量、臀围或腰围的变化表示。在另一个实施方案中,用于评估通过应用一项或多项膳食干预而获得的受试者维持体重减轻的倾向性的方法包括确定seqidno:2的第101位处a或g的存在,其中体重减轻由bmi、去脂肪质量、臀围或腰围的变化表示。在另一个实施方案中,用于评估通过应用一项或多项膳食干预而获得的受试者维持体重减轻的倾向性的方法包括确定seqidno:3的第101位处a或g的存在,其中体重减轻由bmi、脂肪质量、去脂肪质量、臀围或腰围的变化表示。在另一个实施方案中,用于评估通过应用一项或多项膳食干预而获得的受试者维持体重减轻的倾向性的方法包括确定seqidno:1的第101位处a或g的存在以及seqidno:2的第101位处a或g的存在,其中体重减轻由bmi、脂肪质量、去脂肪质量、臀围或腰围表示。在一个特别优选的实施方案中,基因型包含seqidno:1的第101位处的g和seqidno:2的第101位处的a的受试者被鉴定为在膳食干预后具有维持体重减轻的倾向性。所谓“第101位处的g”是指受试者在所述位点处具有至少一个拷贝的g,即受试者可具有基因型g/g(纯合)或g/a(杂合)。所谓“第101位处的a”是指受试者在所述位点处具有至少一个拷贝的a,即受试者可具有基因型a/a(纯合)或g/a(杂合)。优选地,用于评估受试者维持体重减轻的倾向性的方法包括确定选自以下的基因型的存在:(i)seqidno:1的第101位处的g/g(纯合)和seqidno:2的第101位处的a/a(纯合);(ii)seqidno:1的第101位处的g/g(纯合)和seqidno:2的第101位处的g/a(杂合);(iii)seqidno:1的第101位处的g/a(杂合)和seqidno:2的第101位处的g/a(杂合);以及(iv)seqidno:1的第101位处的g/a(杂合)和seqidno:2的第101位处的a/a(纯合);并且其中所述基因型的存在表明受试者在膳食干预后具有维持体重减轻的倾向性。优选地,用于评估受试者维持体重减轻的倾向性的方法包括确定选自以下的基因型的存在:(i)seqidno:1的第101位处的a/a(纯合)和seqidno:2的第101位处的a/a(纯合);(ii)seqidno:1的第101位处的a/a(纯合)和seqidno:2的第101位处的a/g(杂合);(iii)seqidno:1的第101位处的a/a(纯合)和seqidno:2的第101位处的g/g(纯合);(iv)seqidno:1的第101位处的a/g(杂合)和seqidno:2的第101位处的g/g(纯合);以及(v)seqidno:1的第101位处的g/g(纯合)和seqidno:2的第101位处的g/g(纯合);并且其中所述基因型的存在表明受试者在膳食干预后具有不会维持体重减轻的倾向性。优选地,通过测量bmi的变化来评估体重减轻的维持状况。在另一个实施方案中,用于评估可通过应用一项或多项膳食干预而获得的受试者体重减轻的倾向性的方法包括确定seqidno:1的第101位处a或g的存在,并且其中体重减轻由臀围的变化表示。在另一个实施方案中,用于评估可通过应用一项或多项膳食干预而获得的受试者体重减轻的倾向性的方法包括确定seqidno:2的第101位处a或g的存在,并且其中体重减轻由bmi或臀围的变化表示。在另一个实施方案中,用于评估可通过应用一项或多项膳食干预而获得的受试者体重减轻的倾向性的方法包括确定seqidno:3的第101位处a或g的存在,并且其中体重减轻由bmi或臀围的变化表示。在另一个实施方案中,用于评估可通过应用一项或多项膳食干预而获得的受试者体重减轻的倾向性的方法包括检测seqidno:1的第101位处a或g的存在以及seqidno:2的第101位处a或g的存在,并且其中体重减轻由bmi的变化或臀围的变化表示。在一个特别优选的实施方案中,基因型包含seqidno:1的第101位处的g和seqidno:2的第101位处的a的受试者被鉴定为具有减轻体重的倾向性。所谓“第101位处的g”是指受试者在所述位点处具有至少一个拷贝的g,即受试者可具有基因型g/g(纯合)或g/a(杂合)。所谓“第101位处的a”是指受试者在所述位点处具有至少一个拷贝的a,即受试者可具有基因型a/a(纯合)或g/a(杂合)。优选地,用于评估可通过应用一项或多项膳食干预而获得的受试者体重减轻的倾向性的方法包括确定选自以下的基因型的存在:(i)seqidno:1的第101位处的g/g(纯合)和seqidno:2的第101位处的a/a(纯合);(ii)seqidno:1的第101位处的g/g(纯合)和seqidno:2的第101位处的g/a(杂合);(iii)seqidno:1的第101位处的g/a(杂合)和seqidno:2的第101位处的g/a(杂合);以及(iv)seqidno:1的第101位处的g/a(杂合)和seqidno:2的第101位处的a/a(纯合);并且其中所述基因型的存在表明受试者在膳食干预后具有减轻体重的倾向性。优选地,用于评估可通过应用一项或多项膳食干预而获得的受试者体重减轻的倾向性的方法包括确定选自以下的基因型的存在:(i)seqidno:1的第101位处的a/a(纯合)和seqidno:2的第101位处的a/a(纯合);(ii)seqidno:1的第101位处的a/a(纯合)和seqidno:2的第101位处的a/g(杂合);(iii)seqidno:1的第101位处的a/a(纯合)和seqidno:2的第101位处的g/g(纯合);(iv)seqidno:1的第101位处的a/g(杂合)和seqidno:2的第101位处的g/g(纯合);以及(v)seqidno:1的第101位处的g/g(纯合)和seqidno:2的第101位处的g/g(纯合);并且其中所述基因型的存在表明受试者在膳食干预后具有不会减轻体重的倾向性。在本发明的另一个方面,提供了一种用于评估可通过向受试者应用一项或多项膳食干预而获得的受试者体重减轻的倾向性和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性的方法,所述方法包括确定受试者在图7中所示的一个或多个多态性位置(seqidno10至101)处的核苷酸。本发明在另一个方面提供了一种用于评估可通过应用一项或多项膳食干预而获得的受试者体重减轻的倾向性和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性的方法,所述方法包括确定fat4基因或其调节元件的序列。本文提及的膳食干预优选地为低热量膳食。膳食干预可包括向受试者施用至少一种膳食产品。低热量膳食可包含降低的脂肪消耗和/或增加的低脂肪食品消耗。仅以举例说明的方式,低脂肪食品可包括全麦面粉和面包、燕麦片、高纤维早餐谷类、全粒大米和面食、蔬菜和水果、干豆和扁豆、烤马铃薯、果干、核桃、白鱼、鲱鱼、鲭鱼、沙丁鱼、腌鱼、皮尔彻德鱼、鲑鱼和精白肉。在一个实施方案中,低热量膳食可包括约600至约1200千卡/天的热量摄入和/或可包括施用至少一种膳食产品。低热量膳食还可以包括施用最多例如约400g蔬菜/天。优选地,膳食产品是或因此,膳食可包括诸如或之类的产品。膳食可补充有三份非淀粉类蔬菜,以使得总的能量摄入为约2.5mj(600千卡/天)。膳食还可补充有每天至少2l水或其他无能量饮料。在另一个实施方案中,膳食可包括例如含46.4%碳水化合物、32.5%蛋白和20.1%脂肪、维生素、矿物质及微量元素(热量为2.1mj/天(510千卡/天))的组合物。膳食可补充有三份非淀粉类蔬菜,以使得总的能量摄入为约2.5mj(600千卡/天)。膳食还可补充有每天至少2l水或其他无能量饮料。在一个实施方案中,低热量膳食持续最多12周,例如6至12周。本文提及的方法还可包括确定受试者的一个或多个人体测量指标和/或生活方式特征。人体测量指标可选自例如性别、体重、身高、年龄和身体质量指数,生活方式特征可为例如受试者是吸烟者还是不吸烟者。根据本发明的另一个方面,提供了一种用于优化受试者的一项或多项膳食干预的方法,所述方法包括:根据本文所定义的方法评估可通过一项或多项膳食干预而获得的受试者体重减轻的倾向性和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性;以及向受试者应用一项或多项膳食干预。根据本发明的另一个方面,提供了一种用于选择对受试者的生活方式进行改变的方法,所述方法包括:(a)执行根据本文所定义的方法评估可通过一项或多项膳食干预而获得的受试者体重减轻的倾向性和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性的方法;以及(b)基于从(a)预测的可获得的体重减轻和/或预测的体重减轻维持来选择对生活方式进行合适的改变。在一个实施方案中,对生活方式进行的改变包括膳食干预,优选地为本文所定义的膳食干预。根据本发明的另一个方面,提供了一种用于减轻体重的低热量膳食,其中将该膳食产品施用给通过本文所定义的方法预测将获得体重减轻的受试者。根据本发明的另一个方面,提供了一种用作用于减轻体重的低热量膳食的一部分的膳食产品,其中将该膳食产品施用给通过本文所定义的方法预测将获得体重减轻或维持的受试者。根据本发明的另一个方面,提供了一种用作用于减轻体重的低热量膳食的一部分的膳食产品,其中将该膳食产品施用给通过本文所定义的方法预测将同时获得体重维持和体重减轻的受试者。根据本发明的另一个方面,提供了一种用于治疗肥胖或肥胖相关疾病的膳食产品,其中将该膳食产品施用给通过本文所定义的方法预测将获得体重维持和/或体重减轻的受试者。在一个实施方案中,膳食产品可包括诸如或之类的产品。在另一个实施方案中,膳食产品可包括例如含46.4%碳水化合物、32.5%蛋白和20.1%脂肪、维生素、矿物质及微量元素(热量为2.1mj/天(510千卡/天))的组合物。膳食可补充有三份非淀粉类蔬菜,以使得总的能量摄入为约2.5mj(600千卡/天)。膳食还可补充有每天至少2l水或其他无能量饮料。根据本发明的另一个方面,提供了一种能够检测fat4基因或其调节元件内的多态性位置的等位基因特异性寡核苷酸探针。根据本发明的另一个方面,提供了一种能够检测fat4基因或其调节元件内的多态性位置的等位基因特异性寡核苷酸引物。根据本发明的另一个方面,提供了一种能够检测选自以下的多态性位置的等位基因特异性寡核苷酸探针:(i)seqidno:1的第101位;(ii)seqidno:2的第101位;或者(iii)seqidno:3的第101位。根据本发明的另一个方面,提供了一种能够检测选自以下的多态性位置的等位基因特异性寡核苷酸引物:(i)seqidno:1的第101位;(ii)seqidno:2的第101位;或者(iii)seqidno:3的第101位。根据本发明的另一个方面,提供了一种包含本文所定义的等位基因特异性寡核苷酸引物和/或等位基因特异性寡核苷酸探针的诊断试剂盒。根据本发明的另一个方面,提供了一种包含以下组分的诊断试剂盒:(i)能够检测seqidno:1的第101位处g的存在的等位基因特异性寡核苷酸引物和/或等位基因特异性寡核苷酸探针,以及(ii)能够检测seqidno:2的第101位处a的存在的等位基因特异性寡核苷酸引物和/或等位基因特异性寡核苷酸探针。在一个实施方案中,该诊断试剂盒还包含能够检测seqidno:3的第101位处a的存在的等位基因特异性寡核苷酸引物和/或等位基因特异性寡核苷酸探针。在本发明的又一个方面,提供了一种计算机程序产品,所述计算机程序产品包括用于使可编程计算机根据本文所述的方法预测受试者可获得的体重减轻程度的计算机可执行指令。附图说明图1:位于fat4基因附近的snp与体重维持期间bmi的变化之间的关联。x轴示出了snp在4号染色体上的坐标,y轴示出了由等位测试得出的-log10p值。该图展示了在基因分型阵列芯片上分析的snp结果。图2:位于fat4基因附近的snp与体重维持期间bmi的变化之间的关联。x轴示出了snp在4号染色体上的坐标,y轴示出了由等位基因剂量测试得出的-log10p值。该图展示了在基因分型阵列芯片上分析的或通过填补得出基因型的snp结果。图3:rs953211(seqidno:1)、rs1509290(seqidno:2)和rs1509289(seqidno:3)的序列。多态性位置在方括号中示出,即[a/g]。图4:fat4基因(seqidno:4)的基因组序列。多态性位置rs953211、rs1509290和rs1509289被标识为加有下划线的序列内的粗体碱基。图5a、图5b和图5c:三种fat4转录物(seqidno:5、seqidno:6和seqidno:7)的序列图6a和图6b:两种fat4蛋白质序列(seqidno:8和seqidno:9)的序列。图7:位于fat4基因附近(+/-10kb)的snp。示出了九十二个p值小于5%的snp。具体实施方式预测体重减轻和体重维持本发明提供了一种用于评估可通过向受试者应用一项或多项膳食干预而获得的受试者体重减轻的倾向性和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性的方法。本发明允许在体重减轻干预之前和/或在体重维持干预之前准确预测患者的体重轨迹。所述方法可用于对受试者减轻体重和/或维持体重减轻的能力进行有根据的预测,并相应地选择或调整一项或多项膳食干预。例如,本发明可用于预测体重管理计划的结果,可用于调整体重管理计划,并且可用于在这种计划后将患者分成具有成功或不太成功特性的组。例如,所述方法可用于鉴定通过应用膳食干预而“较高”或“较低”可能性实现体重减轻的受试者。类似地,所述方法可用于鉴定在膳食干预后“较高”或“较低”可能性维持体重减轻的受试者。在膳食干预是低热量膳食的情况下,所述方法可用于帮助为受试者选择适当的膳食或者调整每日热量摄入或特定膳食持续时间,并且帮助为受试者设立切合实际的期望。具体地讲,所述方法可为技术人员提供用于评估哪些受试者将最可能受益于特定膳食干预(例如,低热量膳食)的有用手段。本发明的方法因此使得能够优化膳食干预(诸如,低热量膳食)并改变生活方式。本文所定义的体重减轻可以指诸如体重(例如,单位为千克)、身体质量指数(kgm-2)、腰臀比(例如,单位为厘米)、脂肪质量(例如,单位为千克)、臀围(例如,单位为厘米)或腰围(例如,单位为厘米)等参数的降低。体重减轻可通过以下方式计算:将膳食干预结束时上述参数中一个或多个的值从膳食干预开始时所述参数的值中减去。体重减轻的程度可表示为上述体重表型参数之一的百分比变化(例如,受试者体重(例如,单位为千克)或身体质量指数(kgm-2)的百分比变化)。例如,在膳食干预后倾向于减轻体重的受试者可能减轻其初始体重的至少10%、其初始体重的至少8%或其初始体重的至少5%。仅以举例说明的方式,受试者可能减轻其初始体重的5%至10%。在一个实施方案中,体重减轻程度为初始体重的至少10%可导致肥胖相关并存病的风险显著降低。本文所定义的体重维持可以指在膳食干预后诸如体重(例如,单位为千克)、身体质量指数(kgm-2)、腰臀比(例如,单位为厘米)、脂肪质量(例如,单位为千克)、臀围(例如,单位为厘米)或腰围(例如,单位为厘米)等参数的维持。体重维持的程度可通过以下方式来计算:确定在膳食干预结束后的一段时间内一种或多种上述参数的变化。该段时间可为例如膳食干预结束后的至少4、8、12、16、20、24、28、32、36、40、44、48或52周。体重维持的程度可表示为膳食干预结束后的一段时间内体重变化的百分比。例如,倾向于维持体重的受试者可能恢复在膳食干预期间所减轻的体重的不到50%、40%、30%、20%、10%、5%或1%。生物标志物术语“多态性”是指基因座群体中核苷酸序列不同或具有可变数量重复核苷酸单元的两种或更多种替代形式(等位基因)。多态性出现在基因的编码区(外显子)、非编码区或基因的外部(基因间隔区)。“等位基因”是可与其他形式的基因、遗传标志物或其他基因座区分开的特定形式的基因、遗传标志物或其他基因座。术语“等位基因”包括(例如但不限于)一种形式的单核苷酸多态性(snp)。个体对于二倍体细胞中的特定等位基因可为纯合的,即,两个配对染色体上的等位基因是相同的;或者对于所述等位基因可为杂合的,即,两个配对染色体上的等位基因是不相同的。术语“基因”是指执行一种功能的dna单元。通常,所述功能等同于产生一种rna或一种蛋白质。基因可包含编码区、内含子、非翻译区和控制区。如本文所用,短语“遗传标志物”是指与所关注的一个或多个基因座相关联的个体基因组的特征(例如,存在于个体基因组中的核苷酸或多核苷酸序列)。通常,遗传标志物具有多态性,并且遗传标志物的变体形式包括例如单核苷酸多态性(snp)、插入缺失(即,插入/缺失)、简单重复序列(ssr)、限制性片段长度多态性(rflp)、随机扩增多态性dna(rapd)、酶切扩增多态性序列(caps)标志物、多样性阵列技术(dart)标志物和扩增片段长度多态性(aflp)、微卫星或简单重复序列(ssr)以及许多其他示例。“单核苷酸多态性(snp)”是基因组(或物种的各个体之间共用的其他序列)中的单个核苷酸(a(腺嘌呤)、t(胸腺嘧啶)、c(胞嘧啶)或g(鸟嘌呤))在物种的各个体之间(或在个体的配对染色体之间)有所差异时出现的dna序列变化。如本文所用,“基因型”是指个体中配对(同源)染色体的单个基因座上的遗传标志物(例如,不限于snp)的两个等位基因的组合。术语“单体型”是指来自共同位于同一染色体上的不同标志物(例如,snp)的变体或等位基因。“连锁不平衡”(也称“等位基因关联”)是指两个或更多个基因座处的特定等位基因在从亲代向子代分离时,趋于以比它们在给定群体中的独立频率的预期值更大的频率一起保留在连锁群中这一现象。在一个方面,本发明提供了一种用于评估可通过向受试者应用一项或多项膳食干预而获得的受试者体重减轻的倾向性和/或在一项或多项膳食干预后受试者维持体重减轻的倾向性的方法,所述方法包括确定受试者在选自以下的一个或多个多态性位置处的核苷酸:(i)seqidno:1(rs953211)的第101位(ii)seqidno:2(rs1509290)的第101位(iii)seqidno:3(rs1509289)的第101位以及/或者检测与所述多态性位置遗传连锁的一种或多种生物标志物。优选地,所述方法包括确定seqidno:1的第101位处a或g、和/或seqidno:2的第101位处a或g、和/或seqidno:3的第101位处a或g的存在。应当注意,在本申请中,snp被称为(例如是指)seqidno:1中的位置(例如,第101位)、seqidno:2中的位置(例如,第101位)或seqidno:3中的位置(例如,第101位)。然而,当提及这些时,应当理解,本发明不限于以seqidno示出的确切序列,而是包括其变体和衍生物。相反,可以设想在相似序列中鉴定snp位置(即,技术人员将考虑的位置处的snp对应于在seqid编号中鉴定的位置)。本领域技术人员可易于对相似序列进行比对,并定位相同的snp位置。还应当注意,seqidno:1、seqidno:2或seqidno:3的互补链中与seqidno:1、seqidno:2或seqidno:3的第101位处的核苷酸进行碱基配对的核苷酸的检测,当然在要求保护的发明的范围内。在本发明的上下文中,检测fat4基因或其调节元件中生物标志物的存在包括确定在个体的fat4编码基因或其调节元件中的多态性位点处的一个或多个核苷酸的类别。类似地,检测与seqidno:1的多态性第101位、seqidno:2的第101位和seqidno:3的第101位遗传连锁的生物标志物的存在包括确定与所述位置遗传连锁的多态性位点处的一个或多个核苷酸的类别。多态性位点将为与以下有关联的位点:在受试者群体中可通过应用一项或多项膳食干预而获得的体重减轻和/或在一项或多项膳食干预后体重减轻的维持。这是指多态性位点处的特定核苷酸或核苷酸序列与所述体重减轻和/或维持相关联。对本领域技术人员来说显而易见的是,大量分析程序可用于检测本发明的一个或多个多态性位置处变体核苷酸的存在或不存在。一般来讲,等位变异的检测需要突变辨别技术、任选的扩增反应以及任选的信号生成系统。等位基因的检测可对从样品中获取的核酸进行基因分型,以鉴定标志物基因座所存在的特定等位基因。可获取足量的样品,以允许从样品中直接检测标志物等位基因。或者,可从受试者中获取较少样品,并在检测之前对核酸进行扩增。任选地,在检测标志物等位基因之前对核酸样品进行纯化(或部分纯化)。下面给出了等位基因检测方法的示例:等位基因特异性pcr等位基因特异性pcr在变异或多态性存在与否方面有所差异的靶区域之间不同。pcr扩增引物基于它们与靶序列(诸如,本文所公开的序列)的互补性来选择。引物仅结合到靶序列的某些等位基因。等位基因特异性寡核苷酸筛选方法进一步筛选方法采用等位基因特异性寡核苷酸(aso)筛选方法(例如,参见saikietal.,nature324:163-166,1986(saiki等人,《自然》,第324卷,第163-166页,1986年))。针对任何特定等位基因生成具有一个或多个碱基对错配的寡核苷酸。aso筛选方法检测靶基因组或pcr扩增的dna中的一个等位基因与另一个等位基因之间的错配,表明寡核苷酸相对于第二等位基因(即,另一个等位基因)寡核苷酸的结合力下降。寡核苷酸探针可被设计成在低严格性下将结合到等位基因的两种多态性形式,但在高严格性下将结合到它们所对应的等位基因。或者,严格性条件可被设计成在这些条件下获得基本上二元响应,即与靶基因的变体形式对应的aso将与该等位基因杂交,而不会与野生型等位基因杂交。连接酶介导的等位基因检测方法连接酶也可用于检测连接扩增反应中的点突变(诸如snp)(例如,如wuetal.,genomics4:560-569,1989(wu等人,《基因组学》,第4卷,第560-569页,1989年)中所述)。连接扩增反应(lar)采取的方法是使用连续多轮模板依赖性连接来扩增特异性dna序列(例如,如wu(出处同上)以及barany,proc.nat.acad.sci.88:189-193,1990(barany,《美国国家科学院院刊》,第88卷,第189-193页,1990年)中所述)。变性梯度凝胶电泳可通过使用变性梯度凝胶电泳来分析使用聚合酶链反应所生成的扩增产物。基于不同的序列依赖性解链性质以及dna在溶液中的电泳迁移,可鉴定不同的等位基因。dna分子在温度升高或变性的条件下解链为片段,称为解链结构域。每个解链结构域在不同的碱基特异性解链温度(tm)下协同解链。解链结构域的长度为至少20个碱基对,最多可为几百个碱基对。可使用聚丙烯酰胺凝胶电泳,来评估基于序列特异性解链结构域差异的等位基因之间的差异,如erlich,ed.,pcrtechnology,principlesandapplicationsfordnaamplification,w.h.freemanandco.,newyork(1992)(erlich编辑,《pcr技术:dna扩增的原理与应用》,纽约w.h.弗里曼出版社,1992年)的第7章中所述。一般来讲,使用位于靶区域两侧的pcr引物来扩增将通过变性梯度凝胶电泳分析的靶区域。将扩增的pcr产物施加到具有线性变性梯度的聚丙烯酰胺凝胶上,如myersetal.,meth.enzymol.155:501-527,1986(myers等人,《酶学方法》,第155卷,第501-527页,1986年)以及myersetal.,ingenomicanalysis,apracticalapproach,k.daviesed.irlpresslimited,oxford,pp.95139,1988(myers等人,《基因组分析:实用方法》,k.davies编辑,牛津irl出版社,第95-139页,1988年)中所述。电泳系统保持在略低于靶序列解链结构域的tm的温度下。在变性梯度凝胶电泳的替代方法中,靶序列最初可附接到一段gc核苷酸(称为gc夹子),如erlich(出处同上)的第7章中所述。在一个示例中,gc夹子中至少80%的核苷酸是鸟嘌呤或胞嘧啶。在另一个示例中,gc夹子的长度为至少30个碱基。这种方法特别适于tm较高的靶序列。一般来讲,通过如上所述的聚合酶链反应来扩增靶区域。寡核苷酸pcr引物中的一个引物在其5’端携带gc夹子区域,该gc夹子区域为至少30个碱基的富gc序列,在扩增期间掺入靶区域的5’端。在如上所述的变性梯度条件下使所得的扩增靶区域在电泳凝胶上进行电泳。因单个碱基变化而不同的dna片段将通过该凝胶迁移至不同位置,这可通过溴化乙锭染色观测到。温度梯度凝胶电泳温度梯度凝胶电泳(tgge)基于与变性梯度凝胶电泳相同的基本原理,不同的是变性梯度是由温度差异产生的,而不是由化学变性剂的浓度差异产生的。标准tgge采用的电泳装置具有沿着电泳路径运行的温度梯度。当样品迁移通过化学变性剂浓度均匀的凝胶时,它们会遇到不断升高的温度。tgge的一种替代方法是时相温度梯度凝胶电泳(ttge或ttgge),其使用在整块电泳凝胶中稳定升高的温度来获得相同的结果。当样品迁移通过凝胶时,整块凝胶的温度升高,从而导致样品在迁移通过凝胶时遇到不断升高的温度。样品的制备(包括掺入gc夹子的pcr扩增)以及产物的可视化与变性梯度凝胶电泳相同。单链构象多态性分析可利用单链构象多态性分析来区分靶序列或等位基因,该分析通过单链pcr产物在电泳迁移中的变化来鉴定碱基的差异,例如在oritaetal.,proc.nat.acad.sci.85:2766-2770,1989(orita等人,《美国国家科学院院刊》,第85卷,第2766-2770页,1989年)中所述。可如上所述那样生成扩增的pcr产物,并对其进行加热或以其他方式变性,从而形成单链的扩增产物。单链核酸可再折叠或形成二级结构,所述二级结构部分依赖于碱基序列。因此,单链扩增产物的电泳迁移率可检测等位基因或靶序列之间的碱基序列差异。错配的化学裂解或酶促裂解也可通过错配碱基对的差异化学裂解来检测靶序列之间的差异,例如在grompeetal.,am.j.hum.genet.48:212-222,1991(grompe等人,《美国人类遗传学杂志》,第48卷,第212-222页,1991年)中所述。在另一种方法中,可通过错配碱基对的酶促裂解来检测靶序列之间的差异,如nelsonetal.,naturegenetics4:11-18,1993(nelson等人,《自然遗传学》,第4卷,第11-18页,1993年)中所述。简而言之,来自动物或受影响的家族成员的遗传物质可用于生成无错配的异源杂种dna双链体。如本文所用,“异源杂种”意指包含来自一个动物的一条dna链和来自另一个动物的第二条dna链的dna双链,所述另一个动物的所关注的性状通常具有不同的表型。非凝胶系统其他可能的技术包括非凝胶系统,诸如taqmantm(珀金埃尔默公司(perkinelmer))。在这种系统中,将寡核苷酸pcr引物设计成位于所研究的突变的两侧,并允许对该区域进行pcr扩增。然后将第三寡核苷酸探针设计成与含有在该基因的不同等位基因之间发生变化的碱基的区域杂交。这种探针的5’和3’端均用荧光染料标记。将这些染料选择成使得当它们彼此接近时,其中一者的荧光被另一者淬灭,从而不能检测到。在taqdna聚合酶作用下从相对于探针位于模板上5’端的pcr引物延伸,导致附接到退火后的探针5’端的染料通过taqdna聚合酶的5’核酸酶活性被裂解。由此消除了淬灭效应,从而能够检测探针3’端的染料的荧光。通过如下事实辨别不同的dna序列:如果探针与模板分子不完全杂交,即存在某种形式的错配,则不会发生染料的裂解。因此,只有当寡核苷酸探针的核苷酸序列与其结合的模板分子完全互补时,淬灭才会被消除。反应混合物可含有两种不同的探针序列,每种探针针对可能存在的不同等位基因设计而成,从而允许在一个反应中检测两个等位基因。用于检测等位变异的许多当前方法由以下文献进行综述:nollauetah,clin.chem.43,1114-1120,1997(nollau等人,《临床化学》,第43卷,第1114-1120页,1997年);以及标准教科书,例如“laboratoryprotocolsformutationdetection”,ed.byu.landegren,oxforduniversitypress,1996(《突变检测的实验室规程》,u.landegren编辑,牛津大学出版社,1996年)和“pcr”,2ndeditionbynewton&graham,biosscientificpublisherslimited,1997(《pcr》,第2版,newton和graham,bios科学出版社,1997年)。样品核酸的测试样品最好是从个体获取的血液、口腔拭子、活检物或者其他体液或组织样品。应当理解,测试样品可同样为与测试样品中的序列对应的核酸序列,也就是说,可在分析等位变异之前先使用任何方便的技术(例如,pcr)来扩增样品核酸中的全部或部分区域。本发明包括对由已从个体取出的核酸样品(其可如上所定义)确定的多态性进行检测。引物/探针根据本发明的一个方面,提供了一种能够检测fat4基因(或其互补链)或其调节区域中的多态性(例如,snp)的等位基因特异性寡核苷酸引物或等位基因特异性寡核苷酸探针。根据本发明的另一个方面,提供了一种能够检测选自以下的多态性位置的等位基因特异性寡核苷酸引物或等位基因特异性寡核苷酸探针:(i)seqidno:1的第101位;(ii)seqidno:2的第101位;或者(iii)seqidno:3的第101位。应当注意,提及能够检测选自seqidno:1的第101位、seqidno:2的第101位或seqidno:3的第101位的多态性位置的等位基因特异性寡核苷酸引物或等位基因特异性寡核苷酸探针,包括能够分别检测seqidno:1的第101位、seqidno:2的第101位或seqidno:3的第101位处多态性的互补序列的等位基因特异性寡核苷酸引物或等位基因特异性寡核苷酸探针。本发明的等位基因特异性引物一般与通用引物一起用于扩增反应(诸如pcr反应)中,从而通过选择性扩增特定序列位置处的一个等位基因来辨别等位基因,例如,如arms(tm)测定法所用。本发明的等位基因特异性引物优选地为15至50个核苷酸,更优选地为约15至35个核苷酸,还更优选地为约17至30个核苷酸。能够检测seqidno:1的第101位处的多态性的等位基因特异性引物可在扩增反应(诸如pcr反应)中辨别在seqidno:1的第101位处包含碱基a的序列(或与这个基因或片段互补的序列)和在seqidno:1的第101位处包含碱基g的序列(或与这个基因或片段互补的序列或片段)。能够检测seqidno:2的第101位处的多态性的等位基因特异性引物可在扩增反应(诸如pcr反应)中辨别在seqidno:2的第101位处包含碱基a的序列(或与这个基因或片段互补的序列)和在seqidno:2的第101位处包含碱基g的序列(或与这个基因或片段互补的序列或片段)。类似地,能够检测seqidno:3的第101位处的多态性的等位基因特异性引物可在扩增反应(诸如pcr反应)中辨别在seqidno:3的第101位处包含碱基a的序列(或与这个基因或片段互补的序列)和在seqidno:3的第101位处包含碱基g的序列(或与这个基因或片段互补的序列或片段)。可使用任何方便的合成方法来制备引物。此类方法的示例可见于标准教科书,例如“protocolsforoligonucleotidesandanalogues;synthesisandproperties,”methodsinmolecularbiologyseries;volume20;ed.sudhiragrawal,humanaisbn:0-89603-247-7;1993;1stedition(“寡核苷酸和类似物规程:合成和特性”,《分子生物学方法丛书》,第20卷,sudhiragrawal编辑,humanaisbn:0-89603-247-7,1993年,第1版)。如果需要,可对引物进行标记,以便于检测。探针的设计对于具有普通技能的分子生物学家来说将是显而易见的。这些探针具有任何合适的长度,例如长度多达100个碱基、多达50个碱基、多达40个碱基以及多达30个碱基。例如,探针的长度可为10至30个碱基,优选地为18至30个碱基。探针可包含与靶序列完全互补的碱基序列。但是,如果需要,可引入一个或多个错配,只要寡核苷酸探针的辨别能力不会受到过度影响即可。本发明的探针可携带一个或多个标记,以便于检测。能够检测seqidno:1的第101位处的多态性的等位基因特异性探针可在杂交反应中辨别在seqidno:1的第101位处包含碱基a的序列(或与这个基因或片段互补的序列)和在seqidno:1的第101位处包含碱基g的序列(或与这个基因或片段互补的序列或片段)。能够检测seqidno:2的第101位处的多态性的等位基因特异性探针可在杂交反应中辨别在seqidno:2的第101位处包含碱基a的序列(或与这个基因或片段互补的序列)和在seqidno:2的第101位处包含碱基g的序列(或与这个基因或片段互补的序列或片段)。能够检测seqidno:3的第101位处的多态性的等位基因特异性探针可在杂交反应中辨别在seqidno:3的第101位处包含碱基a的序列(或与这个基因或片段互补的序列)和在seqidno:3的第101位处包含碱基g的序列(或与这个基因或片段互补的序列或片段)。本发明的引物和/或探针通常为核酸的形式(例如,dna或cdna)。或者,引物和/或探针可为核酸类似物的形式,例如pna(肽核酸)、lna(锁核酸)或bna(桥接核酸)。引物或探针可为已被lna或pna部分取代的核酸。引物设计策略随着越来越多地使用聚合酶链反应(pcr)方法,促进了许多程序的开发,以帮助设计或选择用作pcr引物的寡核苷酸。可从互联网免费获得的这类程序的四个示例是:由怀特黑德研究所(whiteheadinstitute)的markdaly和stevelincoln开发的primer(unix、vms、dos和macintosh)、由圣路易斯华盛顿大学(washingtonuniversityinst.louis)的philgreen和ladeanahiller开发的寡核苷酸选择程序(oligonucleotideselectionprogram,osp)(unix、vms、dos和macintosh)、由yoshi开发的pgen(仅用于dos),以及由威斯康星大学(universityofwisconsin)的billengels开发的amplify(仅用于macintosh)。一般来讲,这些程序通过搜寻已知重复序列元件的字节然后通过分析推定引物的长度和gc含量来优化tm,从而帮助设计pcr引物。可用的商业软件有35款,最常用的序列分析软件包正快速将引物选择程序包括在其中。设计用作引物的寡核苷酸需要选择特异性识别靶位的合适序列,然后测试该序列以消除寡核苷酸具有稳定二级结构的可能性。可使用重复体鉴定程序或rna折叠程序来鉴定序列中的反向重复。如果观察到可能的茎结构,则可将该引物的序列在任一方向上移动几个核苷酸以使所预测的二级结构最小化。对于用来扩增基因组dna的pcr引物,应将引物序列与genbank数据库中的序列进行比较,以确定是否发生任何明显的匹配。如果寡核苷酸序列存在于任何已知的dna序列中,或者更重要地存在于任何已知的重复元件中,则应更换引物序列。试剂盒根据本发明的另一个方面,提供了一种包含本发明的等位基因特异性寡核苷酸探针和/或本发明的等位基因特异性引物的诊断试剂盒。在一个实施方案中,该诊断试剂盒包含(i)能够检测seqidno:1的第101位处g的存在的等位基因特异性寡核苷酸引物和/或等位基因特异性寡核苷酸探针,以及(ii)能够检测seqidno:2的第101位处a的存在的等位基因特异性寡核苷酸引物和/或等位基因特异性寡核苷酸探针。在一个实施方案中,该诊断试剂盒包含(i)能够检测seqidno:1的第101位处g的存在的等位基因特异性寡核苷酸引物,以及(ii)能够检测seqidno:2的第101位处a的存在的等位基因特异性寡核苷酸引物。在一个实施方案中,该诊断试剂盒包含(i)能够检测seqidno:1的第101位处g的存在的等位基因特异性寡核苷酸探针,以及(ii)能够检测seqidno:2的第101位处a的存在的等位基因特异性寡核苷酸探针。该诊断试剂盒可包含适当的包装以及用于本发明的方法中的说明书。此类试剂盒还可包含适当的缓冲液和聚合酶,诸如热稳定聚合酶,例如taq聚合酶。fat4基因原钙粘蛋白fat4也被称为钙粘蛋白家族成员14(cdhf14)或fat肿瘤抑制因子同源物4(fat4),它是一种在人类中由fat4基因编码的蛋白质。fat4与hippo信号通路相关联。hippo通路以保守信号通路的形式出现,该通路对于在果蝇和脊椎动物中适当调节器官生长是必不可少的(halder,2011,development,jan;138(1):9-22(halder,2011年1月,《发育》,第138卷第1期,第9-22页))。本文提及的fat4基因可包含编码区、内含子、非翻译区和控制区。图4示出了fat4基因(seqidno:4)的基因组序列。图5a、图5b和图5c示出了三种fat4转录物(seqidno:5、seqidno:6和seqidno:7)的示例。图6a和图6b示出了从fat4基因翻译的两个蛋白质序列(seqidno:8和seqidno:9)的示例。最近已证实,果蝇fat(ft)钙粘蛋白对调节线粒体形态和代谢具有直接作用(singetal.,2014,cell,158,1293-1308(sing等人,2014年,《细胞》,第158卷,第1293-1308页))。研究表明,ft经蛋白水解裂解所释放的可溶性片段(ftmito)会被转运到线粒体中,并且这种裂解充当协调细胞周期和代谢的开关机制。研究指出,改变ftmito的水平可允许生物体按照变化的能量要求来直接调节代谢速率。应当理解,提及fat4并不限于上文所公开的确切序列,而是包括其变体和衍生物。可以设想在相似序列中鉴定多态性如snp(即,技术人员将考虑的位置处的snp对应于本文所鉴定的位置)。受试者优选地,受试者是哺乳动物,优选为人。或者,受试者可以是非人哺乳动物,包括例如马、牛、绵羊或猪。在一个实施方案中,受试者是伴侣动物,诸如狗或猫。膳食干预所谓术语“膳食干预”是指应用于受试者且引起受试者的膳食变化的外部因素。在一个实施方案中,膳食干预是低热量膳食。低热量膳食是适应动物初始体重的膳食。优选地,低热量膳食包含约600至约1500千卡/天、更优选约600至约1200千卡/天、最优选约800千卡/天的热量摄入。在一个实施方案中,低热量膳食可包括每天预定量(单位为克)的蔬菜。优选地,最多约400g蔬菜/天,例如约200g蔬菜/天。低热量膳食可包括施用至少一种膳食产品。膳食产品可以是可例如抑制受试者食欲的餐食代替产品或补充产品。膳食产品可包括食品产品、饮料、宠物食品产品、食品补充剂、营养品、食品添加剂或营养配方。在一个实施方案中,膳食可包括诸如或之类的产品。膳食可补充有三份非淀粉类蔬菜,以使得总的能量摄入为约2.5mj(600千卡/天)。膳食还可补充有每天至少2l水或其他无能量饮料。在另一个实施方案中,膳食可包括例如含46.4%碳水化合物、32.5%蛋白和20.1%脂肪、维生素、矿物质及微量元素(热量为2.1mj/天(510千卡/天))的组合物。膳食可补充有三份非淀粉类蔬菜,以使得总的能量摄入为约2.5mj(600千卡/天)。膳食还可补充有每天至少2l水或其他无能量饮料。在一个实施方案中,低热量膳食持续最多12周。优选地,低热量膳食持续6至12周、优选地8至10周,例如8周。将基因分型与人体测量指标和/或生活方式特征相结合在一个实施方案中,本发明的方法还包括将本文提及的确定基因型(例如,确定多态性标志物如snp的存在/确定fat4基因或其调节元件的序列)与受试者的一个或多个人体测量指标和/或生活方式特征相结合。如本领域已知的是,人体测量指标是受试者的测量结果。在一个实施方案中,人体测量指标选自性别、年龄(岁)、体重(公斤)、身高(厘米)和身体质量指数(kg/m-2)。其他人体测量指标也将是本领域技术人员已知的。所谓术语“生活方式特征”是指受试者做出的任何生活方式选择,这包括所有膳食摄入数据,活动指标,或得自生活方式、动机或偏好问卷的数据。在一个实施方案中,生活方式特征是受试者是吸烟者还是不吸烟者。这在本文也称为受试者的吸烟状况。受试者分类通过本发明的方法所预测的体重减轻和/或体重维持的倾向性可用于将受试者分成多个类别。可将受试者分成不同类别,表示所预测的体重减轻/维持的程度。这种分类可用于确定哪些受试者将最受益于某些干预。以此方式,可以对膳食干预和生活方式的改变进行优化,并且可以设立受试者将获得的对体重减轻的切合实际的期望。在一个实施方案中,类别包括体重减轻耐性受试者和体重减轻敏感性受试者。所谓术语“体重减轻耐性”是指低于预定值的预测体重减轻程度。在一个实施方案中,“体重减轻耐性”被定义为受试者的体重减轻百分比劣于预定值,例如预测受试者的体重减轻小于预期体重减轻的第10、第15、第20或第30个百分位数。所谓术语“体重减轻敏感性”是指预测的体重减轻程度大于预定值。在一个实施方案中,“体重减轻敏感性”被定义为受试者的体重减轻百分比优于预定的阈值。例如,预测受试者的体重减轻大于预期体重减轻的第85、第80或第75个百分位数。“预期的体重减轻”可得自已经接受了与所测试的膳食干预相同的膳食干预的一群受试者的数据。在另一个实施方案中,类别包括体重维持耐性受试者和体重维持敏感性受试者。所谓术语“体重维持耐性”是指预测的体重维持程度小于预定值。在一个实施方案中,“体重维持耐性”被定义为受试者的体重维持程度劣于预定阈值,例如预测受试者在膳食干预后维持体重减轻的程度小于受试者人群的第10、第15、第20或第30个百分位数。所谓术语“体重维持敏感性”是指预测的体重维持程度大于预定值。在一个实施方案中,“体重维持敏感性”被定义为受试者的体重维持程度优于预定阈值,例如预测受试者在膳食干预后维持体重减轻的程度大于受试者人群的第10、第15、第20或第30个百分位数。受试者群体可得自已经接受了与所测试的膳食干预相同的膳食干预的一群受试者的数据。用于选择对受试者生活方式的改变的方法在又一个方面,本发明提供用于改变受试者的生活方式的方法。受试者中生活方式的改变可以是如本文所述的任何变化,例如,膳食的变化、更多的锻炼、不同的工作和/或生活环境等。优选地,该改变是如本文所述的膳食干预。更优选地,膳食干预包括施用至少一种膳食产品。该膳食产品优选地在之前未被该受试者消费,或被该受试者以不同的方式消费。该膳食产品可以如本文所述。改变受试者的生活方式还包括指出受试者需要改变其生活方式,例如,规定更多的运动或停止吸烟。例如,如果预测受试者不能通过低热量膳食减轻体重,则改变可包括受试者生活方式中更多的运动。膳食产品的使用在一个方面,本发明提供用作用于减轻体重的低热量膳食的一部分的膳食产品。将该膳食产品施用给通过本文所述的方法预测将实现体重减轻的受试者。在另一个方面,本发明提供了一种用于治疗肥胖或肥胖相关疾病的膳食产品,其中将该膳食产品施用给通过本文所述的方法预测将获得体重减轻的受试者。肥胖相关疾病可选自糖尿病(例如,2型糖尿病)、中风、高胆固醇、心血管疾病、胰岛素耐性、冠心病、代谢综合征、高血压和脂肪肝。在又一个方面,本发明提供了膳食产品在用于减轻体重的低热量膳食中的用途,其中将该膳食产品施用给通过本文所述的方法预测将获得体重减轻的受试者。计算机程序产品本文所述的方法可实施为在通用硬件诸如一个或多个计算机处理器上运行的计算机程序。在一些实施方案中,本文所述的功能可通过诸如智能手机、平板终端或个人计算机的装置实施。在一个方面,本发明提供了一种计算机程序产品,该计算机程序产品包括用于使可编程计算机基于本发明的基因分型以及任选地来自用户的人体测量指标和/或生活方式特征来预测体重减轻/维持程度的计算机可执行指令。如本文所述,人体测量指标包括年龄、体重、身高、性别和身体质量指数及生活方式特征,包括吸烟状况。在一个特别优选的实施方案中,用户向装置输入本发明的基因分型的结果,任选地连同年龄、身体质量指数、性别和吸烟状况一起输入。然后装置对该信息进行处理并提供对用户可从膳食干预获得的体重减轻/维持程度的预测。该装置通常可以为网络上的服务器。然而,可以使用任何装置,只要其可使用处理器、中央处理单元(cpu)等处理生物标志物数据和/或人体测量及生活方式数据即可。该装置可以例如为智能手机、平板终端或个人计算机,并输出指示用户可获得的体重减轻程度的信息。本领域的技术人员将理解,在不脱离本文所公开的本发明范围的前提下,他们可以自由地组合本文所述的本发明的所有特征。现将通过非限制性实施例来描述本发明的各优选特征和实施方案。除非另外指明,本发明的实践将采用常规化学、分子生物学、微生物学、重组dna和免疫学技术,这些技术均在本领域普通技术人员的能力范围之类。此类技术在文献中有所阐述。参见例如j.sambrook,e.f.fritsch,andt.maniatis,1989,molecularcloning:alaboratorymanual,secondedition,books1-3,coldspringharborlaboratorypress(j.sambrook、e.f.fritsch和t.maniatis,1989年,《分子克隆:实验室手册》,第二版,第1-3册,冷泉港实验室出版社);ausubel,f.m.etal.(1995andperiodicsupplements;currentprotocolsinmolecularbiology,ch.9,13,and16,johnwiley&sons,newyork,n.y.)(ausubel,f.m.等人,1995年和定期增补;《分子生物学实验指南》,第9、13和16章,纽约州纽约约翰·威利父子公司);b.roe,j.crabtree,anda.kahn,1996,dnaisolationandsequencing:essentialtechniques,johnwiley&sons(b.roe,j.crabtree和a.kahn,1996年,《dna分离和测序:基本技术》,约翰·威利父子公司);j.m.polakandjameso’d.mcgee,1990,insituhybridization:principlesandpractice;oxforduniversitypress(j.m.polak和jameso’d.mcgee,1990年,《原位杂交:原理和实践》,牛津大学出版社);m.j.gait(editor),1984,oligonucleotidesynthesis:apracticalapproach,irlpress(m.j.gait编,1984年,《寡核苷酸合成:实用方法》,irl出版社);d.m.j.lilleyandj.e.dahlberg,1992,methodsofenzymology:dnastructureparta:synthesisandphysicalanalysisofdnamethodsinenzymology,academicpress(d.m.j.lilley和j.e.dahlberg,1992年,《酶学方法:dna结构a部分:dna合成及物理分析》,《酶学方法》,学术出版社);以及e.m.shevachandw.strober,1992andperiodicsupplements,currentprotocolsinimmunology,johnwiley&sons,newyork,ny(e.m.shevach和w.strober,1992年和定期增补,《免疫学实验指南》,纽约州纽约约翰·威利父子公司)。这些一般性文本中的每一个以引用的方式并入本文。实施例背景资料diogenes(膳食基因和肥胖)是一项对超重/肥胖患者(bmi介于27与45kg/m2之间)开展的干预研究(larsen,t.m.etal.n.engl.j.med.363,2102–2113(2010)(larsen,t.m.等人,《新英格兰医学杂志》,第363卷,第2102-2113页,2010年))。据我们所知,diogenes是有关体重管理的最大型且最全面的研究。患者摄取为期8周的低热量膳食(约800千卡/天)。然后按2×2因子设计,将减轻其初始体重至少8%的参与者随机分配到五种随意取用的饮食中的一种中,以防止体重在26周时间内恢复:低蛋白质且低血糖指数的膳食、低蛋白质且高血糖指数的膳食、高蛋白质且低血糖指数的膳食、高蛋白质且高血糖指数的膳食或者对照膳食。我们分析的目的是分析遗传标志物(snp)是否是体重减轻和体重维持的分类物。实施例1-材料和方法研究设计diogenes(clinicaltrials.gov标识符:nct00390637)由两个主要阶段构成:·摄取为期8周的低热量膳食(800千卡/天)的体重减轻阶段·摄取为期26周的体重维持膳食在不同时间点采集多个临床测量值和生物样品。本发明重点关注以下四个时间点:1.筛选访视2.临床干预第1天(cid1):体重减轻阶段的开始3.cid2:体重减轻阶段的结束,随机分配到一种体重维持膳食中,以及体重维持阶段的开始4.cid3:体重维持阶段的结束基因型数据使用ilumina660-quad微珠芯片进行全基因组扫描(无预先假设的方法)。illumina技术基于dna芯片,可以对每名受试者的大约660,000个单核苷酸多态性(snp)进行基因分型。snp分布在所有染色体上,用作对应基因组区域的标记标志物。该方法和实验方案的细节遵循制造商的建议(www.illumina.com)。基因型数据的质量控制如果满足以下至少一点,则从分析中排除snp:·检出率低(<95%)·违背哈迪-温伯格平衡(fdr<20%)·等位基因频率较小<0.28%如果满足以下至少一点,则从分析中排除受试者:·检出率低(<95%)·常染色体杂合性异常(fdr<1%)·对于状态同一性>0.95的一对受试者而言,排除检出率最低的受试者。·个体在遗传结构方面具有离群值:基于对来自diogenes和hapmap计划(采用ceu、yri和chb+jpt人群)的基因型执行的主成分分析(2个第一成分),得出远离diogenes聚类中心的6个标准偏差·个体在来自临床数据库的记录与由基因型数据推断的性别之间存在性别差异qc之后,最终的基因型数据集包括516,636个snp和869名受试者。关于基因组坐标本文档中使用的所有基因组坐标(snp,基因)对应于人类基因组拼接序列hg19。全基因组关联性测试使用以下线性模型进行全基因组扫描:bmi3~bmi2+snp+中心+性别+年龄+ε在该模型中,bmi2和bmi3是指分别在体重维持阶段前后(即,cid2和cid3)测量的身体质量指数(bmi)。中心是指一个diogenes中心。年龄是指在筛选访视时测量的受试者年龄。ε对应于该模型的残差项。使用genabelqtscore函数独立地测试每个snp的关联性(aulchenkoetal.,genetics177,577–585(2007)(aulchenko等人,《遗传学》,第177卷,第577-585页,2007年))。该方法首先在协变量的所关注性状上进行回归,然后使用最小二乘法测试所得残差与snp之间的关联性。该函数同时实施等位测试和基因型测试。由于等位测试比基因型测试更为强效,因此数据解读时仅考虑等位测试。当去除存在bmi或任何回归测试协变量(包括被建模的snp)的缺失值的受试者时,受试者的初始数量(n=869)变得小得多。用于516,636个模型每一者的、没有任何缺失值的受试者的平均数量为n=461。tagsnp鉴定我们使用了carlson提出的ldselect方法(carlsonetal.,am.j.hum.genet.74,106–120(2004)(carlson等人,《美国人类遗传学杂志》,第74卷,第106-120页,2004年))。按照以下设置进行标签snp分析:maf阈值为10%和r平方阈值为50%。单体型分型使用mach软件(li,etal.,genet.epidemiol.34,816–834(2010)(li等人,《遗传流行病学》,第34卷,第816–834页,2010年))并按照以下参数对基因型数据进行分型:--轮数为50--状态数为200--分型。单体型关联性测试在用mach进行分型之后,使用以下模型测试体重维持期间bmi的变化与所有单体型区块之间的关联性:bmi3~bmi2+中心+性别+年龄+aac+aat+agc+gac+gat+ggc+ε中心是指一个diogenes中心。年龄是指在筛选访视时测量的受试者年龄。ε对应于该模型的残差项。同时测试了加性(单体型的数量:0、1或2)和显性(不存在/存在)遗传模型。然后对拟合模型进行后向选择(r中的stepaic函数(rdevelopmentcoreteam(2013).r:alanguageandenvironmentforstatisticalcomputing.rfoundationforstatisticalcomputing,vienna,austria.isbn3-900051-07-0,urlhttp://www.r-project.org(rfoundationforstatisticalcomputing,vienna,austria(r核心开发小组,2013年,r:用于统计计算的语言和环境,r统计计算基金会,奥地利维也纳,isbn3-900051-07-0,urlhttp://www.r-project.org)))。其他线性模型使用线性模型测试在所关注阶段(体重维持或体重减轻阶段)内给定性状的变化。这些模型用于以若干体重相关表型测试单个snp或上位性效应(两个snp之间的相互作用)。这些模型可按照wilkinson-roger记法书写如下:时间(i)时的性状~时间(i-1)时的性状+遗传效应+中心+性别+年龄+ε性状可以是任意数量性状,如bmi、腰围或臀围、去脂肪质量或脂肪重量。时间点(i-1)和(i)是指给定阶段(例如,体重维持或体重减轻阶段)之前和之后。这种建模允许在时间(i)时预测性状,同时在时间(i-1)时调节基线值。中心是指一个diogenes中心。年龄是指在筛选访视时测量的受试者年龄。ε对应于该模型的残差项。遗传效应可以是单个snp或两个snp(snp1和snp2)之间的相互作用。对于单个snp效应而言,使用基因型模型。当测试snp相互作用时,snp1和snp2各自针对给定等位基因的存在/不存在进行编码。ε项对应于该模型的残差。使用iii型方差分析来测试该模型中每项的显著性(fox&weisberg,anrcompaniontoappliedregression.(sage,2011)(fox和weisberg,《r应用回归指南》,赛吉出版公司,2011年),参见http://socserv.socsci.mcmaster.ca/jfox/books/companion)。对bmi随时间推移的变化进行二分体重维持研究中的一个经常性挑战是限定可将患者分类为良好和不良体重维持者组的阈值。通常使用两个最极端的患者组(例如,具有低于第10个百分位数或高于第90个百分位数的响应的患者)进行这种分类。然而,有关该使用哪个百分位数的决策完全是任意的。无法排除可能根据所选择的截止值得出不同结论的风险(在进行分析之前)。不同于作出这种任意决策,我们决定使用如下所述的若干截止值。首先,我们将体重维持期间bmi的变化表示为:其中bmi2和bmi3代表体重维持阶段前后(即,cid2和cid3)的bmi。该差值(δ)对应于相对于基线bmi(体重维持之前的bmi)而言体重维持期间bmi变化的百分比。δ的解读如下:·δ<0意味着bmi2<bmi3,因此患者恢复体重(即,不良体重维持特性)·δ>0意味着bmi2>bmi3,因此患者继续减轻体重(即,良好体重维持特性)接下来,我们计算二分成“良好”和“不良”体重维持者特性组的δ。这种二分法可通过选择具有最极端δ值的患者进行。这通过选择高于或低于给定百分位数的δ来实现。例如,我们使用第10和第90个百分位数限定了以下组:·“不良体重维持者”,即δ低于第10个百分位数的患者·“良好体重维持者”,即δ高于第90个百分位数的患者在该实施例中,δ介于第10与第90个百分位数之间的受试者不包括在分析中。这些受试者可指这样的受试者,他们具有“中间特性”但无法决定应将他们分类到一个组还是另一个组中。为了避免因选择任意百分位数阈值而带来的偏差,我们分析了来自以下若干分类方案的数据:·δ低于第10个百分位数或高于第90个百分位数的患者·δ低于第25个百分位数或高于第75个百分位数的患者·δ低于第33个百分位数或高于第66个百分位数的患者·δ低于第50个百分位数或高于第50个百分位数的患者应当注意,前三种方案放弃了具有中间特性(例如,δ介于第10与第90个百分位数之间)的患者,因此仅使用该数据的子集,而最后一种方案将所有患者分类为两个不同组。因此仅使用了该数据的有限子集这一事实对使用前三种方案进行的分类提出了挑战。相比之下,第四种方案使用所有可用数据,但这两个组均具有高异质性,并且由于未放弃具有“中间特性”的患者,这两个组之间的区别可能不清晰。因此预期最后一种分类方案是最复杂的情况。使用下式对体重减轻阶段期间的bmi应用类似的二分法:在该式中,bmi1和bmi2分别对应于体重减轻阶段前后(即,cid1和cid2)。分类分析在将bmi二分成“不良”和“良好”体重维持者组之后,我们用如下简单列联表评估了分类性能:不良体重维持者较差体重维持者保护基因型ab非保护基因型cd其中a、b、c和d对应于来自给定类别的患者的数量。根据该表,可以计算分类的精度:然后用二项检验得出分类精度的九十五置信区间(95%ci)(r中的binom.test函数(rdevelopmentcoreteam(2013).r:alanguageandenvironmentforstatisticalcomputing.rfoundationforstatisticalcomputing,vienna,austria.isbn3-900051-07-0,urlhttp://www.r-project.org(rfoundationforstatisticalcomputing,vienna,austria(r核心开发小组,2013年,r:用于统计计算的语言和环境,r统计计算基金会,奥地利维也纳,isbn3-900051-07-0,urlhttp://www.r-project.org)))。还进行了单侧二项检验,以检验分类精度是否好于“无信息率”,“无信息率”被视为数据中的最大类别百分比(参见caret软件包中的confusionmatrix函数(kuhnj.stat.softw.28,1–26(2008)(kuhn,《统计软件杂志》,第28卷,第1-26页,2008年)))。我们旨在通过非保护基因型来鉴定具有低δ的受试者(恢复体重的患者)。相反,我们旨在利用保护基因型来鉴定具有高δ的受试者(恢复较少体重或甚至还减轻体重的患者)。保护和非保护基因型定义如下:对体重减轻阶段期间的bmi变化应用相同方法。对填补后的snp进行分析使用填补获得来自位于fat4附近的其他snp的基因型。使用mach软件(lietal.,genet.epidemiol.34,816–834(2010)(li等人,《遗传流行病学》,第34卷,第816-834页,2010年))并使用来自千人基因组计划(consortium,t.1000g.p.nature491,56–65(2012)(千人基因组计划协作组,《自然》,第491卷,第56-65页,2012年))(ii期)的欧洲血统受试者作为参考数据,以进行填补。利用mach软件按照建议的步骤进行填补。使用probabel(aulchenko,etal.,bmcbioinformatics11,134(2010)(aulchenko等人,《bmc生物信息学》,第11卷,第134页,2010年))并使用以下模型来进行等位基因剂量值分析:bmi3~bmi2+中心+性别+年龄+snp+εbmi3和bmi2分别指在cid3和cid2时测得的bmi。中心是指一个diogenes中心。年龄是指在筛选访视时测量的受试者年龄。ε对应于该模型的残差项。snp项对应于填补后的等位基因剂量值。实施例2-详细结果体重维持的gwa扫描根据全基因组分析,发现位于4号染色体上的snp与体重维持阶段期间的bmi轨迹有着显著关联性。具体地讲,以下两个snp:rs953211和rs1509289(表1)(均是fat4基因中的内含子变体)在全基因组列表内以排在最前面的snp出现。由于这两个snp仅处于中度连锁不平衡(ld)(r平方=50.61%),并且考虑到存在关联性p值小于5%(图1)的其他邻近snp,我们搜索了所观察到的关联性是否可归因于单体型。体重维持的单体型检验为了限制用于单体型分型的snp列表,进行了tagsnp搜索。该搜索将以下三个snp:rs953211、rs1509290和rs13136889鉴定为其他snp的最佳标签snp,所述其他snp仅与体重维持期间的bmi存在很小关联性(p值<5%,参见图1)。可使用这三个snp定义六个不同的单体型:aat、gac、ggc、aac、gat和agc(每个单体型中的snp顺序为rs953211、rs1509290和rs13136889)。接下来,测试这些单体型与体重维持期间的bmi变化的关联性。考虑了显性遗传模型和加性遗传模型。结果在表2-5中示出。这些分析将gac和gat单体型的存在鉴定为与体重维持干预后更有利的bmi有着显著关联性。具体地讲,与不携带任何这些单体型的患者相比,携带至少一个拷贝的任一单体型的患者不太容易恢复体重。使用单变量法(秩和检验)并利用plink软件(purcell,s.etal.plink:atoolsetforwhole-genomeassociationandpopulation-basedlinkageanalyses.am.j.hum.genet.81,559-575(2007)(purcell,s.等人,plink:一种用于全基因组关联分析和基于群体的连锁分析的工具集,《美国人类遗传学杂志》,第81卷,第559-575页,2007年))再现这些结果。由于在gac和gat单体型的情况下均观察到有利的体重维持,第三snp似乎贡献不大,因此保护单体型可简化为rs953211-g和rs1509290-a。相反,该结果也暗示rs953211-a和rs1509290-g单体型为不利的单体型。体重维持的上位性测试鉴于这些单体型结果,我们定义了以下基因型组:·rs953211-g携带者和rs1509290-a携带者·rs953211-g携带者和rs1509290-g/g·rs953211-a/a和rs1509290-a携带者·rs953211-a/a和rs1509290-g/g发现如以上组定义的rs953211和rs1509290之间的相互作用与体重维持期间的bmi变化有着显著关联性(p=7.96e-05,有关该方法的详细内容,参见“其他线性模型”)。由线性模型及随后的方差分析得出的全部结果分别在表6和表7中示出。这些结果表明,rs953211-g携带者和rs1509290-a携带者具有比其余三个基因型组显著更好的bmi特性。为了有利于遗传诊断,这四个基因型组可简化为“rs953211-g携带者和rs1509290-a携带者”以及汇集在一起的其余三个组的受试者。这种遗传二分法也与体重维持阶段期间的bmi轨迹有着显著关联性。由线性模型得出的结果在表8中示出。该表还包括该模型每项的回归系数的95%置信区间(ci)。这些95%ci通过进行线性模型的自举(davison.&hinkleybootstrapmethodsandtheirapplications.(cambridgeuniversitypress,1997)(davison和hinkley,《自举法及其应用》,剑桥大学出版社,1997年),参见http://statwww.epfl.ch/davison/bma)而获得(n=1,000重采样并使用r中的boot软件包(canty,a.&ripley,b.d.boot:bootstrapr(s-plus)functions.(2014)(canty,a.和ripley,b.d.,boot:自举r(s-plus)函数,2014年)))。这些系数的解读如下:当调节协变量诸如年龄、性别、参与中心及体重维持前的bmi时;“rs953211-g携带者和rs1509290-a携带者”对体重维持后的bmi的效应为-0.76kg/m2。换句话讲,对于作为“rs953211-g携带者和rs1509290-a携带者”的受试者而言,与来自任何其他基因型组合的受试者相比,其对体重维持阶段后的bmi的效应为-0.76kg/m2(95%ci=[-1.1,-0.43])。看待这些结果的甚至更简单的方法是仅考虑在不调节协变量(年龄、性别等…)的情况下体重维持前后的bmi变化。在未进行任何遗传分类的情况下(即,未获知受试者的基因型),bmi的中值变化为+0.38kg/m2(表9)。这意味着受试者趋于恢复体重。当可获知rs953211和rs1509290的基因型时;来自这三个基因型组的受试者:“rs953211a/a;rs1509290a携带者”;“rs953211a/a;rs1509290g/g”和“rs953211g携带者;rs1509290g/g”都恢复体重(中值bmi变化分别为+0.57、+0.63和+0.68kg/m2)。相比之下,作为“rs953211g携带者;rs1509290a携带者”的受试者往往不会恢复体重(中值bmi变化=0)。因此这两种遗传标志物的这种组合能够:·鉴定具有比其他三组更好的体重维持的一组患者(“rs953211g携带者;rs1509290a携带者”)·证实获知这两种标志物的基因型相较于未获知基因型可带来附加信息。这四个基因型组各自的bmi变化不同于全球人群的平均bmi变化。体重减轻的上位性测试发现snp的相同组合与体重减轻阶段期间的bmi变化相关联(p=0.0243,有关该方法的详细内容,参见“其他线性模型”)。由线性模型及其随后的方差分析得出的结果分别在表10和表11中示出。与体重维持分析一样,将这两个snp的组合二分成“rs953211-g携带者和rs1509290-a携带者”以及汇集在一起的其余所有受试者。对体重减轻阶段期间的bmi变化进行建模的线性模型所得出的结果在表12中示出。该表显示,与具有其他基因型组合的受试者相比,受试者“rs953211g携带者;rs1509290a携带者”具有更强的体重减轻(bmi变化=-0.20kg/m2,95%ci[-0.34,-0.06])。通过计算体重减轻阶段前后的bmi中值变化,可以观察到“rs953211g携带者;rs1509290a携带者”比其他遗传组减轻更多体重(中值bmi变化=-3.69kg/m2)(参见表13)。基线bmi的上位性测试未在由rs953211和rs1509290定义的不同基因型组之间发现基线(cid1)时bmi的差异(秩和p=0.93)。这表明该标志物组合对干预后的体重(例如,体重减轻或体重维持)具有预测性,但没有预后性。fat4snp与体重相关表型之间的关联性由于fat4snp与bmi变化相关联,因此我们测试了相同snp是否也与其他体重相关表型(诸如,脂肪质量、去脂肪质量、臀围和腰围的变化)相关联(有关该方法的详细内容,参见“其他线性模型”)。实际上,这些snp:rs1509289、rs1509290、rs953211;不论单独还是相结合,都与体重维持期间的体重相关表型变化有着显著关联性(表14)。在体重减轻阶段期间,发现若干snp(rs1509289、rs1509290以及rs953211与rs1509290之间的相互作用)与bmi和臀围的变化有着显著关联性(表15)。这些分析证实,fat4snp与体重维持和体重减轻阶段的bmi和若干其他体重相关表型相关联。使用fat4snp作为二分的bmi变化的分类物为了估计在预测体重减轻或体重维持干预的结果方面的性能,我们计算了bmi变化的百分比并将该值二分为“良好”和“不良”特性组(有关方法的详细内容,参见“对bmi随时间推移的变化进行二分”一节)。我们还将我们的遗传标志物的基因型二分为预期将鉴定具有“良好”或“不良”特性的受试者的组合(参见“分类分析”)。虽然分析二分的值会造成统计功效的明显损失,但这种简单方案给出了有关预测性能的非常保守的估计值。我们对体重维持和体重减轻阶段期间的bmi进行了此类分析。另外,为了避免因选择对bmi进行二分的截止值而引入偏差,我们利用若干截止值重复了该分析。不同遗传标志物的预测性能在表16和表17中示出。这两个表分别给出了体重维持或体重减轻干预之后的bmi结果。这两个表包括处于其95%置信区间的比值比,以及根据对列联表(由该分类得出)进行的费希尔精确检验得出的p值。这些值评估分类物(基因型)与真实结果(二分的bmi变化百分比)之间的关联性。p值低于5%表示分类物与结果之间存在显著关联性。这两个表还包括性能度量,诸如分类精度(即,正确分类受试者的分率)以及精度的95%置信区间。随机分类物(例如,抛硬币来预测结果)将具有50%的精度。当最低置信区间高于50%时,这意味着该分类物显著优于随机分类物。评价精度是否显著优于随机分类物的另一种度量称为精度的p值。该单侧p值(用二项检验得出)评估所观察到的精度是否优于无信息率(kuhn,j.stat.softw.28,1–26(2008)(kuhn,《统计软件杂志》,第28卷,第1-26页,2008年))。这些分析表明,fat4遗传标志物(rs1509289、rs150929和rs953211)不论独立使用还是结合使用,在预测体重维持阶段之后的bmi结果时都具有比随机分类物显著更高的精度(所有精度p值均<5%,参见表16)。就体重减轻阶段之后的bmi结果而言,rs953211的分类精度不优于随机分类物(精度p值>5%,参见表17)。这是预料之中的,因为rs953211与体重减轻期间的bmi变化不存在显著关联性。对于另外两个snp(rs1509289和rs150929)及rs953211与rs1509290的组合而言,大多数二分方案的分类精度显著优于随机分类物(表17)。因此,fat4标志物不论单独使用还是结合使用,都可用作体重减轻或体重维持阶段后的体重结果的分类物。寻找其他snp作为潜在更佳的分类物只要已经鉴定一些候选区,就可直截了当地细调关联性信号。研究人员已经撰写了大量综述,说明细调关联性信号的方法(mccarthy&hirschhornhum.mol.genet.17,r156–r165(2008)(mccarthy和hirschhorn,《人类分子遗传学》,第17卷,第r156–r165页,2008年);ioannidisetal.,nat.rev.genet.10,318–329(2009)(ioannidis等人,《自然综述:遗传学》,第10卷,第318-329页,2009年);以及mccarthyetal.nat.rev.genet.9,356-369(2008)(kuhn,《统计软件杂志》,第28卷,第1-26页,2008年))。下文概括了一些策略。基因重测序和其他基因分型随着新一代测序(ngs)的出现,现在可对大规模组群的外显子组或全基因组进行测序。按照供应商的实验方案并使用学术界近期的分析流程(mckennaetal.genomeres.20,1297–1303(2010)(mckenna等人,《基因组研究》,第20卷,第1297-1303页,2010年)),可鉴定未用传统snp芯片分析的其他snp。可以使用宽泛范围的技术对其他标志物进行基因分型。这包括但不限于测序(外显子组测序、全基因组测序、靶向测序、rna测序、焦磷酸测序)、基因分型检测(高密度snp芯片、定制snp芯片、采用taqman的靶向基因分型(富鲁达公司(fluidigm)))、基于pcr的检测、质谱基因分型技术等。一旦对其他标志物进行基因分型,就可直截了当地测试这些标志物各自与性状(例如,bmi变化)的关联性并鉴定有因果关系的标志物。特别是通过对位于我们所鉴定的snp附近(例如,+/-100kb)的其他标志物进行基因分型并重复上述相同分析,可鉴定可用于预测体重减轻或体重维持阶段的体重相关表型变化的另外标志物或标志物的组合。使用填补后的snp还可使用填补技术来获得其他标志物(marchini&howie,nat.rev.genet.11,499–511(2010)(marchini和howie,《自然综述:遗传学》,第11卷,第499-511页,2010年))。由于可获得公共参考数据集(consortium,t.i.h.3.,nature467,52–58(2010)(国际单体型图计划3协作组,《自然》,第467卷,第52-58页,2010年);consortium,t.1000g.p.nature491,56–65(2012)(千人基因组计划协作组,《自然》,第491卷,第56-65页,2012年);gibbsetal.,nature426,789–796(2003)(gibbs等人,《自然》,第426卷,第789-796页,2003年);theinternationalhapmapconsortium.ahaplotypemapofthehumangenome.nature437,1299–1320(2005)(国际单体型图计划协作组,人类基因组单体型图,《自然》,第437卷,第1299-1320页,2005年))以及沿用已久的填补工具(lietal.,genet.epidemiol.34,816–834(2010)(li等人,《遗传流行病学》,第34卷,第816-834页,2010年);marchini&howie,nat.rev.genet.11,499–511(2010)(marchini和howie,《自然综述:遗传学》,第11卷,第499-511页,2010年)),该策略常用于全基因组关联性研究中,以增加待测试标志物的数量。将这种策略应用于我们的数据之后,获得了位于fat4基因附近(+/-10kb)的475个snp的基因型数据(参见“对填补后的snp进行分析”一节)。随后,测试这些snp与体重维持期间的bmi变化的关联性。结果在图2中示出。九十二个snp的p值小于5%,表明位于fat4附近的其他遗传变体与体重管理相关联(参见图7中的列表)。但是与已检测的snp(图2)相比,这些snp均没有更强的关联性(p值更低)。这表明,虽然这些填补后的snp可充当rs953211、rs1509290或rs1509289的代理snp,但它们不太可能具有显著更好的预测价值。元分析元分析旨在将来自多个研究的结果组合,使得可在这些研究之中找到一致的模式。具体地讲,gwa研究的元分析旨在鉴定可能在分析单个研究时缺失的与所关注性状相关联的snp。用于执行遗传元分析的统计方法沿用已久(ioannidisetal.,nat.rev.genet.10,318–329(2009)(ioannidis等人,《自然综述:遗传学》,第10卷,第318-329页,2009年);evangelou&ioannidisnat.rev.genet.14,379–389(2013)(evangelou和ioannidis,《自然综述:遗传学》,第14卷,第379-389页,2013年))并且常用于大型协作组中(yangetal.nat.genet.44,369–375,s1–3(2012)(yang等人,《自然遗传学》,第44卷,第369-375页,第s1-3页,2012年);diabetesgeneticsreplicationandmeta-analysis(diagram)consortiumetal.nat.genet.46,234–244(2014)(糖尿病遗传学重复性和元分析(diagram)协作组等,《自然遗传学》,第46卷,第234-244页,2014年))以鉴定其他标志物。通过组合来自多项研究的结果并着重研究位于fat4基因之内和附近(例如,+/-100kb)的snp,可以找到其他snp。多因子降维分析另一种方法是一旦检测到所关注的区域,就使用多因子降维法筛选所有可能的2-snp或甚至更高阶的snp相互作用(例如,3-snp相互作用)(mooreadv.genet.72,101–116(2010)(moore,《遗传学进展》,第72卷,第101-116页,2010年);panetal.,methodsmol.biol.cliftonnj1019,465–477(2013)(pan等人,《分子生物学方法》,新泽西州克利夫顿,第1019卷,第465-477页,2013年))。可使用多因子降维法(mdr)在病例对照研究中测试snp-snp相互作用。mdr是非参数、无模型方法,旨在鉴定在预测二元分类时具有最佳精度的snp组合。当使用mdr时,建议通过交叉验证对训练数据集和验证数据集进行精度评价。这些精度分别称为训练和验证数据集的分类精度和预测精度(有关更多详情,参见motsinger和ritchie的综述(hum.genomics2,318–328(2006)(《人类基因组学》,第2卷,第318-328页,2006年)))。还可评价交叉验证一致性。即,当对数据进行若干次随机分割而划分为训练和验证集时,有多少次可鉴定到相同最佳分类物(snp的组合)。我们将此技术应用于位于fat4基因内(+/-10kb)的snp,并旨在鉴定最佳snp或两个snp的最佳组合,以预测体重维持或体重减轻期间的二分的bmi变化(参见章节:对bmi随时间推移的变化进行二分)。对于体重维持期间或体重减轻期间的bmi变化而言,结果分别在表18和表19中示出。这些分析基本上得出rs1509289、rs953211、rs1509290snp为bmi变化的最佳分类物。这符合我们此前的分析,说明这些是用于预测体重减轻/维持的优选标志物。实施例3-优选实施方案的汇总fat4snp及体重维持期间的bmi变化我们的各种分析证实,位于fat4附近的snp与体重维持阶段期间的bmi变化相关联。这些结果可概括如下:·患者趋于在体重维持阶段恢复体重。可在不考虑任何snp的基因型的情况下得出这一观察结果。·当技术人员获知fat4snp处(在我们的优选实施方案中,为rs953211和rs1509290snp处)的基因型时,可作出以下预测:ο作为“rs953211g携带者;rs1509290a携带者”的患者趋于具有bmi的中性变化(bmi的中值变化接近0)ο来自任何其他基因型组的患者趋于比“rs953211g携带者;rs1509290a携带者”恢复显著更多的体重;并且也趋于恢复比人群(即,在不考虑患者基因型的情况下汇集在一起的所有患者)中的平均(或中值)变化更多的体重。因此,当获知fat4snp的基因型时,可作出以下预测:fat4snp及体重减轻期间的bmi变化我们的分析还发现,预测体重维持结果的相同fat4snp也可用于预测体重减轻结果。结果可如下使用:组别体重减轻后的预测结果总人群(未获知基因型)减轻体重“rs953211a/a;rs1509290a携带者”减轻体重“rs953211a/a;rs1509290g/g”减轻体重“rs953211g携带者;rs1509290a携带者”比所有其他组减轻更多体重“rs953211g携带者;rs1509290g/g”减轻体重fat4snp及体重维持期间的其他体重相关表型变化fat4snp与体重维持期间其他体重相关表型(诸如,臀围和腰围、脂肪质量和去脂肪质量)的变化存在关联性。按基因型组分类的这些表型的中值变化在表20中示出。这些结果可如下使用:fat4snp及体重减轻期间的其他体重相关表型变化fat4snp与体重维持期间的臀围变化存在显著关联性。按基因型组分类的臀围和其他体重相关表型的中值变化在表21中示出。臀围的结果可概括如下:当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1