一种大腹园蛛包卵丝蛋白全长基因及其制备方法与流程
本发明属于蜘蛛丝蛋白领域,特别涉及一种大腹园蛛包卵丝蛋白全长基因及其制备方法。
背景技术:
蜘蛛丝是一种天然的蛋白丝纤维,具有优异的机械性能,如强度高,质地轻,弹性好,耐高低温以及良好的生物学相容性,其各项品质都优于蚕丝以及现阶段人工合成纤维。由于其具有巨大的潜在应用价值,因此在未来可用于航天航空,军事以及生物医学领域。但直到目前,蛛丝的大规模生产及仿生应用仍面临一些挑战,主要在于:1.蜘蛛是肉食动物,独居,无法像蚕那样大规模养殖;2.天然蜘蛛丝的组分及其成丝机理现在还不清楚;3.通过基因工程的方法在异源宿主中表达蛛丝蛋白被认为是目前为止最有潜力的方法。但同样面临着很多困难,由于蛛丝蛋白基因大小通常为10kb以上,并且中央有高度重复的序列,因此只有少数几种完整的蛛丝蛋白基因被获得。其次,由于重复区富含甘氨酸和丙氨酸,以及存在密码子使用偏好性问题,因此通过基因重组所生产出来的蛛丝蛋白在大小,产量以及性能上都不及天然蛛丝蛋白。
技术实现要素:
本发明所要解决的技术问题是提供一种大腹园蛛包卵丝蛋白全长基因及其制备方法,该基因包含有完整的NT端,重复区以及完整的CT端,用于编码大腹园蛛包卵丝蛋白;该丝纤维在力学性能以及生物相容性上都优于蚕丝以及人造纤维,而且对环境无污染,为在基因工程上大规模生产蛛蛋白奠定了基因基础。
本发明的一种大腹园蛛包卵丝蛋白全长基因,所述基因全长大小为5763bp,共编码1921个氨基酸,具体序列如SEQ ID NO.1所示,蛋白分子量为180.0kDa。
所述全长基因中NT端大小为618bp,编码206个氨基酸,具体序列如SEQ ID NO.2所示。
重复区大小为4689p,共编码1529个氨基酸,具体序列如SEQ ID NO.3所示。
CT端大小为456bp,共编码152个氨基酸,具体序列如SEQ ID NO.4所示。
本发明的一种大腹园蛛包卵丝蛋白全长基因的制备方法,包括如下步骤:
(1)根据现有的蜘蛛包卵丝的Tusp1蛋白基因,分别在NT端和CT端设计2条简并引物,在重复区设计2条特异性引物,通过简并PCR扩增得到部分的NT端和CT端基因序列;
(2)根据步骤(1)所获得的序列分别设计2对特异性引物及1条锚定引物,通过锚定PCR将NT端和CT端补齐;
(3)根据步骤(2)所获得的包含完整NT端和CT端的序列,在NT端5’末端设计1条正向引物,在CT端3’末端设计1条反向引物,用于扩增全长Tusp1基因;
(4)对步骤(3)所得PCR产物进行琼脂糖凝胶切胶回收后,利用Taq酶对产物进行加A反应,之后进行产物回收;
(5)对步骤(4)的基因片段进行TA克隆,使用热击的转化方法,转入DH5α感受态细胞中进行转化,将转化后的菌液涂布在具有Amp抗性的LB固体培养基上,过夜培养后,挑取单克隆进行PCR检测;
(6)将步骤(5)中得到的阳性单克隆的质粒进行完整测序,最终得到大腹园蛛包卵丝蛋白全长基因。
所述步骤(1)中用于扩增部分NT端的正向引物:ATGGTHTGGCTNATNAG;
反向引物:CAAACGAGGAGGCCAGGGAAC;
用于扩增部分CT端的正向引物:TCAAGTAGCAGTGCCTCC;
反向引物:AGNGCNGCDATNCCYTC。
用简并PCR获得的部分NT端序列的正向引物为简并引物,反向引物为重复区的特异性引物,用于获得部分CT端序列的正向引物为重复区特异性引物,反向引物为简并引物。
所述步骤(1)中的简并PCR的条件为:95℃预变性5min,95℃变性30s,53℃退火30s,72℃延伸30s,循环30次。
所述步骤(2)中的用于补全NT端的特异性引物1:TGTTATAGGCTTAGGTGGCGTTGC;
用于补全NT端的特异性引物2:GAAGGTATTGCAGCCCTCTTGCAAG;
用于补全CT端的特异性引物1:GGAGATCTGGTGAAGATGCGAGT;
用于补全CT端的特异性引物2:CTGAAGGAAACTGCTTGCCAAGG;
锚定引物:ACTCCTGTGGAACCATCGGACGGGGGG。4条特异性引物中,2条用于补全Tusp基因的NT端,2条用于补全Tusp基因的CT端。
所述锚定PCR条件为:在第一轮PCR中,使用特异性引物1进行单引物扩增,条件:95℃预变性5min,95℃变性30s,60℃退火30s,72℃延伸30s,循环30次;之后将PCR产物进行琼脂糖凝胶切胶回收,用TdT酶进行加C反应,产物回收后进行第二轮PCR;使用特异性引物2以及锚定引物进行扩增,条件同上。
所述步骤(3)中用于扩增全长Tusp1基因的正向引物:GCCGCATATGATGGTTTGGCTGACTAGCGTAGCGGTCC;
反向引物:ATCGCTCGAGACCAGAAAGAGCACTCAGCAATGCCTGTG。1对引物分别位于Tusp基因的5’末端以及3’末端,以获得完整的Tusp基因。
所述步骤(3)中用于扩增全长Tusp1基因的酶为Q5高保真酶;扩增条件为:98℃预变性1min,98℃变性5s,72℃延伸8min,循环30次。
所述步骤(4)中的加A反应条件为:72℃反应30min。
所述步骤(5)中连接产物和DH5α感受态细胞的体积比为1:10,复苏时间为1h。用于TA克隆的载体为pMD-18T。
所述步骤(5)中的LB固体培养基含有氨苄青霉素,氨苄青霉素在培养基中的浓度为100μg/mL。
本发明首先通过简并PCR扩增出Tusp1的NT端和CT端部分序列,然后利用锚定PCR将其补全,之后采用长距离PCR方法扩增得到完整的Tusp1基因并进行TA克隆,得到含有全长Tusp1基因的克隆。
有益效果
本发明整个基因包含有完整的NT端,重复区以及完整的CT端,用于编码大腹园蛛包卵丝蛋白;该丝纤维在力学性能以及生物相容性上都优于蚕丝以及人造纤维,而且对环境无污染;制备方法工艺简单,条件温和,易于操作,为在基因工程上大规模生产蛛蛋白奠定了基因基础。
附图说明
图1为本发明大腹园蛛Tusp1蛋白的结构域(A)和序列(B);
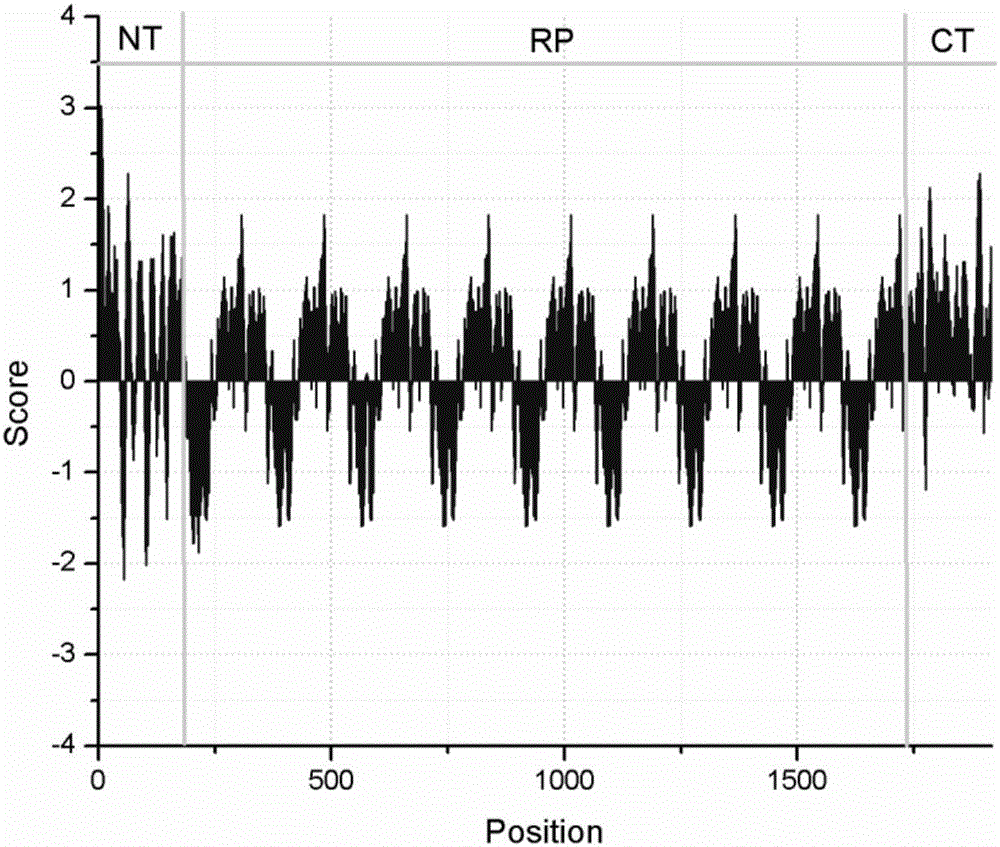
图2为本发明大腹园蛛Tusp1蛋白的疏水性分析图;其中,图2a为完整的Tusp1蛋白疏水性图,图2b为NT端疏水性图,图2c为CT端疏水性图,图2d为repeat 5疏水性图。
图3为本发明大腹园蛛Tusp1蛋白的二级结构预测图;其中,图3a为NT端二级结构预测图,图3b为CT端二级结构预测图,图3c为repeat 5二级结构预测图。
图4为本发明大腹园蛛Tusp1蛋白的序列比对图;其中,图4a为7种蜘蛛的Tusp1的NT端序列比对图,图4b为13种蜘蛛的Tusp1的CT端序列比对图,图4c为大腹园蛛Tusp1重复区内的9个重复单元之间的序列比对图。
具体实施方式
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。此外应理解,在阅读了本发明讲授的内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
实施例1
(1)根据其它蜘蛛的Tusp1的NT端和CT端的保守序列,分别在NT端和CT端各设计1条简并引物:NT端简并引物:ATGGTHTGGCTNATNAG,CT端简并引物:AGNGCNGCDATNCCYTC,并在重复区设计1条特异性正向引物和一条特异性反向引物,特异性正向引物:TCAAGTAGCAGTGCCTCC;
特异性反向引物:CAAACGAGGAGGCCAGGGAAC;然后进行PCR,条件为95℃预变性5min,95℃变性30s,53℃退火30s,72℃延伸30s,循环30次。
(2)根据所获得的序列分别在NT端和CT端各设计2条特异性引物以及1条锚定引物用于补全NT端和CT端。
NT端特异性引物1:TGTTATAGGCTTAGGTGGCGTTGC;
NT端特异性引物2:GAAGGTATTGCA GCCCTCTTGCAAG;
CT端特异性引物1:GGAGATCTGGTGAAGATGCGAGT;
CT端特异性引物2:CTGAAGGAAACTGCTTGCCAAGG;
锚定引物:ACTCCTGTGGAACC ATCGGACGGGGGG;
锚定PCR条件为:在第一轮PCR中,使用特异性引物1进行单引物扩增,条件:95℃预变性5min,95℃变性30s,60℃退火30s,72℃延伸30s,循环30次。之后将PCR产物进行琼脂糖凝胶切胶回收,用TdT酶进行加C反应,产物回收后进行第二轮PCR。使用特异性引物2以及锚定引物进行扩增,条件同上。
(3)之后设计1对特异性引物用于扩增全长Tusp1基因片段,
正向引物:GCCGCATATGATGGTTTGGCT GACTAGCGTAGCGGTCC;
反向引物:ATCGCTCGAGACCAGAAAGAGCACTCAGC AATGCCTGTG;
PCR条件为98℃预变性1min,98℃变性5s,72℃延伸8min,循环30次。之后琼脂糖凝胶切胶回收。
(4)利用Taq酶对产物进行加A反应,72℃反应30min;
(5)之后与pMD-18T进行连接,条件为:连接反应温度16℃,时间12h。转入DH5α感受态细胞,体积比为1:10,复苏时间为1h。之后将菌液涂布在氨苄青霉素的固体培养基上,37℃培养12h。然后对菌落进行筛选,测序。
使用SignalP4.1、Expasy tools、PSIPRED等在线软件以及Geneious、DNA star软件对该基因进行分析,SignalP4.1用于寻找基因的信号肽,Expasy tools用来对Tusp的疏水性进行分析,PSIPRED对Tusp1的二级结构进行预测,Geneious用来进行多序列比对,DNA star用来对Tusp1基因的碱基组成和氨基酸组成进行分析。
分析结果如下:
SignalP4.1以及DNA star分析结果:图1为Tusp1蛋白的结构域以及序列图谱,从图1A中可以看出,整个Tusp1基因划分为三个区域,NT端,重复区以及CT端。其中重复区内由9个在序列上极其相似的重复单元组成,并占据了整个蛋白序列的85%以上。其中前8个重复单元大小为176个氨基酸,第9个重复单元为155个氨基酸。在图1B中可以发现蛋白的信号肽存在于NT端中(斜体,加粗),并且存在2个Cys残基。
Expasy tools分析结果:图2为整个Tusp1蛋白的疏水性分析图以及NT端,Repeat unit 5(R5),CT端的疏水性图。从图2中可以看到,疏水性区域以及亲水性区域在整个蛋白序列中反复变化,整个蛋白分子表现为疏水性。CT端的平均疏水性值大于NT端,但NT端的疏水性峰值大于CT端。
PSIPRED分析结果:图3为Tusp1蛋白二级结构预测图,从图3a可以看出,在NT端中含有5个α螺旋结构,而且2个Cys残基分别处于第1个和第4个螺旋中。在图3b中可以发现CT端中同样含有5个螺旋结构另外含有3个折叠片结构。图3c中重复单元内有6个螺旋结构。
Geneious分析结果:图4为Tusp1蛋白的对比图。从图4a中可以看出Tusp1的NT端有较高的相似度,其中在ALSTALASS这段序列的相似度为100%,说明可能这段序列有某种重要的功能。图4b中Tusp1的CT端之间的保守性比NT端强,图4c中为这9个重复单元之间的比对,发现重复单元之间相似度极高,结合图3c可以发现,6个螺旋结构的氨基酸序列之间的保守性最高,说明这6个螺旋结构对丝的性能具有关键作用。
- 还没有人留言评论。精彩留言会获得点赞!