核酸序列信息的空间定位的制作方法
用于检测和分析组织样品中的核酸(例如mRNA或基因组DNA)的现有技术通常一次提供一种或有限数目的基因的空间或局部信息,或在没有期望位置信息的情况下提供样品中所有基因的信息。最近的兴趣聚焦于技术的发展,该技术可以在保存关于组织的空间信息的情况下对组织中的转录物组和/或基因组变异进行表征。需要表征组织样品背景下的核酸的方法。
2.发明概述
本公开提供了方法和组合物,其有助于在保存与组织中靶核酸起源有关的空间信息的情况下表征组织中的转录物组和/或基因组变异。例如,本文中公开的方法可以使得能够鉴定携带异常突变的组织生检中的细胞或细胞簇的位置。因此,本文中提供的方法可用于诊断目的,例如用于癌症的诊断,并且可能有助于选择靶向疗法。
本公开提供了用于空间检测和分析组织样品中的核酸的捕捉阵列,所述捕捉阵列包含捕捉位点,所述捕捉位点包含表面上固定化的捕捉探针对,其中捕捉探针对的第一捕捉探针包含第一引物结合区和空间地址区,并且其中捕捉探针对的第二捕捉探针包含第二引物结合区和捕捉区。
本公开还提供了用于空间检测和分析组织样品中的核酸的方法,所述方法包括(a)提供捕捉阵列,所述捕捉阵列包含捕捉位点,所述捕捉位点包含表面上固定化的捕捉探针对,其中捕捉探针对的第一捕捉探针包含第一引物结合区和空间地址区,且其中捕捉探针对的第二捕捉探针包含第二引物结合区和捕捉区。
本公开还提供了用于空间检测和分析组织样品中的核酸的方法,所述方法包括提供包含固定化捕捉探针的磁性纳米颗粒,所述固定化捕捉探针包含捕捉区。
本公开还提供了用于空间检测和分析组织样品中的核酸的方法,所述方法包括提供包含固定化捕捉探针的磁性纳米颗粒,所述固定化捕捉探针包含捕捉区。
本公开还提供了用于空间检测和分析组织样品中的核酸的捕捉阵列,所述捕捉阵列包含含有捕捉探针的捕捉位点,所述捕捉探针包含空间地址区和转座子末端(TE)区。
本公开还提供了用于空间检测和分析组织样品中的核酸的方法,所述方法包括提供包含含有捕捉探针的捕捉位点的捕捉阵列,所述捕捉探针包含空间地址区和转座子末端(TE)区。
3.附图简述
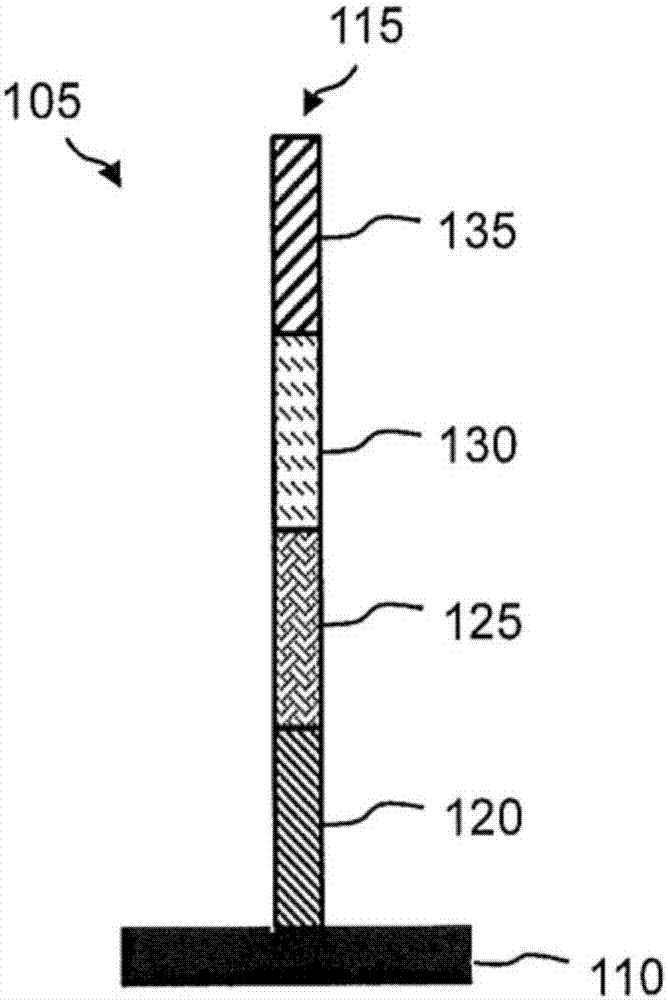
图1A显示了用于捕捉组织样品中的核酸的捕捉阵列的示例性实施方案的平面视图。
图1B显示了捕捉阵列的一个捕捉位点的侧视图,其中一个捕捉位点包含至少一个用于捕捉组织样品中的核酸的捕捉探针。
图1C显示了用于捕捉组织样品中的核酸的通用捕捉珠的示例性实施方案的侧视图。
图2显示了空间检测和分析组织样品中的核酸的方法的示例性实施方案的流程图。
图3显示了图1A的阵列的侧视图并且显示了将组织样品中的总mRNA捕捉到阵列上的过程的示例性实施方案。
图4显示了图1的阵列的侧视图并且显示了将组织样品中的靶定mRNA捕捉到阵列上的过程的示例性实施方案。
图5显示了通过原位逆转录组织样品中的RNA来产生cDNA以捕捉到阵列上的方法的示例性实施方案的流程图。
图6显示了图5的方法的步骤的示例性实施方案。
图7显示了图5的方法的步骤的另一个示例性实施方案。
图8显示了将DNA扩增子捕捉到阵列上的方法的示例性实施方案的流程图,其中通过靶核酸的原位扩增产生DNA扩增子。
图9显示了图8的方法的步骤的示例性实施方案。
图10显示了将DNA扩增子捕捉到阵列上的方法的示例性实施方案的流程图,其中通过靶核酸的原位扩增产生DNA扩增子。
图11显示了图10的方法的步骤。
图12显示了通过单链连接将cDNA捕捉到阵列上的方法的示例性实施方案的流程图,其中通过靶RNA分子的原位逆转录产生cDNA。
图13A和13B显示了图12的方法的步骤。
图14显示了将DNA扩增子捕捉到阵列上的方法的示例性实施方案的流程图,其中通过靶核酸的原位扩增产生DNA扩增子。
图15A、15B、和15C显示了图14的方法的步骤。
图16显示了配置用于空间检测和分析组织样品中的核酸的电泳转移系统的部分的侧视图。
图17显示了捕捉阵列(例如图1A的捕捉阵列)上的一个捕捉位点的侧视图,其中一个捕捉位点包含不同的两组固定化捕捉探针。
图18显示了将核酸从组织样品转移到捕捉阵列以产生空间寻址测序文库的方法的一个实施方案的流程图,其中捕捉阵列包含捕捉位点,所述捕捉位点包含不同的固定化捕捉探针对,例如如图17中显示。
图19A、19B、19C、和19D显示了图18的方法的步骤。
图20显示了捕捉组织样品中的核酸以后续锚定到阵列上的方法的示例性实施方案。
图21A和21B分别显示了用于空间检测和分析组织样品中的核酸的一维索引化(indexing)方案的网格阵列和二维索引化方案的网格阵列。
图22显示了使用组合索引化系统以生成空间寻址cDNA测序文库的方法的示例性实施方案的流程图。
图23A和23B显示了图21的方法的步骤。
图24显示了使用组合索引化系统以产生空间寻址cDNA测序文库的备选方法的示例性实施方案的流程图。
图25A、25B、和25C显示了图24的方法的步骤。
图26A显示了用于将逆转录(RT)引物递送到组织样品以原位合成cDNA的阵列的示例性实施方案的平面视图。
图26B显示了图26A的阵列的一个递送位点的部分的侧视图,其中递送位点的部分包含至少一个用于从组织样品中的mRNA合成cDNA的RT引物。
图27A显示了用于捕捉使用图26B的RT引物原位合成cDNA的捕捉阵列的示例性实施方案的平面视图。
图27B显示了图27A的捕捉阵列的一个捕捉位点的部分的侧视图,其中捕捉位点的部分包含至少一个用于捕捉合成的cDNA的捕捉探针。
图28显示了产生空间寻址测序文库的方法的示例性实施方案的流程图,其中第一阵列用于原位合成第一链cDNA,并且第二阵列用于捕捉cDNA以进行随后的文库产生。
图29A和29B显示了图28的方法的步骤。
图30显示了使用可释放的捕捉探针产生空间寻址cDNA文库的方法的示例性实施方案的流程图。
图31A和31B分别显示了包含5二硫化物修饰的空间寻址捕捉探针和包含5光可切割接头的空间寻址捕捉探针的示例性示意图。
图32A、32B、和32C显示了将图31A的空间寻址捕捉探针可逆锚定到玻璃盖玻片表面上的过程的示例性实施方案。
图33A、33B、和33C显示了将图31B的空间寻址捕捉探针可逆锚定到玻璃盖玻片表面上的过程的示例性实施方案。
图34A和34B显示了图30的方法的步骤。
图35显示了用于捕捉感兴趣的基因组DNA区的捕捉探针对的示例性实施方案的示意图。
图36显示了使用可释放捕捉探针产生空间寻址基因组扩增子文库的方法的示例性实施方案的流程图。
图37显示了图36的方法的步骤。
图38显示了使用磁性纳米颗粒从组织样品捕捉核酸产生空间寻址测序文库的方法的示例性实施方案的流程图。
图39显示了图38的方法的步骤。
图40A和40B显示了使用捕捉探针形成组织样品中的互补核酸并且随后将互补核酸固定化到纳米颗粒的过程的实例。
图41A和41B分别显示了包含第一部分空间地址区的颗粒结合捕捉探针和包含第二部分地址区的第二阵列捕捉探针的实例的示意图,用于空间检测和分析组织样品中的核酸。
图42A和42B分别显示了颗粒结合捕捉探针和包含空间地址区的第二阵列捕捉探针的实例的示意图,用于空间检测和分析组织样品中的核酸。
图43A和43B分别显示了颗粒结合捕捉探针和第二阵列捕捉探针的实例的示意图,用于空间检测和分析组织样品中的核酸。
图44显示了配置用于空间检测和分析组织样品中的核酸的基于磁性的转移系统的透视图。
图45显示了图44的捕捉阵列上的一个捕捉位点的侧视图,其中一个捕捉位点包含多个捕捉探针。
图46显示了使用图44和图45的基于磁性的转移系统将cDNA从组织样品转移到捕捉阵列以产生空间寻址测序文库的方法的实例的流程图。
图47A、47B、和47C以图像显示了图46的方法的步骤。
图48显示了使用图44的基于磁性的转移系统将cDNA从组织样品转移到捕捉阵列以产生空间寻址测序文库的方法的实例的流程图。
图49A、49B、和49C以图像显示了图48的方法的步骤。
图50显示了对组织样品中的基因组DNA进行序型分析的实例的流程图。
图51显示了用于全基因组DNA的预扩增和空间索引化的空间寻址PCR引物的图。
图52A和52B以图像显示了图50的方法的步骤。
图53显示了微孔反应器覆盖的实例的透视图。
图54显示了图53的单一微孔的透视图。
图55A和55B显示了制作图53的微孔基底的过程的实例。
图56显示了用于捕捉和空间区室化来自组织样品的核酸的微孔结构的实例的侧视图。
图57显示了使用图56的微孔结构制备测序文库从组织切片捕捉核酸的方法的实例的流程图。
图58A显示了用于组织切割和制备空间寻址核酸文库的插针系统(pin system)的实例的侧视图。
图58B显示了图58A的插针结构上的插针的不同切割表面的实例。
图59显示了使用图58的插针系统制备测序文库从组织切片捕捉核酸的方法的实例的流程图。
图60显示了图58的插针系统的侧视图并且以图像显示了图59的方法的步骤5910和5915。
图61显示了使用图58的插针系统制备测序文库从组织切片捕捉核酸的方法的另一个实例的流程图。
图62显示了图58的插针系统的侧视图并且以图像显示了图61的方法的步骤6110和6115。
图63显示了用于从组织切片捕捉核酸以制备空间寻址核酸文库的毛细管“微反应器”系统的透视图。
图64显示了使用图63的毛细管微反应器系统制备测序文库从组织切片捕捉核酸的方法的实例的流程图。
图65显示了配置用于空间检测和分析组织样品中的核酸的液滴致动器的部分的侧视图。
图66显示了图65的孔片层的侧视图。
图67A和67B显示了图65的液滴致动器的侧视图并且显示了分离组织样品中的核酸以进行空间检测和分析的过程。
图68显示了使用全基因组DNA的标签片段化产生空间寻址基因组扩增子文库的方法的示例性实施方案。
图69A、69B、和69C显示了图68的方法的步骤。
图70显示了空间地址覆盖的平面视图。
图71显示了图70的基底上单一空间特征的平面视图。
4.发明详述
本文中描述了多种方法和组合物,其允许在保持与组织中的靶分析物起源相关的空间信息的情况下表征组织中的分析物。在各个实施方案中,阵列包含基底,所述基底上固定化多个捕捉探针,使得每个捕捉探针占据阵列上的独特位置。除别的序列和/或分析外,每个捕捉探针包含独特的位置核酸标签(即,空间地址或索引化序列)。每个空间地址对应于阵列上的捕捉探针位置。阵列上捕捉探针的位置可以与组织样品中的位置相关联。
组织样品中的分析物的实例包括基因组DNA、甲基化DNA、特定的甲基化DNA序列、信使RNA(mRNA)、多聚A mRNA、片段化mRNA、片段化的DNA、线粒体DNA、病毒RNA、微小RNA、原位合成的PCR产物、RNA/DNA杂合物、脂质、碳水化合物、蛋白质、糖蛋白、脂蛋白、磷蛋白、蛋白质的特定磷酸化或乙酰化变体或病毒壳体蛋白。
核酸标签编码位置(即,空间地址或索引化序列)可以与核酸捕捉区或任何其它结合靶分析物的分子偶联。可以与核酸标签偶联的其它分子的实例包括抗体、抗原结合结构域、蛋白质、肽、受体、半抗原等。
本文中描述了允许在保留与组织中靶核酸的起源有关的空间信息的情况下表征组织中转录物组和/或基因组变异的多种方法和组合物。例如,本文公开的方法可以使得能够鉴定携带异常突变的组织生检中的细胞或细胞簇的位置。因此,本文提供的方法可以用于诊断目的,例如用于癌症的诊断,并且可能有助于选择靶向疗法。
本公开部分基于以下认识:在制备用于测序的核酸的过程中可以在核酸中编码与组织样品中的核酸的空间起源有关的信息。例如,来自组织样品的核酸可以通过包括位置特异性序列信息(“空间地址”)的探针进行标签化(tagging)。然后,可以对来自组织样品的空间寻址核酸分子批量测序。源自组织样品中不同区域的序列相同的核酸分子可以基于其空间地址来区分,并且可以定位到组织样品中的其起源区域上。
本公开还部分基于以下认识:基于组织样品中其空间起源对相关核酸进行区分可以提高复杂组织中罕见突变的检测灵敏性。例如,发现核酸的空间寻址可以提高组织样品中单核苷酸变异(SNV)的检测灵敏性。
在本文描述的一些方法中,用于空间标签化的探针可以包括例如空间地址区和基因特异性捕捉区的组合。可以使空间寻址和基因特异性探针作为捕捉阵列上的固定化探针与组织样品接触。或者,可以从捕捉阵列中释放空间寻址探针,并与组织样品中溶液中的核酸相互作用,例如原位相互作用。
本公开还部分基于以下认识:可以使用将空间地址区与基因特异性捕捉区分开的探针来进行空间标签化。在两个或更多个探针中将捕捉区与空间地址区分开的能力可以增加测序文库设计和文库制备方案的灵活性。
本公开还部分基于以下认识:通过促进将核酸从组织样品转移到捕捉阵列,例如空间寻址捕捉探针的捕捉阵列上,可以增强空间转录组学实验的稳健性(robustness)和数据质量。例如,可以使用核酸的电泳转移来改善核酸的转移收率和转移动力学。从组织样品到捕捉区上的高收率核酸转移可以促进罕见SNV的检测。快速转移动力学可用于限制转移过程期间的核酸扩散,并有助于提高空间寻址的分辨率。本文描述的其它方法牵涉使用中间核酸底物,如颗粒(例如电磁性纳米颗粒)、膜(例如尼龙膜)或微孔板以促进组织样品中的核酸捕捉,以促进核酸转移到捕捉阵列上,以及用于限制扩散并且改善空间分辨率。描述了牵涉例如基因组DNA的标签片段化(tagmentation)的其它方法,其可用于有效地将空间地址添加到核酸上,例如在捕捉阵列的表面上。
本公开还部分基于以下的认识:来自组织样品的核酸的空间寻址可以牵涉二维空间寻址,例如以将二维捕捉阵列上的核酸的位置与二维组织切片中的核酸位置相关联。也可以在额外的维度上进行空间寻址。例如,可以将空间地址序列添加到核酸以描述核酸在第三维度或第四维度上的相对空间位置,例如通过描述组织生检中组织切片的位置或者受试者器官中组织生检的位置进行。可以将时间地址序列添加到来自组织样品的核酸以表示在时间进程实验中的时间点,例如调查响应物理或化学刺激物,如临床试验期间的药物治疗的细胞中基因表达变化。
必须注意的是,如在本说明书和所附权利要求书中所用,除非内容另有明确规定,否则单数形式“一种”、“一个”和“该/所述”包括复数指代物。因此,例如,对“捕捉探针”的引用包括两个/种或更多个/种捕捉探针的混合物等。
术语“约”特别就给定量而言意指包含±5%的偏差。
如本文所用,术语“包括”、“包含”、“含有”及其任何变型旨在涵盖非排他性的纳入,使得包括、包含或含有一项要素或一批要素的工艺、方法、副产物或物质组合物不仅仅包括那些要素,而是可以包括没有明确列出的或者对于此类工艺、方法、副产物或物质组合物固有的其它要素。
如本文所用,术语“地址”、“标签”或“索引”在参考核苷酸序列使用时旨在意指与其它索引以及与样品内含有的多核苷酸内的其它核苷酸序列可区分的独特核苷酸序列。核苷酸“地址”、“标签”或“索引”可以是随机或特定设计的核苷酸序列。“地址”、“标签”或“索引”可以有任何期望的序列长度,只要其具有足够的长度以成为群体中的多个索引内和/或分析或询问的多个多核苷酸内的独特核苷酸序列。本公开内容的核苷酸“地址”、“标签”或“索引”可用于例如附着于靶多核苷酸以标签化或标记特定种类,用于鉴定群体内标签化种类的全部成员。因此,索引作为条形码是有用的,其中相同分子种类的不同成员可以包含相同的索引,并且其中不同多核苷酸的群体内的不同种类可以具有不同索引。
如本文所用,当参考核苷酸序列使用时,“空间地址”、“空间标签”或“空间索引”意指编码与组织样品中的寻址、标签化或索引化核酸的起源的区域或位置相关的空间信息的地址、标签或索引。
如本文所用,术语“基底”旨在意指固体支持物。该术语包括可以充当固体或半固体底座以创建特征(诸如孔)以及沉积生物聚合物(包括核酸、多肽和/或其它聚合物)的任何材料。例如,通过对于本领域技术人员公知的多种方法,修饰或可以修饰如本文所提供的基底以适应生物聚合物的附着。基底材料的示例性类型包括玻璃、改性玻璃、功能化玻璃、无机玻璃、微球(包括惰性和/或磁性颗粒)、塑料、多糖、尼龙、硝酸纤维素、陶瓷、树脂、二氧化硅、基于二氧化硅的材料、碳、光纤或光纤束、除上文例示的材料以外的多种聚合物和多孔微量滴定板。示例性塑料的具体类型包括丙烯酸树脂(acrylics)、聚苯乙烯、苯乙烯和其它材料的共聚物、聚丙烯、聚乙烯、聚丁烯、聚氨基甲酸酯和TeflonTM。示例性的基于二氧化硅的材料的具体类型包括硅和改性硅的各种形式。
本领域技术人员应当知道或理解,如本文提供的基底的组成和几何形状可以随意图的用途和用户的偏好而变化。因此,尽管为了阐述而在本文中参考微阵列例示了诸如载玻片、芯片或晶片的平面基底,但是鉴于本文提供的教导和指导,本领域技术人员应当理解,本文例示的或本领域中公知的极其多种其它基底也可以用于本文的方法和/或组合物中。
在一些实施方案中,固体支持物包括流动池的一个或多个表面。如本文所用,术语“流动池”是指包含可以使一种或多种流体试剂流过的固体表面的腔室。在本公开的方法中可以容易地使用的流动池和相关的流体系统和检测平台的例子例如记载于Bentley et al.,Nature 456:53-59(2008),WO 04/018497;US 7,057,026;WO 91/06678;WO 07/123744;US 7,329,492;US 7,211,414;US 7,315,019;US 7,405,281,和US 2008/0108082,每篇通过引用并入本文。
在一些实施方案中,固体载体包括图案化的表面。“图案化表面”是指固体支持物的暴露层中或上的不同区域的排列。例如,一个或多个区域可以是存在一个或多个扩增引物的特征。这些特征可以以不存在扩增引物的间隙区域分开。在一些实施方案中,图案可以是行和列中的特征的x-y格式。在一些实施方案中,图案可以是特征和/或间隙区域的重复排列。在一些实施方案中,图案可以是特征和/或间隙区域的随机排列。可用于本文阐述的方法和组合物的示例性图案化表面记载于美国流水号13/661,524或美国专利申请公开号2012/0316086 A1,每篇通过引用并入本文。
如本文所用,术语“间隙区域”是指在基底中或表面上分隔基底或表面的其它区域的区域。例如,间隙区域可以将阵列的一个特征与阵列的另一个特征分开。两个彼此分开的区域可以是离散的,彼此缺乏接触。在另一个实例中,间隙区域可以将特征的第一部分与特征的第二部分分开。由间隙区域提供的分开可以是部分或全部分开。间隙区域通常会具有与表面上的特征的表面材料不同的表面材料。例如,阵列的特征可以具有超过存在于间隙区域的量或浓度的捕捉剂或引物的量或浓度。在一些实施方案中,捕捉剂或引物可以不存在于间隙区域。
在一些实施方案中,固体支持物包括表面中的孔或凹陷的阵列。这可以如本领域公知的那样使用多种技术来制造,所述技术包括但不限于光刻法(photolithography)、冲压技术(stamping techniques)、模制技术(molding techniques)和微蚀刻技术(microetching techniques)。如本领域技术人员应当理的,所使用的技术将取决于阵列基底的组成和形状。
图案化表面中的特征可以是在玻璃、硅、塑料或具有图案化的共价连接的凝胶如聚(N-(5-叠氮基乙酰胺基戊基)丙烯酰胺-共-丙烯酰胺)(PAZAM,参见例如美国临时专利申请序列号61/753,833,其通过引用并入本文)的其它合适的固体支持物上的孔(例如微孔或纳米孔)阵列中的孔。该过程产生用于测序的凝胶垫,其在具有大量循环的测序运行中可以是稳定的。聚合物与孔的共价连接有助于在多种用途期间在结构化基底的整个寿命期间维持结构化特征中的凝胶。然而,在许多实施方案中,凝胶不需要共价连接至孔。例如,在某些条件下,不共价附着到结构化基底的任何部分的不含硅烷的丙烯酰胺(SFA,参见例如美国专利申请公开号2011/0059865 A1,其通过引用并入本文)可以用作凝胶材料。
在具体的实施方案中,可以如下生成结构化基底,即通过用孔(例如微孔或纳米孔)对固体支持物材料进行图案化,用凝胶材料(例如PAZAM、SFA或其化学改性的变体,例如SFA的叠氮化(azidolyze)形式(叠氮基-SFA))包被图案化支持物,并且例如通过化学或机械抛光来抛光经凝胶包被的支持物,从而在孔中保留凝胶,但从孔之间的结构化基底表面上的间隙区域除去或灭活基本上所有的凝胶。可以将引物核酸附着到凝胶材料。然后,可以使靶核酸(例如片段化的人基因组)的溶液与经抛光的基底接触,使得单独的靶核酸将通过与附着于凝胶材料的引物的相互作用而接种单独的孔;然而,由于凝胶材料不存在或无活性,靶核酸将不占据间隙区域。靶核酸的扩增将限制于孔,因为间隙区域中凝胶的不存在或无活性阻止了生长的核酸集落的向外迁移。该工艺可方便地制造,可扩展并利用常规的微米或纳米制造方法。
图案化的基底可以包括例如蚀刻到载玻片或芯片中的孔。孔的蚀刻和几何形状的图案可以呈现多种不同的形状和尺寸,只要此类特征在物理上或功能上彼此可分开。具有此类结构特征的特别有用的基底是图案化的基底,其可以选择诸如微球的固体支持物颗粒的尺寸。具有这些特性的示例性图案化基底是与BeadArray技术(Illumina,Inc.,San Diego,CA)结合使用的蚀刻基底。进一步的实例记载于美国专利No.6,770,441,其通过引用并入本文。
如本文所用,当就核酸而言使用时,术语“固定化”旨在意指通过共价或非共价键直接或间接附着到固体支持物。在某些实施方案中,可以使用共价附着,但是所需要的只是核酸在意图使用支持物的条件下,例如在需要核酸扩增和/或测序的应用中保持静止或附着于支持物。可以将要用作捕捉引物或扩增引物的寡核苷酸固定化,使得3’端可用于酶促延伸,并且该序列的至少一部分能够与互补序列杂交。通过与表面附着的寡核苷酸杂交可以发生固定化,在这种情况下,固定化的寡核苷酸或多核苷酸可以是3’-5’方向。或者,可通过不同于碱基配对杂交的手段(如上文阐述的共价附着)发生固定化。
某些实施方案可以使用由惰性基底或基质组成的固体支持物(例如玻璃载玻片,聚合物珠等),所述固体支持物已经被官能化,例如通过施加包含允许共价附着到生物分子,如多核苷酸的反应性基团的中间材料的层或涂料进行。此类支持物的实例包括但不限于惰性基底如玻璃上的聚丙烯酰胺水凝胶,特别是如WO 2005/065814和US 2008/0280773(其通过引用完整并入本文)中所述的聚丙烯酰胺水凝胶。在此类实施方案中,生物分子(例如多核苷酸)可以直接共价附着到中间材料(例如水凝胶),但中间材料本身可以非共价附着到基底或基质(例如玻璃基底)。因此,术语“共价附着到固体支持物”解释为涵盖此类排列。
示例性的共价连接包括例如由于使用点击化学技术而产生的那些连接。示例性的非共价连接包括但不限于非特异性相互作用(例如氢键键合、离子键键合、范德华相互作用等)或特异性相互作用(例如亲和相互作用、受体-配体相互作用、抗体-表位相互作用、抗生物素蛋白-生物素相互作用、链霉亲合素-生物素相互作用、凝集素-碳水化合物相互作用等)。示例性的连接在美国专利号6,737,236;7,259,258;7,375,234和7,427,678;和美国专利公开号2011/0059865 A1中阐述,每篇通过引用并入本文。
如本文所用,术语“阵列”是指可根据相对位置而彼此区分的位点群体。位于阵列不同位置的不同分子可以根据阵列中位点的位置来彼此区分。阵列的单独位点可以包括一个或多个特定类型的分子。例如,位点可以包括具有特定序列的单一靶核酸分子,或者位点可以包括具有相同序列(和/或其互补序列)的几个核酸分子。阵列的位置可以是位于同一基底上的不同特征。示例性的特征包括但不限于基底中的孔、基底中或基底上的珠(或其它颗粒)、基底上的突出物、基底上的脊或基底上的通道。阵列的位点可以是各自带有不同分子的不同基底。可以根据与基底相关联的表面上的基底位置或者根据液体或凝胶中的基底位置来鉴定附着到不同基底的不同分子。不同基底位于表面上的示例性阵列包括但不限于在孔中具有珠的那些阵列。
如本文所用,术语“多个/复数个”旨在意指两个或更多个不同成员的群体。多个/复数个的范围可以从小、中等、大、到非常大。例如,较小的多个/复数个的大小可以为几个成员到几十个成员的范围。例如,中等大小的多个/复数个可以为几十个成员到约100个成员或几百个成员的范围。例如,较大的多个/复数个可以为约几百个成员到约1000个成员,到几千个成员和多达数万个成员。例如,非常大的多个/复数个可以为几万名成员到约几十万、一百万、几百万、几千万、和直至或大于几亿成员。因此,多个/复数个的大小范围可以为2至完全超过1亿个成员以及其间和大于上文示例性范围的谁有大小,如通过成员数目测量。微阵列内的特征的示例性数目包括在1.28cm2内的多个约500,000个或更多个离散特征。示例性的核酸多个/复数个包括例如约1×105、5×105和1×106或更多不同核酸种类的群体。因此,该术语的定义旨在包括大于2的所有整数值。多个/复数个的上限可以通过例如核酸样品中的核苷酸序列的理论多样性来设定。
如本文所用,术语“核酸”旨在与其在本领域中的使用一致,并且包括天然存在的核酸或其功能类似物。特别有用的功能类似物能够以序列特异性方式与核酸杂交或能够用作特定核苷酸序列复制的模板。天然存在的核酸通常具有含磷酸二酯键的主链。类似物结构可以具有包括本领域技术人员已知的多种中任一种的交替主链连接。天然存在的核酸通常具有脱氧核糖糖(例如在脱氧核糖核酸(DNA)中发现)或核糖糖(例如在核糖核酸(RNA)中发现)。核酸可含有本领域已知的这些糖部分的多种类似物中的任一种。核酸可以包括天然或非天然碱基。就这点而言,天然脱氧核糖核酸可具有选自腺嘌呤、胸腺嘧啶、胞嘧啶或鸟嘌呤的一个或多个碱基,而核糖核酸可具有选自尿嘧啶、腺嘌呤、胞嘧啶或鸟嘌呤的一个或多个碱基。可以包含在核酸中的有用的非天然碱基是本领域已知的。当就核酸而言使用时,术语“靶物”意图作为本文阐述的方法或组合物的背景下的核酸的语义标识符,并且不一定将核酸的结构或功能限制为超出另有明确的指示。核酸的特定形式可以包括生物体中发现的所有类型的核酸以及合成核酸,如通过化学合成产生的多核苷酸。通过掺入由如本文提供的方法产生的微阵列中适用于分析的核酸的具体实例包括基因组DNA(gDNA)、表达序列标签(EST)、DNA复制信使RNA(cDNA)、RNA复制信使RNA(cRNA)、线粒体DNA或基因组、RNA、信使RNA(mRNA)和/或其它RNA群体。这些示例性核酸的片段和/或部分当其在本文中使用时也包括该术语的意义内。
如本文所用,当就核酸分子而言使用时,术语“双链”是指核酸分子中的基本上全部核苷酸与互补核苷酸氢键键合。部分双链核酸可以具有与互补核苷酸氢键键合的其核苷酸的至少10%、25%、50%、60%、70%、80%、90%或95%。
如本文所用,当就核酸分子而言使用时,术语“单链”是指核酸分子中基本上无一核苷酸与互补核苷酸氢键结合。
如本文所用,术语“捕捉引物”旨在意指下述的寡核苷酸,其具有能够在例如扩增或测序反应的引物退火步骤中遇到的条件下与要分析或进行核酸询问的单链多核苷酸序列特异性退火的核苷酸序列。术语“核酸”,“多核苷酸”和“寡核苷酸”在本文中可互换使用。除非另外特别指出,不同的术语不意图表示大小、序列或其它性质上的任何特定的差异。为了描述的清楚,当描述包括几种核酸种类的特定方法或组合物时,术语可用于将一种核酸与另一种核酸进行区分。
如本文所用,术语“基因特异性”或“靶物特异性的”当就捕捉探针或其它核酸使用时,意图是指捕捉探针或其它核酸,其包括对靶定核酸,例如来自组织样品的核酸特异性的核苷酸序列,即能够选择性退火到靶定核酸的鉴定区域的核苷酸序列。基因特异性捕捉探针可以具有单一种类的寡核苷酸,或者可以包括具有不同序列的两种或更多种。因此,基因特异性捕捉探针可以是两个或更多个序列,包括3、4、5、6、7、8、9或10个或更多个不同的序列。基因特异性捕捉探针可以包括基因特异性捕捉引物序列和通用捕捉探针序列。其它序列如测序引物序列等也可以包含在基因特异性捕捉引物中。
相比之下,当就参考捕捉探针或其它核酸使用时,术语“通用”意指在多个捕捉探针中具有共同核苷酸序列的捕捉探针或核酸。例如,共同序列可以是与相同的衔接头序列互补的序列。通用捕捉探针适用于在不必区分不同的种类的情况下询问多个不同的多核苷酸,而基因特异性捕捉引物适用于区分不同的种类。
在各个实施方案中,可以间隔捕捉元件(例如捕捉引物或捕捉探针或其它核酸序列)以A)空间分辨单室的几何形状内的核酸,即每个室的多个捕捉位点;B)在约单室水平上空间分辨核酸,即每个室的约1个捕捉位点。此外,捕捉元件可以如上面的A或B中那样间隔开,并且可以:I)间隔开来以规则时间间隔从样品中取样核酸,例如以网格或图案隔开,使得采样约每隔一个或每隔5或10个室,或者采样约每隔一组或者每隔5或者每10组的2、3、4、5、6、7、8、9、10或者更多个室;II)间隔开来以从样品的一个或多个区域中的基本上全部可用的室捕捉样品,或III)间隔开来以捕捉来自样品中基本上全部可用的室的样品。
如本文所用,当就核酸而言使用时,术语“扩增子”是指复制核酸的产物。其中该产物具有与核酸的核苷酸序列的至少一部分相同或互补的核苷酸序列。扩增子可以通过使用核酸或其扩增子作为模板的多种扩增方法中的任何一种来产生,所述方法包括例如聚合酶延伸、聚合酶链式反应(PCR)、滚环扩增(RCA)、连接延伸或连接链式反应。扩增子可以是具有特定核苷酸序列(例如PCR产物)的单一拷贝或核苷酸序列的多个拷贝(例如RCA的多联体产物)的核酸分子。靶核酸的第一扩增子可以是互补拷贝。随后的扩增子是第一扩增子产生后从靶核酸或从第一扩增子产生的拷贝。随后的扩增子可以具有与靶核酸基本互补或与靶核酸基本上相同的序列。
可以产生的模板拷贝或扩增子的数目可以通过适当修饰扩增反应来调节,包括例如改变扩增循环运行的数目、使用扩增反应中不同持续合成能力的聚合酶和/或改变扩增反应运行的时间长度,以及修饰本领域已知影响扩增产率的其它条件。核酸模板的拷贝数可以是至少1、10、100、200、500、1000、2000、3000、4000、5000、6000、7000、8000、9000和10,000拷贝,并且可以随特定的应用而变化。
如本文所用,术语“每个/各自(each)”在就项目集合而言使用时旨在鉴定集合中的单独项目,但不一定是指集合中的每个项目,除非上下文另外明确指出。
本文提供了空间检测和分析(例如突变分析或单核苷酸变异(SNV)检测)组织样品中核酸的阵列和方法。本文描述的阵列可以包含基底,该基底上固定化了多个捕捉探针,使得每个捕捉探针占据阵列上的独特位置。多个捕捉探针中的一些或全部可以包含独特的位置标签(即,空间地址或索引化序列)。空间地址可以描述阵列上捕捉探针的位置。阵列上捕捉探针的位置可以与组织样品中的位置相关联。
如本文所用,术语“组织样品”是指已经从受试者获得,固定,切片并且安装在平面表面,例如显微镜载玻片上的一片组织。组织样品可以是福尔马林固定的石蜡包埋(FFPE)组织样品或新鲜组织样品或冷冻组织样品等。本文公开的方法可以在染色组织样品之前或之后进行。例如,在苏木精和曙红染色之后,组织样品可以按照本文提供的方法进行空间分析。方法可以包括分析样品的组织学(例如使用苏木精和曙红染色),然后空间分析组织。
如本文所用,术语“福尔马林固定的石蜡包埋(FFPE)组织切片”是指一片组织,例如已经从受试者获得,在甲醛(例如磷酸盐缓冲盐水中的3%-5%甲醛)或Bouin溶液中固定,包埋到蜡中,切成薄切片,然后安装在平面表面,如显微镜载玻片中的生检。
在一些实施方案中,将组织样品中的核酸转移并捕捉到阵列上。例如,使组织切片与阵列接触,将核酸捕捉到阵列上并用空间地址标签化。空间标签化的DNA分子从阵列释放,并且例如通过高通量下一代测序(NGS)如合成测序(SBS)进行分析。在一些实施方案中,将组织切片(例如福尔马林固定的石蜡包埋(FFPE)组织切片)中的核酸转移到阵列,并通过与捕捉探针杂交捕捉到阵列上。在一些实施方案中,捕捉探针可以是通用捕捉探针,其例如与核酸测序文库中的衔接子区域,或mRNA的多聚-A尾杂交。在一些实施方案中,捕捉探针可以是基因特异性捕捉探针,其例如与样品中特异性靶定的mRNA或cDNA杂交,如TruSeqTM定制扩增子(TSCA)寡核苷酸探针(Illumina,Inc.)。捕捉探针可以是多个捕捉探针,例如多个相同或不同的捕捉探针。
在一些实施方案中,将组织切片(例如FFPE切片)中的核酸转移至阵列,并通过与通用衔接子寡核苷酸的单链连接捕捉到阵列上。例如,可以在珠阵列上固定化包含空间地址的通用衔接子寡核苷酸。可以将单链核酸靶物连接到衔接子上用于捕捉。核酸可以包含cDNA或基因组DNA扩增子。通用衔接子可用于捕捉基因特异性cDNA或DNA扩增子。可以控制阵列(例如珠阵列)上的通用衔接子的方向以捕捉靶核酸的3’和5’区域。
在一些实施方案中,可以将捕捉阵列(即捕捉位点阵列)与电泳系统整合,以便于将核酸分子从组织切片转移到阵列上的捕捉位点上。核酸的电泳转移可以通过限制核酸分子在转移过程中远离其起源位置的扩散,并由此减少捕捉位点之间的核酸的损失来维持关于组织背景的空间分辨。
在一些实施方案中,使用组合索引化(寻址)系统来提供空间信息用于分析组织样品的核酸。组合索引化系统可以牵涉使用两个或更多个空间地址序列(例如2、3、4、5或更多个空间地址序列)。
在一些实施方案中,在制备测序文库的过程中,将两个空间地址序列掺入核酸中。可以使用第一空间地址来定义捕捉阵列上的X维度上的某个位置(即捕捉位点),并且可以使用第二空间地址序列来定义捕捉阵列上的Y维度上的位置(即,捕捉位点)。在文库制备过程中,可以确定X和Y空间地址序列两者,并且可以分析序列信息来定义捕捉阵列上的具体位置。
在一些实施方案中,在制备测序文库过程中,将三个空间地址序列掺入核酸中。可以使用第一空间地址来定义捕捉阵列上的X维度上的某个位置(即捕捉位点),可以使用第二空间地址序列来定义捕捉阵列上的Y维度上的位置(即捕捉位点),并且可以使用第三空间地址序列来定义样品(例如组织生检)中的二维样品切片的位置(例如组织样品的切片的位置)以提供样品的第三维度(Z维度)上的位置空间信息。在文库测序过程中,可以确定X、Y、Z空间地址序列,并可以对序列信息进行分析以定义捕捉阵列上的具体位置。
在一些实施方案中,在制备测序文库过程中,任选地将时间地址序列(T)掺入核酸中。在一些实施方案中,时间地址序列可以与两个或三个空间地址序列组合。例如,时间地址序列可以在时间过程实验的背景中用于确定组织样品中基因表达的时间依赖性变化。例如,响应于化学、生物或物理刺激(例如毒素、药物或热),基因表达的时间依赖性变化可以发生在组织样品中。可以将来自可比较的组织样品(例如组织样品的近端切片)的不同时间点获得的核酸样品合并并批量测序。可以使用任选的第一空间地址来定义捕捉阵列上的X维度上的某个位置(即捕捉位点),可以使用第二任选的空间地址序列来定义捕捉阵列上的Y维度上的位置(即捕捉位点),并且可以使用第三任选的空间地址序列来定义样品(例如组织生检)中的二维样品切片的位置(例如组织样品的切片的位置)以提供样品的第三维(Z维度)的位置空间信息。在文库测序过程中,确定T、X、Y和Z地址序列,并且分析序列信息以在每个时间点(T)定义捕捉阵列上特定的X、Y(和任选Z)位置。
地址序列X,Y以及任选地Z和/或T可以是连续的核酸序列,或者地址序列可以被一个或多个核酸(例如2或更多,3或更多,10或更多,30或更多,100或更多,300或更多,或1,000或更多)分开。在一些实施方案中,X,Y和任选地Z和/或T地址序列可以各自单独为组合核酸序列。
在一些实施方案中,地址序列(例如X,Y,Z或T)的长度可以各自独立地并且独立地为100个核酸或更少,90个核酸或更少,80个核酸或更少,70个核酸或更少,60个核酸或更少,50个核酸或更少,40个核酸或更少,30个核酸或更少,20个核酸或更少,15个核酸或更少,10个核酸或更少,8个核酸或更少,6个或更少的核酸,或4个或更少的核酸。核酸中两个或更多个地址序列的长度可以相同或不同。例如,如果地址序列X的长度是10个核酸,则地址序列Y的长度可以是8个核酸,10个核酸或12个核酸。
地址序列,例如空间地址序列,如X或Y可以是部分或完全简并序列。
在一些实施方案中,可以将阵列上的空间寻址捕捉探针从阵列释放到组织切片上以产生空间寻址测序文库。在一些实施方案中,捕捉探针包含随机引物序列,用于从组织切片中的RNA原位合成空间标签化的cDNA。在一些实施方案中,捕捉探针是TruSeqTM定制扩增子(TSCA)寡核苷酸探针(Illumina,Inc.),用于捕捉和空间标签化组织切片中的基因组DNA。从组织切片回收空间标签化的核酸分子(例如cDNA或基因组DNA),并且在单管反应中加工以生成空间标签化的扩增子文库。
在一些实施方案中,可以使用磁性纳米颗粒来捕捉组织样品中的核酸(例如原位合成cDNA),以生成空间寻址文库。
在一些实施方案中,可以对液滴致动器进行空间检测和分析组织样品中的核酸。
4.1阵列的生成
在一方面,本文提供了包含空间编码的捕捉探针的捕捉阵列。可以将捕捉探针上的空间编码的捕捉探针固定化,例如在平面玻璃基底上或在多个珠上。
图1A显示了用于捕捉组织样品中的核酸的捕捉阵列100的示例性实施方案的平面图。捕捉阵列100包含固体支持物110上捕捉位点105的排列(例如行和列)。在一些实施方案中,固体支持物110是平面玻璃基底。在一些实施方案中,固体支持物110是珠(例如参见图1C)。在捕捉阵列100的每个捕捉位点105处固定化至少一个捕捉探针115,如图1B中显示。即,图1B显示了捕捉阵列100的一个捕捉位点105的侧视图,其中一个捕捉位点105包含至少一个捕捉探针115,用于捕捉组织样品中的核酸。图1B显示固体支持物110表面上固定化的捕捉探针115。在此实施方案中,显示了单一捕捉探针115,但是可以在固体支持物110上在每个捕捉位点105处固定化任何数目的捕捉探针115。任选地,捕捉探针115可以包含可切割区域120中的可切割序列、SBS引物结合位点125中的SBS引物序列、空间地址区130中的空间地址序列、和捕捉区135中的捕捉序列。可以使用可切割区域120从捕捉阵列100释放捕捉的核酸,使得在释放的核酸中包含空间地址区130,并且将核酸“标签化”。SBS引物区125可以包含SBS引物序列(例如SBS12或SBS3),其可以用于合成测序(SBS)过程中。或者,随后可以添加SBS引物序列125或其某个部分,例如通过连接或通过PCR合成。也可以在扩增反应中使用SBS引物区125以生成测序文库,如参考图12和图13更为详细描述。空间地址区130对应于捕捉阵列100中捕捉探针115的位置。捕捉位点105处的每个捕捉探针115包含独特的空间地址区130。捕捉阵列100中的捕捉探针115的位置可以与组织样品中的位置相关联。
例如,捕捉区135可以是通用(一般)捕捉区。在一些实施方案中,捕捉区135包含多聚-T寡核苷酸,其可以用于捕捉组织样品中的总mRNA,如参考图3更为详细描述。在一些实施方案中,捕捉区135是通用捕捉区,其可以用于捕捉通过RNA的原位逆转录合成的cDNA,如参考图6更为详细描述。在一些实施方案中,捕捉区135是通用捕捉区,其可以用于捕捉基因组DNA扩增子,如参考图9和图10更为详细描述。
在一些实施方案中,捕捉区135是基因特异性或靶物特异性捕捉区,其可以用于捕捉组织样品中的特定核酸。捕捉阵列100上的每个捕捉探针115可以包含一个或多个独特的基因特异性捕捉区135。美国专利公开号2015148239(Peter等人于2014年9月22日提交并且通过引用并入本文)描述了可切割PCR引物,其中每个探针包含多个可切割引物,并且其可以在本文中描述的此种和其它实施方案中采用。捕捉阵列110上的不同捕捉探针115可以具有相同基因特异性捕捉区或者它们可以是不同基因特异性捕捉区。在一些实施方案中,组织样品中的核酸是基因特异性mRNA,如参考图4更为详细描述。在一些实施方案中,组织样品中的核酸是通过RNA的原位逆转录合成的基因特异性cDNA,如参考图7更为详细描述。在一些实施方案中(未显示),捕捉区135是可以捕捉基因组DNA扩增子的基因特异性区域。
可如下使探针与组织接触:通过将组织直接放置在包含探针的表面上;将组织放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织与探针分开,使得靶核酸可以从组织扩散过物质到探针;将组织放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织与探针分开,使得探针可从包含探针的表面扩散过物质到靶物;将目标从组织中提取到中间基底(例如凝胶,滤器,固体基底或前述的组合)上,然后将其放置在支持探针的表面上;以及前述的组合。在每种情况下,选择该技术以基本上维持编码样品中靶物的空间取向的信息。
图1C显示了用于捕捉组织样品中的核酸的通用捕捉珠150的实施方案的侧视图。在一些实施方案中,在捕捉位点105of捕捉阵列100的每个捕捉位点105处具有通用捕捉珠150。例如,可以将通用捕捉珠150沉积到固体支持物110(例如玻璃基底)上的孔中。通用捕捉珠150包含珠155。在珠155表面上固定化通用衔接子寡核苷酸160。通用衔接子寡核苷酸160与图1B的捕捉探针115基本上相同,只是省略了捕捉区135,即,通用衔接子寡核苷酸160仅包含可切割区域120、SBS引物区125、和空间地址区130。SBS引物区125可以包含例如SBS12序列或SBS3序列。在此实施方案中,显示了单一通用衔接子寡核苷酸160,但是在珠155上固定化任何数目的通用衔接子寡核苷酸160。可以通过将靶核酸(例如cDNA或基因组DNA扩增子)与通用衔接子寡核苷酸160单链连接,使用通用捕捉珠150捕捉组织样品中的核酸,如参考图12到15更为详细描述。
可以使用基于连接的方法生成基因特异性捕捉阵列,如在每个珠上具有多个基因特异性捕捉探针的珠阵列。例如,可以将珠阵列设计为在珠上具有100万个空间地址。可以将阵列设计为捕捉来自1,000种基因的核酸。为了捕捉来自1,000种基因的核酸,在设计为具有100万个空间地址的珠上会需要10亿个捕捉探针(即,1,000种基因x 100万个空间地址=10亿个捕捉寡核苷酸)。为了避免合成10亿个捕捉探针,可以将代表基因特异性捕捉区(例如捕捉区135)的寡核苷酸合并物连接到空间寻址捕捉探针上,所述空间寻址捕捉探针包含切割区120、SBS引物区125、和空间地址区130(例如代表1,000个基因特异性捕捉区的寡核苷酸+100万个空间地址区=110万个捕捉探针)。在一些实施方案中,使用酶促连接方法将基因特异性捕捉区的合并物连接到空间寻址捕捉探针。在一些实施方案中,使用化学连接方法将基因特异性捕捉区合并物连接到空间寻址捕捉探针。
也可以使用基于连接的方法来生成用于珠阵列的多个空间地址。生成空间寻址珠阵列的本方法需要以不依赖于每个独特空间地址(例如100万个空间地址需要合成100万个寡核苷酸)的方式合成每个寡核苷酸。为了避免合成100万个寡核苷酸,可以使用组合方法。例如,合成具有独特序列的寡核苷酸的三个独特亚组(例如具有100个独特序列的亚组A、具有100个独特序列的亚组B、和具有100个独特序列的亚组C),并且在组合连接反应中使用,例如100个亚组A x 100个亚组B x 100个亚组C=具有独特的空间地址的100万个寡核苷酸。组合方法需要合成仅300个不同的寡核苷酸。
在一些实施方案中,可以使用杂交和延伸方法来生成空间寻址基因特异性捕捉探针。例如,合成具有独特空间地址的1,000个寡核苷酸的集合“X”。合成1000个寡核苷酸的第二集合“Y”,其单独捕捉独特的基因并且可以与集合“X”寡核苷酸杂交。使集合“Y”寡核苷酸的每个单独的寡聚物与集合“X”寡核苷酸杂交,并且进行延伸反应。使用此方法,需要合成2,000个寡核苷酸以生成100万个不同捕捉探针(1,000个独特的空间地址序列与1,000个不同基因特异性捕捉序列单独配对)。使用通用寡核苷酸合成,生成各自单独具有1,000个不同空间地址的1,000个基因特异性捕捉探针将需要合成100万个寡核苷酸(1,000个基因x 1,000地址)。
可以使包含探针的珠与组织接触,即通过将组织直接放置在包含珠的表面上;将组织放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织与珠分开,使得靶核酸可以从组织扩散过物质到探针;将组织放置在诸如滤器或凝胶或缓冲层的物质上,将组织与探针分开,使得探针可从珠扩散过物质到靶物;将靶物从组织中提取到中间基底(例如凝胶,滤器,固体基底或前述的组合)上,然后将其放置在支持珠的表面上;将珠直接沉积到组织中;以及前述的组合。在每种情况下,选择该技术以基本上维持编码样品中靶物的空间取向的信息。
4.2空间检测和分析组织样品中的核酸
在另一个方面,本文提供了用于空间检测和分析样品中的核酸的方法。
图2显示了空间检测和分析组织样品中的核酸的方法200的实施方案的流程图。方法200可以包括但不限于一些或所有以下步骤。
在步骤210,制备组织样品以进行分析。在一些实施方案中,组织样品是切片到载玻片上的FFPE组织样品。其它实例包括新鲜组织、冷冻组织等。
在步骤215,对组织切片进行原位生化以促进样品中核酸的后续操作。在一些实施方案中,使用原位逆转录反应来从组织样品中的靶定mRNA合成cDNA。在一些实施方案中,可以使用原位扩增反应来从组织样品中的靶定基因生成多个基因组DNA扩增子。在一些实施方案中,不存在原位生化步骤,并且在从组织捕捉或延伸RNA后进行cDNA合成。
在步骤220,将组织切片中的靶核酸转移到阵列上,使得可以将阵列上的核酸位置与组织切片中的位置相关联。在一些实施方案中,靶核酸包含mRNA。在一些实施方案中,靶核酸包含原位合成的cDNA。在一些实施方案中,核酸包含通过原位扩增生成的基因组DNA扩增子。在一些实施方案中,阵列是捕捉位点的阵列,诸如图1A和图1B中显示的捕捉阵列100的捕捉位点105。在一些实施方案中,阵列是包含多个捕捉探针的珠(例如图1C的通用捕捉珠150)的阵列。阵列的其它实例包括包含多个捕捉探针的孔或孔隙(pores)或突出物或测序流动池的阵列。可以将核酸捕捉到阵列上,例如,通过将核酸与阵列上的捕捉探针杂交进行。在一些实施方案中,可以通过将核酸单链连接到通用衔接子寡核苷酸将核酸捕捉到阵列上。
在本文所述的此种和其它实施方案中,可以如下使探针与靶核酸接触,即通过将组织直接放置在包含探针的表面上;将组织放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织与探针分开,使得靶核酸可以从组织扩散过物质到探针;将组织放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织与探针分开,使得探针可以从包含探针的表面扩散过物质到靶核酸;将靶核酸从组织中提取到中间基底(例如凝胶,滤膜,固体基质或前述的组合)上,然后将其放置在支持探针的表面上;以及前述的组合。在每种情况下,选择该技术以基本上维持编码样品中靶物的空间取向的信息。
在步骤225,制备测序文库。在一些实施方案中,制备测序文库以进行合成测序。可以在捕捉阵列基底上实现文库制备,或者可以从基底切割核酸并且合并,使得可以分开实现文库制备,例如使用NeoPrepTM Library Prep System(Illumina,Inc.,San Diego)进行。
在步骤230,对文库测序。可以使用任何测序技术来完成测序。合适的测序仪的实例包括从或由Illumina,Inc.,F.Hoffmann-La Roche AG,Life Technologies,Inc.,Beckman Coulter,Inc.可购得或开发的那些测序仪。
在步骤235,分析序列数据(例如突变和/或变体调用)并解码空间信息。空间信息可用于提供关于组织切片中核酸位置的信息。
(a)基于杂交的核酸捕捉
在一些实施方案中,使用基于杂交的捕捉将组织样品中的靶核酸捕捉到阵列上的捕捉探针上。例如,阵列可以是包含多个捕捉探针的珠或孔或孔隙或突出物、平面表面或测序流动池的阵列。在一个实例中,使组织样品与阵列表面上固定的捕捉探针接触。可以将组织样品直接放置在包含捕捉探针的表面上或者可以将组织样品放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织样品与捕捉探针分开,使得靶核酸可以从组织扩散过物质到捕捉探针。
在另一个实例中,使组织样品与阵列接触,并且将阵列上的捕捉探针释放到组织样品中以与组织样品中的核酸杂交。可以将组织样品直接放置在包含捕捉探针的表面上或者可以将组织样品放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织样品与捕捉探针分开,使得释放的捕捉探针可以从阵列扩散过物质到组织样品中的核酸。可以使用可释放基团或选择性可切割部分或接头在阵列上锚定捕捉探针。例如,可以使用化学切割、酶促切割或光切割从阵列释放捕捉探针。在另一个实例中,可以将捕捉探针打印到阵列的表面上,并且干燥。可以通过再水合从阵列释放捕捉探针。在又一个实例中,可以使用在存在某种处理的情况下溶解的物质将捕捉探针打印到阵列上。然后,在将组织样品放置在阵列上前应用释放捕捉探针的处理。
在一些实施方案中,核酸是总mRNA。在一些实施方案中,核酸是基因特异性mRNA。
图3显示了图1A的捕捉阵列100的捕捉位点105的另一个实施方案的侧视图,并且显示了将组织样品中的总mRNA捕捉到阵列上的过程的实施方案。在此实施方案中,捕捉探针115包含多聚-T捕捉区310。使包含多个mRNA分子315的核酸样品,诸如包含源自组织样品的核酸靶物的组织样品或基底(未显示)与捕捉位点105接触。mRNA分子315包含多聚-A尾部320。突变325可以存在于mRNA分子315中。例如,突变325可以是单核苷酸多态性(SNP)。通过将多聚-A尾部320与多聚-T捕捉区310杂交在捕捉探针115上捕捉mRNA分子315。多聚-T捕捉区310也可以发挥逆转录酶引物功能以从捕捉的mRNA分子315(由虚线箭头标示)合成第一链cDNA。
在此实例中,使组织样品与捕捉位点105表面上固定的捕捉探针115接触。通过与多聚T捕捉序列310杂交将mRNA分子315捕捉到阵列上。在另一个实例(未显示)中,使组织样品与阵列接触,并且通过切割任选的可切割序列120将捕捉探针115从捕捉位点105释放到组织样品中。
在一些实施方案中(未显示),捕捉区310是随机寡核苷酸(“随机聚体(randomer)”)捕捉区,其可以用于捕捉RNA分子的随机合并物。例如,随机寡核苷酸捕捉区可以包含随机序列,其具有降低的复杂性,使得实质性降低组织样品中核糖体RNA的捕捉。
图4显示了图1A的捕捉阵列100的捕捉位点105的一些实施方案的侧视图,并且显示了将组织样品中单独靶定的(即,基因特异性)mRNA捕捉到阵列上的过程的实施方案。在此实施方案中,显示了两个捕捉探针115,即,捕捉探针115a和115b。捕捉探针115a包含基因特异性捕捉区410a。类似地,捕捉探针115b包含不同基因特异性捕捉区410b。使包含多个不同mRNA分子415的核酸样品,诸如包含源自组织样品的核酸靶物的组织样品或基底(未显示)与捕捉位点105接触。在此实施方案中,第一mRNA分子415a是来自第一基因的转录物,并且第二mRNA分子415b是来自第二基因的转录物。mRNA分子415a可以包含突变420a。mRNA分子415b可以包含突变420b。突变420可以例如是SNP。通过将互补的mRNA序列与基因特异性捕捉区410杂交在捕捉探针115a上捕捉mRNA分子415a。类似地,通过将互补的mRNA序列与基因特异性捕捉区410b杂交在捕捉探针115b上捕捉mRNA分子415b。基因特异性捕捉区410也可以发挥逆转录酶引物功能以从捕捉的mRNA分子415合成第一链cDNA。
在此实例中,使组织样品与捕捉位点105表面上固定的捕捉探针115接触。通过与基因特异性捕捉序列410杂交将mRNA分子415捕捉到阵列上。在另一个实例中(未显示),使组织样品与阵列接触,并且通过切割任选的可切割序列120将捕捉探针115从捕捉位点105释放到组织样品中。
在一些实施方案中,使用基于杂交的捕捉来捕捉通过将组织样品中的RNA原位逆转录到阵列上生成的cDNA。
图5显示了通过原位逆转录组织样品中的RNA以捕捉到阵列(例如图1A的捕捉阵列100)上产生cDNA的方法500的实施方案的流程图。方法500包含但不限于一些或所有以下步骤。
在步骤510,通过原位逆转录从组织样品中的靶mRNA合成基因特异性cDNA。例如,可以使用包含第一基因特异性引物和通用捕捉序列的寡核苷酸序列来引发第一链cDNA合成。
在任选的步骤515,通过原位等温扩增在组织样品中扩增cDNA。例如,可以使用包含第二基因特异性引物和SBS引物序列,例如SBS12的寡核苷酸序列进行等温扩增。
在步骤520,将扩增的cDNA捕捉到阵列上。通过将cDNA与阵列上的捕捉探针杂交将cDNA捕捉到阵列上。在一些实施方案中,捕捉探针包含通用捕捉序列,并且可以用于捕捉使用基因特异性引物合成的cDNA,所述基因特异性引物包含互补的捕捉序列,如参考图6更为详细描述。在一些实施方案中,捕捉探针包含基因特异性捕捉序列,并且可以用于捕捉使用基因特异性引物或包括SBS引物序列的随机引物的合并物合成的cDNA,如参考图7更为详细描述。
图6显示了图5的方法500的步骤的实施方案。在此实施方案中,在将DNA转移到阵列前使用原位等温扩增步骤(即,任选的步骤515)。即,组织切片(未显示)包含靶mRNA分子610。靶mRNA分子610可以包含突变615。在步骤510,使用逆转录(RT)引物620原位逆转录靶mRNA分子610。RT引物620包含第一基因特异性引物区625和通用捕捉区630。RT引物620也可以包含独特的分子标识符(UMI)区(未显示)。使用RT引物620合成的cDNA分子635包含通用捕捉区630。在步骤515,通过使用扩增引物640的原位等温扩增来扩增cDNA分子635。扩增引物640包含第二基因特异性引物区645和SBS测序引物区650,其包含例如SBS12序列。使用扩增引物640生成的DNA分子655包含通用捕捉区630和SBS测序引物650。在步骤520,将DNA分子655捕捉到捕捉位点105上。例如,使含有DNA分子655的组织样品或包含源自组织样品的DNA分子655的基底(未显示)与捕捉位点105接触。在一些实施方案中,捕捉位点105上的捕捉探针115包含与通用捕捉区630互补的捕捉区660;SBS测序引物130是SBS3。通过将通用捕捉区630与捕捉区660杂交将DNA分子655捕捉到捕捉位点105上。
在此实例中,捕捉区660和通用捕捉区630两者也可以发挥引物功能以进行延伸反应。当使用捕捉区660作为引物时,复制DNA分子655中的突变615。当使用通用捕捉区630作为引物时,复制空间地址区130和SBS引物区125。这两种延伸产物可以用于下游文库生成。
在此实例中,使组织样品与捕捉位点105表面上固定的捕捉探针115接触。通过将通用捕捉区630与捕捉区660杂交将DNA分子655捕捉到阵列上。在另一个实例中(未显示),使组织样品与阵列接触,并且通过切割任选的可切割序列120将捕捉探针115从捕捉位点105释放到组织样品中。
图7显示了图5的方法500的步骤的实施方案。在此实施方案中,使用基因特异性捕捉探针捕捉特定cDNA,并且省略图5的任选的等温扩增步骤515。即,组织切片(未显示)包含靶mRNA分子610。靶mRNA分子610可以包含突变615。在步骤510,使用RT引物710原位逆转录靶mRNA分子610。RT引物710包含基因特异性引物区625和SBS引物区720,例如SBS3。RT引物710还可以包含UMI序列(未显示)。使用RT引物710合成的cDNA分子725包含SBS引物区720。在步骤520,将cDNA分子725捕捉到捕捉位点105上。例如,使含有DNA分子725的组织样品或包含源自组织样品的DNA分子725的基底(未显示)与捕捉位点105接触。在此实施方案中,捕捉位点105上的捕捉探针115包含cDNA分子725的3’端的序列互补的基因特异性捕捉区730;SBS引物区125是SBS12。通过使捕捉区730与cDNA分子725的3’端的互补序列杂交在捕捉位点105上捕捉cDNA分子725。
在一些实施方案中,可以使用基于杂交的捕捉将通过原位扩增组织样品中的基因组DNA生成的扩增子转移到阵列。在一些实施方案中,使用“TSCA样”扩增方法产生基因组DNA扩增子。在一些实施方案中,使用DNA-挂锁(Padlock)方法生成基因组DNA扩增子。
图8显示了将DNA扩增子捕捉到阵列(例如图1A的捕捉阵列100)上的方法800的实施方案的流程图,其中通过原位扩增靶核酸生成DNA扩增子。在此实施方案中,扩增反应是TSCA样扩增(TruSeq定制扩增子装配体,Illumina)。方法800包括但不限于一些或所有以下步骤。
在步骤810,将在感兴趣的区域侧翼的一对基因特异性捕捉寡核苷酸与基因组DNA原位杂交。例如,在感兴趣的区域的5’杂交的第一捕捉寡核苷酸可以包含基因特异性序列和通用捕捉序列。在感兴趣的区域的3’杂交的第二捕捉寡核苷酸可以包含第二基因特异性序列和SBS引物序列(例如SBS12)。
在步骤815,在感兴趣的区域间的侧翼捕捉寡核苷酸之间进行原位延伸/连接反应。
在步骤820,通过原位等温扩增来扩增侧翼有捕捉寡核苷酸的DNA以生成多个拷贝的感兴趣的区域,即,多个基因组扩增子。例如,可以使用与通用捕捉序列互补的引物序列和SBS引物序列进行等温扩增。
在步骤825,将基因组扩增子转移到阵列上,并且通过与阵列上的通用捕捉区杂交来捕捉。
图9显示了图8的方法800的步骤的实施方案。即,组织切片(未显示)包含靶基因组DNA分子910。靶DNA分子910可以包含突变915。在步骤810,将在感兴趣的区域侧翼的第一基因特异性捕捉寡核苷酸920和第二基因特异性捕捉寡核苷酸925与DNA分子910原位杂交。捕捉寡核苷酸920包含基因特异性区域930和通用捕捉区935。捕捉寡核苷酸920也可以包含UMI区(未显示)。捕捉寡核苷酸925包含第二基因特异性区域940和SBS引物区945(例如SBS12)。在步骤815,在感兴趣的区域间的侧翼捕捉寡核苷酸920和925之间原位进行延伸/连接反应。延伸/连接反应中形成的DNA分子950包含通用捕捉区935和SBS引物区945。在步骤820,通过原位等温扩增来扩增DNA分子950以生成靶定的感兴趣的区域的多个拷贝(多个扩增子)。例如,可以使用与通用捕捉区935互补的引物区935a和与SBS引物区945互补的引物区945a进行等温扩增。在步骤825,将基因组扩增子950捕捉到捕捉位点105上。例如,使含有基因组扩增子950的组织样品或包含源自组织样品的基因组扩增子950的基底(未显示)与捕捉位点105接触。在此实例中,捕捉位点105上的捕捉探针115包含与通用捕捉区935互补的捕捉区960;SBS引物区130是SBS3。通过将通用捕捉区935与捕捉区960杂交在捕捉位点105上捕捉DNA扩增子950。
在此实例中,使组织样品与捕捉位点105表面上固定的捕捉探针115接触。通过使通用捕捉序列935与捕捉序列960杂交将基因组扩增子950捕捉到阵列上。在另一个实例中(未显示),使组织样品与阵列接触,并且通过切割任选的可切割序列120将捕捉探针115从捕捉位点105释放到组织样品中。
图10显示了将DNA扩增子捕捉到阵列(例如图1A的捕捉阵列100)上的方法1000的实施方案的流程图,其中通过原位扩增靶核酸生成DNA扩增子。在一些实施方案中,扩增反应可以是DNA挂锁扩增。方法1000可以包括但不限于一些或所有以下步骤。
在步骤1010,将挂锁捕捉探针与基因组DNA原位杂交。例如,挂锁捕捉探针可以包括第一基因特异性序列和SBS引物序列,其经由接头序列连接到通用捕捉序列和第二基因特异性序列。第一基因特异性序列和第二基因特异性序列在基因组DNA中在感兴趣的靶区域侧翼。
在步骤1015,在靶定区域间在挂锁捕捉探针中的侧翼基因特异性序列之间进行原位延伸/连接反应以生成环状分子。
在步骤1020,通过原位滚环扩增来扩增侧翼有挂锁捕捉探针的DNA以生成靶定扩增子的多联体。例如,可以使用与挂锁捕捉探针中的SBS引物序列互补的引物序列进行滚环扩增。
在步骤1025,将靶定扩增子多联体捕捉到包含通用捕捉序列的阵列上。
图11显示了图10的方法1000的步骤。即,组织切片(未显示)包含靶基因组DNA分子1110。靶DNA分子1110可以包含突变1115。在步骤1010,将挂锁捕捉探针1120与DNA分子1110原位杂交。挂锁捕捉探针1120可以包含第一基因特异性区域1125和SBS引物区1130(例如SBS12),其可以经由接头区1135连接到通用捕捉区1140和第二基因特异性区域1145。挂锁捕捉探针1120也可以包含独特的分子标识符(UMI)区(未显示),以促进测序误差的校正。第一基因特异性区域1125和第二基因特异性区域1145在DNA分子1110中的感兴趣的靶区域侧翼。在步骤1015,在靶定的感兴趣的区域间在侧翼第一基因特异性区域1125和第二基因特异性区域1145之间进行原位延伸/连接反应以生成环状分子1150。在一些实施方案中,可以在扩增前使用CircLigaseTM(Epicentre,Illumina)连接环状分子1150。在步骤1020,通过原位滚环扩增来扩增环状分子1150以生成多联体1155,其包含多个拷贝的环状分子1150。使用与环状分子1150上的SBS引物区1130互补的引物序列(未显示)进行滚环扩增。在步骤1025,将多联体1155捕捉到包含可切割区域120、SBS测序引物区125(例如SBS3)、空间地址区130、和捕捉区135(例如通用捕捉序列)的捕捉位点105上。例如,使含有多联体1155的组织样品或包含源自组织样品的多联体1155的基底(未显示)与捕捉位点105接触。在一些实施方案中,捕捉位点105上的捕捉探针115包含与通用捕捉区1140互补的捕捉区1160;SBS引物区包含SBS3序列。通过使通用捕捉区1140与捕捉区1160杂交在捕捉位点105上捕捉多联体1155。
在此实例中,使组织样品与捕捉位点105表面上固定的捕捉探针115接触。通过使通用捕捉序列1140与捕捉序列1160杂交将多联体1155捕捉到阵列上。在另一个实例中(未显示),使组织样品与阵列接触,并且通过切割任选的可切割序列120将捕捉探针115从捕捉位点105释放到组织样品中。
(b)基于连接的核酸捕捉
在一些实施方案中,使用基于连接的捕捉来将组织样品中的靶核酸捕捉到阵列上的捕捉探针上。例如,阵列可以是包含多个捕捉探针的珠或孔或空隙或突出物、平面表面或测序流动池的阵列。在一个实例中,使组织样品与阵列表面上固定的捕捉探针接触。可以将组织样品直接放置在包含捕捉探针的表面上或者可以将组织样品放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织样品与捕捉探针分开,使得靶核酸可以从组织扩散过物质到捕捉探针。
在另一个实例中,使组织样品与阵列接触,并且将阵列上的捕捉探针释放到组织样品中,用于与组织样品中的核酸杂交。可以将组织样品直接放置在包含捕捉探针的表面上或者可以将组织样品放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织样品与捕捉探针分开,使得释放的靶核酸可以从阵列扩散过物质到组织样品中的核酸。可以使用可释放基团或选择性可切割部分或接头在阵列上锚定捕捉探针。可以使用例如化学切割、酶促切割或光切割从阵列释放捕捉探针。在另一个实例中,可以将捕捉探针打印到阵列的表面上并且干燥。可以通过再水合从阵列释放捕捉探针。在又一个实例中,可以使用在存在某种处理的情况下溶解的物质将捕捉探针打印到阵列上。然后,在将组织样品放置到阵列上前应用释放捕捉探针的处理。
在一个实例中,核酸是通过原位逆转录组织样品中的RNA合成的cDNA。在另一个实例中,核酸是通过原位扩增组织样品中的基因组DNA生成的DNA扩增子。
图12显示了通过单链连接将cDNA捕捉到阵列(例如图1A的捕捉阵列100)上的方法1200的实施方案的流程图,其中通过原位逆转录靶RNA分子生成cDNA。方法1200包括但不限于一些或所有以下步骤。
在步骤1210,使用基因特异性引物原位合成cDNA。例如,可以使用包含第一基因特异性引物区和独特的分子标识符(UMI)区的基因特异性逆转录引物(RT)来引发第一链cDNA合成。可以在5’或3’端上修饰cDNA以阻止自身连接。在一些实施方案中,可以在原位cDNA合成前将阻止cDNA自身连接的修饰预先掺入UMI序列中。在一些实施方案中,可以在cDNA合成后添加修饰,诸如“尾部”寡核苷酸区的添加以阻止cDNA的自身连接。
在步骤1215,将cDNA转移到珠阵列,并且通过将cDNA连接到珠阵列上的通用衔接子寡核苷酸捕捉。通用衔接子寡核苷酸可以包含可切割区域、SBS引物序列(例如SBS3)、和空间地址,如参考图1A和图1C描述。
在步骤1220,从珠阵列切割cDNA。
在步骤1225,使用基因特异性引物合成第二链cDNA。基因特异性引物可以包含例如基因特异性序列和SBS测序引物序列(例如SBS12)。
在步骤1230,使用一对SBS引物扩增cDNA以生成测序文库。例如,第一SBS引物可以包含SBS12互补序列和P7序列。第二SBS引物可以包含SBS3互补序列和P5序列。所得的文库扩增子可以在5’端侧翼有P7序列和SBS引物序列,并且在3’端侧翼有UMI、空间地址、SBS测序引物、和P5序列。可以使用P7和P5序列来使DNA扩增子结合流动池表面,用于随后的簇扩增和测序。
在步骤1235,对文库测序。
图13A和13B显示了图12的方法1200的步骤。在一些实施方案中,可以将原位合成的cDNA捕捉到包含图1C的通用捕捉珠150的珠阵列上。即,组织切片(未显示)可以包含靶RNA分子1310。靶RNA分子1310可以包含突变1315。在步骤1210,使用基因特异性RT引物1320原位合成cDNA。RT引物1320可以包含基因特异性区域1325和UMI区1330。使用RT引物1320合成的cDNA分子1335包含UMI区1330。在步骤1215,将cDNA分子1335捕捉到通用捕捉珠150上。例如,使含有cDNA分子1335的组织样品或包含源自组织样品的cDNA分子1335的基底(未显示)与通用捕捉珠150接触。通过将UMI 1330连接到通用衔接子寡核苷酸160中的空间地址区130在通用捕捉珠150上捕捉cDNA分子1335。在步骤1220,从通用捕捉珠150切割cDNA 1335。cDNA分子1335现在包含SBS引物区125(例如SBS3)和空间地址区130。在步骤1225,使用基因特异性引物1340合成第二链cDNA。基因特异性引物1340包含基因特异性区域1345和SBS引物区1350(例如SBS12)。使用基因特异性引物1340合成的第二链cDNA分子1355包含SBS测序引物区1350、UMI区1330、空间地址区130、和SBS引物区125。在步骤1230,使用第一SBS引物1360和第二SBS引物1365扩增cDNA分子1355。SBS引物1360包含与SBS测序引物1350互补的SBS互补区1350a和P7引物区1370。SBS引物1365包含与SBS引物区125互补的SBS互补区125a和P5引物区1375。使用SBS引物1360和1365合成的文库扩增子1380在5’端侧翼有P7引物区1370和SBS引物区1350,并且在3’端侧翼有UMI区1330、空间地址区130、SBS测序引物区125、和P5引物区1375。在步骤1235(未显示),对文库测序。
在此实例中,使组织样品与通用捕捉珠150表面上固定的通用衔接子寡核苷酸160接触。通过将UMI 1330连接到通用衔接子寡核苷酸160中的空间地址130将cDNA分子1335捕捉到阵列上。在另一个实例中(未显示),使组织样品与阵列接触,并且通过切割任选的可切割序列120将通用衔接子寡核苷酸160从阵列释放到组织样品中。
在一些实施方案中,任选地,可以在步骤1225前进行富集靶cDNA序列的锚定PCR步骤(未显示)。例如,首先可以使用没有SBS引物序列1350的基因特异性引物进行锚定PCR扩增。在锚定PCR扩增步骤后,可以使用包含基因特异性区域1345和SBS测序引物区1350的基因特异性引物1340进行第二扩增。
图14显示了将DNA扩增子捕捉到阵列(例如图1A的捕捉阵列100)上的方法1400的实施方案的流程图,其中可以通过原位扩增靶核酸生成DNA扩增子。方法1400可以包括但不限于一些或所有以下步骤。
在步骤1410,使在感兴趣的区域侧翼的一对基因特异性捕捉探针与基因组DNA原位杂交。例如,在感兴趣的区域的5’杂交的第一捕捉寡核苷酸可以包含第一基因特异性序列和通用序列。在感兴趣的区域的3’杂交的第二捕捉寡核苷酸可以包含第二基因特异性序列、UMI、和SBS引物序列(例如SBS3)。
在步骤1415,在感兴趣的区域间在侧翼捕捉探针之间进行原位延伸/连接反应。
在步骤1420,通过原位等温扩增来扩增侧翼有捕捉探针的DNA以生成多个拷贝的感兴趣的区域,即,多个扩增子。例如,可以使用与通用序列互补的引物序列和SBS引物序列进行等温扩增。
在步骤1425,可以将3’尾部寡核苷酸添加到DNA扩增子上以阻止自身连接。
在步骤1430,将DNA扩增子转移到珠阵列(例如图1C中显示的通用捕捉珠150的阵列),并且通过连接捕捉到珠阵列上的通用捕捉寡核苷酸上。例如,可以将DNA扩增子变性,转移到珠阵列,并且连接到通用捕捉寡核苷酸。通用捕捉寡核苷酸可以包含可切割序列、SBS引物序列(例如SBS12)、和空间地址序列,如参考图1C描述。变性的扩增子的这两条链(即,顶部链和底部链)可以连接到珠阵列上的捕捉寡核苷酸。
在步骤1435,从珠阵列切割DNA扩增子。
在步骤1440,使用一对SBS引物扩增DNA扩增子以生成测序文库。例如,第一SBS引物可以包含SBS12互补序列和P7序列。第二SBS引物可以包含SBS3互补序列和P5序列。在一些实施方案中,仅扩增侧翼有SBS12和SBS3序列的靶定序列(即,DNA扩增子的顶部链)。所得的文库扩增子在5’端侧翼有P7和SBS12引物序列,并且在3’端侧翼有UMI、空间地址、SBS3引物、和P5序列。
在步骤1445,对文库测序。
图15A、15B、和15C显示了图14的方法1400的步骤。即,组织切片(未显示)包含靶定DNA区1510。靶DNA分子1510可以包含突变1515。在步骤1410,使在感兴趣的区域侧翼的第一基因特异性捕捉探针1520和第二基因特异性捕捉探针1525与DNA分子1510原位杂交。基因特异性捕捉探针1520包含基因特异性区域1530和通用区1535。基因特异性捕捉探针1525可以包含第二基因特异性区域1540、UMI区1545、和SBS引物区1550(例如SBS3)。在步骤1415,在感兴趣的区域间在侧翼基因特异性捕捉探针1520和1525之间进行原位延伸/连接反应以生成DNA分子1555。DNA分子1555包含通用区1535、UMI区1545、和SBS引物区1550。在步骤1420,扩增(例如原位等温扩增)DNA 1555以生成感兴趣的靶定区域的多个拷贝(即,多个扩增子)。使用与通用区1535互补的引物区1535a和与SBS引物区1550互补的引物区1550a进行扩增,例如等温扩增。在步骤1425,将3’尾部寡核苷酸1560添加到DNA扩增子上以阻止自身连接。在步骤1430,将DNA分子1555变性,并且转移到通用捕捉珠150。例如,使含有DNA分子1555的组织样品或包含源自组织样品的DNA分子1555的基底(未显示)与通用捕捉珠150接触。通过将3’尾部寡核苷酸1560与通用衔接子寡核苷酸160中的空间地址区130连接在通用捕捉珠150上捕捉DNA分子1555的每条链(即,顶部链“A”和底部链“B”)。在步骤1435,从通用捕捉珠150切割DNA分子1555。形成DNA分子1555的两个不同构造:A)DNA分子1565,其包含(以3’至5’方向)SBS引物区125(例如SBS12)、空间地址区130、3’尾部寡核苷酸1560、通用区1535、DNA分子1555、UMI区1545、和SBS引物区1550(例如SBS3)和B)DNA分子1570,其包含(以3’至5’方向)SBS引物区125(例如SBS12)、空间地址区130、3’尾部寡核苷酸1560、SBS引物区1550(例如SBS3)、UMI区1545、DNA分子1555、和通用区1535。在步骤1440,使用第一SBS引物1575和第二SBS引物1580扩增DNA分子1565(A)和1570(B)。第一SBS引物1575包含与SBS引物序列125互补的SBS引物序列125a和P7序列1585。第二SBS引物1580包含与SBS引物序列1550互补的SBS引物序列1550a和P5序列。使用SBS引物1575和1580从DNA分子1565扩增的文库扩增子1595在3’端侧翼有P7区1585、SBS引物区125(例如SBS12)、空间地址区130、3’尾部寡核苷酸1560、和通用区1535,并且在5’端侧翼有UMI区1545、SBS引物区1550(例如SBS3)、和P5引物区1590。由于DNA分子1570中的SBS序列的构造,不扩增DNA分子1570。
在此实例中,使组织样品与通用捕捉珠150表面上固定的通用衔接子寡核苷酸160接触。通过将3’尾部寡核苷酸1560与空间地址130连接在通用捕捉珠150上捕捉DNA分子1555。在另一个实例中(未显示),使组织样品与阵列接触,并且通过切割任选的可切割序列120将通用衔接子寡核苷酸160从阵列释放到组织样品中。
4.3将核酸转移到捕捉阵列上
在本文中所述的方法的一些实施方案中,可以通过被动扩散将核酸分子从样品,诸如组织切片转移到捕捉阵列上。
在一些实施方案中,例如,可以经由电泳或离心促进将核酸分子从样品转移到捕捉阵列上。
在一些实施方案中,可以将核酸分子从样品,诸如组织切片直接转移到捕捉阵列上。例如,可以将组织切片直接放置到捕捉阵列上。
在一些实施方案中,可以将核酸分子从样品,诸如组织切片间接转移到捕捉阵列上。例如,首先可以将来自组织切片的核酸转移到一个或多个中间基底,例如除捕捉阵列以外的任何基底,使得中间基底上的核酸的相对空间取向反映组织切片中的相对空间取向。然后,可以将核酸从中间基底转移到捕捉阵列,使得捕捉阵列上的核酸的相对空间取向反映组织切片中的核酸的相对空间取向。例如,可以经由被动扩散或经由促进转移(例如电泳或离心)发生间接转移。中间基底可以是例如膜,诸如尼龙膜,或凝胶,或微孔板。在一些实施方案中,首先可以将来自组织样品的核酸转移到凝胶,然后转移到一个或多个膜,然后到捕捉阵列。在一些实施方案中,可以将中间基底配制为使得它稳定组织切割的不同空间区域中的核酸的分开。例如,可以将组织切片中的不同空间区域中的核酸永久彼此分开,即通过将组织切片(或包含来自组织切片的核酸的凝胶或膜)的不同片段放入微孔板的不同孔中,使得微孔板中的组织切片片段的相对空间取向可以与组织切片中片段的相对空间取向相关联。随后,可以将微孔板中的组织片段中的核酸从微孔板转移到捕捉阵列。
在一些实施方案中,可以使用中间基底来产生两个或更多个核酸拷贝,其相对空间取向可以与其在组织切片中的相对空间取向相关联。例如,可以将核酸从组织切片转移到几个膜上,例如通过将组织切片放置到形成几个成层膜的第一层的膜上。可以发生从组织切片转移到两个或更多个成层的膜,例如经由被动扩散或它可以被促进。两个或更多个成层膜中每个上的核酸的空间取向对应于组织切片中核酸的相对空间取向。随后,可以将两个或更多个成层膜上的核酸转移到两个或更多个捕捉阵列。
在一些实施方案中,可以使用磁响应性纳米颗粒促进将核酸分子从样品转移到捕捉阵列上。
(a)核酸到捕捉阵列上的促进转移
在一些实施方案中,与在其它方面相当的实验条件(例如转移温度、转移缓冲液等)下被动扩散的核酸转移相比,促进的核酸转移可导致从样品到捕捉阵列的核酸转移的更大产率。在一些实施方案中,与通过被动扩散进行核酸转移相比,促进的核酸转移可以导致大至少2倍,至少3倍,至少4倍,至少5倍,至少6倍,至少7倍,至少8倍,至少9倍,至少10倍,至少15倍,至少20倍,至少25倍,至少50倍或至少100倍的产率。用于分析核酸转移效率的方法是本领域中公知的,例如使用放射性同位素标记的或荧光标记的核酸,或比较下一代测序反应的产率或效率。
在一些实施方案中,与在其它方面相当的实验条件下被动扩散的核酸转移相比,促进的核酸转移可以允许转移时间的减少(例如,转移时间从超过12小时,超过24小时,超过36小时,或超过48小时减少到小于6小时,小于4小时,小于2小时或小于1小时)。在一些实施方案中,与通过被动扩散进行核酸转移相比,促进的核酸转移可以导致至少2倍,至少3倍,至少4倍,至少5倍,至少6倍,至少7倍,至少8倍,至少9倍,至少10倍的转移时间减少。用于分析或比较转移时间的方法在本领域中是公知的。例如,如通过使用放射性同位素标记的或荧光标记的核酸,或者通过比较下一代测量反应的产率或效率测定,转移时间可以表示将一定量的核酸从样品转移到捕捉阵列所需要的时间。
在一些实施方案中,与在其它方面相当的实验条件下被动扩散的核酸转移相比,促进的核酸转移可以允许将核酸从较大的样品(例如较厚的组织切片)转移到捕捉阵列上。在一些实施方案中,与通常在通过被动扩散转移核酸时应用的样品的厚度(例如厚度约10μm至约100μm)相比,促进的核酸转移可以允许从具有大至少2倍,至少3倍,至少4倍,至少5倍,至少6倍,至少7倍,至少8倍,至少9倍或至少10倍厚度的组织切片转移核酸。在一些实施方案中,组织切片的厚度可以小于约5μm。
在一些实施方案中,就捕捉阵列上的某些捕捉位点而言,可以促进从组织样品的核酸转移,而就捕捉阵列上的某些其它捕捉位而言,可以通过被动扩散发生从组织样品的核酸转移。在一些实施方案中,就捕捉位点组的选定亚组而言,可以促进从组织样品到捕捉位点上的核酸转移,例如捕捉位点的至少1%,至少3%,至少5%,至少10%,至少20%,至少30%,至少40%,至少50%,至少60%,至少70%,至少80%,至少90%,至少95%,至少98%,或至少99%的亚组。
(b)用于空间检测和分析组织样品中的核酸的电泳系统
在一些实施方案中,捕捉阵列(即,捕捉位点的阵列)可以与电泳系统整合以迫使核酸分子从组织切片直接移动到捕捉探针上。在一些实施方案中,核酸是RNA。在一些实施方案中,核酸是DNA(例如cDNA或DNA扩增子)。
图16显示了配置用于空间检测和分析组织样品中的核酸的示例性电泳转移系统1600的部分的侧视图。电泳转移系统1600包含捕捉阵列1610。捕捉阵列1610可以是图1A的捕捉阵列100。捕捉阵列1610包含固体支持物1615。在一些实施方案中,固体支持物1615是平面玻璃基底。在固体支持物1615上形成捕捉位点1620的排列(例如行和列)。在一些实施方案中,显示了一行6个捕捉位点1620(即,捕捉位点1620a到1620f),但是可以使用捕捉位点1620的任何数目和构造。在每个捕捉位点1620处固定化多个寡核苷酸(未显示)。与每个捕捉位点1620结合的是底部电极1625(例如六个底部电极1625a到1625f)。在此实例中,显示了每个捕捉位点1620的一个底部电极1625,但是可以使用每个捕捉位点1620的任何数目的底部电极1625。例如,捕捉阵列1610可以包含每个捕捉位点162的2个底部电极1625,或者每个捕捉位点1620的10个底部电极1625,或者每个捕捉位点1620的100个底部电极1625,或者每个捕捉位点1620的任何数目的底部电极1625。样品基底1630位置捕捉阵列1610上。在一个实例中,样品基底1630是平面玻璃基底。样品基底1630包含顶部电极1635的排列。在此实例中,顶部电极1635的排列对应于捕捉阵列1610上底部电极1625的排列,即,每个底部电极1625(例如底部电极1625a到1625f)的一个顶部电极1635(例如顶部电极1635a到1635f)。在另一个实例中(未显示),样品基底1630包含单一顶部电极1635。在面向捕捉阵列1610的捕捉位点1620的样品基底1630表面上安装组织样品1640。
配置捕捉位点1620、底部电极1625和顶部电极1635以进行电泳转移和捕捉来自组织样品的核酸,使得维持空间取向,并且消除或实质性降低来自组织样品的核酸的扩散和捕捉位点1620之间的核酸损失。可以单独寻址(装载)每个捕捉位点1620或者可以以单一单元共同寻址所有或选定组的捕捉位点1620。在底部电极1625和顶部电极1635间连接电压源1645。在存在由电压源1645供应的电场的情况下,将多个核酸1650从组织样品1640转移到捕捉位点1620。通过与捕捉位点1620处固定的捕捉探针(未显示)杂交在捕捉位点1620处捕捉核酸1650。
4.4使用捕捉探针组来空间检测和分析组织样品中的核酸
根据本文中所述的方法,可以使用两个或更多个捕捉探针(例如3或更多个、4或更多个、5或更多个、6或更多个、7或更多个、8或更多个、9或更多个、或10或更多个捕捉探针)的组进行空间检测和分析组织样品中的核酸。通常,至少在捕捉阵列上固定化捕捉探针组中的第一捕捉探针。在一些实施方案中,可以与第一捕捉探针在相同捕捉阵列上固定化第二捕捉探针,例如在第一捕捉探针附近,例如在相同捕捉位点中。在一些实施方案中,可以在颗粒,诸如磁性颗粒或磁性纳米颗粒上固定化第二捕捉探针。参见例如5.6节。在一些实施方案中,第二捕捉探针可以在溶液中,例如以用于与组织样品中的核酸进行原位反应。
单独且独立地,捕捉探针组中的捕捉探针可以具有多个不同区域,例如捕捉区(例如通用或基因特异性捕捉区)、引物结合区(例如SBS引物区,诸如SBS3或SBS12区,或另一个通用区,诸如P5或P7区)、空间地址区(例如部分或组合空间地址区)、或可切割区域。
在一些实施方案中,捕捉探针组中仅一个捕捉探针包含捕捉区。在一些实施方案中,捕捉探针组中两个或更多个捕捉探针包含捕捉区。
在一些实施方案中,捕捉探针组中仅一个探针包含空间地址区,例如诸如完整的空间地址区,其描述捕捉阵列上捕捉位点的位置。在一些实施方案中,捕捉探针组中的两个或更多个探针可以包含空间地址区,例如两个或更多个探针可以各自包含部分空间地址区(即,组合地址区),其中每个部分地址区描述了捕捉阵列上捕捉位点的位置,例如沿着x-轴或y-轴。
在一些实施方案中,捕捉探针组可以包含至少一个捕捉探针,其包含捕捉区和空间地址区(例如完整或部分空间地址区)。在一些实施方案中,捕捉探针组中无捕捉探针包含捕捉区和空间地址区两者。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的捕捉阵列,其包括包含捕捉探针组的捕捉位点。在一些实施方案中,捕捉探针组包含至少两个捕捉探针(即,至少捕捉探针对)。在一些实施方案中,捕捉探针组包含3或更多个、4或更多个、5或更多个、6或更多个、7或更多个、8或更多个、9或更多个、或10或更多个捕捉探针。在一些实施方案中,捕捉阵列可以在本文中所述的电泳转移系统中整合。参见例如5.3节。
图17显示了捕捉阵列(例如图1A的捕捉阵列100)上的一个捕捉位点1700的侧视图,其中一个捕捉位点1700包含不同的两组固定化捕捉探针。在一些实施方案中,固定化捕捉探针组包含两个捕捉探针(即,捕捉探针对).在一些实施方案中,固定化捕捉探针组包含三个或更多个捕捉探针。举例而言,
图17显示了捕捉阵列的捕捉位点1700a。第一组捕捉探针1710包含SBS引物区1715(例如SBS3)和空间地址区1720。捕捉位点1700a处固定化的每个捕捉探针1710包含相同的独特空间地址区1720。其它捕捉位点1700(未显示),例如捕捉位点1700b到1700f处固定化的捕捉探针1710各自包含其自身的独特空间地址区1720(例如空间地址区1720b到1720f),即,每个捕捉位点1700具有独特的空间地址区。第二组捕捉探针1725包含第二SBS引物区1730(例如SBS12)和基因特异性捕捉区1735,例如基因特异性捕捉区1735a和基因特异性捕捉区1735b。在一些实施方案中,第二组捕捉探针不包含空间地址序列。在此实例中,显示了两个空间寻址捕捉探针1710和捕捉探针1725,但是可以在捕捉位点1700a处固定化任何数目的空间寻址捕捉探针1710和捕捉探针1725。在一些实施方案中,图17中显示的捕捉阵列可以在图16的电泳转移系统1600中整合。
由于使用不同的至少两组捕捉探针,例如在图17中显示的实施方案中,实现基因特异性捕捉需要的寡核苷酸的数目实质性减少。例如,在常规方法中,对于20,000个捕捉位点处来自100个不同基因的RNA,将需要200万个不同空间寻址捕捉寡核苷酸。然而,根据本文中所述的方法,为了在20,000个捕捉位点处捕捉来自100个不同基因的RNA,仅需要20,000个空间寻址寡核苷酸和100个捕捉寡核苷酸。
在一些实施方案中,捕捉阵列包含捕捉位点(例如1700a),其包含表面上固定化的捕捉探针对(例如1710和1725a),其中捕捉探针对的第一捕捉探针(例如1710)包含第一引物结合区(例如SBS引物结合区1715;例如SBS3)和空间地址区(例如空间地址区1720),且其中捕捉探针对的第二捕捉探针(例如1725a)包含第二引物结合区(例如SBS引物结合区1730;例如SBS12)和捕捉区(例如1735a)。
在一些实施方案中,第一捕捉探针不包含基因特异性区域。
在一些实施方案中,第二捕捉探针不包含空间地址区。
在一些实施方案中,捕捉位点是多个捕捉位点。在一些实施方案中,多个捕捉位点是2或更多个、10或更多个、30或更多个、100或更多个、300或更多个、1,000或更多个、3,000或更多个、10,000或更多个、30,000或更多个、100,000或更多个、300,000或更多个、1,000,000或更多个、3,000,000或更多个、或10,000,000或1,000,000,000或更多个捕捉位点。
在一些实施方案中,捕捉阵列包含每平方厘米(cm2)的1或更多个、2或更多个、10或更多个、30或更多个、100或更多个、300或更多个、1,000或更多个、3,000或更多个、10,000或更多个、100,000或更多个、1,000,000或更多个捕捉位点的捕捉位点密度。
在一些实施方案中,捕捉位点中的捕捉探针对是多个捕捉探针对。在一些实施方案中,多个捕捉探针是2或更多个、10或更多个、30或更多个、100或更多个、300或更多个、1,000或更多个、3,000或更多个、10,000或更多个、30,000或更多个、100,000或更多个、300,000或更多个、1,000,000或更多个、3,000,000或更多个、或10,000,000或更多个、100,000,000或更多个、或1,000,000,000或更多个捕捉探针.
在一些实施方案中,捕捉阵列的捕捉位点中的捕捉探针对是多个捕捉探针对。在一些实施方案中,相同捕捉位点内的多个捕捉探针对中的每个第一捕捉探针包含相同空间地址序列。在一些实施方案中,不同捕捉位点中的多个捕捉探针对中的每个第一捕捉探针包含不同空间地址序列。
在一些实施方案中,捕捉阵列的一个或多个捕捉位点具有相同数目的第一捕捉探针和第二捕捉探针。在一些实施方案中,一个或多个捕捉位点具有比第二捕捉探针多的第一捕捉探针。例如,在一些实施方案中,一个或多个捕捉位点具有比第二捕捉探针多至少2倍、至少3倍、至少10倍、至少30倍、至少100倍、至少300倍、至少1,000倍、至少3,000倍、或至少10,000倍的第一捕捉探针。在一些实施方案中,一个或多个捕捉位点具有比第一捕捉探针多的第二捕捉探针。例如,在一些实施方案中,一个或多个捕捉位点具有比第一捕捉探针多至少2倍、至少3倍、至少10倍、至少30倍、至少100倍、至少300倍、至少1,000倍、至少3,000倍、或至少10,000倍的第二捕捉探针。
在一些实施方案中,捕捉阵列整合到电泳系统中。在一些实施方案中,电泳系统是如5.3节中描述的电泳系统(参见例如图16)。在一些实施方案中,每个捕捉位点在电泳系统中独立可电寻址。在一些实施方案中,电泳系统中的捕捉位点配置用于转移并捕捉来自组织样品的核酸,使得相对于其它方面相同的条件下在缺乏电泳系统的情况下,例如通过扩散发生的被动核酸转移,来自组织样品的核酸的扩散和捕捉位点间的核酸损失实质性减少。
在一些实施方案中,捕捉阵列表面是平面表面,例如玻璃表面。参见例如图1B。在一些实施方案中,捕捉阵列表面包含一个或多个孔。在一些实施方案中,一个或多个孔对应于一个或多个捕捉位点。在一些实施方案中,捕捉阵列表面是珠表面,例如如图1C中显示。
在一些实施方案中,第二捕捉探针中的捕捉区是基因特异性捕捉区。在一些实施方案中,第二捕捉探针中的基因特异性捕捉区包含TruSeqTM定制扩增子(TSCA)寡核苷酸探针(Illumina,Inc.)的序列。例如,捕捉位点中的多个第二捕捉探针中的基因特异性捕捉区可以包含多个TSCA寡核苷酸探针序列。
在一些实施方案中,第二捕捉探针中的捕捉区是通用捕捉区。在一些实施方案中,第二捕捉探针中的通用捕捉区包含随机引物序列。例如,捕捉位点中的多个第二捕捉探针中的捕捉区可以包含随机化序列。在一些实施方案中,第二捕捉探针中的通用捕捉区包含多聚T捕捉序列。例如,捕捉位点中的多个第二捕捉探针中的一些或所有通用捕捉序列可以包含多聚T捕捉序列。
在一些实施方案中,捕捉位点的一个或多个第二捕捉探针中的捕捉区在捕捉阵列的两个或更多个捕捉位点中可以是基本上相同的捕捉区。在一些实施方案中,至少1%、至少5%、至少10%、至少15%、至少20%、至少25%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、或至少99%的第二捕捉探针在捕捉阵列的两个或更多个捕捉位点中具有相同的捕捉区(例如在捕捉阵列上的至少1%、至少5%、至少10%、至少15%、至少20%、至少25%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、或至少99%的捕捉位点中)。
在一些实施方案中,捕捉阵列的一个或多个第二捕捉探针中的捕捉区在捕捉阵列上的至少1%、至少5%、至少10%、至少15%、至少20%、至少25%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%,或至少99%的捕捉位点中可以是基本上相同的捕捉序列。在一些实施方案中,捕捉阵列的一个或多个第二捕捉探针中的捕捉区在捕捉阵列上的基本上所有捕捉位点中可以是基本上相同的捕捉序列。
在一些实施方案中,空间地址区包含可以以组合方式(例如X*Y*Z)组合的两个或更多个部分空间地址区(例如第一、第二和任选地第三部分地址区)。在一些实施方案中,空间地址区包含第一和第二部分地址区以在第一(X)和第二(Y)维度上鉴定捕捉阵列上捕捉位点的位置。在一些实施方案中,空间地址区还包含第三部分地址区以在第三(Z)维度上鉴定组织样品中组织切割(和从组织切片转移的核酸)的位置。
在一些实施方案中,捕捉阵列上的第一或第二捕捉探针还包含时间地址区(T)以鉴定在时间过程实验(例如测定响应化学、生物学或物理刺激物随时间的组织中基因表达的变化的时间过程实验)的过程中获得样品的时间点的相对顺序。
在一些实施方案中,捕捉探针中的两个或更多个地址区(例如空间或时间地址区)是连续的。在一些实施方案中,两个或更多个地址区以一个或多个核酸(例如2或更多个、3或更多个、10或更多个、30或更多个、100或更多个、300或更多个、或1,000或更多个核酸)分开。
图18显示了将核酸从组织样品转移到捕捉阵列以生成空间寻址测序文库的方法1800的实施方案的流程图,其中捕捉阵列包含捕捉位点,其包含不同的固定化捕捉探针对,例如如图17中显示。在此实施方案中,核酸是RNA。在图18中显示的方法的一些实施方案中,捕捉阵列整合到电泳转移系统诸如图16的电泳转移系统1600中。方法1800包括但不限于一些或所有以下步骤。
任选地,在步骤1810,可以将来自组织样品的核酸电泳转移到捕捉阵列。例如并且现在参考图16和图17,可以在捕捉阵列1610上放置其上具有组织样品1640的样品基底1630。在此实例中,捕捉阵列1610的捕捉位点1620包含不同的固定化捕捉探针对,例如如图17中显示。将电场施加到捕捉位点1620。由于活化了捕捉位点1620,将来自组织样品1640的核酸1650转移到捕捉阵列1610,并且与捕捉位点1620处固定化的捕捉探针1725杂交。在此实施方案中,捕捉探针1725是设计为捕捉特定mRNA的基因特异性捕捉探针。
在一些实施方案中,在步骤1810,可以通过被动扩散发生来自样品,诸如组织样品1640的核酸的转移。在一些实施方案中,在步骤1810,通过除电泳以外的方法促进来自样品,诸如组织样品1640的核酸的转移。
在步骤1815,合成第一链cDNA。例如,基因特异性捕捉区1735可以发挥逆转录酶引物的功能以从捕捉的mRNA分子合成第一链cDNA。
在步骤1820,通过将cDNA与空间地址区1720单链连接将第一链cDNA与第二捕捉探针1710共价连接。
在步骤1825,使用与SBS引物区1715互补的引物合成第二链cDNA。
在步骤1830,通过变性从捕捉位点1700释放第二链cDNA分子。
在步骤1835,扩增cDNA以生成测序文库。
图19A、19B、19C、和19D以图像显示了图18的方法1800的步骤。即,在步骤1810,将多个mRNA分子1910从组织切片(未显示)转移(例如电泳)到捕捉位点1700上,并且与捕捉探针1725杂交。在一些实施方案中,第一mRNA分子1910a是来自第一基因的转录物,并且第二mRNA分子1910b是来自第二基因的转录物。mRNA分子1910a可以包含突变1915a。mRNA分子1910b可以包含突变1915b。通过使互补mRNA序列与基因特异性捕捉区1735a杂交在捕捉探针1725a上捕捉mRNA分子1910a。类似地,通过使互补mRNA序列与基因特异性捕捉区1735b杂交在捕捉探针1735b上捕捉mRNA分子1910b。
在步骤1815,使用基因特异性捕捉区1735作为引物合成第一链cDNA。cDNA分子1920(即,cDNA分子1920a和1920b)包含SBS引物区1730。
在步骤1820,通过将cDNA分子1920与空间地址区1720单链连接将cDNA分子1920与空间寻址寡核苷酸1710共价连接。
在步骤1825,使用与SBS引物区1715互补的引物区1715a合成第二链cDNA。
在步骤1830,通过变性从捕捉位点1700释放第二链cDNA分子1920。cDNA分子1920现在包含SBS引物区1715、空间地址区1720、和SBS引物区1730。
在步骤1835,扩增cDNA分子1920以生成测序文库。在第一扩增反应中,使用第一SBS引物1925扩增cDNA分子。SBS引物1925包含与SBS引物区1730互补的SBS互补区1730a和P7区1930。扩增子1935(即,扩增子1935a和1935b)在5’端侧翼有P7区1930和SBS引物区1730,并且在3’端侧翼有空间地址区1720和SBS引物区1715。使用第二SBS引物1940扩增扩增子1935。SBS引物1940包含与SBS引物区1715互补的SBS互补区1715a和P5区1945。扩增子1935(即,扩增子1935a和1935b)现在侧翼有P7区1930和SBS引物区1730并且侧翼有空间地址区1720、SBS引物区1715、和P5区1945。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的方法,包括提供本文中所述的捕捉阵列。在一些实施方案中,捕捉阵列包含捕捉位点,其包含捕捉探针组。在一些实施方案中,捕捉探针组包含两个捕捉探针(即,捕捉探针对)。在一些实施方案中,捕捉探针组包含3个或更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、或10个或更多个捕捉探针。在一些实施方案中,捕捉阵列可以在本文中所述的电泳转移系统中整合。参见例如5.3节。
在一些实施方案中,方法包括(a)提供捕捉阵列,所述捕捉阵列包含捕捉位点,所述捕捉位点包含表面上固定化的捕捉探针对(例如1710和1725a),其中捕捉探针对的第一捕捉探针包含第一引物结合区(例如1715)和空间地址区(例如1720),且其中捕捉探针对的第二捕捉探针包含第二引物结合区(例如1730)和捕捉区(例如1735a)。
在一些实施方案中,第一捕捉探针不包含捕捉区。
在一些实施方案中,第二捕捉探针不包含空间地址区。
在一些实施方案中,所述方法还包括以下步骤中的任何一个或多个:(b)使所述捕捉阵列与组织样品接触,使得所述阵列上的捕捉位点的位置可以与组织样品中的位置相关联;(c)允许组织样品的核酸与第二捕捉探针的捕捉区杂交;(d)延伸第二捕捉探针的捕捉区以形成与基因特异性序列杂交的核酸的固定化第一互补链;(e)将固定化第一互补链与第一捕捉探针的空间地址序列连接以在两个末端固定化第一互补链;(f)使用与所述第一捕捉探针的第一引物结合序列互补的引物合成第二互补链;(f)从捕捉阵列的表面释放第二互补链;(g)分析释放的第二互补链的序列,并且(h)将释放的第二互补链的序列与组织样品中的核酸位置相关联。
在一些实施方案中,允许组织样品的核酸与第二捕捉探针的捕捉区杂交包括将核酸从组织样品电泳转移到捕捉阵列上。
在一些实施方案中,允许组织样品的核酸与第二捕捉探针的捕捉区杂交包括将核酸从组织样品被动扩散到捕捉阵列上。
在一些实施方案中,分析释放的互补链的序列包括下一代测序,例如通过合成测序。
在一些实施方案中,组织样品的核酸包括信使核糖核酸(mRNA)。
在一些实施方案中,第一或第二引物结合区包含SBS引物序列。在一些实施方案中,SBS引物序列是SBS3或SBS12序列。
在一些实施方案中,第二捕捉探针的捕捉区包含单核苷酸变异(SNV)。在一些实施方案中,方法具有至少0.00025%SNV(1/400,000个细胞)的SNV检测灵敏性。下文参考表3更为详细描述了用于检测单核苷酸变异的空间NGS的灵敏性。
在一些实施方案中,第二捕捉探针中的捕捉区是通用捕捉区。在一些实施方案中,第二捕捉探针中的通用捕捉区包含随机引物序列。在一些实施方案中,捕捉位点中的多个第二捕捉探针中的捕捉区包含10或更多个、100或更多个、1,000或更多个、10,000或更多个、100,000或更多个、或1,000,000或更多个随机化捕捉序列。在一些实施方案中,第二捕捉探针中的通用捕捉区包含多聚T捕捉序列。
在一些实施方案中,第二捕捉探针中的捕捉区是基因特异性捕捉区。在一些实施方案中,第二捕捉探针中的基因特异性捕捉区包含TSCA寡核苷酸探针的序列。在一些实施方案中,捕捉位点中的多个第二捕捉探针中的捕捉区包含10或更多个、100或更多个、1,000或更多个、10,000或更多个、100,000或更多个、或1,000,000或更多个TSCA捕捉序列。
在一些实施方案中,捕捉探针组中的至少一个捕捉探针在溶液中,例如与组织样品中的核酸杂交。图20显示了捕捉组织样品中的核酸以随后锚定到阵列上的过程2000的示例性实施方案。在此实施方案中,捕捉探针2010包含核酸捕捉区2015和通用阵列捕捉区2020。在一个实例中,核酸捕捉区2015是随机引物序列。在另一个实例中,核酸捕捉区2015是基因特异性引物序列。通用阵列捕捉区2020是通用序列,其用于将核酸分子锚定到捕捉阵列上。可以在基于溶液的杂交反应中使用捕捉探针2010以捕捉组织切片中的核酸分子2025。在一个实例中,核酸分子2025是基因组DNA分子。捕捉探针2010与来自组织样品的核酸分子2025杂交,并且延伸以形成与核酸分子2025(通过箭头标示)互补的核酸,并且将通用阵列捕捉区2020掺入互补链中。然后,使用通用阵列捕捉区2020将复制的核酸分子锚定到捕捉阵列(未显示)上。在另一个实例中,核酸分子2025是RNA分子。捕捉探针2010与核酸分子2025杂交,并且在逆转录反应中用作引物以合成第一链cDNA(通过箭头标示),并且将通用阵列捕捉区2020掺入cDNA分子中。然后,使用通用阵列捕捉区2020将cDNA分子锚定到捕捉阵列(未显示)上。
在一些实施方案中,方法包括(a)提供捕捉阵列,其包含捕捉位点,所述捕捉位点包含表面上固定化的第一捕捉探针,其中所述捕捉探针包含可切割区域、第一引物结合区和空间地址区。
在一些实施方案中,第一捕捉探针不包含捕捉区。
在一些实施方案中,所述方法还包括以下一个或多个步骤:(b)使组织样品与第二捕捉探针接触,其中第二捕捉探针包含第二引物结合区和捕捉区(例如基因特异性或通用捕捉区);(c)允许组织样品核酸与第二捕捉探针的捕捉区杂交;(d)延伸第二捕捉探针的捕捉区以形成与核酸杂交的核酸的第一互补链。
在一些实施方案中,第二捕捉探针不包含空间地址区。
在一些实施方案中,所述方法还包括以下一个或多个步骤:(e)任选地,使互补寡核苷酸与捕捉探针的空间地址区杂交以形成双链空间地址区;(f)使捕捉阵列与包含核酸的第一互补链的组织样品接触使得所述阵列上的捕捉位点的位置可以与组织样品中的位置相关联;(g)允许核酸的第一互补链从组织样品转移到捕捉阵列上;(h)将核酸的第一互补链(其任选与核酸杂交)连接到第一捕捉探针(例如通过平端连接,诸如双链片段连接)以形成空间标签化双链核酸,其包含第一和第二引物结合位点和可切割域;(g)从捕捉阵列表面释放双链核酸;(h)分子释放的双链核酸的序列,并且(i)将释放的核酸的序列与组织样品中的核酸的位置相关联。
在一些实施方案中,允许核酸的第一互补链从组织样品转移到捕捉阵列上包括将核酸从组织样品电泳转移到捕捉阵列上。
在一些实施方案中,允许核酸的第一互补链从组织样品转移到捕捉阵列上包括将核酸从组织样品被动扩散到捕捉阵列上。
在一些实施方案中,释放的双链核酸包括下一代测序,例如通过合成测序。
在一些实施方案中,组织样品的核酸包含信使核糖核酸(mRNA)。
在一些实施方案中,第一或第二引物结合区包含SBS引物序列。在一些实施方案中,SBS引物序列是SBS3或SBS12序列。
4.5组合索引化系统
在另一个实施方案中,使用组合索引化(寻址)系统来提供空间信息以分析组织样品中的核酸。在此方法中,在测序文库制备过程中将两个或更多个空间地址序列掺入核酸中。使用第一空间地址在捕捉阵列上在X维度上限定某个部分(即,捕捉位点),并且使用第二空间地址序列在捕捉阵列上在Y维度上限定部分(即,捕捉位点)。在文库测序期间,测定X和Y空间地址序列两者,并且分析序列信息以限定捕捉阵列上的特定部分。
在一个实例中,使组织样品与阵列表面上固定的捕捉探针接触。可以将组织样品直接放置在包含捕捉探针的表面上或者可以将组织样品放置在诸如滤器或凝胶或薄缓冲层的物质上,将组织样品与捕捉探针分开,使得靶核酸可以从组织扩散过物质到捕捉探针。
图21A和21B分别显示了一维索引化方案的网格阵列2100和二维索引化方案的网格阵列2105,用于空间检测和分析组织样品中的核酸。参考图21A,为了在一维索引化方案中空间寻址100位置,需要100个独特的空间地址。现在参考图21B,为了在二维索引化方案中空间寻址100位置,对于X维度需要10个独特的空间地址并且对于Y维度需要10个独特的空间地址,即,需要的总共独特空间地址序列是20。组合索引化基本上降低空间检测和分析组织样品中的核酸需要的空间地址的数目。
在一些实施方案中,阵列上的捕捉位点包含对于X维度具有第一空间地址序列的第一捕捉探针和对于Y维度具有第二空间地址序列的第二捕捉探针。在一些实施方案中,第一和第二捕捉探针在捕捉位点上以相反方向取向,使得捕捉RNA分子的5和3端两者。在随后的文库制备步骤期间,将第一和第二空间地址序列掺入文库扩增子中,如参考图22和图23A和23B更为详细描述。
在一些实施方案中,第一和第二捕捉探针在捕捉位点上以相同方向取向,使得仅仅捕捉mRNA分子的3端。在随后的文库制备步骤期间,将第一和第二空间地址序列掺入文库扩增子中,如参考图24和图25A和25B更为详细描述。
在一些实施方案中,可以将两个或更多个部分地址区(例如第一和第二部分地址区)掺入单一捕捉探针中。两个或更多个部分地址区可以形成连续区或者以一个或多个核酸分开。例如,可以将两个或更多个部分地址区掺入捕捉探针对的单一第一捕捉探针中,根据例如图17-19中显示的捕捉阵列和方法。
图22显示了使用组合索引化系统以生成空间寻址cDNA测序文库的方法2200的实例的流程图。方法2200包括但不限于一些或所有以下步骤。
在步骤2210,使包含多个mRNA分子的组织样品与捕捉阵列接触。捕捉阵列可以是例如图1A的捕捉阵列100。对于每个基因特异性(即,特异性靶定)mRNA分子,使用两个捕捉探针,即,包含对mRNA分子的3端特异性的序列的第一捕捉探针和包含对mRNA分子的5端特异性的序列的第二捕捉探针。通过将mRNA与捕捉探针上的基因特异性捕捉区将mRNA分子捕捉到阵列上。参考图23A更为详细描述了捕捉阵列100上的捕捉探针的实例。
在步骤2215,合成第一链cDNA。例如,使用第一捕捉探针上的基因特异性捕捉区作为逆转录酶引物以从捕捉的mRNA分子合成第一链cDNA。然后,将cDNA分子连接到第二捕捉探针的5端。
在步骤2220,从捕捉阵列释放cDNA分子。例如,使用切割反应从捕捉阵列释放cDNA分子。
在步骤2225,扩增cDNA以生成测序文库。
图23A和23B显示了图22的方法2200的步骤。在此实例中,显示了图1A的捕捉阵列100的单一捕捉位点105。捕捉位点105包含第一捕捉探针2310a和第二捕捉探针2310b。捕捉探针2310包含可切割域2315、SBS引物区2320、空间地址区2325、独特的分子标识符(UMI)2330、和捕捉区2335。例如,捕捉探针2310a包含可切割区域2315;SBS引物区2320a,其包含例如SBS3序列;空间地址区2325a,其对于X维度包含独特的空间地址序列;UMI区2330a,其包含捕捉探针2310a的独特序列;和基因特异性捕捉区2335a,其对mRNA分子的3端是特异性的。以5至3取向在捕捉位点105处固定化捕捉探针2320a(即,将捕捉探针2320a的5端附着到捕捉位点105表面)。类似地,捕捉探针2310b包含可切割序列2315;SBS引物区2320b,其包含SBS12序列;空间地址区2325b,其对于Y维度包含独特空间地址区;UMI区2330b,其包含捕捉探针2310b的独特序列;和基因特异性捕捉区2335b,其对mRNA分子的5端是特异性的。以3至5取向在捕捉位点105处固定化捕捉探针2320b(即,将捕捉探针2320a的3端附着到捕捉位点105表面)。
在步骤2210,在捕捉探针2310上捕捉组织样品中的mRNA分子2340。例如,使含有mRNA分子2340的组织样品或包含源自组织样品的mRNA分子2340的基底(未显示)与捕捉探针2310接触。mRNA分子2340可以包含突变2345。通过将mRNA分子2340的3端与捕捉区2335a杂交并且将mRNA分子2340的5端与捕捉区2335b杂交在捕捉位点105处捕捉mRNA分子2340。
在步骤2215,在逆转录反应中使用捕捉区2335a作为引物合成cDNA分子2350。然后,将cDNA分子2350的3端连接到捕捉区2335b的5’端。
在步骤2220,通过切割可切割区域2315从捕捉位点105释放cDNA分子2350。cDNA分子2350包含SBS引物区2320a(即,SBS3)、空间地址区2325a(即,对于X维度的独特空间地址)、UMI区2330a、UMI区2330b、空间地址区2325b(即,对于Y维度的独特空间地址)、和SBS引物区2320b(即,SBS12)。
在步骤2225,使用SBS引物2355和第二SBS引物2360扩增cDNA分子2350。SBS引物2355包含与SBS引物区2320a互补的SBS序列2320a和P5区2365。SBS引物2360包含与SBS引物区2320b互补的SBS区2320b和P7区2370。使用SBS引物2355和2360合成的文库扩增子2375在5端侧翼有P5区2365、SBS引物区2320a(即,SBS3)、空间地址区2325a(即,对于X维度的独特空间地址)、UMI区2330a,并且在3端侧翼有UMI区2330b、空间地址区2325b(即,对于Y维度的独特空间地址)、SBS引物区2320b(即,SBS12)、和P7区2370。
图24显示了使用组合索引化系统生成空间寻址cDNA测序文库的备选方法2400的实例的流程图。方法2400包括但不限于一些或所有以下步骤。
在步骤2410,使包含多个mRNA分子的组织样品与捕捉阵列接触。捕捉阵列可以是例如图1A的捕捉阵列100。对于每个基因特异性mRNA分子,使用两个捕捉探针,即,包含对mRNA分子的3端特异性的序列的第一捕捉探针和包含对相应第一链cDNA分子的3端特异性的序列的第二捕捉探针。通过将mRNA的3端与第一捕捉探针上的基因特异性区域杂交将mRNA分子捕捉到阵列上。参考图25A更为详细描述了捕捉阵列100上的捕捉探针的实例。
在步骤2415,合成第一链cDNA。例如,第一捕捉探针上的基因特异性捕捉区用作逆转录酶引物以从捕捉的mRNA分子合成第一链cDNA。
在步骤2420,在第二捕捉探针上捕捉第一链cDNA。通过将cDNA的3端与第二捕捉探针上的基因特异性区域杂交在第二捕捉探针上捕捉第一链cDNA。
在步骤2425,合成第二链cDNA。例如,第二捕捉探针上的基因特异性区域用作引物以合成第二链DNA分子。
在步骤2430,从捕捉阵列释放第二链cDNA分子。例如,使用切割反应从捕捉阵列释放cDNA分子。
在步骤2435,扩增cDNA分子以生成测序文库。
图25A、25B、和25C显示了图24的方法2400的步骤。在此实例中,显示了图1A的捕捉阵列100的单一捕捉位点105。捕捉位点105基本上是相同的,如上文参考图23A描述,只是捕捉探针2310在捕捉位点105处以相同的方向取向,即,以5至3方向在捕捉位点105处固定化捕捉探针2310a和捕捉探针2310b两者(即,将捕捉探针2310a和2310b的5端附着到捕捉位点105表面)。捕捉区2335a对mRNA分子的3端是特异性的并且捕捉区2335b对相应的第一链cDNA分子的3端是特异性的。
在步骤2410,在捕捉探针2310a上捕捉组织样品中的mRNA分子2510。例如,将含有mRNA分子2510的组织样品或包含源自组织样品的mRNA分子2510的基底(未显示)与捕捉探针2310接触。mRNA分子2510可以包含突变2515。通过将mRNA分子2510的3端与捕捉区2335杂交在捕捉位点105处捕捉mRNA分子2510。
在步骤2415,在逆转录反应中使用捕捉区2335a作为引物合成第一链cDNA。
在步骤2420,通过将cDNA分子2520的3端与捕捉区2335b杂交在捕捉探针2310b上捕捉第一链cDNA分子2520。
在步骤2425,在使用捕捉区2335b作为引物在延伸反应中合成第二链cDNA。
在步骤2430,通过切割可切割区域2315从捕捉位点105释放cDNA分子2525。cDNA分子2525包含SBS引物区2320a(即,SBS3)、空间地址区2325a(即,对于X维度的独特的空间地址)、UMI区2330a、UMI区2330b、空间地址区2325b(即,对于Y维度的独特的空间地址)、和SBS引物区2320b(即,SBS12)。
在步骤2435,使用SBS引物2530和第二SBS引物2535扩增cDNA分子2525。SBS引物2530包含与SBS引物区2320a互补的SBS区2320a和P5区2540。SBS引物2535包含与SBS引物区2320b互补的SBS区2320b和P7区2545。使用SBS引物2530和2535合成的文库扩增子2550在5端侧翼有P5区2540、SBS引物区2320a(即,SBS3)、空间地址区2325a(即,对于X维度的独特的空间地址)、UMI区2330a,并且在3端侧翼有UMI区2330b、空间地址区2325b(即,对于Y维度的独特的空间地址)、SBS引物区2320b(即,SBS12)、和P7区2545。
在一些实施方案中,组合索引化系统可以涉及使用两种不同的阵列,即,包含对于X维度的空间地址序列的第一阵列和包含对于Y维度的空间地址序列的第二阵列。在一个实例中,可以使用第一阵列来将逆转录(RT)引物递送到组织样品以原位合成cDNA,并且使用第二阵列来捕捉cDNA以生成空间寻址测序文库。
图26A显示了用于将RT引物递送到组织样品以原位合成cDNA的阵列2600的实例的平面图。阵列2600包含固体支持物2610上的递送位点2605的排列,例如行。在此实例中,在固体支持物2610上排列10个递送位点2605(例如递送位点2605a到2605j)。在一个实例中,固体支持物2610是玻璃盖玻片。沿着每个递送位点2605的长度以条沉积(例如打印)多个RT引物2615。在每个递送位点2605处沉积RT引物2615,使得它们可以容易从阵列2600释放到组织样品上。
图26B显示了阵列2600的一个递送位点2605的部分的侧视图,其中递送位点2605的部分包含至少一个RT引物2615以从组织样品中的mRNA合成cDNA。在此实例中,显示了单一RT引物2615,但是可以在每个递送位点2605处在固体支持物2610上沉积任何数目的RT引物2615。RT引物2615包含SBS引物区2620(例如SBS3)、空间地址区2625(即,对于X维度的独特的空间地址)、和基因特异性引物区2630。RT引物2615具有对于每个递送位点2605的独特的空间地址区2625,即,递送位点2605a具有独特的空间地址区2625a,递送位点2605b具有独特的空间地址区2625b,等等。基因特异性引物区2630可以是相同的基因特异性区域或者它们可以是不同的基因特异性序列。
图27A显示了用于捕捉使用图26B的RT引物2615原位合成的cDNA的捕捉阵列2700的实例的平面图。捕捉阵列2700包含固体支持物2710上捕捉位点2705的排列,诸如列。在此实例中,在固体支持物2710上排列10个捕捉位点2705(例如捕捉位点2705a到2705j)。在一个实例中,固体支持物2710是平面玻璃表面。沿着每个捕捉位点2705的长度以条沉积多个捕捉探针2715。
图27B显示了捕捉阵列2700的一个捕捉位点2705的部分的侧视图,其中捕捉位点2705的部分包含至少一个捕捉探针2715以捕捉使用图26B的RT引物2615原位合成的cDNA。在此实例中,显示了单一捕捉探针2715,但是可以在每个捕捉位点2705处在固体支持物2710上固定化任何数目的捕捉探针2715。捕捉探针2715包含可切割区域2720、SBS引物区2725(例如SBS12)、空间地址区2730、独特的分子标识符(UMI)区2735、和基因特异性捕捉区2740。捕捉探针2715具有每个捕捉位点2705的独特的空间地址区2730,即,捕捉位点2705a具有独特的空间地址区2730a,捕捉位点2705b具有独特的空间地址区2730b,等等。捕捉区2740与图26B的捕捉探针2615的捕捉区2630互补。
图28显示了生成空间寻址测序文库的方法2800的实例的流程图,其中使用第一阵列原位合成第一链cDNA,并且使用第二阵列捕捉cDNA以进行随后的文库生成。方法2800包括但不限于以下步骤。
在步骤2810,使包含多个mRNA分子的组织样品与第一阵列接触。阵列是例如图26A的阵列2600,其包含多个RT引物2615。将RT引物2615从阵列2600释放到组织样品上。
在步骤2815,原位合成第一链cDNA。例如,使用RT引物上的基因特异性引物序列从组织样品中的靶定mRNA分子引发第一链cDNA。在合成第一链cDNA后,从组织样品表面取出阵列2600。
在步骤2820,将第一链cDNA捕捉到第二阵列上,并且合成第二链cDNA。例如,将包含多个捕捉探针2715的图27A的捕捉阵列2700与组织样品接触。通过将cDNA的3端与捕捉探针2715上的基因特异性区域杂交将第一链cDNA捕捉到捕捉阵列2700上。在延伸反应中使用捕捉探针2715上的基因特异性区域作为引物合成第二链cDNA。
在步骤2825,从捕捉阵列释放第二链cDNA分子。例如,使用切割反应从捕捉阵列释放cDNA分子。
在步骤2830,扩增cDNA分子以生成测序文库。
图29A和29B显示了图28的方法2800的步骤。在步骤2810,使包含mRNA分子2910的组织样品(未显示)与阵列2600(未显示)接触。例如,使含有mRNA分子2910的组织样品或包含源自组织样品的mRNA分子2910的基底(未显示)与阵列2600接触。mRNA分子2910可以包含突变2915。将RT引物2615从阵列2600释放到组织样品上,并且经由基因特异性区域2630与mRNA分子2910杂交。
在步骤2815,在逆转录反应中使用基因特异性区域2630作为引物原位合成第一链cDNA分子2920。
在步骤2820,使包含多个捕捉探针2715的图27A捕捉阵列2700与组织样品接触。通过将第一链cDNA 2920的3端与基因特异性捕捉区2740杂交在捕捉位点2705处捕捉第一链cDNA 2920。在延伸反应中使用基因特异性捕捉区2740作为引物合成第二链cDNA。
在步骤2825,通过切割可切割区域2720从捕捉位点2705释放第二链cDNA 2925。cDNA分子2925包含SBS引物区2725(SBS12)、空间地址区2730(即,对于Y维度的独特的空间地址)、UMI区2735、空间地址区2625(即,对于X维度的独特的空间地址)、和SBS引物区2620(SBS3)。
在步骤2830,使用SBS引物2930和SBS引物2935扩增cDNA分子2925。SBS引物2930包含与SBS引物区2725互补的SBS区2725a和P5区2940。SBS引物2935包含与SBS引物区2620互补的SBS区2620a和P7区2945。使用SBS引物2930和2935合成的文库扩增子2950在5端侧翼有P5区2940、SBS引物区2725(即,SBS3)、空间地址区2730(即,对于Y维度的独特的空间地址)、UMI区2735,并且在3端侧翼有空间地址区2625(即,对于X维度的独特的空间地址)、SBS引物区2620(即,SBS3)、和P7区2945。
在此实例中,将组织样品与捕捉位点2705表面上固定的捕捉探针2715接触。通过将第一链cDNA 2920的3端与基因特异性捕捉序列2740杂交在捕捉位点2705处捕捉第一链cDNA 2920。在另一个实例中(未显示),使组织样品与捕捉阵列2700接触,并且通过切割可切割序列2720将捕捉探针2715从捕捉位点2705释放到组织样品中。使用基因特异性捕捉序列2740作为引物在延伸反应中合成第二链cDNA。
4.5使用可释放捕捉探针空间检测和分析组织样品中的核酸
在其它实施方案中,使用空间寻址阵列将捕捉探针释放到组织切片中以生成空间寻址测序文库。在此方法中,在独特的捕捉位点或“特征”处在基底(例如玻璃盖玻片)表面上沉积空间寻址捕捉探针。在一个实例中,通过形成可切割的键将空间寻址捕捉探针锚定到基底的表面上。通过切割可逆键将空间寻址捕捉探针释放到组织切片中,并且在随后的生化加工步骤中掺入核酸中。在一些实施方案中,在基质,诸如BioGel基质中悬浮的基底上沉积空间寻址捕捉探针。将BioGel中悬浮的空间寻址捕捉探针释放到组织切片中,例如,通过应用热处理或化学处理。使用可切割键或BioGel悬浮在基底表面上固定化空间寻址捕捉探针消除了将核酸(即,RNA、cDNA或基因组DNA)从组织切片捕捉到基底表面上以生成空间寻址文库的需要。
在一个实例中,空间寻址捕捉探针包含随机引物序列,其用于从组织样品中的总RNA原位合成cDNA。
图30显示了使用可释放捕捉探针生成空间寻址cDNA文库的方法3000的实例的流程图。方法3000包括但不限于以下步骤。
在步骤3010,给盖玻片打印空间寻址捕捉探针以形成空间特征的阵列。在一个实例中,在2cm x 2cm盖玻片上打印空间寻址捕捉探针以形成直径100μm且35μm间距的空间特征的阵列。空间寻址捕捉探针包含用于在逆转录反应中合成cDNA的随机引物序列、空间地址序列和生物素化的SBS引物序列,如参考图31A和31B更为详细描述。空间寻址捕捉探针还包含分子的5’端的修饰以可逆附着到盖玻片。在一个实例中,空间寻址捕捉探针包含5二硫化物修饰,如参考图32A、32B、和32C更为详细描述。在一些实施方案中,空间寻址捕捉探针包含5光可切割接头,如参考图33A、33B、和33C更为详细描述。
在步骤3015中,将盖玻片置于载玻片上安装的半透化FFPE组织切片的顶上,使得其上带有空间寻址捕捉探针的盖玻片的表面与组织切片接触。
在步骤3020,在盖玻片上固定化的空间寻址捕捉探针从盖玻片的表面释放到组织切片的细胞空间中。在一个实例中,通过使二硫苏糖醇(DTT)溶液流经半透化组织切片释放包含5'二硫化物修饰的空间寻址捕捉探针。在一些实施方案中,使用紫外光照射释放包括5'光可切割接头的捕捉探针。
在步骤3025,使用逆转录反应原位合成第一链cDNA。例如,将逆转录主混合物溶液在盖玻片和载玻片之间流入半透化组织切片。盖玻片作用为防止反应期间蒸发的屏障。由于逆转录反应中使用的空间寻址捕捉探针中的内部生物素标记物,将第一链cDNA生物素化。
在步骤3030,解离RNA:cDNA杂合物并破坏细胞基质。在一个实例中,解离RNA:cDNA双链体,并使用NaOH溶液破坏细胞基质。在一些实施方案中,RNA:cDNA双链体解离并且使用热处理方案破坏细胞基质。
在步骤3035中,将其中具有RNA和cDNA的半透化组织样品从载玻片表面取出并收集在收集管中。在一个实例中,通过刮入Eppendorf管从玻璃载玻片取出其中具有RNA和第一链cDNA的半透化组织样品。在一些实施方案中,通过将载玻片置于50mL离心管中并离心以将物质在管底部收集在容器中,从载玻片中取出其中具有RNA和第一链cDNA的半透化组织样品。
在步骤3040中,使用两轮基于链霉亲合素珠的纯化方案来纯化生物素化的第一链cDNA。将纯化的第一链cDNA收集在PCR管中用于随后的处理步骤。cDNA分子包括SBS引物序列(例如SBS12),空间地址序列和随机引物序列。
在步骤3045,使用在例如一个或多个靶定SNV侧翼的正向和反向引物对的混合物在多重反应中扩增第一链cDNA。例如,正向引物包含靶向感兴趣的SNV的基因特异性序列,SBS引物序列(例如SBS3)和P5序列。基因特异性序列设计为在靶定的SNV上游约50bp。反向引物包含SBS12互补序列和P7序列。
在步骤3050,对文库扩增子进行测序。例如,SBS反应的读段1(例如从约50bp到约75bp)提供了靶定SNV的序列信息,而读段2(约25bp)提供了空间地址的序列信息。
图31A和图31B分别显示了包括5'二硫化物修饰的空间寻址捕捉探针3100和包括5'光可切割接头的空间寻址捕捉探针3150的示意图。参考图31A,空间寻址捕捉探针3100包括随机引物区域3110,空间地址区域3115和SBS引物区域3120(例如SBS12)。随机引物区域3110可以包括例如可用于在逆转录反应中从细胞的全部转录组引发cDNA合成的6bp序列。空间地址区3115包括用于阵列上的每个空间特征的独特序列。SBS引物区域3120包含内部生物素标记物3125。内部生物素标记物3125用于在后续处理步骤中纯化cDNA。空间寻址捕捉探针3100还包括一个5'二硫化物修饰3130。二硫化物修饰3130用于将空间寻址捕捉探针3100可逆地锚定玻璃基底(例如玻璃盖玻片)的表面上,如参考图32A,32B和32C更为详细描述。
参考图31B,除了空间寻址捕捉探针3150的5'端用可光切割的氨基接头3155修饰之外,空间寻址捕捉探针3150与空间寻址捕捉探针3100基本相同。可光切割的氨基接头3155用于将空间寻址捕捉探针3150可逆地锚定到玻璃基底(例如玻璃盖玻片)的表面上,如参考图33A,33B和33C更详细描述的。
图32A,32B和32C显示将图31A的空间寻址捕捉探针3100可逆地锚定到玻璃盖玻片表面上的过程的实例。
在第一步中并且现在参考图32A,提供用多个巯基(SH)基团3215官能化的玻璃盖玻片3210。巯基官能化盖玻片是商品化的。
在下一步中并且现在参考图32B,将空间寻址捕捉探针3100沉积(例如打印)到玻璃盖玻片3210的表面上。
在下一步中并且现在参考图32C,通过空间寻址捕捉探针3100上巯基3215和二硫化物修饰3130之间的巯基-二硫化物交换反应中形成二硫键将空间寻址捕捉探针3100锚定到玻璃盖玻片3210的表面上。可以随后通过使用还原剂切割二硫键从玻璃盖玻片3210释放空间寻址捕捉探针3100。在一个实例中,二硫苏糖醇(DTT)用于切割二硫键并释放空间寻址捕捉探针3100。
图33A,33B和33C显示了将图31B的空间寻址捕捉探针3150可逆地锚定到玻璃盖玻片的表面上的过程的实例。
在第一步中并且现在参照图33A,提供了用多个醛基3315官能化的玻璃盖玻片3310。醛官能化玻璃基底是商品化的。
在下一步中并且现在参考图33B,将空间寻址捕捉探针3150沉积在玻璃盖玻片3310的表面上。
在下一步中并且现在参考图33C,通过空间寻址捕捉探针3150上醛基3315和光可切割氨基接头3155之间形成共价键将空间寻址捕捉探针3150锚定到玻璃盖玻片3310的表面上。随后,可以通过使用紫外光照射(例如350nm,约5分钟)切割共价键而从玻璃盖玻片3310释放空间寻址捕捉探针3150。
图34A和34B以图像显示了图30的方法3000的步骤。在步骤3010中,给盖玻片3410打印空间寻址捕捉探针(未显示)以形成空间特征3415的阵列。在一个实例中,在2cmx2cm盖玻片上打印空间寻址捕捉探针以形成直径为100μm且间距为35μm的空间特征3415的阵列。空间寻址捕捉探针是例如图31的空间寻址捕捉探针3100,其用于捕捉细胞的整个转录物组。
在步骤3015中,将盖玻片3410置于安装在载玻片3425上的半透化组织切片3420的顶上。组织切片3420包含多个细胞3430。每个细胞含有一个或多个RNA分子3435。一个或多个RNA分子3435可以包括单核苷酸变异(SNV)3440。
在步骤3020中,在盖玻片3410上固定化的空间寻址捕捉探针(用箭头标示)从盖玻片表面释放到细胞空间中。
在步骤3025中,使用空间寻址捕捉探针3100上的随机引物序列3110在逆转录反应中原位合成第一链cDNA。例如,将逆转录主混合物溶液(未显示)在盖玻片3410和载玻片3425之间流入半透化组织切片3420中。
在步骤3030中,解离RNA:cDNA杂合物并破坏组织样品3420中的细胞基质以释放cDNA分子3445。在一个实例中,解离RNA:cDNA双链体并使用NaOH溶液破坏细胞基质。在一些实施方案中,解离RNA:cDNA双链体并且使用热处理方案破坏细胞基质。
在步骤3035中,将其中具有细胞3430,RNA分子3435和cDNA分子3445的半透化组织样品3420从载玻片3425的表面取出并收集在收集管3450中。在一个实例中,通过刮入Eppendorf管中将半透化组织样品从玻璃载片3425取出其中具有细胞3430,RNA分子3435和cDNA分子3445的半透化组织样品3420。在一些实施方案中,通过将载玻片3425放入50mL离心管中并离心以在50ml管的底部在容器中收集取出其中具有细胞3430,RNA分子3435和cDNA分子3445的半透化组织样品3420。
在步骤3040中,使用两轮基于链霉亲合素珠的纯化方案纯化生物素化的cDNA分子3445。
在步骤3045中,使用位于靶定SNV侧翼的正向和反向引物对的混合物在多重反应中扩增cDNA分子3445。例如,正向引物3455包含靶向感兴趣的SNV(例如SNV 3440)的基因特异性区域3460,SBS引物区3465(例如SBS3)和P5区3470。基因特异性区域3460设计为靶定SNV上游约50bp。反向引物3475包含SBS12互补区3120a和P7区3480。使用正向引物3455和反向引物3475合成的文库扩增子3485包含P5区3470,SBS引物区3465,SNV 3440,随机引物区3110,空间地址区3115,SBS引物区3120和P7区3480。
在步骤3050,对文库扩增子测序。例如,SBS反应的读段1(例如从约50bp到约75bp)提供了靶定SNV的序列信息,并且读段2(约25bp)提供了空间地址的序列信息。
在一些实施方案中,空间寻址捕捉探针包含组织样品中基因组DNA的原位靶向捕捉和扩增的序列。在一个实例中,使用TSCA样方法(TruSeq定制扩增子装配体,Illumina)进行靶定基因组DNA区域的捕捉和扩增。在TSCA样的方法中,在感兴趣的靶定区域(例如SNV)侧翼的捕捉探针对用于捕捉基因组DNA。图35显示了用于捕捉感兴趣的基因组DNA区域的捕捉探针对3500的实例的示意图。捕捉探针对3500包括在感兴趣区域5'杂交的第一捕捉探针3510和感兴趣区域3'杂交的第二捕捉探针3515。第一捕捉探针3510包括SBS引物区3520a(例如SBS12),空间地址区3525,基因特异性区域3530a。第二捕捉探针3515包括SBS引物区3520b(例如SBS3)和基因特异性区域3530b。
图36显示了使用可释放捕捉探针生成空间寻址基因组扩增子文库的方法3600的实例的流程图。在此实例中,使用TSCA样的方法(TruSeq定制扩增子装配体,Illumina)进行靶定基因组DNA区域的捕捉和扩增。方法3600包括但不限于以下步骤。
在步骤3610中,将其上具有空间寻址捕捉探针的盖玻片放置在安装在载玻片上的半透化FFPE组织切片的顶上。在一个实例中,空间寻址捕捉探针悬浮在沉积到盖玻片表面上的BioGel基质中。空间寻址捕捉探针是一对探针,其包括位于基因组DNA中感兴趣区域(例如SNV)侧翼的DNA序列。如上参照图35所述,捕捉探针还包括用于后续PCR扩增的序列(例如SBS3和SBS12序列)。捕捉探针中的一个(或两个)还可以包括用于随后纯化靶定DNA区域的内部生物素标记物。使用例如热处理方案从BioGel基质将捕捉探针释放到组织切片上。
在步骤3615中,将捕捉探针与基因组DNA杂交,并且在侧翼捕捉探针之间在感兴趣的靶定区域间进行原位延伸/连接反应。
在步骤3620中,将延伸/连接产物纯化。例如,将其中具有延伸/连接产物的组织样品从载玻片表面取出并收集在收集管中。然后使用一轮或多轮纯化方案(例如基于链霉亲合素珠的纯化方案)纯化延伸/连接产物。
在步骤3625,将扩增/连接产物进行PCR扩增以添加索引和测序引物。
在步骤3630中,对文库扩增子测序。
图37显示了图36的方法3600的步骤。在步骤3610中,将其上具有BioGel基质中的空间寻址捕捉探针的盖玻片3710置于安装在载玻片3720上的半透化FFPE组织切片3715上。组织切片3715包含细胞3725。细胞3725包含靶定基因组DNA3730的区域。基因组DNA 3730可以包括SNV 3735。使用热处理方案将第一捕捉探针3510和第二捕捉探针3515释放到组织切片3715中。
在步骤3615中,将第一捕捉探针3510和第二捕捉探针3515与基因组DNA杂交,并在在感兴趣的靶定区域间在侧翼捕捉探针之间进行原位延伸/连接反应,以产生延伸/连接产品3740。
在步骤3620中,纯化延伸/连接产物3740。例如,将其中具有延伸/连接产物3740的组织样品从载玻片的表面取出并收集在收集管3745中。然后使用一轮或多轮纯化方案,诸如基于链霉亲合素珠的纯化方案纯化延伸/连接产物3740。
在步骤3625中,使用正向引物3750和反向引物3755对延伸/连接产物3740进行PCR扩增以添加测序衔接头。正向引物3750包含与SBS引物区3520b互补的SBS区3520b'和P5区3760。反向引物3755包含与3520a互补的SBS区3520a'和P7区3765。
4.6基于核酸的核酸捕捉
在一些实施方案中,可以首先通过在颗粒,诸如纳米颗粒上固定化的探针捕捉组织样品中的核酸,然后转移到本文所述的捕捉阵列。基于颗粒的核酸转移可以提高核酸从组织样品转移到捕捉阵列的效率,例如收率或动力学。
可以通过将包含核酸的颗粒从组织样品转移到捕捉阵列将核酸从组织样品转移到捕捉阵列。例如,可以通过使用磁响应颗粒,诸如磁响应纳米颗粒,并且通过向组织样品和捕捉阵列施加磁场以促进将磁响应纳米颗粒从组织样品转移到捕捉阵列来促进颗粒转移。在一些实施方案中,例如,可以通过使用分子相互作用,例如配体结合相互作用(例如链霉亲合素蛋白-生物素相互作用)促进从组织样品到捕捉阵列的颗粒转移。例如,可以通过使用链霉亲合素包被的颗粒,如链霉亲合素包被的纳米颗粒和生物素包被的捕捉阵列促进颗粒转移。或者,可以使用任何蛋白质-蛋白质,蛋白质-小分子或核酸-核酸相互作用或任何特定的化学反应(例如“点击化学”)来促进包含核酸的颗粒从组织样品快速或完全转移样品到捕捉阵列。
可以将多种探针固定化在颗粒上以从组织样品中捕捉核酸,并与本文所述的捕捉阵列上的多种探针组合。在一些实施方案中,颗粒上的探针基本上由捕捉区组成,以从组织样品中捕捉核酸(此外,例如将探针固定到颗粒的附加元件)。在一些实施方案中,颗粒上的探针可以包括捕捉区和空间地址区(例如部分或组合空间地址区或完整的空间地址区)。本文所述的探针包被的颗粒可以组合使用,例如与捕捉阵列组合使用,所述捕捉阵列包括具有探针的捕捉位点,所述探针基本上包含可切割区域,或可切割区域和空间地址区(例如部分或组合空间地址区或完整的空间地址区)或可切割区域,空间地址区和捕捉区(例如颗粒上捕捉核酸的区域)或任何其它区域组合。
在一些实施方案中,磁响应纳米颗粒用于捕捉组织样品中的核酸(例如原位合成的cDNA)以产生空间寻址文库。
在一些实施方案中,磁响应纳米颗粒可以包含基本上包含捕捉区(例如基因特异性或通用捕捉区)的固定化探针。在一些实施方案中,探针可以进一步包含SBS引物区(例如SBS3或SBS12区)或其他通用区,如P5或P7区。在一些实施方案中,纳米颗粒与包括捕捉探针的捕捉阵列组合使用,所述捕捉探针包括可切割区域,空间地址区域和捕捉区(例如基因特异性捕捉区)。在一些实施方案中,捕捉阵列上的探针还包括SBS引物区(例如SBS3或SBS12区域)或其他通用区域,如P5或P7区域。
图38显示了使用颗粒,诸如磁响应纳米颗粒从组织样品中捕捉核酸来生成空间寻址测序文库的方法3800的实例的流程图。
在步骤3810中,通过原位逆转录从组织样品中的靶mRNA合成cDNA。在一些实施方案中,cDNA是基因特异性(即,靶定)cDNA。在一些实施方案中,cDNA是随机cDNA或代表整体mRNA的cDNA。例如,在一些实施方案中,结合到颗粒,例如磁响应纳米颗粒表面的基因特异性RT引物可用于在逆转录反应中引发第一链cDNA合成。
在步骤3815中,将结合到例如磁响应纳米颗粒表面的第一链cDNA捕捉到阵列上。例如,该阵列是用空间寻址捕捉探针打印以形成捕捉位点阵列的玻璃基底。空间寻址捕捉探针可以包括例如可切割的多接头序列,空间地址序列和与第一链cDNA序列互补的基因特异性捕捉序列。空间寻址捕捉探针可以通过可切割多接头序列附着到玻璃基底。空间地址序列通常是阵列上每个空间特征的独特序列。每个空间特征可以包括具有不同基因特异性捕捉序列的多个空间寻址捕捉探针。磁体可以放置在阵列附近。可将反应加热至约95℃的温育温度达约1分钟以使RNA:cDNA杂合物变性。在磁体附近,结合到磁响应纳米颗粒表面的第一链cDNA可以锚定到阵列的表面上。可以通过在空间寻址捕捉探针上与基因特异性捕捉序列杂交(例如在约60℃约10分钟)将第一链cDNA分子捕捉到阵列上。
在步骤3820中,可以合成第二链cDNA。例如,从阵列附近除去磁体,并且可以通过清洗除去未与空间寻址捕捉探针中的基因特异性捕捉序列杂交的第一链cDNA分子。使用第二基因特异性捕捉序列作为引物在延伸反应中合成第二链cDNA。
在步骤3825中,可以通过切割可切割的多接头序列从捕捉阵列释放双链cDNA。
在步骤3830中,可任选地将双链cDNA进行末端修复并连接至测序衔接头以产生测序文库。在捕捉阵列上的RT引物和捕捉引物包括SBS引物区的实施方案中,末端修复。
图39显示了图38的示例性方法3800的步骤。即,组织切片(未显示)包含靶RNA分子3910。靶RNA分子3910可以包含突变3915。在步骤3810中,使用基因特异性RT引物3920在逆转录反应中原位合成cDNA。将RT引物3920附着到磁响应纳米颗粒3925的表面。将使用RT引物3920合成的cDNA分子3930附着到磁响应纳米颗粒3925。
在步骤3815,将cDNA分子3930捕捉到包括多个捕捉位点3935的阵列上。在此实例中,显示了单一捕捉位点3935。捕捉位点3935包括空间寻址捕捉探针3940。空间寻址捕捉探针3940包含可切割多接头区域3945、空间地址区3950、和与第一链cDNA中的序列互补的基因特异性捕捉区3955。通过可切割的多接头区3945将空间寻址捕捉探针3940附着到捕捉位点3935。磁体3960靠近捕捉位点3935放置。将反应加热到约95℃的温育温度约1分钟以使RNA:cDNA杂合物变性。在磁体3960附近,将与磁响应纳米颗粒3925表面结合的第一链cDNA分子3930锚定到捕捉位点3935表面上。通过与空间寻址捕捉探针3940中的基因特异性捕捉区3955杂交(例如在约60℃达约10分钟)将第一链cDNA分子3930捕捉到捕捉位点3935上。
在步骤3820中,合成第二链cDNA。例如,从捕捉位点3935附近除去磁体3960,并且通过清洗除去未与空间寻址捕捉探针3940中的基因特异性捕捉序列3955杂交的第一链cDNA分子3930。利用基因特异性捕捉区3955作为引物在延伸反应中合成第二链cDNA。
在步骤3825中,通过切割可切割多接头区3945从捕捉位点3935释放现在包含空间地址区3950的双链cDNA分子3965。
在步骤3830(图39中未显示),任选地将双链cDNA分子3965进行末端修复并连接至测序衔接头以产生空间寻址测序文库。
在一些实施方案中,捕捉阵列上的RT引物和捕捉引物可以任选地且独立地包含附加区域,如SBS引物区(例如SBS3或SBS12区)或通用区(例如P5或P7区),其可以例如被掺入到双链cDNA分子3965中。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的方法,包括提供包含固定化捕捉探针的磁响应纳米颗粒(例如3925),所述固定化捕捉探针包含捕捉区(例如3920)。
在一些实施方案中,所述捕捉区是基因特异性捕捉区(包含例如TSCA序列)。在一些实施方案中,所述捕捉区是通用捕捉区(包含例如多聚-T序列或随机化核酸序列)。
在一些实施方案中,固定化捕捉探针不包含空间寻址区。
在一些实施方案中,所述方法还包括使磁响应纳米颗粒与组织样品接触,使得组织样品上磁响应纳米颗粒的位置可以与组织样品中的核酸位置相关联,并且允许所述核酸与所述固定化捕捉探针的所述捕捉区杂交。
在一些实施方案中,所述方法还包括延伸固定化捕捉探针的捕捉区以形成与所述捕捉区杂交的核酸的固定化第一互补链(例如3930)。
在一些实施方案中,所述方法还包括使所述组织样品与捕捉阵列接触,使得所述捕捉阵列上的捕捉位点的位置可以与所述组织样品中的位置相关联,其中捕捉阵列包含捕捉位点(例如3935),其包含表面上固定化的捕捉探针(例如3940),其中所述捕捉探针包含可切割区域(例如3945)、空间地址区(例如3950)和基因特异性区域(例如3955)。
在一些实施方案中,所述方法还包括对所述捕捉阵列和组织样品施加磁场以将具有固定化第一互补链的磁响应纳米颗粒转移到捕捉阵列,并且允许固定化第一互补链与捕捉阵列上的捕捉探针的捕捉区杂交。
在一些实施方案中,所述方法还包括延伸所述捕捉阵列上所述捕捉探针的所述捕捉区以形成所述核酸的固定化第二互补链(例如3965的新合成链)。
在一些实施方案中,所述方法还包括切割可切割域处所述捕捉阵列上的所述捕捉探针以从所述捕捉阵列的表面释放空间标签化第二互补链。
在一些实施方案中,所述方法还包括分析释放的空间标签化第二互补链的序列。
在一些实施方案中,所述方法还包括将所述释放的空间标签化第二互补链的序列与所述组织样品中的核酸位置相关联。
在一些实施方案中,可以将颗粒(例如纳米颗粒)结合探针与来自组织样品的核酸杂交,之后将探针固定化到纳米颗粒,然后可以将探针-核酸杂合物固定化到纳米颗粒。在一些实施方案中,可以延伸与来自组织样品的核酸杂交的探针以形成与来自组织样品的核酸互补的核酸,然后可以将互补核酸固定化到纳米颗粒。在一些实施方案中,探针可以包含用于将探针-核酸杂合物或互补核酸连接至纳米颗粒的接头元件。
图40A和40B显示了使用捕捉探针在组织样品中形成互补核酸并随后将互补核酸固定化到纳米颗粒的过程4000的实例。
在第一步中并且现在参考图40A,将捕捉探针4010与来自组织样品的核酸4015杂交。捕捉探针4010包括捕捉区4015和接头元件4020。在一个实例中,捕捉区4015包括随机引物序列。在另一个实例中,捕捉区4015包含基因特异性引物序列。接头元件4020是用于将探针-核酸杂合物或互补核酸连接至纳米颗粒的元件。在一个实例中,核酸分子4025是基因组DNA分子。在另一个实例中,核酸分子4025是RNA分子。捕捉探针4010与来自组织样品的核酸分子4025杂交并延伸形成互补核酸分子4025a。
在下一步中并且现在参考图40B中,经由捕捉探针4010中的连接元件4020将互补核酸4025a固定化到纳米颗粒4030。在一个实例中,纳米颗粒4030是磁响应纳米颗粒。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的方法,包括提供包含捕捉区和接头元件(例如生物素基团,硫醇基团或另一种化学接头)的引物。
在一些实施方案中,捕捉区是基因特异性捕捉区(包括例如TSCA序列)。在一些实施方案中,捕捉区是通用捕捉区(包括例如多聚-T序列或随机化核酸序列)。
在一些实施方案中,所述方法还包括将组织样品与引物接触并使引物与来自组织样品的核酸杂交。
在一些实施方案中,所述方法还包括将引物延伸以形成与引物杂交的核酸的固定化第一互补链。
在一些实施方案中,所述方法还包括使组织样品与磁响应纳米颗粒(例如链霉亲合素包被的磁响应纳米颗粒)接触,使得组织样品上的磁响应纳米颗粒的位置可以与组织样品中的延伸引物的位置相关联,以及将包含核酸的第一固定化链的延伸引物在接头元件处固定到磁响应纳米颗粒。
在一些实施方案中,所述方法还包括使所述组织样品与捕捉阵列接触使得所述捕捉阵列上的捕捉位点的位置可以与所述组织样品中的位置相关联其中所述捕捉阵列包含捕捉位点,所述捕捉位点包含表面上固定化的捕捉探针,其中所述捕捉探针包含可切割区域、空间地址区和基因特异性区域。
在一些实施方案中,所述方法还包括将磁场施加到捕捉阵列和组织样品以将具有固定化第一互补链的磁响应纳米颗粒转移到捕捉阵列,并且允许固定化第一互补链与捕捉阵列上的捕捉探针的基因特异性区域杂交。
在一些实施方案中,所述方法还包括延伸捕捉阵列上的捕捉探针的基因特异性区域以形成所述核酸的固定化第二互补链。
在一些实施方案中,所述方法还包括切割捕捉阵列上的捕捉探针以从所述捕捉阵列的表面释放空间标签化第二互补链。
在一些实施方案中,所述方法还包括分析释放的空间标签化第二互补链的序列。
在一些实施方案中,所述方法还包括将所述释放的空间标签化第二互补链的序列与所述组织样品中的核酸位置相关联。
在一些实施方案中,颗粒(例如磁响应纳米颗粒)可以包含固定化探针,其包含捕捉区(例如基因特异性或通用捕捉区)和第一部分地址区。在一些实施方案中,颗粒结合探针可以进一步包含SBS引物区(例如SBS3或SBS12区)或另一通用区,如P5或P7区。在一些实施方案中,颗粒可以与捕捉阵列结合使用,该捕捉阵列包括捕捉探针,所述捕捉探针包含可切割区域、第二空间地址区域和捕捉区(例如基因特异性捕捉区域)。在一些实施方案中,捕捉阵列上的探针还可以包括SBS引物区(例如SBS 3或SBS12区域)或另一通用区域,如P5或P7区域。
图41A和41B分别显示了包括第一部分空间地址区的颗粒结合捕捉探针和包含第二部分地址区的第二阵列捕捉探针用于空间检测和分析组织样品中的核酸的实例的示意图。现在参照图41A,颗粒结合捕捉探针4110包括在颗粒4125上固定化的捕捉区4115(例如基因特异性或通用捕捉区)和第一部分地址区4120。在一个实例中,颗粒4125是磁响应纳米颗粒。捕捉探针4110与来自组织切片的核酸分子4130杂交,并延伸(通过箭头标示)以形成与核酸分子4130互补的核酸。互补核酸包含第一部分地址区4120和颗粒4125。第一部分地址区4120鉴定沿着捕捉阵列的第一维度的捕捉位点位置。在一些实施方案中,使用磁场来促进包含互补核酸分子的颗粒4125转移到包含阵列捕捉探针的阵列上。
现在参考图41B,阵列捕捉探针4135包括捕捉区4140(例如基因特异性捕捉区),第二部分地址区4145和SBS区4150以及可切割区域4155。可以通过可切割区域4155将阵列捕捉探针4135在捕捉阵列(未显示)的表面上固定化。可以使捕捉阵列(未显示)上的阵列捕捉探针4135与包含用图41A的第一部分地址区4120标记的核酸分子的组织样品接触,使得阵列捕捉探针4135的位置可以与组织样品中的位置相关联。第二部分地址区4145沿捕捉阵列的第二维度鉴定捕捉位点的位置。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的方法,包括提供包含固定化捕捉探针的磁响应纳米颗粒,所述固定化捕捉探针包含捕捉区和第一部分空间地址区。
在一些实施方案中,捕捉区是基因特异性捕捉区(包括例如TSCA序列)。在一些实施方案中,捕捉区是通用捕捉区(包括,例如多聚-T序列或随机化核酸序列)。
在一些实施方案中,第一部分空间地址区鉴定沿着捕捉阵列的第一维度的捕捉位点位置。
在一些实施方案中,所述方法还包括使磁响应纳米颗粒与组织样品接触,使得组织样品上磁响应纳米颗粒的位置可以与组织样品中核酸的位置相关,并且允许所述核酸与所述固定化捕捉探针的所述捕捉区杂交。
在一些实施方案中,所述方法还包括延伸固定化捕捉探针的捕捉区以形成与所述捕捉区杂交的核酸的固定化第一互补链,其中固定化第一互补链包含第一部分地址区。
在一些实施方案中,所述方法还包括使所述组织样品与捕捉阵列接触使得所述捕捉阵列上的捕捉位点的位置可以与所述组织样品中的位置相关联其中所述捕捉阵列包含捕捉位点,所述捕捉位点包含表面上固定化的捕捉探针,其中所述捕捉探针包含可切割区域、第二部分地址区和基因特异性区域。
在一些实施方案中,所述第二部分空间地址区鉴定沿着捕捉阵列的第二维度的捕捉位点位置。
在一些实施方案中,所述方法还包括对所述捕捉阵列和组织样品施加磁场以将具有固定化第一互补链的磁响应纳米颗粒转移到捕捉阵列的捕捉位点并且允许所述固定化第一互补链与所述捕捉阵列的所述捕捉位点上的所述捕捉探针的捕捉区杂交。
在一些实施方案中,所述方法还包括延伸所述捕捉位点上的所述捕捉探针的所述捕捉区以形成所述核酸的固定化第二互补链,其中所述核酸的所述第二互补链包含所述第一和第二部分空间地址区。
在一些实施方案中,所述第一和第二部分空间地址区的组合限定所述捕捉阵列上所述捕捉位点的位置。
在一些实施方案中,所述方法还包括切割可切割域处所述捕捉阵列上的所述捕捉探针以从所述捕捉阵列的表面释放空间标签化第二互补链。
在一些实施方案中,所述方法还包括分析释放的空间标签化第二互补链的序列。
在一些实施方案中,所述方法还包括将所述释放的空间标签化第二互补链的序列与所述组织样品中的核酸位置相关联。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的方法,其包括提供包含固定化捕捉探针的磁响应纳米颗粒,所述固定化捕捉探针包含捕捉区。
在一些实施方案中,所述固定化捕捉探针不包含空间地址区。
在一些实施方案中,所述捕捉区是基因特异性捕捉区(包含例如TSCA序列)。在一些实施方案中,所述捕捉区是通用捕捉区(包含例如多聚-T序列或随机化核酸序列)。
在一些实施方案中,所述方法还包括使磁响应纳米颗粒与组织样品接触,使得组织样品上的磁响应纳米颗粒的位置可以与组织样品中核酸的位置相关联,并且允许所述核酸与所述固定化捕捉探针的所述捕捉区杂交。
在一些实施方案中,所述方法还包括延伸固定化捕捉探针的捕捉区以形成与所述捕捉区杂交的核酸的固定化第一互补链。
在一些实施方案中,固定化第一互补链不包含空间地址区。
在一些实施方案中,所述方法还包括使所述组织样品与捕捉阵列接触使得所述捕捉阵列上的捕捉位点的位置可以与所述组织样品中的位置相关联其中所述捕捉阵列包含捕捉位点,所述捕捉位点包含表面上固定化的探针,其中所述探针包含空间地址区。
在一些实施方案中,所述探针不包含捕捉区。
在一些实施方案中,所述方法还包括对所述捕捉阵列和组织样品施加磁场以将具有固定化第一互补链的磁响应纳米颗粒转移到捕捉阵列的捕捉位点并将固定化第一互补链连接到捕捉阵列上的探针的空间地址区,以在捕捉阵列上和磁响应纳米颗粒上在两个末端上固定化第一互补链。
图42A和图42B分别显示了颗粒结合捕捉探针和包括空间地址区的第二阵列捕捉探针用于空间检测和分析组织样品的核酸的实例的示意图。现在参考图42A,颗粒结合捕捉探针4210包括在磁响应纳米颗粒4220上固定化的捕捉区4215(例如基因特异性或通用捕捉区)。捕捉探针4210与来自组织切片的核酸分子4225杂交并延伸(通过箭头标示)以形成与核酸分子4225互补的核酸。互补核酸包含磁响应纳米颗粒4220。在一些实施方案中,使用磁场来促进包含互补核酸分子的磁响应纳米颗粒4220转移到包含阵列捕捉探针的阵列上。
现在参考图42B,阵列捕捉探针4230包括空间地址区4235,可切割区域4240。可以通过可切割区域4240在捕捉阵列(未显示)的表面上固定化阵列捕捉探针4230。可以使捕捉阵列表面(未显示)上的阵列捕捉探针4230与包含互补核酸分子的组织样品接触,使得阵列捕捉探针4230的位置可以与在组织样品中的位置,所述互补核酸分子用图42A的磁响应纳米颗粒4220加标签。可以通过连接到空间地址区4235将包含磁响应纳米颗粒4220的互补核酸捕捉到捕捉探针4230上。空间地址区4240鉴定捕捉位点在阵列上的位置。
在一些实施方案中,第一互补链包括捕捉阵列上和磁响应纳米颗粒上在两个末端上固定化时的空间地址区。
在一些实施方案中,磁响应纳米颗粒上的固定化捕捉探针和空间地址区任选各自还包含引物结合区(例如SBS引物结合区,诸如SBS3或SBS12区)。在一些实施方案中,所述方法还包括使用与所述第一互补链中的所述引物结合区互补的引物对合成第二互补链,其中所述第二互补链包含所述空间地址区,并且从所述捕捉阵列的表面释放所述第二互补链。在一些实施方案中,所述方法还包括分析释放的第二互补链的序列,并且将所述释放的第二互补链的序列与所述组织样品中的所述核酸的位置相关联。
在一些实施方案中,磁响应纳米颗粒上的固定化捕捉探针和空间地址区任选地各自还包含可切割区域(例如相同可切割区域或不同可切割区域)。在一些实施方案中,所述方法还包括通过切割所述固定化第一互补链中的所述可切割区域释放所述固定化第一互补链。在一些实施方案中,所述方法还包括分析释放的第一互补链的序列,并且将所述释放的第二互补链的序列与所述组织样品中的所述核酸的位置相关联。
在一些实施方案中,颗粒(例如磁性响应纳米颗粒)可以包含固定化的探针,其包含捕捉区(例如基因特异性或通用捕捉区),第一引物结合区(例如SBS引物区,如SBS3或SBS12区域)和空间地址区。在一些实施方案中,颗粒结合探针可以进一步包含另一个通用区域,例如P5或P7区域。在一些实施方案中,颗粒可以与包含捕捉探针的捕捉阵列组合使用,所述捕捉探针基本上包含第二引物结合区(例如SBS引物区,诸如SBS 3或SBS12区)和任选地另一个通用区,诸如P5或P7区。
图43A和43B分别显示了颗粒结合捕捉探针和第二阵列捕捉探针用于空间检测和分析来自组织样品的核酸的实例的示意图。现在参考图43A,颗粒结合捕捉探针4310包括磁响应纳米颗粒4330上固定化的捕捉区4315(例如基因特异性或通用捕捉区),空间地址区4320和第一引物结合位点4325(例如SBS引物区域SBS3)。捕捉探针4310可以与组织样品(未显示)接触,使得组织样品上的捕捉探针4310的位置可以与组织样品中核酸分子4335的位置相关联。捕捉探针4310与来自组织切片的核酸分子4335杂交,并延伸(通过箭头标示)以形成与核酸分子4335互补的核酸。互补核酸(未显示)包含空间地址区4320,第一引物结合位点4325和磁响应纳米颗粒4330。在一些实施方案中,使用由磁体产生的磁场(未显示)以促进将包含互补核酸分子的磁响应纳米颗粒4330转移到包含阵列捕捉探针的阵列上。
现在参考图43B,阵列捕捉探针4340包括与图43A的第一引物结合位点4325不同的第二引物结合位点4325a(例如SBS引物区SBS12)。阵列捕捉探针4340可以固定化在捕捉阵列(未显示)的表面上。在捕捉阵列表面(未显示)上的阵列捕捉探针4340可以与包含用图43A的磁响应纳米颗粒4330加标签的互补核酸分子的组织样品接触。可通过与第二引物结合位点4325a连接将包含磁响应纳米颗粒4330的互补核酸捕捉到捕捉探针4340上。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的方法,包括提供磁响应纳米颗粒,所述磁响应纳米颗粒包含固定化捕捉探针,所述固定化捕捉探针包括捕捉区,第一引物结合区和空间地址区。
在一些实施方案中,所述捕捉区是基因特异性捕捉区(包括例如TSCA序列)。在一些实施方案中,所述捕捉区是通用捕捉区(包括,例如多聚-T序列或随机化核酸序列)。
在一些实施方案中,所述方法还包括使磁响应纳米颗粒与组织样品接触,使得组织样品上磁响应纳米颗粒的位置可以与组织样品中核酸的位置相关联,并且允许所述核酸与所述固定化捕捉探针的所述捕捉区杂交。
在一些实施方案中,所述方法还包括延伸固定化捕捉探针的捕捉区以形成与所述捕捉区杂交的核酸的固定化第一互补链,其中固定化第一互补链包含空间地址区。
在一些实施方案中,所述方法还包括使所述组织样品与捕捉阵列接触使得所述捕捉阵列上的捕捉位点的位置可以与所述组织样品中的位置相关联其中所述捕捉阵列包含捕捉位点,所述捕捉位点包含表面上固定化的捕捉探针,其中捕捉探针基本上包含第二引物结合区(例如SBS引物区,诸如SBS3或SBS12区)。
在一些实施方案中,所述方法还包括对所述捕捉阵列和组织样品施加磁场以将具有固定化第一互补链的磁响应纳米颗粒转移到捕捉阵列的捕捉位点,并且将固定化第一互补链与捕捉阵列的捕捉位点上的捕捉探针连接以在两个末端上固定化第一互补链。
在一些实施方案中,所述方法还包括使用与所述第一捕捉探针的第一引物结合序列互补的引物合成第二互补链并且从所述捕捉阵列的表面释放所述第二互补链。
在一些实施方案中,所述方法还包括分析释放的第二互补链的序列并且将所述释放的第二互补链的序列与所述组织样品中的所述核酸的位置相关联。
图44显示了配置用于空间检测和分析组织样品中的核酸的基于磁性的转移系统4400的透视图。基于磁性的转移系统4400包括捕捉阵列4410。捕捉阵列4410包括固体支持物4415。在一个实例中,固体支持物4415是平面玻璃基底。在固体支持物4415的表面上打印多个独特的捕捉位点(“空间特征”)4420,其是含有空间寻址寡核苷酸的区域。参照图45更详细地描述单一捕捉位点4420的实例。在捕捉阵列4410上覆盖样品基底4425。在一个实例中,样品基底4425是玻璃载玻片。将组织样品4430安装在面向捕捉阵列4410上的捕捉位点4420的样品基底4425的表面上。在一个实例中,组织切片4430是FFPE组织切片。用磁响应纳米颗粒将组织样品4430中的核酸(未显示)加标签。位于捕捉阵列4410下方和附近的是磁体4435。磁体4435可以是永磁体或电磁体。在一个实例中,磁体4435是可移动的磁体。即,磁体4435可以接近或远离捕捉阵列4410移动。由磁体4435产生的磁场用于将来自组织切片4430的加纳米颗粒标签的核酸分子(未显示)吸引到捕捉阵列4410上的捕捉位点4420上。由磁体4435产生的磁场被配置为使得消除或显著减少来自组织样品的核酸的转移和捕捉位点4420之间的核酸损伤。
在另一个实施方案(未显示)中,捕捉位点4420是固体支持物4415中的微孔。在每个微孔的底部表面上打印空间寻址寡核苷酸。在存在磁场的情况下,微孔发挥功能以捕捉加纳米颗粒标签的核酸并消除或基本上减少磁响应纳米颗粒的聚集。
图45显示了捕捉阵列4410上的一个捕捉位点4420的侧视图,其中一个捕捉位点4420包括多个捕捉探针。在一个实例中,多个捕捉探针4510包括SBS引物序列4515(例如SBS3)和空间地址序列4520。在另一个实例中(未显示),捕捉4510包括P7序列,SBS引物序列(例如SBS12)和空间地址序列,如参考图49A更详细描述的。在单一捕捉位点4420处固定化的每个捕捉探针4510包括相同的空间地址序列4520(例如空间地址序列4520a)。例如,图45中,捕捉探针4510a和捕捉探针4510b两者都具有相同的独特空间地址序列4520a。其他捕捉位点4420(未显示)各自包含其自身的独特空间地址序列4520(例如空间地址序列4520b,4520c,4520d,4520e等)。因此,在其他捕捉位点4420处固定化的捕捉探针4510各自包括其自身的独特的空间地址序列4520。SBS引物序列4515用于随后处理步骤以进行文库制备。
图46显示了使用图44和图45的基于磁性的转移系统4100将cDNA从组织样品转移到捕捉阵列以产生空间寻址测序文库的方法4600的实例的流程图。方法4600包括但不限于以下步骤。
在步骤4610,在逆转录(RT)反应中从组织样品中的RNA原位合成第一链cDNA。例如,使用与磁响应纳米颗粒表面结合的RT引物来从安装在样品基底4425上的组织样品4430中的mRNA引发第一链cDNA。在一个实例中,RT引物包括基因特异性引物序列和SBS引物序列(例如SBS3)。在另一个实例中,RT引物包括随机引物序列和SBS引物序列(例如SBS3)。由于RT引物与磁响应纳米颗粒的表面结合,因此第一链cDNA用磁响应纳米颗粒加标签。
在步骤4615,利用磁场将第一链cDNA转移到空间寻址捕捉阵列上。例如,其上带有组织样品4430的样品基底4425放置在捕捉阵列4410的顶部。将磁体4435置于非常接近捕捉阵列4410。由磁体4435产生的磁场用于吸引来自组织切片4430的加纳米颗粒标签的第一链cDNA吸引到捕捉阵列4410上的捕捉位点4420上。因此,通过磁体4435在捕捉位点4420处固定化第一链cDNA。
在步骤4620,通过将cDNA的3’末端单链连接至空间地址序列4520将第一链cDNA共价连接到捕捉探针4510。
在步骤4625,将磁体4435从捕捉阵列4410移开,使得捕捉阵列4410不再位于磁体4435的磁场内。随着捕捉阵列4410处的磁场减弱,从捕捉位点4420释放第一链cDNA分子的5’末端。现在通过捕捉探针4510将第一链cDNA锚定在捕捉位点4420处。
在步骤4630,利用与捕捉探针4510上的SBS引物序列4515互补的引物合成第二链cDNA。
在步骤4635,从捕捉位点4420释放第二链cDNA。在一个实例中,使用热变性方案从捕捉位点4420释放第二链cDNA。在另一个实例中,使用化学(例如NaOH)变性方案释放第二链cDNA。
在步骤4640,扩增第二链cDNA以产生测序文库。
图47A,47B和47C以图像显示了图46的方法4600的步骤。即,组织切片(未显示)包括RNA分子4710。RNA分子4710可以包括突变4715。在步骤4610,使用RT引物4720原位合成第一链cDNA。RT引物4720包括随机引物序列4725和SBS引物序列4730(例如SBS3)。RT引物4720结合到磁响应纳米颗粒4735的表面。使用RT引物4720合成的第一链cDNA分子4740包括SBS引物序列4730和磁响应纳米颗粒4735。
在步骤4615,将第一链cDNA分子4740从组织切片(未显示)转移到捕捉位点4420上。将磁体4435置于非常接近捕捉阵列4410。由磁体4435产生的磁场用于将来自组织切片(未显示)的加纳米颗粒标签的第一链cDNA分子转移到捕捉位点4420。因此,通过磁体4435在捕捉位点4420处固定化第一链cDNA分子4740。
在步骤4620,通过将cDNA分子4740单链连接至空间地址序列4520将第一链cDNA分子4740共价连接至捕捉探针4510。
在步骤4625,将磁体4435从捕捉阵列4410移开,使得捕捉阵列4410不再位于磁体4435的磁场内。随着捕捉阵列4410处的磁场减弱,从捕捉位点4420释放第一链cDNA分子4740的5'末端。现在通过捕捉探针4510将第一链cDNA分子4740锚定在捕捉位点4420处。
在步骤4630,使用与捕捉探针4510上的SBS引物序列4515互补的引物4515a合成第二链cDNA分子4745。
在步骤4635,从捕捉位点4420释放第二链cDNA分子4745。第二链cDNA分子4745包括SBS引物序列4730(例如SBS3),突变4715,空间地址序列4520和SBS引物序列4515(例如SBS12)。
在步骤4640,使用第一SBS引物4750和第二引物4755扩增第二链cDNA分子4745以产生测序文库。第一SBS引物4750包括与SBS引物序列4730互补的SBS互补序列4730a和P5序列4760。第二SBS引物4755包括与SBS引物序列4515互补的SBS互补序列4515a和P7序列4765。使用SBS引物4750和4755合成的文库扩增子4770包括P5序列4760,SBS引物序列4730(例如SBS3),突变4715,空间地址序列4520,SBS引物序列4515(例如SBS12)和P7序列4765。
在另一个实施方案中,用磁响应纳米颗粒对组织样品(例如FFPE组织样品)中的RNA原位加标签,随后转移到捕捉阵列以产生空间寻址cDNA文库。
图48显示了使用图44的基于磁性的转移系统4400将RNA从组织样品转移到捕捉阵列以生成空间寻址测序文库的方法4800的实例的流程图。方法4800包括但不限于以下步骤。
在步骤4810,用磁响应纳米颗粒对组织样品中的RNA加标签。例如,将结合到磁响应纳米颗粒表面的SBS引物寡核苷酸与组织样品中的RNA连接。然后修饰(即阻断)RNA分子的3'末端以防止在随后的加工步骤中不需要的连接。
在步骤4815,使用磁场将加标签的RNA转移到捕捉位点4420上,如上文在图46的方法4600的步骤4615中针对cDNA描述。
在步骤4820,在RT反应中从转移的RNA合成第一链cDNA。例如,使用包含基因特异性引物序列和连接寡核苷酸的RT引物来引发第一链cDNA。在另一个实例中,使用包含随机引物序列和连接寡核苷酸的RT引物来引发第一链cDNA。
在步骤4825,通过单链连接将第一链cDNA与捕捉位点4420上的捕捉探针共价连接。参考图49A更详细地描述捕捉探针。
在步骤4830,从捕捉位点4420释放用于产生cDNA的RNA模板。例如,从捕捉位点4410移开磁体4435。由此减小捕捉位点4420处的磁场,并将磁响应纳米颗粒从表面捕捉位点4420释放。使用热处理方案解离RNA:cDNA双链体。通过与捕捉探针连接将第一链cDNA锚定在捕捉位点4420处。
在步骤4835中,在延伸反应中合成第二链cDNA。例如,引物包括与cDNA中SBS引物序列互补的序列,并且使用P5序列引发第二链cDNA合成。
在步骤4840,通过变性从捕捉位点4420释放cDNA文库分子。
图49A,49B和49C以图像显示了图48的方法4800的步骤。即,组织切片(未显示)包括RNA分子4910。RNA分子4910可以包括突变4915。在步骤4810,将结合到磁响应纳米颗粒4925的表面的SBS引物寡核苷酸4920原位连接到RNA分子4910。然后修饰(即阻断)RNA分子4910的3'末端以防止在随后的加工步骤中不需要的连接。
在步骤4815,使用磁场将其上具有磁响应纳米颗粒4925的RNA分子4910从组织切片(未显示)转移至捕捉位点4420,如上文在图46的方法4600的步骤4615中针对cDNA所述。在此实例中,捕捉位点4420包括捕捉探针4935。捕捉探针4935包括P7序列4940,SBS引物序列4945(例如SBS12)和空间地址序列4950。
在步骤4820,在RT反应中从RNA分子4910合成cDNA。例如,使用包含基因特异性引物序列4960和连接寡核苷酸4965的RT引物4955引发第一链cDNA。
在步骤4825,通过将连接寡核苷酸4965单链连接到空间地址序列4950将包含SBS引物序列4920(例如SBS3)和连接寡核苷酸4965的cDNA分子4970共价连接至捕捉探针4935。
在步骤4830,从捕捉位点4420释放RNA分子4910。例如,从捕捉位点4420移开磁体4435。由此减小捕捉位点4420处的磁场并从表面捕捉位点4420释放与RNA分子4910结合的磁响应纳米颗粒4925。然后使用热处理方案解离RNA:cDNA双链体(即,RNA分子4910:cDNA分子4970双链体)。现在通过捕捉探针4935将cDNA分子4970锚定在捕捉位点4420处。
在步骤4835,在延伸反应中复制cDNA分子4970。例如,使用包括与SBS引物序列4920(例如SBS3)互补的序列4920a和P5序列4980的引物4975引发第二链cDNA合成。cDNA分子4970现在包括P7序列4940,SBS引物序列4945(例如SBS12),空间地址序列4950,连接寡核苷酸4965,突变4915,SBS引物序列4920(例如SBS3)和P5序列4980。
在步骤4840,通过变性(例如热或化学变性)从捕捉位点4420释放cDNA分子4970。cDNA分子4970现在准备好进行测序。
4.7基于DNA的空间组织序型分析(Profiling)
所公开的技术提供了组织样品中基因组DNA的空间检测和分析(例如突变分析或单核苷酸变异(SNV)检测)的方法。在一个实例中,组织样品是FFPE组织样品。组织样品(例如FFPE样品)中的DNA的空间检测和分析与组织样品中的RNA的空间检测和分析相比有几个优点:(1)DNA比RNA更稳定;(2)与FFPE组织样品中的RNA片段(例如100-200bp)相比,FFPE组织样品中的DNA片段更长(例如300-400bp);(3)以相对较低水平表达的RNA分子可能检测不到;以及(4)在DNA中检测到肿瘤抑制基因的变化,而在RNA中检测不到它们。
使用DNA进行空间组织序型分析的缺点是对于大多数基因,每个细胞可能只有2个基因拷贝。本文公开的方法包括初始原位全基因组预扩增步骤,其用于在进行其他生化处理步骤之前增加基因拷贝数。
图50显示对组织样品中的基因组DNA进行序型分析的方法5000的实例的流程图。在一个实例中,方法5000用于对基因组DNA中感兴趣的SNV进行序型分析。方法5000包括但不限于以下步骤。
在步骤5010,给玻璃基底打印空间寻址PCR引物以形成空间特征阵列。在一个实例中,在2cmx2cm的盖玻片上打印空间寻址PCR引物以形成直径为100μm,间距为35μm的空间特征阵列。在另一个实例中,将空间寻址PCR引物打印在载玻片表面上制作的微孔中。使用例如商品化打印技术将空间寻址PCR引物打印在盖玻片或玻璃载玻片上。如参考图51更详细描述的,空间寻址PCR引物包括随机引物序列,空间地址序列,SBS引物序列和生物素标记物。空间寻址PCR引物也可以在分子的5'末端包含修饰以可逆地附着于盖玻片上。在一个实例中,空间寻址PCR引物可以包括5'二硫化物修饰,如对图32A,32B和32C的过程3200的空间寻址捕捉探针3100所述。在另一个实例中,空间寻址PCR引物可以包括5'光可切割接头,如对图33A,33B和33C的过程3300的空间寻址捕捉探针3100所述。
在步骤5015,将PCR主混合物溶液分配到安装在玻璃载玻片上的半透化FFPE组织切片的表面上。PCR主混合物溶液包括例如dNTP,DNA聚合酶,MgCl2和反应缓冲液。
在步骤5020,将其上带有空间寻址PCR引物的玻璃基底置于半透化FFPE组织切片的顶上,使得其上带有空间寻址PCR引物的玻璃基底的表面与组织切片。将空间寻址PCR引物从玻璃基底表面释放到组织切片的细胞空间中。
在步骤5025中,通过原位等温扩增来扩增基因组DNA。在一个实例中,扩增反应是重组酶聚合酶扩增(RPA)反应。下面的表1显示了可用于扩增基因组DNA的等温DNA扩增方法的其他实例。在另一个实例中,使用常规的基于PCR的全基因组扩增反应来扩增基因组DNA。在一实施例中,基于PCR的常规方法是改进的引物延伸预扩增PCR(iPEP PCR)。在另一个实例中,基于PCR的常规方法是简并寡核苷酸引发的PCR(DOP-PCR;例如Rubicon Picoplex试剂盒)。
在步骤5030,将其中具有扩增的基因组DNA的半透化组织样品从玻璃载玻片表面取出并收集在收集管中。在一个实例中,通过刮入Eppendorf管从玻璃载玻片上取出其中具有扩增的基因组DNA的半透化组织样品。由于扩增反应中使用的PCR引物上的生物素标记物,将扩增的DNA生物素化。使用基于链霉亲合素珠的纯化方案纯化扩增的生物素化DNA。
在步骤5035,通过使用3'-5'外切核酸酶消化除去残留的单链PCR引物。
在步骤5040,在靶向感兴趣的SNV的多重PCR反应中扩增DNA。例如,正向引物包括靶向感兴趣的SNV的基因特异性序列,SBS引物序列(例如SBS3)和P5序列。反向引物包括SBS12互补序列和P7序列。在另一个实例中,使用TSCA样方法来靶向感兴趣的DNA区域。
在步骤5045,对PCR产物进行测序。
图51显示了用于全基因组DNA的预扩增和空间索引化的空间寻址PCR引物5100的图。空间寻址PCR引物5100包括随机引物序列5110,空间地址序列5115和SBS引物序列5120(例如SBS12)。随机引物序列5110是9bp的序列,其用于在全基因组预扩增反应中扩增基因组DNA。空间地址序列5115是阵列上每个捕捉位点(空间特征)的独特序列。SBS引物序列5120包括生物素标记物5125。生物素标记物5125用于在随后的加工步骤中纯化扩增的DNA。
图52A和52B以图像显示了图50的方法5000的步骤。在步骤5010,给玻璃基底5210打印空间寻址PCR引物(未显示)以形成空间特征5215阵列。在此实例中,玻璃基底5210是具有直径为100μm的空间特征的2cm x 2cm的盖玻片。空间寻址PCR引物是例如图51的空间寻址PCR引物5100。
在步骤5015,安装在玻璃载玻片5225上的半透化组织切片5220覆盖有PCR主混合物溶液5230。半透化组织样品5220包括细胞5235。细胞5235包括基因组DNA分子5240。基因组DNA分子5240可以包括单核苷酸变异(SNV)5245。
在步骤5020,将玻璃基底5210放置在组织切片5220和PCR主混合物溶液5230的顶上,使得在其上具有空间寻址PCR引物5100(未显示)的表面玻璃基底5210与组织切片5220接触。将PCR引物5100(未显示)从玻璃基底5210的表面释放到组织切片5220的细胞空间中。
在步骤5025,使用PCR引物5100通过原位等温扩增来扩增基因组DNA。
在步骤5030,将其中具有扩增的DNA 5260的组织切片5220从玻璃载玻片5225的表面取出并收集在收集管5255中。在一个实例中,收集管5255是Eppendorf管。由于PCR引物5100上的生物素标记物5125,将扩增的DNA 5260生物素化。
在步骤5035(图52中未显示),使用基于链霉亲合素珠的纯化方案纯化扩增的DNA 5260。
在步骤5040(图52中未显示),通过使用3'-5'外切核酸酶消化来除去残留的单链PCR引物5100。
在步骤5045,在靶物特异性多重PCR反应中扩增DNA分子5260。例如,正向引物5265包括靶向SNV 5240的基因特异性序列5270,SBS引物序列5275(例如SBS3)和P5序列5280。反向引物5285包括SBS12互补序列5120a和P7序列5290。使用正向引物5265和反向引物5285合成的文库扩增子5295包括P5序列5285,SBS引物序列5275,SNV 5240,空间地址序列5115,SBS引物序列5120和P7序列5290。
4.8空间区室化
为了限制组织切片上空间寻址寡核苷酸(和其他反应组分或产物)的扩散并维持空间分辨率,可以使用“微孔反应器”或“微反应器”中的生化反应的区室化(compartmentalization)。空间区室化可以与本文所述的任何生化技术组合以表征组织中的转录物组和/或基因组变异,同时保留与组织中靶核酸起源相关的空间信息。
图53显示了微孔反应器覆盖(overlay)5300的一个实例的透视图。微孔反应器覆盖5300包括玻璃载玻片5310。安装在载玻片5310上的是组织切片5315。在一个实例中,组织切片5315是FFPE组织切片。将反应流体5320分配到玻璃载玻片5310上,使得组织切片5315被反应流体5320覆盖。在一个实例中,反应流体5320是用于扩增反应的PCR主混合物溶液。在另一个实例中,反应流体5320是逆转录混合溶液。将微孔基底5325置于载玻片5320上的反应流体5320和组织切片5315的顶上。微孔基底5325包括多个微孔5330。在每个微孔5330的底表面上打印多个空间寻址寡核苷酸(未显示)。微孔5330发挥进行生化反应(例如PCR扩增)的区室化反应室的功能。参照图54更详细地描述微孔5330。
图54显示了图53的微孔反应器覆盖5300的单个微孔5330的透视图。在此实例中,微孔5330具有约200μm的直径和约100μm的高度。微孔5350的体积为约3nL。在微孔5350的底表面上打印多个空间寻址寡核苷酸5410。空间寻址寡核苷酸5410包含每个微孔5330的独特空间地址序列。
图55A和55B显示了制作图53的微孔反应器覆盖5300的微孔基底5325的过程5500的实例。在此实例中只显示了微孔基底5325的一部分。
在第一步中且现在参考图55A,微孔基底5325包括玻璃基底5510。在一个实例中,玻璃基底5510是约1mm厚的亲水性玻璃基底。聚酰亚胺层5515设置在玻璃基底5510的表面上。在一个实例中,聚酰亚胺层5515是约100μm厚的黑色Kapton层。疏水层5520设置在聚酰亚胺层5515的表面上。在一个实例中,通过硅烷化聚酰亚胺层5515形成疏水层5520。
在下一步中且现在参考图55B中,在聚酰亚胺层5515中形成多个微孔5525。在该实例中,显示了两个微孔5525,但是可以使用微孔5525的任何数目和排列。在一个实例中,利用精确的紫外激光烧蚀在聚酰亚胺层5515中形成微孔5525,以除去聚酰亚胺层5515和疏水层5520的部分。在此实例中,微孔5525的直径为约200μm。下面的表2显示了微孔5525的合适尺寸的其他实例。微孔5525是亲水性区域,其发挥进行生化反应的反应室的功能。由于在微孔5525之间的间隙区域中存在疏水层5520,消除或显著降低反应组分或产物的侧向扩散。微孔5525的相对较小体积提供了小的反应体积和较高的效率反应。在微孔5525内的空间寻址寡核苷酸的区室化(限制)也避免了从玻璃基底表面释放打印的寡核苷酸可能需要的工艺步骤。
图56显示了来自组织样品的核酸的捕捉和空间区室化的微孔结构5600的实例的侧视图。微孔结构5600包括基底5610。在一个实例中,基底5610是玻璃载玻片。基底5610包括微孔5615的阵列。在一个实例中,使用蚀刻工艺,诸如通过精确紫外激光烧蚀在基底5610中形成微孔5615。微孔5615是亲水性区域,而微孔5615之间的间隙区域是疏水性区域。微孔5615发挥进行生化反应(例如RNA至cDNA的逆转录,基因组DNA的扩增)的反应室的功能。因为微孔5615之间的间隙区域是疏水性的,所以消除或显著减少反应组分或产物远离微孔5615的侧向扩散。在一个实例中,微孔5615具有约1mm的直径和约100μm的高度。微孔5615之间的间距约为1mm。
在每个微孔5615中沉积一定量的凝胶材料5620。选择沉积在每个微孔5615中的凝胶材料5620的量,使得随着随后水合凝胶材料5620,凝胶材料5620膨胀以填充微孔5615并从微孔5615突出,而不接触相邻的微孔5615。在一个实例中,凝胶材料5620是用共价连接的空间寻址捕捉寡核苷酸(未显示)功能化的水凝胶材料,如聚(N-(5-叠氮基乙酰胺基戊基)丙烯酰胺-共-丙烯酰胺)(PAZAM)。空间寻址捕捉寡核苷酸(未显示)包括空间地址序列,其对于每个微孔5615是独特的。空间寻址捕捉寡核苷酸还包括用于从组织切片中捕捉核酸(例如RNA或基因组DNA)的捕捉序列。捕捉序列可以是例如基因特异性捕捉序列或通用捕捉序列。可以通过打印(例如接触打印或压电打印)将凝胶材料5620沉积在每个微孔5615中。
图57显示了使用图56的微孔结构5600捕捉来自组织切片的核酸以制备测序文库的方法5700的实例的流程图。方法5700包括但不限于以下步骤。
在步骤5710,将一定量的凝胶材料5620沉积在微孔结构5600的每个微孔5615中,使得每个微孔5615包括其中具有独特空间地址序列的凝胶材料5620。在一些实施方案中,可以在使用前将其中沉积有凝胶材料5620的微孔结构5600贮存一段时间。
在步骤5715,在使用时,将微孔结构5600浸入生化反应溶液中以使凝胶材料5620水合。在一些实施方案中,生化反应溶液包括逆转录酶和反应成分以从组织样品中的靶定RNA合成cDNA。在一些实施方案中,生化反应溶液包括DNA聚合酶和反应组分以从组织样品中的靶定基因产生多个基因组DNA扩增子。将微孔结构5600浸入生化反应溶液达足以使凝胶材料5620水合的时间段。当水合凝胶材料5620时,凝胶材料5620膨胀以填充微孔5615并从微孔5615突出。然后,自生化反应溶液取出微孔结构5600。因为微孔5615之间的间隙区域是疏水性的,所以生化反应溶液不在微孔5615之间保留。与凝胶材料5620共价结合的空间寻址捕捉寡核苷酸位于每个微孔5615处。
在步骤5720,将组织切片置于微孔结构5600的顶上,使得组织切片与其中具有空间寻址捕捉寡核苷酸的水合凝胶材料5620接触。在一个实例中,组织切片是半透化FFPE组织切片。当组织切片接触其上具有空间寻址捕捉寡核苷酸的水合凝胶材料5620时,开始生化反应。因为微孔5615之间的间隙区域是疏水的,所以反应组分和产物位于每个微孔5615处。
在步骤5725,从凝胶材料5620中取出反应产物(例如cDNA或DNA)。可以使用珠纯化方法从PAZAM中纯化反应产物。在步骤5730,制备测序文库。
图58A显示了用于组织切除和制备空间寻址核酸文库的插针系统(pin system)5800的实例的侧视图。插针系统5800包括插针结构5810和微孔块(microwell block)5815。插针结构5810包括基底5820。在一个实例中,基底5820是玻璃基底。从基底5820突出的是插针5825阵列。在一个实例中,插针5825由玻璃材料形成。在一个实例中,插针5825具有约100μm的直径和约10μm至约20μm的高度。插针5825之间的间距为约100μm。在每个插针5825的末端是切割表面5830。切割表面5830可以是便于从组织切片取出组织样品的任何形状。参照图58B更详细地描述不同的切割表面的实例。可以用物质(未显示)包被插针5825以便于组织粘附到插针5825。在一个实例中,可以用聚赖氨酸包被插针5825。在另一个实例中,用粘着材料包被插针5825。在又一个实例中,用PAZAM包被插针5825。基底5820还包括对准孔5835。在此实例中,显示了两个对准孔5835,但是可以使用任何数目的对准孔5835。对齐孔5835用于将插针结构5810与微孔块5815对齐。
微孔块5815包括基底5840。基底5840包括微孔5845阵列。微孔5845排列成与插针结构5810中的插针5825对准。给微孔5845加载一定量的生化反应混合物5850。在各个实施方案中,生化反应混合物5850包括空间寻址捕捉寡核苷酸,用于将组织样品中的核酸靶向和标签化。生化反应混合物5850中的空间寻址捕捉寡核苷酸(未显示)包括对于每个微孔5845而言独特的空间地址序列。在一些实施方案中,生化反应混合物5850包括空间寻址捕捉寡核苷酸,逆转录酶和反应组分以从组织样品中的靶定RNA合成cDNA。在一些实施方案中,生化反应混合物5850包括空间寻址捕捉寡核苷酸,DNA聚合酶和反应组分以从组织样品中的靶定基因产生多个基因组DNA扩增子。在一些实施方案中,生化反应混合物5850是约1uL体积的溶液。在一些实施方案中,生化反应混合物5850是在使用微孔块5815前再水合的脱水反应混合物或其部分。用可刺穿膜5855,例如箔覆盖其中具有生化反应混合物5850的微孔5845。微孔块5815还包括对准插针5860。在此实例中,显示了两个对准插针5860,但是可以使用任何数目的对准插针5860。通过将微孔块5815的对准插针5860装配到插针结构5810的对准孔5835中,使微孔块5815与插针结构5810对齐。
图58B显示了图58A的插针结构5810上插针5825的不同切割表面5830的实例。在一个实例中,切割表面5830a是凹形的。在另一个实例中,切割表面5830b是“v”形。在另一个实例中,切割表面5830c具有相对浅的锯齿形边缘形状。在另一个实例中,切割表面5830d具有相对较深的锯齿形边缘形状。
图59显示了使用图58A的插针系统5800从组织切片捕捉核酸以制备测序文库的方法5900的实例的流程图。在此实例中,插针结构5810用作“触”插针结构来接触安装在固体基底上的组织切片,并取出组织样品以便随后递送至微孔区块5815以捕捉核酸。方法5900包括但不限于以下步骤。
在步骤5910中,使用插针结构5810收集来自组织切片的组织样品。例如,使玻璃载玻片上的组织切片与插针5825接触,使得组织样品粘附到切割表面5830。当从组织切片表面取出插针结构5810时,从组织切片中取出粘附组织样品。在一个实例中,组织切片是半透化FFPE组织切片。
在步骤5915,将其上具有组织样品的插针结构5810移动到微孔块5815。例如,将插针结构5810与微孔块5815对准并匹配。这样做,使可刺穿膜5855破裂并且将其上具有组织样品的插针5825浸入微孔5845内的生化反应混合物5850中。
在步骤5920,在温育期中,进行生化反应。
在步骤5925,从微孔块5815取出插针结构5810,并且从微孔5845收集其中具有反应产物(例如cDNA或基因组DNA扩增子)的生化反应混合物。
在步骤5930,制备测序文库。
图60显示了图58A的插针系统5800的侧视图,并且显示了图59的方法5900的步骤5910和5915。即,在步骤5910,将玻璃载玻片6015上的组织切片6010(例如FFPE组织切片)与插针系统5800的插针结构5815接触。与切割表面5830接触的组织5830粘附到插针5825。当从组织切片6010的表面上取出插针结构5815时,从组织切片上取出粘附组织样品6020。
在步骤5915,将其上具有组织样品6020的插针结构5810移动到微孔块5815。例如,将插针结构5810与微孔块5815对准并匹配。这样做,使可刺穿膜5855破裂并且将其上具有组织样品6020的插针5825浸入微孔5845内的生化反应混合物5850中。
图61显示了使用图58A的插针系统5800从组织切片捕捉核酸以制备测序文库的方法6100的另一个实例的流程图。在此实例中,插针结构5810用作“推”插针结构以接触安装在可刺穿基底上的组织切片,并将组织样品直接推入微孔5845中以捕捉核酸。方法6100包括但不限于以下步骤。
在步骤6110,将安装在可刺穿基底上的组织切片放置在微孔块5815上;即微孔块5815的可穿透膜5855上。在一个实例中,组织切片是半透化FFPE组织切片。
在步骤6115,将插针结构5810移动到微孔块5815。例如,将插针结构5810与微孔块5815对准并匹配。这样做,使其上具有组织切片的可刺穿基底和可刺穿膜5855破裂并且将其上具有组织样品的插针5825浸入微孔5845内的生化反应混合物5850中。
在步骤6120,在温育期中,进行生化反应。
在步骤6125,从微孔块5815取出插针结构5810,并且从微孔5845收集其中具有反应产物(例如cDNA或基因组DNA扩增子)的生化反应混合物。
在步骤6130,制备测序文库。
图62显示了图58A的插针系统5800的侧视图,并且以图示显示了图61的方法6100的步骤6110和6115。即,在步骤6110,将安装在可穿透基底6215上的组织切片6210(例如FFPE组织切片)放置在微孔块5815的顶上。在步骤6115,将插针结构5810移动到微孔块5815。将插针结构5810与微孔块5815对准并匹配。这样做,将其上具有组织切片6210的可刺穿基底6215和可刺穿膜5855破裂并且将其上具有组织样品6225的插针5825浸入微孔5845内的生化反应混合物5850中。
图63显示了用于从组织切片捕捉核酸以制备空间寻址核酸文库的毛细管“微反应器”系统6300的透视图。毛细管微反器系统6300包括多个毛细管6310。在一个实例中,毛细管6310可以单独用于收集和处理样品或者可以捆扎成阵列并用作单元。在此实例中,显示了4个毛细管6310,但是可以使用任何数目的毛细管6310。毛细管6310具有样品接触端6315和非接触端6320。每个毛细管6310的样品接触端6315包括小突出(protrusion)6325。在一个实例中,毛细管6310直径为约100μm。在每个毛细管6310的内表面上干燥一定量的空间寻址捕捉寡核苷酸(未显示),用于将来自组织切片的核酸靶向和标签化。空间寻址捕捉寡核苷酸(未显示)包括对于每个毛细管6310而言独特的空间地址序列。空间寻址捕捉寡核苷酸还包括用于捕捉来自组织切片的核酸(例如RNA或基因组DNA)的捕捉序列。捕捉序列可以是例如基因特异性捕捉序列或通用捕捉序列。
在使用时,给毛细管6310填充生化反应溶液6330。在一个实例中,通过“芯吸”或毛细管作用给毛细管6310填充生化反应溶液6330。在一些实施方案中,生化反应溶液6330包括逆转录酶和反应组分以从组织样品中靶定RNA合成cDNA。在一些实施方案中,生化反应溶液6330包括DNA聚合酶和反应组分以从组织样品中靶定的基因产生基因组DNA扩增子。
将毛细管6310“压印(stamp)”到基底6340上并压入安装在基底6340上的组织切片6335中。在一个实例中,组织切片6335是半透化FFPE组织切片。当将毛细管6310压入组织切片6335中时,通过突出6325将组织样品压入毛细管6310中。在一些实施方案中,在组织切片6335和基底6340之间放置惰性基底(未显示)。惰性基底用于密封毛细管6310的样品接触端6315。
图64图示了使用图63的毛细管微反应器系统6300从组织切片捕捉核酸以制备测序文库的方法6400的实例的流程图。方法6400包括但不限于以下步骤。
在步骤6410,在使用点时,通过毛细管作用将生化反应溶液6330加载到毛细管6310中。
在步骤6415,将其中具有生化反应溶液6330的毛细管6310压印到基底6340上并压入安装在基底6340上的组织切片6335中,以收集组织样品。
在步骤6420,从基底6340取出毛细管6310并在样品接触端6315密封以防止蒸发。在一些实施方案中,从基底6340取出毛细管6310,并将样品接触端6315压印到惰性基底上以密封而防止蒸发。在一些实施方案中,惰性基底位于组织切片6335和基底6340之间。当将毛细管6310压入组织切片6335中并穿过组织切片6335时,将样品接触端6315压入惰性基底中并密封而防蒸发。
在步骤6425,将毛细管6310的非接触端6320密封而防蒸发。在一些实施方案中,将非接触端6320压印到惰性基底上以密封而防蒸发。
在步骤6430,在温育期中,进行生化反应。
在步骤6435,从毛细管6310收集其中具有反应产物(例如cDNA或基因组DNA扩增子)的生化反应溶液并合并。
在步骤6440,制备测序文库。
4.9连续切片DNA/RNA工作流
可以在连续DNA/RNA工作流中使用空间寻址随机引物寡核苷酸的合并物,用于组织样品中DNA和RNA的空间检测和分析。例如,空间寻址随机引物寡核苷酸可以包括随机引物序列,空间地址序列,SBS引物序列和生物素标记物,如上文对图51的空间寻址PCR引物5100所述。在原位全基因组DNA扩增反应中使用空间寻址随机引物寡核苷酸的合并物,以对组织样品的一个连续切片中的DNA扩增和空间索引化。然后,使用扩增的且空间索引化的DNA产生测序文库。在原位RT反应中使用空间寻址随机引物寡核苷酸的相同合并物以从组织样品的第二连续切片中的RNA合成cDNA并且进行空间索引化。然后,使用空间索引化的cDNA产生第二测序文库。组合的DNA/RNA工作流提供单一组织样品的数据输出。
4.10配置用于核酸的空间检测和分析的液滴致动器
图65显示了液滴致动器6500的一部分的侧视图,所述液滴致动器配置用于空间检测和分析组织样品中的核酸。液滴致动器6500包括底部基底6510,所述底部基底通过液滴操作间隙6520与顶部基底6515分开。在液滴操作表面上的液滴操作间隙6520中进行液滴操作。底部基底6510包括液滴操作电极6525(例如电润湿电极)的排列。在液滴操作表面上的液滴操作电极6525上进行液滴操作。顶部基底6515包括参考电极6530。顶部基底6515还包括具有足以容纳孔片层6540的尺寸和形状的凹陷区域6535。参照图66更详细地描述孔片层6540。与凹陷区域6535结合的是电泳电极6545。底部基底6510包括相应的电泳电极6550。电泳电极6545和6550连接到电压源6555。孔片层6540,电泳电极6545和6550配置用于电泳转移并捕捉组织样品中的核酸,使得维持空间取向。此外,在凹陷区域6535中安装孔片层6540,使得孔片层6540的顶部与电泳电极6545之间存在一定量的空间6536。
在顶部基底6515中面对液滴操作间隙6520的表面上布置疏水层6560。类似地,在底部基底5610中面对液滴操作间隙6520的表面上布置另一个疏水层6565。
图66显示了图65的液滴致动器6500的孔片层6540的侧视图。孔片层6540包括具有顶部表面6615和底部表面6620的基底6610。基底6610包括孔6625的阵列。孔6625是基底6610中的通孔(through-holes)(例如微孔),其从顶部表面6615到底部表面6620延伸过基底6610。给每个孔6625填充包括多个独特的空间寻址捕捉探针(未显示)的凝胶或溶液或反应基质。每个空间地址对应于孔片层6540上的捕捉探针的位置。每个孔6625的位置可以与组织样品中的位置相关联。基底6610的底部表面6620可以包括疏水层(未显示)以促进液滴向孔片层6540和远离孔片层6540的运输。
图67A和图67B表示图65的液滴致动器6500的侧视图,并且显示分离组织样品中的核酸用于空间检测和分析的过程6700。过程6700是核酸分离方案的实例,其中将组织样品中的核酸电泳转移到含有用独特的空间地址序列加标签的独特空间寻址捕捉探针的微孔阵列中,并转移到用于液滴致动器上后续处理的液滴。过程6700包括但不限于以下步骤。
在一个步骤中并且现在参考图67A,安装在样品基底6715上的组织样品6710位于凹陷区域6535中;即,在孔片层6540上方的空间6536中。在一个实例中,组织样品6710是FFPE组织切片。在一个实例中,将组织样品6710直接放置在孔片层6540的表面上。在另一个实例中,将组织样品6710放置在分开组织样品6710与孔片层6540的物质(未显示),如凝胶或薄缓冲层上,以促进将核酸自组织样品6710电泳转移到孔片层6540中。使用液滴操作将液滴6720转运到与孔片层6540对准的特定液滴操作电极6525。将电压从电压源6555施加到电泳电极6545和6550,创建电场。在存在电场的情况下,将多个核酸6725从组织样品6710转移到孔片层6540的孔6625中。用空间地址序列(未显示)对孔片层6540的每个孔6625中的核酸6725加标签,所述空间地址序列对应于孔片层6540上的每个孔6625的位置。
在另一个步骤中且现在参考图67B,在足以将核酸6725捕捉到包含在每个孔6625中的空间寻址捕捉探针上的一段时间之后,将核酸6725以电泳转移到液滴6720中。使用液滴操作将其中具有加标签的核酸6725的液滴6720转运得远离孔片层6540,用于液滴致动器6500上的后续处理步骤。例如,液滴致动器6500可以包含反应区(未显示),用于进行测序文库制备方案中的处理步骤(RNA到cDNA的逆转录,外切核酸酶I消化,PCR扩增和Nextera文库制备)。液滴致动器6500还可以包括用于进行DNA测序方案(例如SBS方案)的测序区(未显示)。测序区可以是例如测序流动池。
4.11基于DNA标签片段化的空间组织序型分析
如本文所用,“标签片段化(tagmentation)”是转座酶介导的片段化(fragmentation)和标签化(tagging)的过程。标签片段化常常涉及通过包含与包含转座子末端序列的衔接子复合的转座酶的转座体复合物来修饰DNA。标签片段化导致同时的DNA片段化和衔接子与DNA双链体片段的两条链的5'末端的连接。
本公开部分基于下述认识,即标签片段化可以有效用于在捕捉阵列上空间寻址来自组织样品的核酸。
图68显示了使用DNA的标签片段化对组织样品中的基因组DNA进行序型分析的方法6800的实例的流程图。在此实例中,方法6800用于对基因组DNA中感兴趣的SNV进行序型分析。方法6800可以包括但不限于一些或所有以下步骤。
在步骤6810,给玻璃基底打印空间寻址寡核苷酸以形成空间特征的阵列。在一些实施方案中,可以在2cmx2cm的盖玻片上打印空间寻址寡核苷酸以形成直径为100μm,间距为35μm的空间特征的阵列。在一些实施方案中,可以在玻璃载玻片的表面上制作的微孔中打印空间寻址寡核苷酸。可以使用例如商品化打印技术在盖玻片或玻璃载玻片上打印空间寻址寡核苷酸。空间寻址寡核苷酸可以包含接头序列,SBS引物序列,空间地址序列和19b镶嵌末端(Mosaic End,ME)序列,如参考图69A更详细描述的。在一些实施方案中,ME序列是Tn5转座酶识别序列。在一些实施方案中,ME序列是Mu转座酶识别序列。空间寻址寡核苷酸可以在分子的5'端进一步包含修饰以可逆地附着于盖玻片。在一些实施方案中,空间寻址寡核苷酸可以包含5'二硫化物修饰,如对图32A,32B和32C的过程3200的空间寻址捕捉探针3100所述。在一些实施方案中,空间寻址PCR引物可以包括5'光可切割接头,如对图33A,33B和33C的过程3300的空间寻址捕捉探针3100所述。
在步骤6815,将反向互补寡核苷酸序列与ME序列杂交以形成双链DNA区域。
在步骤6820,将转座酶酶溶液添加到空间寻址寡核苷酸阵列的表面上以在双链DNA的每个区域处形成转座体同二聚体。在一些实施方案中,转座酶酶溶液包含Tn5。在一些实施方案中,转座酶酶溶液包含Mu。
在步骤6825,将组织切片置于阵列上,使得使其上具有空间寻址寡核苷酸和转座体同二聚体的阵列基底的表面与组织切片接触。在一个实例中,组织切片是FFPE组织切片。
在步骤6830,用转座体复合物对双链DNA进行标签片段化。例如,在US2010/0120098和US2011/0287435(其在此通过引用完整并入)中详细描述了用于处理核酸的方法,组合物和试剂盒,以及具体地使用转座子组合物对DNA进行片段化和标签化的方法和组合物。
在步骤6835,使用基因特异性引物和包含与SBS引物序列的互补区的通用引物扩增经标签片段化的DNA,以产生经标签片段化的基因组DNA文库。
在步骤6840,对经标签片段化的基因组DNA文库进行测序。
图69A,69B和69C显示了图68的方法6800的步骤。在步骤6810(参见图69A)中,对阵列表面6910打印空间寻址寡核苷酸6915,用于全基因组DNA的空间索引化和标签片段化。在此实例中,显示了单一空间寻址寡核苷酸6915,但是可以将任何数目的6915寡核苷酸固定化在阵列表面6010上。空间寻址寡核苷酸6015包括接头区6920,SBS引物区6925,空间地址区6930和ME区6935。空间地址区6930包括用于阵列上的每个捕捉位点(空间特征)的独特序列。例如,在US2010/0120098和US2011/0287435中详细描述ME区域6935的19bp ME序列或转座子末端。例如在US2010/0120098和US2011/0287435中详细描述了用于处理核酸的方法,组合物和试剂盒,以及具体地使用转座子组合物对DNA进行片段化和标签化的方法和组合物。
该实例中的接头区域6920包含可切割序列,其可以用于从阵列表面6910释放捕捉的核酸,使得空间地址区域6930包括在释放的核酸中并且将核酸“标签化”。SBS引物区6925包括可用于合成测序(SBS)过程的SBS引物序列(例如SBS12或SBS3)。SBS引物区6925也可用于扩增反应中,以产生测序文库,如参考图12和图13更详细描述。
在步骤6815(参见图69A),ME反向互补序列6940与ME区6935杂交。
在步骤6820(参见图69B),将转座酶酶溶液(未显示)添加到阵列表面6910上以在双链DNA的每个区域处形成转座体同二聚体6945。在一些实施方案中,转座体末端包含Mu转座体末端,并且转座酶是Mu转座酶。在一些实施方案中,转座体末端包含Tn5转座体末端,并且转座酶是Tn5转座酶。
在步骤6825(参见图69B),将组织切片6950放置在阵列表面6910的顶上,使得其上的空间寻址寡核苷酸6915和转座体同二聚体6945与组织切片6950接触。在一些实施方案中,组织切片6950是FFPE组织切片。组织切片6950包括细胞6955。细胞6955包括基因组DNA分子6960。基因组DNA分子6960可以包括单核苷酸变异(SNV)6965。
在步骤6830(参见图69C),将基因组DNA6960进行标签片段化。例如,将基因组DNA 6960进行标签片段化,使得SNV 6965在标签片段化事件上游是“A”或在标签片段化事件下游是“B”。
在步骤6835(参见图69C),扩增经标签片段化的基因组DNA 6960。例如,如果SNV 6965在标签片段化事件上游是“A”,则使用基因特异性引物6970和通用引物6975扩增基因组DNA 6960,所述通用引物6975包含SBS引物区6925的互补区域。如果SNV 6965在标签片段化事件的下游是“B”,则使用基因特异性引物6980和通用引物6975扩增基因组DNA 6960。
在步骤6840(未在图69A,69B和69C中显示)中,对PCR产物进行测序。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的捕捉阵列,其包含含有捕捉探针(例如6915)的捕捉位点,该捕捉探针包含空间地址区(例如6930)和转座子末端(TE)区域(例如6935)。在一些实施方案中,所述捕捉探针还包含可切割区域(例如6920)和SBS引物结合区(例如6925)。在一些实施方案中,转座子末端区域与反向互补寡核苷酸(例如6940)杂交以形成双链转座子末端区域。在一些实施方案中,TE区包含ME序列。
在一些实施方案中,捕捉阵列还包括转座酶以形成转座体(例如6945)。
在一些实施方案中,转座体末端包含Mu转座体末端,并且转座酶为Mu转座酶。在一些实施方案中,转座体末端包含Tn5转座体末端,并且转座酶是Tn5转座酶。
在另一个方面,本文提供了用于空间检测和分析组织样品中的核酸的方法,包括提供本文描述的捕捉阵列。在一些实施方案中,捕捉阵列包括含有捕捉探针(例如6915)的捕捉位点,所述捕捉探针包含空间地址区(例如6930)和转座子末端(TE)区(例如6935)。在一些实施方案中,捕捉探针还包含可切割区域(例如6920)和SBS引物结合区(例如6925)。
在一些实施方案中,方法还包括使捕捉阵列与作为TE区(例如6940)的反向互补物的寡核苷酸接触以形成双链转座子末端区。
在一些实施方案中,方法还包括将捕捉阵列与转座酶接触以形成转座体(例如6945)。在一些实施方案中,转座体末端包含Mu转座体末端,并且转座酶为Mu转座酶。在一些实施方案中,转座体末端包含Tn5转座体末端,并且转座酶是Tn5转座酶。
在一些实施方案中,方法还包括使所述捕捉阵列与组织样品接触,使得阵列上的捕捉位点的位置可以与组织样品中的位置相关联;并且允许组织样品的基因组DNA和所述捕捉位点处的转座酶之间发生标签片段化反应。在一些实施方案中,基因组DNA包含SNV。
在一些实施方案中,所述方法还包括分析标签片段化DNA的序列。在一些实施方案中,对标签片段化的DNA测序包括使用基因特异性引物和通用引物的组合进行测序反应。在一些实施方案中,分析所述标签片段化的DNA的序列包括检测SNV。
在一些实施方案中,所述方法还包括将标签片段化的DNA的序列与组织样品中基因组DNA的位置相关联。在一些实施方案中,将标签片段化的DNA的序列关联包括将SNV与组织样品中的位置相关联。
4.12测序方法
本文中所述的方法可结合多种核酸测序技术使用。特别适用的技术是如下那些,其中在阵列中的固定位置处附着核酸,使得它们的相对位置不改变并且其中将阵列重复成像。在不同颜色通道(例如,与用于将一种核苷酸碱基类型与另一种区分开的不同标记物一致)中获得图像的实施方案是特别适用的。在一些实施方案中,确定靶核酸的核苷酸序列的方法可以是自动化过程。优选实施方案包括合成测序(sequencing-by-synthesis)(“SBS”)技术。
合成测序(“SBS”)技术通常涉及通过对模板链反复加入核苷酸酶促延伸新生核酸链。在SBS的传统方法中,可以在每次递送中在存在聚合酶的情况下将单核苷酸单体提供给靶核苷酸。然而,在本文中所述的方法中,可以在递送中在存在聚合酶的情况下对靶核酸提供超过一类核苷酸单体。
SBS可以利用具有终止剂部分的核苷酸单体或缺少任何终止剂部分的核苷酸单体。使用缺少终止剂的核苷酸单体的方法包括例如焦磷酸测序和使用γ-磷酸标记的核苷酸的测序,如下文更为详细阐述。在使用缺乏终止剂的核苷酸单体的方法中,每个循环中添加的核苷酸数目通常是可变的,并且取决于模板序列和核苷酸递送的模式。对于利用具有终止剂部分的核苷酸单体的SBS技术,终止剂可以在使用的测序条件下是有效不可逆转的,利用双脱氧核苷酸的传统Sanger测序就是如此,或者终止剂可以是可逆的,由Solexa(现为Illumina,Inc.)开发的测序方法就是如此。
SBS技术可以利用具有标记物部分的核苷酸单体或缺乏标记物部分的核苷酸单体。因此,可以基于标记物的特性,如标记物的荧光;核苷酸单体的特征如分子量或电荷;核苷酸掺入的副产物,如焦磷酸盐的释放等来检测掺入事件。在测序试剂中存在两种或更多种不同核苷酸的实施方案中,不同的核苷酸可以彼此区分,或者备选地,两种或更多种不同的标记物在所使用的检测技术下可以是不可区分的。例如,测序试剂中存在的不同核苷酸可以具有不同的标记物,并且可以使用合适的光学来区分它们,如由Solexa(现在的Illumina,Inc.)开发的测序方法例示。
优选实施方案包括焦磷酸测序技术。焦磷酸测序检测无机焦磷酸盐(PPi)的释放,因为特定的核苷酸被掺入新生链中(Ronaghi,M.,Karamohamed,S.,Pettersson,B.,Uhlen,M.and Nyren,P.(1996)“Real-time DNA sequencing using detection of pyrophosphate release.”Analytical Biochemistry 242(1),84-9;Ronaghi,M.(2001)"Pyrosequencing sheds light on DNA sequencing."Genome Res.11(1),3-11;Ronaghi,M.,Uhlen,M.and Nyren,P.(1998)"A sequencing method based on real-time pyrophosphate."Science 281(5375),363;美国专利号6,210,891;美国专利号6,258,568和美国专利号6,274,320,其公开内容通过引用整体并入本文)。在焦磷酸测序中,可以通过ATP硫酸化酶被立即转化为三磷酸腺苷(ATP)释放的PPi,并且通过萤光素酶产生的光子检测产生的ATP水平。可以将要测序的核酸附着到阵列中的特征,并且可以对阵列进行成像以捕捉由于在阵列的特征处掺入核苷酸而产生的化学发光信号。在用特定核苷酸类型(例如A、T、C或G)处理阵列后,可以获得图像。在添加每种核苷酸类型后获得的图像就阵列中检测的那些特征而言将有所不同。图像中的这些差异反映了阵列上特征的不同序列内容。然而,每个特征的相对位置在图像中将保持不变。可以使用本文中阐述的方法来存储、处理和分析图像。例如,可以以下述方式处理用每种不同核苷酸类型处理阵列后获得的图像,所述方式与本文中对从基于可逆终止剂的测序方法的不同检测通道获得的图像的例示相同。
在另一种示例性类型的SBS中,循环测序通过逐步添加可逆终止剂核苷酸来完成,所述可逆终止剂核苷酸含有例如可切割或光可漂白染料标记物,如记载于例如国际专利公开号WO 04/018497和美国专利7,057,026,其公开内容通过引用并入本文。此方法正得到Solexa(现在为Illumina Inc.)商业化,并且还记载于国际专利公开号WO 91/06678和国际专利公开号WO 07/123,744,其各自通过引用并入本文。荧光标记的终止剂(其中既可以逆转终止又可以切割荧光标记物)的可用性有助于有效的循环可逆终止(CRT)测序。聚合酶也可以共工程化改造为有效地掺入这些修饰的核苷酸并且从这些修饰的核苷酸延伸。
优选地,在基于可逆终止剂的测序实施方案中,标记物在SBS反应条件下基本上不抑制延伸。然而,检测标记物可以是可除去的,例如通过切割或降解。可以在将标记物掺入排列的核酸特征后捕捉图像。在具体实施方案中,每个循环涉及将四种不同核苷酸类型同时递送至阵列,并且每种核苷酸类型具有光谱独特的标记物。然后,可以获得四个图像,每个图像使用对四种不同标记物之一具有选择性的检测通道。或者,可以顺序添加不同的核苷酸类型,并且可以在每个添加步骤之间获得阵列的图像。在此类实施方案中,每个图像将显示已经掺入特定类型的核苷酸的核酸特征。由于每个特征的不同序列内容,不同的图像中将存在或不存在不同的特征。然而,特征的相对位置在图像中将保持不变。可以如本文中阐述的那样储存、处理和分析从此类可逆终止剂-SBS方法获得的图像。在图像捕捉步骤后,可以除去标记物,并且可以除去可逆终止剂部分,用于核苷酸添加和检测的随后循环。标记物当它们在特定循环中以及后续循环前被检出后的除去可以提供降低背景信号和循环之间串扰的优点。下文阐述了有用的标记物和除去方法的实例。
在具体实施方案中,一些或全部核苷酸单体可以包括可逆终止剂。在此类实施方案中,可逆终止剂/可切割荧光团(fluors)可以包括通过3’酯连接与核糖部分连接的荧光团(Metzker,Genome Res.15:1767-1776(2005),其通过引用并入本文)。其它方法已经将终止剂化学与荧光标记物的切割分开(Ruparel et al.,Proc Natl Acad Sci USA 102:5932-7(2005),其全部内容通过引用并入本文)。Ruparel等人描述了可逆终止剂的开发,所述可逆终止剂使用小的3’烯丙基封闭延伸,但是可以通过用钯催化剂的短处理容易地解封闭。通过光可切割接头将荧光团附着到基底,该光可切割接头可以通过对长波长UV光的30秒暴露而容易地切割。因此,可以使用二硫化物还原或光切割作为可切割接头。可逆终止的另一种方法是使用在dNTP上放置大体积染料后发生的自然终止。dNTP上带电荷的大体积染料的存在可通过空间和/或静电阻碍起有效终止剂作用。一个掺入事件的存在防止进一步的掺入,除非除去染料。染料的切割除去荧光团,并且有效逆转终止。经修饰的核苷酸的实例也描述于美国专利7,427,673和美国专利7,057,026,其公开的内容通过引用整体并入本文。
可以与本文中所述的方法和系统一起利用的另外的例示性SBS系统和方法记载于美国专利公开号2007/0166705、美国专利公开号2006/0188901、美国专利7,057,026、美国专利公开号2006/0240439、美国专利公开号2006/0281109、国际专利公开号WO 05/065814、美国专利公开号2005/0100900、国际专利公开号WO 06/064199、国际专利公开号WO 07/010,251、美国专利公开号2012/0270305和美国专利公开号2013/0260372,其公开内容通过引用完整并入本文。
一些实施方案可以利用使用少于四种不同标记物检测四种不同核苷酸。例如,可以利用美国专利公开号2013/0079232的并入材料中描述的方法和系统来进行SBS。作为第一个实例,可以在相同波长处检测一对核苷酸类型,但是基于该对中的一名成员与另一成员相比的强度差异,或者基于对该对中的一名成员的改变(例如通过化学修饰、光化学修饰或物理修饰)来区分一对核苷酸类型,所述改变引起与对所述对中的另一名成员检测的信号相比引起明显信号出现或消失。作为第二个实例,可以在特定条件下检测四种不同核苷酸类型中的三种,而第四种核苷酸类型缺乏在那些条件下可检测的标记物,或者在那些条件下被最小程度检测(例如,由于背景荧光引起的最小程度检测,等等)。将前三种核苷酸类型掺入核酸中可以基于其各自信号的存在来确定,并且将第四种核苷酸类型掺入核酸中可以基于任何信号的不存在或最小程度检测来确定。作为第三个实例,一种核苷酸类型可以包括在两个不同通道中检测到的标记物,而仅在通道之一中检测到其它核苷酸类型。认为上述三种示例性配置不是相互排斥的,并且可以以各种组合使用。组合所有三个实例的示例性实施方案是基于荧光的SBS方法,其使用第一通道中检测的第一核苷酸类型(例如,具有当通过第一激发波长激发时在第一通道中检测到的标记物的dATP)、在第二通道中检测到的第二核苷酸类型(例如具有当通过第二激发波长激发时在第二通道中检测到的标记物的dCTP)、在第一和第二通道两者中检测到的第三核苷酸类型(例如具有当通过第一和/或第二激发波长激发时在这两个通道中检测到的标记物的dTTP)和缺乏在任一通道中检测不到或最小程度检测的标记物的第四种核苷酸类型(例如,没有标记物的dGTP)。
此外,如美国专利公开号2013/0079232的并入材料所述,可以使用单通道获得测序数据。在此类所谓的单染色测序方法中,第一种核苷酸类型是标记的,但在产生第一图像之后除去标记物,并且第二种核苷酸类型仅在产生第一图像后被标记。第三种核苷酸类型在第一和第二图像两者中保留其标记物,并且第四种核苷酸类型在这两个图像中保持未标记。
一些实施方案可以利用连接测序技术。此类技术利用DNA连接酶掺入寡核苷酸并鉴定此类寡核苷酸的掺入。寡核苷酸通常具有不同标记物,所述标记物同与寡核苷酸杂交的序列中的特定核苷酸的身份相关。与其它SBS方法一样,可以在用标记的测序试剂处理核酸特征阵列后获得图像。每个图像将显示已经并入特定类型的标记物的核酸特征。由于每个特征的不同序列内容,在不同图像中将存在或不存在不同的特征,但是特征的相对位置在图像中将保持不变。可以存储、处理和分析从基于连接的测序方法获得的图像,如本文阐述的。可以与本文中描述的方法和系统一起使用的示例性SBS系统和方法记载于美国专利6,969,488、美国专利6,172,218、和美国专利6,306,597,其公开内容通过引用整体并入本文。
一些实施方案可以利用纳米孔测序(Deamer,D.W.&Akeson,M."Nanopores and nucleic acids:prospects for ultrarapid sequencing."Trends Biotechnol.18,147-151(2000);Deamer,D.and D.Branton,"Characterization of nucleic acids by nanopore analysis".Acc.Chem.Res.35:817-825(2002);Li,J.,M.Gershow,D.Stein,E.Brandin,and J.A.Golovchenko,"DNA molecules and configurations in a solid-state nanopore microscope"Nat.Mater.2:611-615(2003),其公开内容通过引用整体并入本文)。在此类实施方案中,靶核酸通过纳米孔。纳米孔可以是合成孔或生物膜蛋白,如α-溶血素。当靶核酸通过纳米孔时,可以通过测量孔的电导率的波动来鉴定每个碱基对。(美国专利7,001,792;Soni,G.V.&Meller,"A.Progress toward ultrafast DNA sequencing using solid-state nanopores."Clin.Chem.53,1996-2001(2007);Healy,K."Nanopore-based single-molecule DNA analysis."Nanomed.2,459-481(2007);Cockroft,S.L.,Chu,J.,Amorin,M.&Ghadiri,M.R."A single-molecule nanopore device detects DNA polymerase activity with single-nucleotide resolution."J.Am.Chem.Soc.130,818-820(2008),其公开内容通过引用整体并入本文)。可以存储、处理和分析从纳米孔测序获得的数据,如本文中阐述。特别地,根据本文所阐述的光学图像和其它图像的示例性处理,数据可以作为图像处理。
一些实施方案可以利用涉及DNA聚合酶活性的实时监测的方法。可以通过携带荧光团的聚合酶和γ-磷酸标记的核苷酸之间的荧光共振能量转移(FRET)相互作用来检测核苷酸掺入,如记载于例如美国专利7,329,492和美国专利7,211,414(其各自通过引用并入本文),或者可以用如记载于例如美国专利7,315,019(其通过引用并入本文)的零模式波导,并且使用如例如美国专利7,405,281和美国专利公开号2008/0108082(其各自通过引用并入本文)中所述的荧光核苷酸类似物和工程化聚合酶来检测核苷酸掺入。照明可以限于表面栓系的聚合酶周围的仄升(zeptoliter)量级体积,使得可以在低背景下观察到荧光标记的核苷酸的掺入(Levene,M.J.et al."Zero-mode waveguides for single-molecule analysis at high concentrations."Science 299,682-686(2003);Lundquist,P.M.et al."Parallel confocal detection of single molecules in real time."Opt.Lett.33,1026-1028(2008);Korlach,J.et al."Selective aluminum passivation for targeted immobilization of single DNA polymerase molecules in zero-mode waveguide nano structures."Proc.Natl.Acad.Sci.USA 105,1176-1181(2008),其公开内容通过引用整体并入本文)。可以存储、处理和分析从此类方法获得的图像,如本文中阐述。
一些SBS实施方案包括在将核苷酸掺入延伸产物中时释放的质子的检测。例如,基于释放的质子的检测的测序可以使用可从Ion Torrent(Guilford,CT,Life Technologies子公司)商购的电检测器和相关技术或美国专利公开号2009/0026082;美国专利公开号2009/0127589;美国专利公开号2010/0137143;或美国专利公开号2010/0282617(其各自通过引用并入本文)中描述的测序方法和系统。本文中阐述的使用动力学排除来扩增靶核酸的方法可以容易地应用于用于检测质子的基底。更具体地,本文阐述的方法可用于产生用于检测质子的扩增子的克隆群体。
可以有利地以多重形式进行上述SBS方法,使得同时操作多个不同的靶核酸。在具体实施方案中,可以在常见的反应容器中或在特定基底的表面上处理不同的靶核酸。这允许以多重方式方便地递送测序试剂、除去未反应的试剂和检测掺入事件。在使用表面结合靶核酸的实施方案中,靶核酸可以为阵列形式。在阵列形式中,靶核酸通常可以以空间上可区分的方式结合到表面。靶核酸可以通过直接共价附着、附着于珠或其它颗粒或结合到附着于表面的聚合酶或其它分子来结合。阵列可以包括每个位点(也称为特征)处的靶核酸的单一拷贝,或者可以在每个位点或特征处存在具有相同序列的多个拷贝。可以通过扩增方法如桥式扩增或乳液PCR产生多个拷贝,如下文进一步详述。
本文中阐述的方法可以使用以多种密度之任一具有特征的阵列,包括例如至少约10个特征/cm2、100个特征/cm2、500个特征/cm2、1,000个特征/cm2、5,000个特征/cm2、10,000个特征/cm2、50,000个特征/cm2、100,000个特征/cm2、1,000,000个特征/cm2、5,000,000个特征/cm2、或更高。
本文中阐述的方法的优点在于它们提供并行的多个靶核酸的快速和有效检测。因此,本公开提供了能够使用本领域已知的技术(如上面例示的那些)制备和检测核酸的集成系统。因此,本公开的集成系统可以包括能够将扩增试剂和/或测序试剂递送到一个或多个固定化DNA片段的流体组分,该系统包括诸如泵、阀、储器、流体线等的部件。流动池可以在用于检测靶核酸的集成系统中配置和/或使用。示例性流动池记载于例如美国专利公开号2010/0111768 A1和美国专利申请号13/273,666,其各自通过引用并入本文。如对流动池示例,集成系统的一个或多个流体部件可用于扩增方法和检测方法。以核酸测序实施方案为例,集成系统的一种或多种流体部分可用于本文中阐述的扩增方法并且用于递送测序方法(如上文例示的测试方法)中的测序试剂。或者,集成系统可以包括用于实施扩增方法和实施检测方法的分开的流体系统。能够创建扩增核酸并确定核酸序列的集成测序系统的实例包括但不限于MiSeqTM平台(Illumina,Inc.,San Diego,CA)和美国专利申请号13/273,666中描述的装置,其通过引用并入本文。例如,MiSeqTM平台可以用捕捉探针5'CAACGATCGTCGAAATTCGC[靶引物]3'和5'[靶引物]AGATCGGAAGAGCGTCGTGTA3'进行,其中[靶引物]是与靶核酸互补的序列。
4.13结束评述
实施方案的前述详细描述参考说明本公开的具体实施方案的附图。具有不同结构和操作的其他实施方案不脱离本公开的范围。本说明书分为各部分,仅供读者方便。标题不应解释为限制本文提供的公开范围。这些定义旨在作为本文提供的公开内容的一部分。应该理解,在不脱离所公开的实施方案的范围的情况下,可以改变本公开的各种细节。此外,前面的描述仅用于说明的目的,而不是为了限制的目的。
5.实施例
5.1空间NGS用于检测单核苷酸变异的灵敏性
本实施例证明了与整体测序(bulk sequencing)相比,使用空间寻址测序可以显著提高SNV检测的灵敏性。
使用模拟数据集评估使用整体测序或空间寻址测序数据的SNV检测的灵敏性。用于推导整体测序数据集的计算基于以下假设:(1)典型的FFPE切片是1.5cm x 1.5cm,其为约225,000,000μm2;(2)典型的细胞为20 x 20μm,其面积为约400μm2;(3)典型FFPE切片中的细胞数目为约563,000(即,225,000,000μm2FFPE区块÷400μm2细胞=每个切片的约563K个细胞);和(4)FFPE切片中的细胞堆积密度为约70%,则典型FFPE切片中的细胞数目为约400,000。下面的表3显示了整体测序中SNV检测灵敏性的模拟数据。为了使用整体测序鉴定克隆突变细胞的罕见群体,变体频率(%SNV)需要高于测序错误率,其为约1%。例如,FFPE切片中的1个变体细胞(“局部突变细胞”)具有0.00025(即,(1细胞÷FFPE切片中的400,000细胞)x 100)=0.00025%SNV)的变体频率(%SNV),其完全低于测序误差率,因此在整体测序数据中检测不到。在整体测序数据中检测SNV需要至少约4000个细胞,例如4096个细胞÷FFPE切片中的400,000个细胞)x 100)=1.024,其高于测序误差率。
已经发现使用空间寻址测序可以显著提高SNV检测的灵性,如使用基于2cm x 2cm阵列上覆盖的1.5cm x 1.5cm组织切片的模拟数据集例示。图70显示空间地址覆盖7000的平面图。空间地址覆盖7000包括基底7005。在一个实例中,基底7005是平面玻璃基底,如尺寸为2cm x 2cm的盖玻片。在基底7005表面上打印多个独特的特征(未显示),它们是含有空间寻址寡核苷酸的区域。参考图71更详细地描述单一空间特征的实例。在基底7005上覆盖组织切片7010。在一个实例中,组织切片7005是1.5cm x 1.5cm大小的FFPE组织切片。
图71显示了图70的基底7005上的单一空间特征7100的平面图。在此实例中,空间特征7100是100μm x 100μm正方形(面积=10,000μm2)。空间特征7100具有足够的尺寸以涵盖包含在组织样品中的多个细胞7105。细胞7105可以是正常细胞(例如7105a)或变体(突变)细胞(例如7105b)。在一个实例中,细胞7105的尺寸为20μm x 20μm(面积=400μm2),并且组织切片中的细胞填充密度为约70%。基于这些参数,包含在空间特征7100中的细胞7105的数目是约18(即,(10,000μm2÷400μm2)*0.7)=约18)。
下面的表4显示了基于图70和图71的空间地址覆盖7000的空间寻址测序数据中的SNV检测灵敏性的模拟数据。例如,在单一空间特征3000中的1个变体细胞(“局部突变细胞”)具有约6(即,(1细胞÷18细胞/空间特征)*100=6%SNV)的变体频率(%SNV),其高于1%的测序误差率,并且因此可以在空间寻址测序数据中检出。相比之下,整体测序数据中SNV检测需要约4000个细胞或更多细胞。
可以如下计算实现期望水平的检测灵敏性需要的基底7005上的空间特征7100的数目(x):(X+Y阵列长度)=(空间特征边缘x x)+((x-1)x空间特征间隔),其中阵列长度为20,000μm,空间特征边缘为100μm,并且空间特征间隔为例如50μm;则对于X维度为x=133个空间特征和对于Y维度为133个空间特征。基底7005(2cm x 2cm)上的特征总数为133 x 133=约17,689。
- 还没有人留言评论。精彩留言会获得点赞!