一种pH、温度和盐适应性改良的内切木聚糖酶突变体及其应用的制作方法
本发明涉及基因工程及蛋白质改造技术领域,具体来说是一种pH、温度和盐适应性改良的内切木聚糖酶突变体及其应用。
背景技术:
木聚糖是一种来源丰富的半纤维素,可被内切木聚糖酶降解为木寡糖和/或木糖,降解产物可进一步发酵为其它产物或被直接利用,因而,内切木聚糖酶在食品、酿酒、饲料、纺织及造纸等领域都具有重要价值(Collins et al.FEMS Microbiology Reviews,2005,29:3–23.)。内切木聚糖酶来源广泛,存在于多种多样的细菌、放线菌、真菌等生物中,但直接从自然界生物中筛选到的木聚糖酶往往在某一酶学性质上具有优势,而在另一酶学性质上具有劣势,如霉菌来源的内切木聚糖酶往往在碱性条件不具有催化活性,从耐碱性微生物来源的内切木聚糖酶往往在酸性条件下不具有催化活性,从嗜热微生物来源的内切木聚糖酶往往在低温下不具有催化活性,而从低温环境微生物来源的内切木聚糖酶往往热稳定性差。综合酶学性质较好的内切木聚糖酶具有更大的应用价值,能带来更大的经济效益(Verma and Satyanarayana,Bioresource Technology,2012,117:360–367.)。
技术实现要素:
本发明的目的是提供一种pH、温度和盐适应性改良的内切木聚糖酶突变体S45F07,所述突变体S45F07的氨基酸序列如SEQ ID NO.1所示。
进一步的,所述突变体S45F07的最适pH为5.5,最适温度为80℃,在pH4.0条件下具有55%的酶活,在90℃条件下具有65%的酶活,在3.0-25.0%(w/v)的NaCl中具有54-110%的酶活,在20.0-30.0%(w/v)的KCl中具有84-99%的酶活。
本发明的第二目的是提供一种pH、温度和盐适应性改良的内切木聚糖酶突变体S45F07的编码基因s45f07,其核苷酸序列如SEQ ID NO.2所示。
本发明的第三目的是提供一种包含内切木聚糖酶突变体S45F07编码基因s45f07的重组载体。
本发明的第四目的是提供一种包含内切木聚糖酶突变体S45F07编码基因s45f07的重组菌。
本发明的第五目的是提供一种pH、温度和盐适应性改良的内切木聚糖酶突变体S45F07在食品、饲料、造纸行业,特别是高盐环境生物技术领域中的应用。
制备本发明pH、温度和盐适应性改良的内切木聚糖酶突变体S45F07的方法按以下步骤进行:
1)将s45f07和表达载体pEasy-E2相连接,并将连接产物转化大肠杆菌BL21-Gold(DE3),获得包含s45f07的重组菌株;
2)培养重组菌株,诱导重组突变内切木聚糖酶表达;
3)回收并纯化所表达的突变内切木聚糖酶S45F07。
4)活性测定。
本发明的有益技术效果是:与野生酶相比,突变内切木聚糖酶S45F07的pH、温度和盐适应性发生了改变。纯化的突变酶S45F07、野生酶rXynAGN16L和rXynAHJ3的最适pH分别为5.5、5.5和6.0;在pH4.0条件下,突变酶S45F07具有55%的酶活,而rXynAGN16L和rXynAHJ3无酶活;在pH9.0–11.0条件下,突变酶S45F07的酶活比野生酶rXynAGN16L和rXynAHJ3的酶活高约20%。纯化的突变酶S45F07、野生酶rXynAGN16L和rXynAHJ3的最适温度分别为80℃、50℃和75℃;在90℃时,突变酶S45F07的酶活约为65%,野生酶rXynAHJ3的酶活约为15%,而野生酶rXynAGN16L无酶活。在3.0–25.0%(w/v)的NaCl中,突变体S45F07的酶活比野生酶rXynAHJ3和rXynAGN16L的酶活高10–25%。在20.0–30.0%(w/v)的KCl中,突变体S45F07的酶活比野生酶rXynAHJ3的酶活高12-19%,比野生酶rXynAGN16L的酶活高19–46%。本发明的突变内切木聚糖酶S45F07可应用于食品、饲料、造纸行业,特别是高盐环境生物技术领域。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1:在大肠杆菌中表达的重组内切木聚糖酶rXynAGN16L、rXynAHJ3及其突变体S45F07的SDS-PAGE分析,其中,CK:蛋白质Marker;
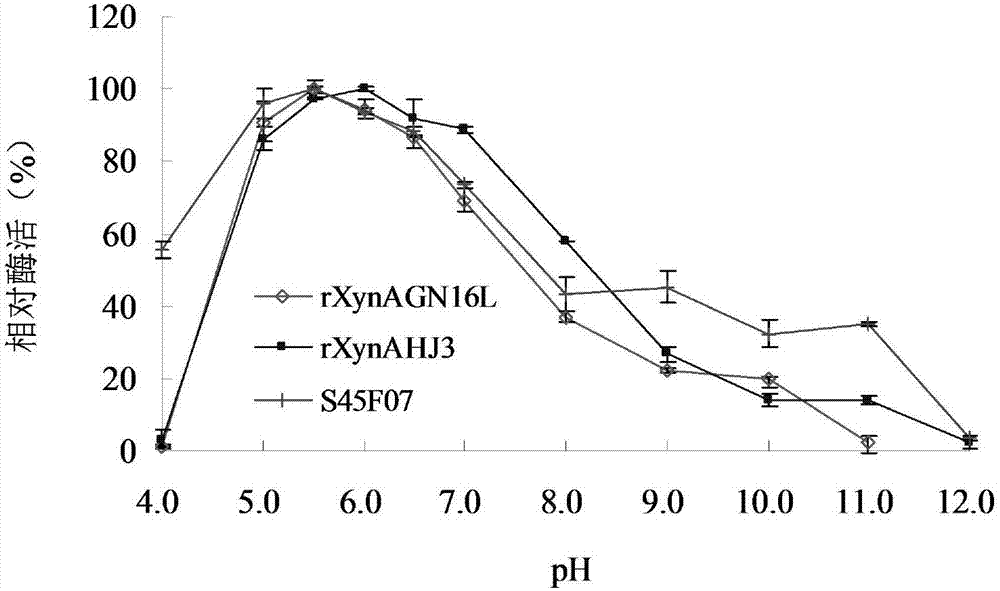
图2:重组内切木聚糖酶rXynAGN16L、rXynAHJ3及其突变体S45F07的pH活性;
图3:重组内切木聚糖酶rXynAGN16L、rXynAHJ3及其突变体S45F07的pH稳定性;
图4:重组内切木聚糖酶rXynAGN16L、rXynAHJ3及其突变体S45F07的热活性;
图5:重组内切木聚糖酶rXynAGN16L、rXynAHJ3及其突变体S45F07的热稳定性;
图6:重组内切木聚糖酶rXynAGN16L、rXynAHJ3及其突变体S45F07在NaCl中的活性;
图7:重组内切木聚糖酶rXynAGN16L、rXynAHJ3及其突变体S45F07在KCl中的活性。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
试验材料和试剂
1、菌株及载体:大肠杆菌Escherichia coli BL21-Gold(DE3)和表达载体pEasy-E2购自北京全式金生物技术有限公司;节杆菌(Arthrobacter sp.)和列舍瓦里尔菌(Lechevalieria sp.)由云南师范大学提供。
2、酶类及其它生化试剂:DNA聚合酶及dNTP购自北京全式金生物技术有限公司,山毛榉木聚糖购自Sigma公司,易错PCR试剂盒购自北京天恩泽基因科技有限公司,细菌基因组提取试剂盒购自GENE STAR公司,PopCultureTM细胞裂解液购自德国默克集团有限公司,其它都为国产试剂(均可从普通生化试剂公司购买得到)。
3、培养基:
LB培养基:Peptone 10g,Yeast extract 5g,NaCl 10g,加蒸馏水至1000mL,pH自然(约为7)。固体培养基在此基础上加2.0%(w/v)琼脂。
说明:以下实施例中未作具体说明的分子生物学实验方法,均参照《分子克隆实验指南》(第三版)J.萨姆布鲁克一书中所列的具体方法进行,或者按照试剂盒和产品说明书进行。
实施例1:突变文库的构建
①按照GENE STAR公司细菌基因组提取试剂盒说明书提取节杆菌(Arthrobacter sp.)和列舍瓦里尔菌(Lechevalieria sp.)基因组。
②根据GenBank记录的节杆菌(Arthrobactersp.)内切木聚糖酶核苷酸序列JQ863105(SEQ ID No.3),设计引物5'GTGCAGCCGGAGGAAAAACG 3'和5'GATGAAGGCAGGATCCGGGGT 3',以节杆菌(Arthrobactersp.)基因组为模板进行PCR扩增,获得内切木聚糖酶基因xynAGN16L;另根据GenBank记录的列舍瓦里尔菌(Lechevalieria sp.)内切木聚糖酶核苷酸序列JF745868(SEQ ID No.4),设计引物5'GTCTCGGCCCCGCCGGACGT 3'和5'GGCTCGCTTCGCCAGCGTGG 3',以列舍瓦里尔菌(Lechevalieria sp.)基因组为模板进行PCR扩增,获得内切木聚糖酶基因xynAHJ3。
③PCR反应参数为:94℃变性5min;然后94℃变性30sec,55℃退火30sec,72℃延伸1min 30sec,30个循环后72℃保温10min。
④以上述PCR产物为模板,利用易错PCR试剂盒,按照试剂盒说明书进行基因突变。
⑤用超声打断仪Biorupter对易错PCR产物进行超声随机打断,打断产物经2%琼脂糖凝胶电泳后切胶纯化。
⑥纯化后的小片段DNA自身互为引物和模板进行DNA Family shuffling PCR,PCR反应参数为:96℃变性1min 30sec;然后94℃变性30sec,依次65℃退火90sec、62℃退火90sec、59℃退火90sec、56℃退火90sec、53℃退火90sec、50℃退火90sec、47℃退火90sec、44℃退火90sec、41℃退火90sec,72℃延伸1min 30sec,35个循环后72℃保温7min。
⑧以纯化的DNA Family shuffling PCR产物为模板,用内切木聚糖酶基因xynAHJ3和xynAGN16L扩增引物和反应条件进行序列全长扩增,扩增产物含突变序列和未突变序列。
⑨将序列全长扩增产物和表达载体pEasy-E2相连接,并将连接产物转化大肠杆菌BL21-Gold(DE3),经过夜培养,从转化平板中挑取单菌落于含有150μL液体LB培养液(含100μg mL-1Amp)的96孔细胞培养板中,于37℃,快速振荡培养约16h后,每孔加入40%(w/w)的甘油50μL,混匀后于-70℃保存。
实施例2:突变体的筛选
1)从保存突变文库的96孔细胞培养板中取2μL菌液,接种于含200μL/孔液体LB培养液(含100μg mL-1Amp)的96深孔板中,于37℃,200rpm振荡培养至OD600>1.0(约20h),加入含2mM IPTG和100μg mL-1Amp的200μL液体LB培养液,于20℃,160rpm过夜诱导。
2)诱导结束后加入40μL/孔的PopCultureTM细胞裂解液,在25℃下,震荡裂解细胞30min。
3)取50μL含1.0%(w/v)山毛榉木聚糖的McIlvaine缓冲液(pH=7.0)及50μL细胞裂解产物,在96深孔板中于70℃恒温箱中反应2h。反应结束后加入150μL DNS试剂终止反应,于140℃恒温箱中保温20min以上并冷却至室温,使用酶标仪读取OD540nm的值,以只含有pEASY-E2空载体的E.coli BL21-Gold(DE3)菌株裂解液反应组作为对照。
4)取有内切木聚糖酶活性的突变体细胞裂解产物10μL,另取90μL含0.5%(w/v)山毛榉木聚糖的McIlvaine缓冲液(pH=7.0),加入10%(w/v)和25%(w/v)的NaCl,在96深孔板中于70℃恒温箱中反应10min。
5)反应结束后加入150μL DNS试剂终止反应,于140℃恒温箱中保温20min以上并冷却至室温,使用酶标仪读取OD540nm的值,以不含NaCl的对应突变体裂解液反应组作为对照。
6)比较突变体与野生重组酶rXynAGN16L和rXynAHJ3的酶活大小,获得在10%(w/v)和25%(w/v)NaCl中酶活提高的1个突变体,编号为S45F07,该突变体氨基酸序列如SEQ ID NO.1所示,其是两个野生酶的杂合体,该突变体核苷酸序列如SEQ ID NO.2所示。
实施例3:突变体S45F07及野生酶rXynAGN16L和rXynAHJ3的酶制备
将含突变体S45F07、野生酶rXynAGN16L和rXynAHJ3的重组菌株以0.1%的接种量分别接种于LB(含100μg mL-1Amp)培养液中,37℃快速振荡16h。
然后将此活化的菌液以1%接种量接种到新鲜的LB(含100μg mL-1Amp)培养液中,快速振荡培养约2–3h(OD600达到0.6-1.0)后,加入终浓度0.1mM的IPTG进行诱导,于20℃继续振荡培养约20h。12000rpm离心5min,收集菌体。用适量的pH=7.0Tris–HCl缓冲液悬浮菌体后,于低温水浴下超声波破碎菌体。以上胞内浓缩的粗酶液经13,000rpm离心10min后,吸取上清并用Nickel-NTA Agarose和0–500mM的咪唑分别亲和和纯化目的蛋白。
SDS-PAGE结果(图1)表明,突变酶S45F07、野生酶rXynAGN16L和rXynAHJ3都获得了纯化,产物为单一条带。
实施例4:突变体S45F07及野生酶rXynAGN16L和rXynAHJ3的纯化酶的性质测定
1)突变体S45F07及野生酶rXynAGN16L和rXynAHJ3的纯化酶的活性分析
活性测定方法采用3,5-二硝基水杨酸(DNS)法:将底物溶于缓冲液中,使其终浓度为0.5%(w/v);反应体系含100μL适量酶液,900μL底物;底物在反应温度下预热5min后,加入酶液后再反应10min,然后加1.5mL DNS终止反应,沸水煮5min,冷却至室温后在540nm波长下测定OD值;1个酶活单位(U)定义为在给定的条件下每分钟分解底物产生1μmol还原糖(以木糖计)所需的酶量。
2)突变体S45F07及野生酶rXynAGN16L和rXynAHJ3的纯化酶的pH活性和pH稳定性测定:
酶的最适pH测定:将酶液置于37℃下和pH=4.0–12.0的缓冲液中进行酶促反应。酶的pH稳定性测定:将酶液置于pH=3.0–12.0的缓冲液中,在37℃下处理1h,然后在pH=7.0及37℃下进行酶促反应,以未处理的酶液作为对照。缓冲液为:McIlvaine buffer(pH=3.0–8.0)和0.1M glycine–NaOH(pH=9.0–12.0)。以山毛榉木聚糖为底物,反应10min,测定纯化的内切木聚糖酶的酶学性质。结果表明:突变酶S45F07、野生酶rXynAGN16L和rXynAHJ3的最适pH分别为5.5、5.5和6.0;在pH4.0条件下,突变酶S45F07具有55%的酶活,而rXynAGN16L和rXynAHJ3无酶活;在pH9.0–11.0条件下,突变酶S45F07的酶活比野生酶rXynAGN16L和rXynAHJ3的酶活高约20%(图2)。在pH4.0和5.0条件下,S45F07和rXynAHJ3稳定而rXynAGN16L不稳定(图3)。
3)突变体S45F07及野生酶rXynAGN16L和rXynAHJ3的纯化酶的热活性及热稳定性测定:
酶的热活性测定:在pH=7.0的缓冲液中,于0–90℃下进行酶促反应。酶的热稳定性测定:将同样酶量的酶液置于37℃处理0–60min后,在pH=7.0及37℃下进行酶促反应,以未处理的酶液作为对照。以山毛榉木聚糖为底物,反应10min,测定纯化的内切木聚糖酶的酶学性质。结果表明:S45F07、rXynAGN16L和rXynAHJ3的最适温度分别为80℃、50℃和75℃;在90℃时,突变酶S45F07的酶活约为65%,野生酶rXynAHJ3的酶活约为15%,而野生酶rXynAGN16L无酶活(图4)。S45F07和rXynAHJ3在50℃时稳定,而rXynAGN16L在50℃时极不稳定(图5)。
4)不同金属离子及化学试剂对突变体S45F07及野生酶rXynAGN16L和rXynAHJ3的纯化酶活力的影响:
在酶促反应体系中加入10.0mM的金属离子及化学试剂,研究其对酶活性的影响。在37℃及pH=7.0条件下,以山毛榉木聚糖为底物测定酶活性。结果(表1)表明:在10.0mM的ZnSO4和FeSO4中,突变体S45F07的酶活比野生酶rXynAHJ3和rXynAGN16L的酶活高约16%。
表1.金属离子及化学试剂对突变体S45F07及野生酶rXynAGN16L和rXynAHJ3活力的影响
5)突变体S45F07及野生酶rXynAGN16L和rXynAHJ3的纯化酶在NaCl和KCl中的活性:
酶在NaCl中的活性测定:在酶促反应体系中加入3.0–30.0%(w/v)NaCl,于pH7.0及37℃下进行酶促反应。以山毛榉木聚糖为底物,反应10min,测定纯化的内切木聚糖酶的酶学性质。结果表明:在3.0–25.0%(w/v)的NaCl中,突变体S45F07的酶活比野生酶rXynAHJ3和rXynAGN16L的酶活高10–25%(图6)。
酶在KCl中的活性测定:在酶促反应体系中加入3.0–30.0%(w/v)KCl,于pH7.0及37℃下进行酶促反应。以山毛榉木聚糖为底物,反应10min,测定纯化的内切木聚糖酶的酶学性质。结果表明:在20.0–30.0%(w/v)的KCl中,突变体S45F07的酶活比野生酶rXynAHJ3的酶活高12-19%,比野生酶rXynAGN16L的酶活高19–46%(图7)。
本技术领域技术人员可以理解,除非另外定义,这里使用的所有术语(包括技术术语和科学术语)具有与本发明所属领域中的普通技术人员的一般理解相同的意义。还应该理解的是,诸如通用字典中定义的那些术语应该被理解为具有与现有技术的上下文中的意义一致的意义,并且除非像这里一样定义,不会用理想化或过于正式的含义来解释。
最后所应说明的是:以上实施例仅用以说明而非限制本发明的技术方案,尽管参照上述实施例对本发明进行了详细说明,本领域的普通技术人员应该理解:依然可以对本发明进行修改或者等同替换,而不脱离本发明的精神和范围的任何修改或局部替换,其均应涵盖在本发明的权利要求范围当中。
SEQUENCE LISTING
<110> 云南师范大学
<120> 一种pH、温度和盐适应性改良的内切木聚糖酶突变体及其应用
<160> 4
<170> PatentIn version 3.5
<210> 1
<211> 344
<212> PRT
<213> 杂合序列
<400> 1
Val Ser Ala Pro Pro Asp Val Ser Gly His Lys Gln Thr Leu Arg Ser
1 5 10 15
Ala Ala Pro Lys Gly Phe His Ile Gly Thr Ala Val Ala Gly Gly Gly
20 25 30
His His Glu Asn Gln Pro Tyr Pro Asp Pro Phe Thr Ser Asp Ser Glu
35 40 45
Tyr Arg Lys Val Leu Ala Ala Glu Phe Asn Ser Val Ser Pro Glu Asn
50 55 60
Gln Met Lys Trp Glu Tyr Ile His Pro Glu Arg Gly Arg Tyr Asn Phe
65 70 75 80
Gly Met Ala Asp Ala Ile Val Arg Phe Ala Lys Gln Asn Arg Gln Val
85 90 95
Val Arg Gly His Thr Leu Met Trp His Ser Gln Asn Pro Glu Trp Leu
100 105 110
Glu Gln Gly Asp Phe Thr Ala Ala Glu Leu Arg Glu Ile Leu Arg Glu
115 120 125
His Ile Met Thr Val Val Gly Arg Tyr Lys Gly Lys Ile Gln Gln Trp
130 135 140
Asp Val Ala Asn Glu Ile Phe Asn Asp Asp Gly Thr Leu Arg Ala Thr
145 150 155 160
Glu Asn Ile Trp Ile Arg Glu Leu Gly Pro Gly Ile Val Ala Asp Ala
165 170 175
Phe Arg Trp Ala His Gln Ala Asp Pro Lys Ala Lys Leu Phe Phe Asn
180 185 190
Asp Tyr Asn Val Glu Ser Val Asn Ala Lys Ser Asp Ala Tyr Tyr Ala
195 200 205
Leu Ile Lys Glu Leu Arg Ala Ala Gly Val Pro Val His Gly Phe Ser
210 215 220
Ala Gln Ala His Leu Ser Leu Asp Tyr Gly Phe Pro Asp Asp Leu Glu
225 230 235 240
Arg Asn Leu Lys Arg Phe Ala Asp Leu Arg Leu Glu Thr Ala Ile Thr
245 250 255
Glu Leu Asp Val Arg Met Thr Leu Pro Ala Ser Gly Val Pro Thr Ala
260 265 270
Ala Gln Leu Gln Gln Gln Ala Asp Tyr Tyr Gln Arg Thr Leu Ala Ala
275 280 285
Cys Leu Lys Val Arg Thr Cys Lys Ser Phe Thr Ile Trp Gly Phe Thr
290 295 300
Asp Lys Tyr Ser Trp Val Pro Val Phe Phe Gln Gly Gln Gly Ala Ala
305 310 315 320
Thr Val Met Trp Asn Asp Phe Gly Arg Lys Gln Ala Tyr Tyr Ala Leu
325 330 335
Arg Ser Thr Leu Ala Lys Arg Ala
340
<210> 2
<211> 1032
<212> DNA
<213> 杂合序列
<400> 2
gtctcggccc cgccggacgt gagcggccac aaacagacgt tgcgctcggc agcgcccaag 60
ggtttccaca tcggcacggc cgtcgcgggc ggcggccacc acgagaacca gccgtacccg 120
gaccccttca cctcggacag cgagtaccgg aaggtgctgg ccgcggagtt caactcggtc 180
tcgcccgaga accagatgaa gtgggagtac atccacccgg agcgcggccg gtacaacttc 240
ggcatggccg acgccatcgt ccggttcgcc aagcagaacc ggcaggtggt ccgcgggcac 300
accctgatgt ggcacagcca gaacccggag tggctggagc agggcgactt caccgcggcc 360
gaactgcgcg agatcctgcg cgagcacatc atgaccgtgg tcggccggta caagggcaaa 420
atccagcagt gggacgtcgc caacgaaatc ttcaatgatg acggaaccct gcgcgccacc 480
gagaacattt ggatccgtga actcggtccg ggcatcgtgg cggacgcgtt ccgctgggcg 540
caccaggccg accccaaggc gaagctgttc ttcaacgact acaacgtcga aagcgtcaac 600
gcgaagagcg acgcgtacta cgcgctgatc aaggagctgc gcgccgcggg tgtgcccgtg 660
cacggcttct ccgcccaggc gcacctcagc ctggactacg gcttcccgga cgacctggag 720
cgcaacctga agcggttcgc cgacctccgg ctggagaccg cgatcaccga gctcgacgtg 780
cggatgaccc tgcccgcgag cggcgtgccg acggcggccc agctgcagca gcaggccgac 840
tactaccagc gcacgctcgc ggcctgcctg aaggtcagga cctgcaagtc gttcaccatc 900
tggggcttca ccgacaagta ctcgtgggtg ccggtcttct tccaggggca gggtgcggcc 960
acggtgatgt ggaacgactt cggtcgcaag caggcgtact acgcgctgcg gtccacgctg 1020
gcgaagcgag cc 1032
<210> 3
<211> 3639
<212> DNA
<213> 节杆菌(Arthrobacter sp.)
<400> 3
atgaaggttc cgcgtttatt aaccgctctg gctgtaacct cggcgctgct gctgccggcg 60
gttccggcgc ttgccgtgca gccggaggaa aaacgtcctc cgggccagtc caaacaggac 120
acgctgcgcc gtgcagcccc caaagacttc aagattggtt ccgccgttgc gggcggaggc 180
catcatgagg cccaggacta ccccgatcct tttacgttcg ataaggaata ccgccggcaa 240
ctggccgccg agttcaattc ggtgtcaccg gagaaccagt cgaagtggga attcatccac 300
ccggaaaagg atgtctaccg cttcacggaa atggacgcca ttgtccgctc cgcccaaaaa 360
aacaagcagg tggtgcgcgg ccacaccctc ttttggcaca gccagaatcc tcagtggctg 420
gagcagggaa acttctccaa agaagaactg cgcggaatcc tcaaagacca cgtccagact 480
gtagtgggca ggtacgccgg caaaatccag cagtgggacg tcgccaacga aatcttcaat 540
gatgacggaa ccctgcgcgc caccgagaac atttggcttc gtgaactggg cccggacatc 600
attgccgacg ttttccgctg ggcgcacgag gccgacccca aggccaagct gttcttcaat 660
gatttcggcg ttgaggacat taatgccaag agtgatgcct acctcgaact catcccccgg 720
cttcaggcac agggcgtgca ggttgacggg tttgccatcc agggccatct gagcacccgc 780
tacggtttcc cttcagggct gcaggccaac ctgcagcgct ttgacgacct ggggctggaa 840
actgccatta cggaaataga cgtccgcatg gatattgcag ccggcacgga gccgacggcc 900
gagcagcttg agcagcaggc ggactactac cagcgcgccc ttgaggcctg cctgtccgtt 960
gcagactgca attcgttcac catttggggc ttcacggaca agtactcgtg ggttccggtc 1020
ttctttgccg gcgagggcga ggcgacagtc atggaggaag acttcacgcg caagcctgcc 1080
tactttgccc tgcgggaaac actgaagcgt ccggtgccga agcccgacga cggcggcccg 1140
tcccagccaa ccccggatcc tgccttcatc cccggcggcg ccgccaaccc gacagcgacg 1200
ccgatcgcag catcccgcgg caccggcaac tccgtggcgc tcaccttcga tgacgggccc 1260
gagcccggcg aaaccacagc tgtcctcgat ttcctcaagg acaagggcat cactgccacc 1320
ttctgcgtca tcggagcgaa catccaggcc cccggcggag ccgagctggt gaagcgcatg 1380
gtcgaggagg gccacacgct gtgcaaccac ggcaccacgt atgcggacat gggttcgtgg 1440
acccaggaac agattaaggc cgacctggtg gaaaacctcc gcatcatccg tgaagccgcc 1500
ggcacgcctg atctgcaggt cccctatttc cgggcaccga acggaagctg gggagtcacg 1560
ggcgaagtag ccgcagcgct tggtatgcag ccgctgggcc tgggcaatgt catctttgac 1620
tgggacggca atgacctcag cgaagccacc ctcacggcaa acctccgtgc cgcgttcacc 1680
cccggcgcgg tggtgctggc gcacgtcggc ggcggtgacc ggaccaacac agtgaaggca 1740
gttacgacgg tcgtgaccga aaagctcgcc caggggtgga cgttcgccct tccgcagggc 1800
ggtgccccgg aggaaccttc cggcggtgtg ccctcggact tcgagaccgg aaccgacggc 1860
tggaccgcgc gcggggactc agtggcggtc aacctcagct ccgacgcccg caccggatcc 1920
ggaagcctgc tggtcacgaa ccggacccag gactggcacg gtgccgcact cgacgtcacg 1980
ggcgccctac cggtcggctc ggccgtaaag atgtccgtct gggccaagct cgcccccggg 2040
cagcagccgg cggcactgaa aatgtccgtt cagcgggaca acggcggcgg gagtgcctat 2100
gaaggcgttg ccggagccgg ggcttcggtc accgccgacg gctggaccga acttgccggg 2160
acttacaccc tcggcgcagc agcggacaaa gcccaggtgt acatcgaagg tgctgtcggc 2220
gtggggttcc tgctcgatga cttcagcctc gccgcatatg ttgagcctcc ccttcaggag 2280
gacatacccg ggttgaaaga cgtccttggc ctgcagggca tcgagcacgt gggagcagca 2340
atcgacgcac gcgagacagc gggcaccgca gcgaacctcc tgcggaaaca cttcaatgcc 2400
ttcactcccg agaacgccgg caggcccgag agcgtgcagc cggtggaggg tcagttcacc 2460
cttacccagc tggaccagct gctggacttc gcagccgcca acaatgtcaa ggtgtacgga 2520
catgtgctgg tctggcattc ccagacccct gagtggttct tcaaggacgg gacccgggac 2580
ctgaccggca accggtccga ccgggcgctg ctgagggcac gcatggaggc acatatcaag 2640
ggcatcgcag atcacatcaa tgcccgctac ccggaggggg acagccccat ttgggcctgg 2700
gacgttgtca acgagaccat tgcggacggt gacacggcca acccgcacga catgcgggac 2760
agccgctggt tccaggtcct cggtgaacgt tttgtcgatg atgccttccg tctcgcggac 2820
aagtacttcc cggaggcaaa gctcttcatc aacgactaca acaccgagat gccccagaaa 2880
cgggccgact atctcgagct gattcgtgcc ctggaagccc ggggcgtacc catcgacggc 2940
gtgggccacc aggcgcacgt cgacgtggca cgtccggtgc agtggctcga ggactcgatc 3000
aaggccgttg agaaggtcaa tcctgacctg atgcaggcga tcactgagct cgacgtgaac 3060
gcgtccaccg agaatcaggg cgcggacgtg gacggtgccc cggtggatcc gtaccagccg 3120
gcattcggga acgacgggga cgccgccgcg gaagtcggat actactaccg cgacttgttc 3180
gccatgctgc gcaagcacag ttcggctatt gattcggtga ccgtctgggg catcagcaac 3240
gcccgcagct ggctgcggac ctggccgatg gcccggccct gggagcagcc gcttccattc 3300
gacgatgatc tgcaggctgc accggcctac tggggaatcg tggatcccgc gaaactgccg 3360
gcccggcctg ccgacgtgct ggcaccccgc atcgccgatc agccggacgt ggtggccttt 3420
tcaaagcgcg ccggacgggt gaaggtggct tacccgttgc cctcggcgat cgacaccctc 3480
gacggcaaag tgccggtgga ctgttctccg cgccgcggca gcacctttgc cgtggggacc 3540
actgcggtca cctgcacggc cacggatgcc gccggcaaca cgaggaccag cagcttcgac 3600
gtggtggtga agaagcaccg gcaccacgga aggcactga 3639
<210> 4
<211> 1104
<212> DNA
<213> 列舍瓦里尔菌(Lechevalieria sp.)
<400> 4
atgaggtcgg ctcgtctggt catcgctttg ttcgctgccg tggcgttgtc ggcgccaccg 60
gcttcggcgg tctcggcccc gccggacgtg agcggccaca aacagacgtt gcgctcggca 120
gcgcccaagg gtttccacat cggcacggcc gtcgcgggcg gcggccacca cgagaaccag 180
ccgtacccgg accccttcac ctcggacagc gagtaccgga aggtgctggc cgcggagttc 240
aactcggtct cgcccgagaa ccagatgaag tgggagtaca tccacccgga gcgcggccgg 300
tacaacttcg gcatggccga cgccatcgtc cggttcgcca agcagaaccg gcaggtggtc 360
cgcgggcaca ccctgatgtg gcacagccag aacccggagt ggctggagca gggcgacttc 420
accgcggccg aactgcgcga gatcctgcgc gagcacatca tgaccgtggt cggccggtac 480
aagggcaagg tccagcagtg ggacgtggcc aacgagatct tcaccgacgc cggcgctctg 540
cggaccacgg agaacatctg gatccgtgaa ctcggtccgg gcatcgtggc ggacgcgttc 600
cgctgggcgc accaggccga ccccaaggcg aagctgttct tcaacgacta caacgtcgaa 660
agcgtcaacg cgaagagcga cgcgtactac gcgctgatca aggagctgcg cgccgcgggt 720
gtgcccgtgc acggcttctc cgcccaggcg cacctcagcc tggactacgg cttcccggac 780
gacctggagc gcaacctgaa gcggttcgcc gacctccggc tggagaccgc gatcaccgag 840
ctcgacgtgc ggatgaccct gcccgcgagc ggcgtgccga cggcggccca gctgcagcag 900
caggccgact actaccagcg cacgctcgcg gcctgcctga aggtcaggac ctgcaagtcg 960
ttcaccatct ggggcttcac cgacaagtac tcgtgggtgc cggtcttctt ccaggggcag 1020
ggtgcggcca cggtgatgtg gaacgacttc ggtcgcaagc aggcgtacta cgcgctgcgg 1080
tccacgctgg cgaagcgagc ctga 1104
- 还没有人留言评论。精彩留言会获得点赞!