基于酶切组合基因分型测序技术的褐菖鲉基因组SNP分子标记方法与流程
本发明涉及褐菖鲉基因组分子标记开发技术领域,尤其是涉及基于酶切组合基因分型测序技术的褐菖鲉基因组snp分子标记方法。
技术背景
分子标记是能区分生物种间、种内群体间及群体内个体间遗传差异的dna序列片段。利用分子标记能够开展物种鉴别、遗传多样性评估、遗传分化监测和个体分类聚群等科学研究。目前使用较为广泛的分子标记有线粒体dna(mitochondrialdna)、微卫星标记(simplesequencerepeat,ssr)、单核苷酸多态位点(singlenucleotidepolymorphism,snp)等。随着高通量测序技术和生物信息学分析方法的发展,分子遗传学研究要求分子标记需具有成本低、遗传信息量大、基因分型效率高等特点。因而snp标记越来越成为重要的分子标记。相对于线粒体dna和ssr标记而言,snp标记具有数据量大、覆盖度高、分型效率高等特点。snp广泛分布在基因组编码区和非编码区,借助高通量测序技术能够在低成本的情况下开发得到数以万计的标记位点。随着测序技术的发展和测序成本的降低,snp标记已经成为群体遗传、基因分型和物种鉴定的主要标记方法。基因分型测序技术(genotyping-by-sequencing,gbs)是一种低成本的简化基因组测序技术(reduced-representationsequencing),能够在基因组范围内开发大量snp标记用于基因分型。由于不依赖参考基因组、测序流程简单、测序成本低等特点,gbs技术已广泛应用于非模式生物分子标记开发、遗传图谱绘制、关联分析和群体基因组学等研究。褐菖鲉是一种广泛分布于西北太平洋海域的岩礁性卵胎生鱼类,具有重要的经济价值和生态价值。目前褐菖鲉的分子标记仅局限于线粒体dna和微卫星标记,分子标记的局限性严重影响了褐菖鲉群体遗传多样性和遗传结构的监测,导致划分渔业管理单元和制定管理策略出现偏差,最终影响褐菖鲉渔业资源的合理开发利用。基于上述原因,有必要提供一种新的开发褐菖鲉基因组snp位点的分析方法,在降低成本的前提下开发大量分子标记,为后续基因分型和遗传学研究提供便利。
技术实现要素:
本发明的目的在于提供一种技术稳定、重复性高、能够在降低成本的情况下大量开发基因组分子标记的基于酶切组合基因分型测序技术的褐菖鲉基因组snp分子标记方法。
本发明针对背景技术中提到的问题,采取的技术方案为:
基于酶切组合基因分型测序技术的褐菖鲉基因组snp分子标记方法,包括以下步骤:
褐菖鲉基因组dna提取:利用苯酚/氯仿/异戊醇方法提取褐菖鲉基因组dna;
adapter接头设计:合成一对通用接头序列、样品标签序列和一对pcr引物序列;
褐菖鲉基因组dna用msei和nlaiii进行酶切;
连接adapter;
混合样品和pcr扩增;
回收序列片段和高通量测序;
数据分析开发snp位点。本发明分子标记方法可以在褐菖鲉基因组水平上开发snp分子标记,snp覆盖基因组编码区和非编码区,基因分型测序技术是一种低成本的简化基因组测序技术,能够在基因组范围内开发大量snp标记用于基因分型,而使用msei和nlaiii的基因分型测序技术大大降低了分子标记开发的成本,开发得到的snp位点可应用于褐菖鲉的种质资源评估、遗传多样性和遗传结构监测等研究,该snp分子标记方法技术稳定,重复性高。
作为优选,褐菖鲉基因组dna提取步骤为:
(a)将肌肉样品去除酒精后置于1.5ml离心管中,加入600-700μl消化缓冲液和18-22μl蛋白酶k溶液,震荡混匀后置于50-60℃烘箱内消化4-6h,每30min摇晃一次;
(b)将步骤a消化好的溶液冷却至室温后,于离心管中加入等体积的平衡酚,摇晃混匀至乳状溶液,在10000-15000rpm条件下离心10-15min,然后重复上述步骤2-3次;
(c)将步骤b得离心上清液体中加入等体积-20℃预冷酒精,振动至dna沉淀下来,然后在2-5℃、10000-15000rpm条件下离心5-10min后,离心沉淀用250-350μl浓度为70-80%酒精溶液漂洗两次,然后在10000-15000rpm条件下离心5-10min后弃上清液,晾干后在离心管中加入100μl去离子水,溶解dna,最后将提取的dna溶液置于4℃冰箱保存,待用。本发明利用苯酚/氯仿/异戊醇方法提取褐菖鲉基因组dna,对褐菖鲉基因组dna的损伤比较小,容易获得完整性相对更好的褐菖鲉基因组dna,同时可在加异丙醇后冰浴10-20分钟后离心,效果更佳。
作为优选,adapter接头的设计与msei和nlaiii的酶切末端和后续illuminahiseq测序平台要求的引物序列一致,所述adapter接头序列由illumina测序平台接头序列、样品标签序列两部分组成。
进一步优选,样品标签序列设计原则如下:
(a)样品标签序列为6碱基序列且不能含有连续两个相同的碱基;
(b)样品标签序列间必须有至少两个差异碱基;
(c)样品标签不能与酶切位点相同或相近。
作为优选,褐菖鲉基因组dna用msei和nlaiii进行酶切的具体步骤为:取0.1-1μg褐菖鲉基因组dna,用msei和nlaiii酶切组合进行酶切,以得到适合的标记密度。采用msei和nlaiii酶切组合酶的酶切位点分布广泛且均匀,对褐菖鲉基因组dna进行酶切,产生的酶切标签具有较高的多样性,可满足筛查snp以进行遗传作图的要求。
作为优选,连接adapter的具体步骤为:adapter接头连接与褐菖鲉基因组dna用msei和nlaiii进行酶切在同一试管中进行,接头通过连接酶与dna酶切末端相连接。
作为优选,混合样品和pcr扩增的具体步骤为:将连接adapter接头的不同样品dna片段等量混合,加入pcr引物序列和dna合成酶进行pcr扩增。
作为优选,回收序列片段和高通量测序的具体步骤为:电泳回收步骤(4)中扩增的产物,利用illuminahiseq2000测序平台进行测序。高通量测序开发方法简单易行、清晰准确、假阳性低并且试验成本低,扩增区内已知和未知snp都能检测;灵敏度和精确度达到近100%。
作为优选,数据分析开发snp位点的具体步骤为:测序得到的原始数据按照样品标签序列进行分选聚类,按样品个体进行数据质控并删除adapter接头序列,利用短序列比对软件bwa将序列比对到参考基因组上,使用samtools软件提取snp位点,使用vcftools软件筛选用于群体遗传结构分析的snp位点。
与现有技术相比,本发明的优点在于:本发明分子标记方法可以在褐菖鲉基因组水平上开发snp分子标记,snp覆盖基因组编码区和非编码区,基因分型测序技术是一种低成本的简化基因组测序技术,能够在基因组范围内开发大量snp标记用于基因分型,而使用msei和nlaiii的基因分型测序技术大大降低了分子标记开发的成本,开发得到的snp位点可应用于褐菖鲉的种质资源评估、遗传多样性和遗传结构监测等研究,该snp分子标记方法技术稳定,重复性高。
附图说明
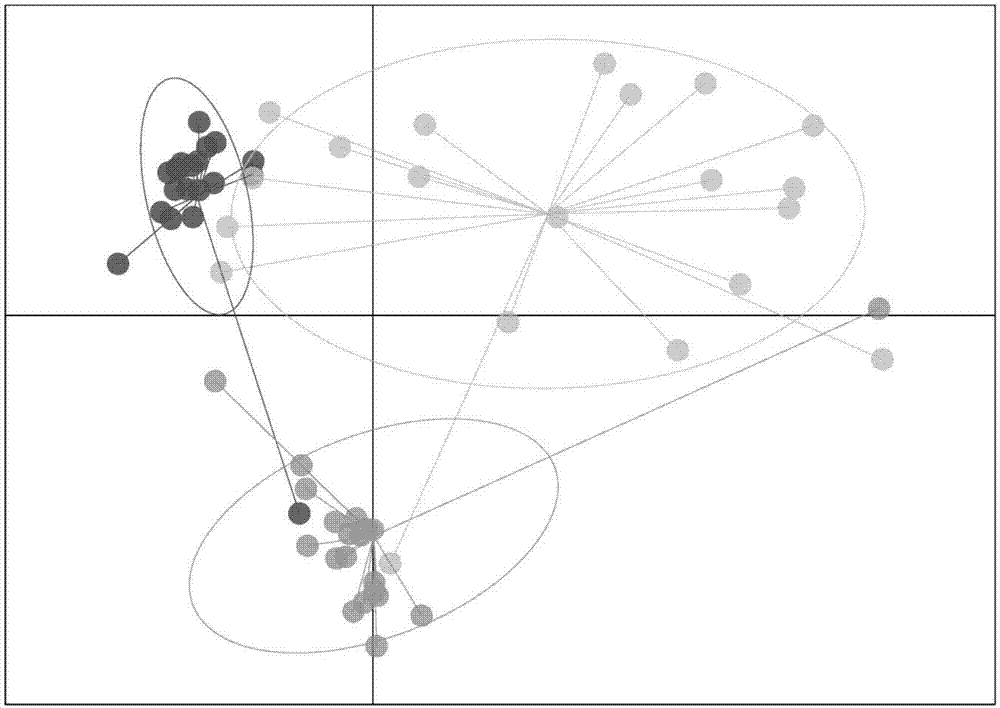
图1:本发明实例例2中利用开发的褐菖鲉基因组snp位点进行群体遗传结构分析。
具体实施方式
下面通过实施例对本发明方案作进一步说明:
实施例1:
基于酶切组合基因分型测序技术的褐菖鲉基因组snp分子标记方法,包括以下步骤:
褐菖鲉基因组dna提取:利用苯酚/氯仿/异戊醇方法提取褐菖鲉基因组dna;
adapter接头设计:合成一对通用接头序列、样品标签序列和一对pcr引物序列;
褐菖鲉基因组dna用msei和nlaiii进行酶切;
连接adapter;
混合样品和pcr扩增;
回收序列片段和高通量测序;
数据分析开发snp位点。本发明分子标记方法可以在褐菖鲉基因组水平上开发snp分子标记,snp覆盖基因组编码区和非编码区,基因分型测序技术是一种低成本的简化基因组测序技术,能够在基因组范围内开发大量snp标记用于基因分型,而使用msei和nlaiii的基因分型测序技术大大降低了分子标记开发的成本,开发得到的snp位点可应用于褐菖鲉的种质资源评估、遗传多样性和遗传结构监测等研究,该snp分子标记方法技术稳定,重复性高。
上述褐菖鲉基因组dna提取步骤为:
(a)将肌肉样品去除酒精后置于1.5ml离心管中,加入600μl消化缓冲液和18μl蛋白酶k溶液,震荡混匀后置于50℃烘箱内消化4h,每30min摇晃一次;
(b)将步骤a消化好的溶液冷却至室温后,于离心管中加入等体积的平衡酚,摇晃混匀至乳状溶液,在10000rpm条件下离心10min,然后重复上述步骤2次;
(c)将步骤b得离心上清液体中加入等体积-20℃预冷酒精,振动至dna沉淀下来,然后在2℃、10000rpm条件下离心5min后,离心沉淀用250μl浓度为70%酒精溶液漂洗两次,然后在10000rpm条件下离心5min后弃上清液,晾干后在离心管中加入100μl去离子水,溶解dna,最后将提取的dna溶液置于4℃冰箱保存,待用。本发明利用苯酚/氯仿/异戊醇方法提取褐菖鲉基因组dna,对褐菖鲉基因组dna的损伤比较小,容易获得完整性相对更好的褐菖鲉基因组dna,同时可在加异丙醇后冰浴10分钟后离心,效果更佳。
上述adapter接头的设计与msei和nlaiii的酶切末端和后续illuminahiseq测序平台要求的引物序列一致,所述adapter接头序列由illumina测序平台接头序列、样品标签序列两部分组成。
其中,样品标签序列设计原则如下:
(a)样品标签序列为6碱基序列且不能含有连续两个相同的碱基;
(b)样品标签序列间必须有至少两个差异碱基;
(c)样品标签不能与酶切位点相同或相近。
上述褐菖鲉基因组dna用msei和nlaiii进行酶切的具体步骤为:取0.1μg褐菖鲉基因组dna,用msei和nlaiii酶切组合进行酶切,以得到适合的标记密度。采用msei和nlaiii酶切组合酶的酶切位点分布广泛且均匀,对褐菖鲉基因组dna进行酶切,产生的酶切标签具有较高的多样性,可满足筛查snp以进行遗传作图的要求。
上述连接adapter的具体步骤为:adapter接头连接与褐菖鲉基因组dna用msei和nlaiii进行酶切在同一试管中进行,接头通过连接酶与dna酶切末端相连接。
上述混合样品和pcr扩增的具体步骤为:将连接adapter接头的不同样品dna片段等量混合,加入pcr引物序列和dna合成酶进行pcr扩增。
上述回收序列片段和高通量测序的具体步骤为:电泳回收步骤(4)中扩增的产物,利用illuminahiseq2000测序平台进行测序。高通量测序开发方法简单易行、清晰准确、假阳性低并且试验成本低,扩增区内已知和未知snp都能检测;灵敏度和精确度达到近100%。
上述数据分析开发snp位点的具体步骤为:测序得到的原始数据按照样品标签序列进行分选聚类,按样品个体进行数据质控并删除adapter接头序列,利用短序列比对软件bwa将序列比对到参考基因组上,使用samtools软件提取snp位点,使用vcftools软件筛选用于群体遗传结构分析的snp位点。
实施例2:
基于酶切组合基因分型测序技术的褐菖鲉基因组snp分子标记方法,包括以下步骤:
(1)实验材料
本实验样品于2015年冬季采自乳山、舟山和防城港近海海域,每个群体20尾褐菖鲉样品,共计60尾样品,样品采集时间相近。所有样品取肌肉样品,置于95%酒精溶液中-20℃冷冻保存;
(2)褐菖鲉基因组dna提取
(a)将肌肉样品去除酒精后置于1.5ml离心管中,加入650μl消化缓冲液和20μl蛋白酶k溶液,震荡混匀后置于56℃烘箱内消化5h,每30min摇晃一次;
(b)待溶液冷却至室温后,于离心管中加入650μl平衡酚,摇晃混匀至乳状溶液,在10000rpm条件下离心10min,用1000μl移液枪吸出上层液体;
(c)加入600μl(相同体积)苯酚:氯仿:异戊醇(25:24:1)按照步骤(b)再次提取两次,将上层液体转移到新的1.5ml离心管中,加入1ml-20℃预冷酒精,轻轻振动离心管,可以观察到dna沉淀下来,在10000rpm条件下离心10min后,用300μl75%酒精溶液漂洗两次,在10000rpm条件下离心10min后弃上清液,晾干后在离心管中加入100μl去离子水,溶解dna,提取的dna溶液置于4℃冰箱保存待用;
(3)adapter接头序列
合成一对通用接头序列,60对样品标签序列和一对pcr引物序列;
(4)褐菖鲉基因组dna用msei和nlaiii进行酶切
使用msei和nlaiii酶切组合对褐菖鲉基因组dna进行酶切,反应体系为20μl,包括15μl去离子水,2μl10×cutsmartbuffer,0.5μlmsei,0.5μlnlaiii,200ng样品dna,反应条件为37℃2h。之后将反应体系置于65℃下30min变性内切酶,抑制其活性;
(5)酶切产物与adapter接头连接
酶切结束后连接adapter接头,接头连接与酶切反应在同一试管中进行。加入nebbuffer4缓冲液和atp、正反向接头各100μmol,反应结束后再加入nebbuffer4缓冲液、atp和200u的t4连接酶。整个反应体系保持在22℃下持续2h,之后反应体系置于65℃下20min;
(6)样品混合和pcr扩增
连接产物通过qbit定量后,按照样品等量混入pcr试管中。经过18次重复循环(98℃30s,65℃30s,72℃30s)后得到待测序文库;
(7)高通量测序和基因组snp开发
按照illumina建库和测序流程对步骤(6)中获得的序列在illuminahiseq2000测序平台上进行高通量测序,测序使用双末端150bp测序模式,共得到测序数据约70g。
按照样品标签对测序数据进行聚类,分别得到60个褐菖鲉样品的原始测序数据。对原始数据进行质量控制并去除adapter接头序列后,使用bwa软件的bwa-mem算法比对到褐菖鲉参考基因组序列上,得到bam文件。使用samtools软件对bam文件进行分析,提取snp标记,得到sam文件。使用vcftools软件筛选snp标记,得到用于褐菖鲉群体遗传学分析的snp标记。
本实施例按照上述流程共得到7376个snp标记。使用上述获得的基因组snp标记对3个褐菖鲉群体进行群体遗传结构分析,结果发现相同群体内个体均呈现明显的聚类关系,显示群体内个体间遗传关系较近,而不同群体间呈现一定的遗传分化,如附图1所示。
实施例3:
为了提高褐菖鲉基因组dna提取效果,进一步优化步骤为:
(2)褐菖鲉基因组dna提取
(a)将肌肉样品去除酒精后置于1.5ml离心管中,加入650μl消化缓冲液和20μl蛋白酶k溶液,震荡混匀后置于56℃烘箱内消化5h,每30min摇晃一次;
(b)待溶液冷却至室温后,于离心管中加入650μl平衡酚,其中平衡酚中含有0.43%十六烷基三甲基溴化铵和0.054%正戊醇,摇晃混匀至乳状溶液,在10000rpm条件下离心10min,用1000μl移液枪吸出上层液体,摇晃混匀至乳状溶液,十六烷基三甲基溴化铵和正戊醇的特殊存在能够和平衡酚发挥增益作用,一方面能够溶解未被破坏的细胞膜,同时结合多糖和蛋白质等物质,提高苯酚溶解蛋白质的量,使得在较少平衡酚的情况下也能有效地变性蛋白质,进而有效抑制dnase的降解作用,同时能够降低dna在异戊醇中的溶解量,使得本实施例3改进的dna提取方法dna得率和纯度的高,简便快速,成本低廉,且能够在后续过程中清除干净,对最终得到的dna溶液无影响;
(c)加入600μl(相同体积)苯酚:氯仿:异戊醇(25:24:1)按照步骤(b)再次提取两次,将上层液体转移到新的1.5ml离心管中,加入1ml-20℃预冷酒精,轻轻振动离心管,可以观察到dna沉淀下来,在10000rpm条件下离心10min后,用300μl75%酒精溶液漂洗两次,在10000rpm条件下离心10min后弃上清液,晾干后在离心管中加入100μl去离子水,溶解dna,提取的dna溶液置于4℃冰箱保存待用。
本发明操作步骤中的常规操作为本领域技术人员所熟知,在此不进行赘述。
以上所述的实施例对本发明的技术方案进行了详细说明,应理解的是以上所述仅为本发明的具体实施例,并不用于限制本发明,凡在本发明的原则范围内所做的任何修改、补充或类似方式替代等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!