SNP组合及在制备检测药物反应相关基因的产品中的应用的制作方法
本发明涉及基因检测技术领域。尤其是涉及基于飞行质谱平台的药物反应相关基因的检测技术及用途。
背景技术
药物体内代谢、转运及药物作用靶点基因的遗传变异及其表达水平的变化可通过影响药物的体内浓度和敏感性,导致药物反应性个体差异。近年来随着人类基因组学的发展,药物基因组学领域得到了迅猛发展,越来越多的药物基因组生物标记物及其检测方法相继涌现。药物基因组学已成为指导临床个体化用药、评估严重药物不良反应发生风险、指导新药研发和评价新药的重要工具,部分上市的新药仅限于特定基因型的适应症患者。美国fda已批准在140余种药物的药品标签中增加药物基因组信息,涉及的药物基因组生物标记物42个。此外,部分行业指南也将部分非fda批准的生物标记物及其特性(如mgmt基因甲基化)的检测列入疾病的治疗指南。药物反应相关基因及其表达产物的分子检测是实施个体化药物治疗的前提。
snp分型的方法多种多样,
质谱是带电原子、分子或分子碎片按分子量(质荷比)的大小顺序排列的图谱。质谱仪可通过对物质的分子量(质荷比)进行检测,达到区分、鉴别物质的目的。snp位点两个等位基因之间存在分子量差异,通过分型实验将差异放大,然后以质谱仪进行检测即可达到snp分型的目的。带电物质无电场的真空管(driftregion)中的飞行速度与分子量负相关,分子量越小,飞行速度越快,到达检测器(detector)的时间越早,反之亦然。
技术实现要素:
本发明的技术目的首先是提供一种用于飞行质谱检测的snp位点组合,其利用飞行质谱平台,能够准确检测39个与药物在人体内作用、转运与代谢相关的基因,通过基因检测技术,能够准确判断某些药物可能存在的不良反应、能够准确的预测出每个个体对于某种药物的代谢能力以及药效。本发明共涉及39个变异位点。所述snp位点包括:rs2108622、rs137852343、rs4986893、rs2242480、rs137852342、rs2279343、rs72554664、rs20417、rs1799931、rs3745274、rs2032582、rs1801253、rs1801394、rs1799930、rs137852327、rs137852340、rs1876828、rs1128503、rs4244285、rs1045642、rs1801131、rs1057910、rs1065852、rs137852314、rs137852330、rs137852341、rs1799971、rs1801133、rs267606617、rs267606619、rs28371759、rs3918290、rs4149056、rs4713916、rs671、rs72554665、rs762551、rs776746、rs9923231。这些变异位点是发明人经过多年研究选择得到的组合形式,基因组合的选择依据是,依据国家现行的法律法规、中国及who组织发布的药品清单、中国及who出版的药品处方集和中国发布的药物基因组检测指南等信息,进行重重筛选后得到的,可以基本覆盖常见临床使用药物相关的基因,同时可以快速准确的得到位点突变检测结果。本发明的技术基于基质辅助激光解吸电离飞行时间质谱仪(maldi-tof),是一种基因snp位点的分型技术。该技术的主要原理是首先通过多重pcr技术,扩增出所要检测snp附近的一个片段,然后,通过纯化技术,取出pcr产物中的杂质,再通过单碱基扩增引物,特异性的扩增出snp所在的位点。再将扩增产物点在芯片上,进行结晶。最后将芯片上的产物,通过飞行时间质谱仪,测定其分子量,来最终判断snp位点的类型。
进一步地,本发明提供用于扩增所述snp位点的引物对以及延伸引物。所述引物基于飞行质谱技术的原理和特点,经过多次设计、试验和优化,最终得到优选的扩增引物对,以及延伸引物。所述引物对的上游引物的序列如seqidno.1-39所示,所述引物对的下游引物的序列如seqidno.40-78所示,所述延伸引物的序列如seqidno.79-117所示。用于后续pcr扩增反应以及单碱基延伸反应。所述引物的设计是此技术开发中的一个难点,因为多重pcr是若干条引物组合,在一个反应孔中进行反应,因此不同引物间会发生串扰,影响检测的准确性。本发明的引物由富有经验的发明人开发设计,利用算法精确的引物设计软件,同时经过了多次试验验证,最终才得到了如上所述的最优选的引物组合,可以达到不串扰、同时可以快速高效地扩增出所述位点的效果。
基于上述优化的引物设计和位点选择,本发明提供一种用于检测药物反应相关基因的方法,包括步骤:
1、合成引物;2、提取待检样品的核酸;3、多重pcr;4、纯化pcr产物,sap酶消化反应;5、单碱基延伸反应;6、单碱基延伸反应产物纯化,树脂脱盐反应;7、产物芯片点样,结晶;8、maldi-tof-ms(质谱)检测;9、数据分析。
上述技术,能够快速、准确的检测出导致药物不良反应相关基因、药物代谢基因、和药物靶点基因,并对相应的结果进行遗传解读,这样,能够通过基因检测技术,避免一些药物不良反应的产生,并能够准确的预测出药物的代谢情况。对于药物的临床使用有着重大意义。
再进一步地,本发明优选提供一种用于检测药物反应相关基因的试剂盒,包括上述引物对以及延伸引物,所述引物对的上游引物的序列如seqidno.1-39所示,所述引物对的下游引物的序列如seqidno.40-78所示,所述延伸引物的序列如seqidno.79-117所示。
上述试剂盒,包括引物储备液、pcr反应试剂、sap酶消化试剂以及单碱基延伸反应试剂。
引物储备液为每种引物用te缓冲液或ddh2o配置而成:pcr引物储备液,终浓度为100μmol/l;和单碱基引物储备液,终浓度为500μmol/l。
pcr反应试剂包括:1.8μl的dddh2o;0.5μl的10倍缓冲液;0.4μl的mg2+;0.1μl的dntp(脱氧核糖核苷酸);0.2μl的hotstar(hotstarttaqdna聚合酶)。
sap酶消化试剂包括:1.53μl的dddh2o;0.17μl的sap缓冲液;0.3μl的sap酶(虾碱酶)。
单碱基延伸反应试剂包括:0.619μl的dddh2o、0.2μl的10倍iplex缓冲液(iplextmgold:agena公司的试剂)、0.2μl的terminatormix、0.041μl的单碱基延伸酶。
采用上述试剂盒,可以取得快速、简单、准确的基因型检测,同时具有其他其它snp检测方法所没有的优势:
(1)质谱仪检测的是分子最本质的特征之一分子量,不涉及荧光标记、凝胶电泳等,就能检测一个碱基的差异,准确性高,机器本身出错的概率非常低;
(2)质谱仪的灵敏度非常高,检测窗口内,任何pmol级别的物质都能被检测出来;
(3)通量高:几秒就能检测完一个反应孔;
(4)操作简单,仪器要求简单,除质谱仪外,都是常规pcr仪器;
(5)灵活:每天可以完成几个反应至上万个反应;
(6)便宜:引物不带荧光标记,普通长度(3条引物总长80bp左右),此外在一个反应孔内能完成4个或更多的反应;
(7)一管式操作:即反应体系在实验过程中始终在一个试管内反应,没有多次转移,这样就减少被污染的概率。
附图说明
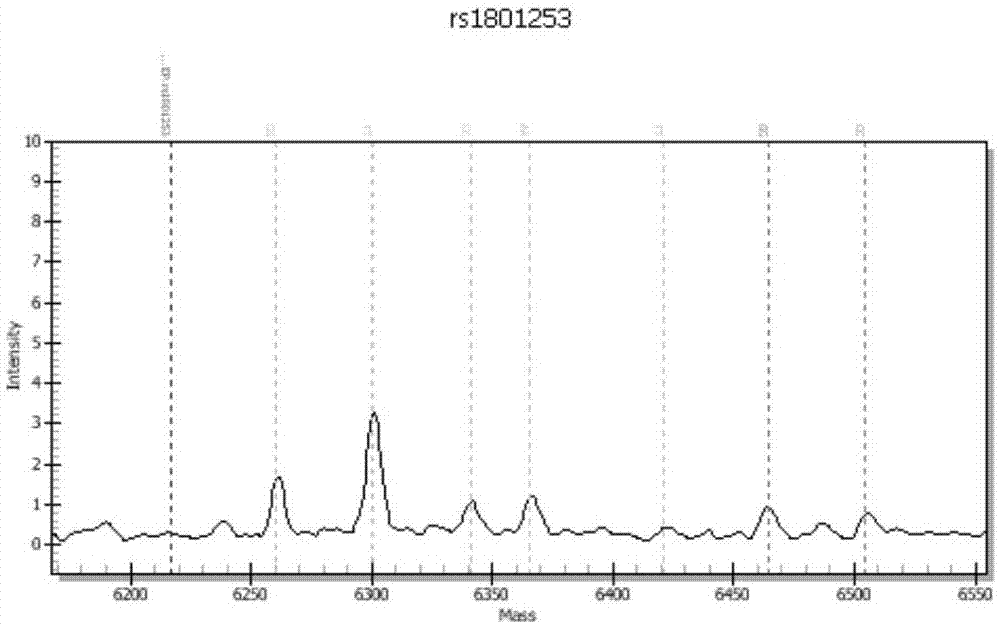
图1-8是实施例3样本的质谱峰图举例,其中:图1:rs1801253;图2:rs4149056;图3:rs671;图4:rs1065852;图5:rs1799930;图6:rs1799931;图7:rs1801131;图8:rs1801133。
具体实施方式
以下实施例进一步说明本发明的内容,但不应理解为对本发明的限制。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本发明的范围。
若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。所用的试剂均为市场上可购得的常规试剂,其中massarray试剂为:agena公司iplexgoldgenotypingreagentset货号10148-2。
实施例1:
一种用于检测药物反应相关基因的试剂盒,包括如下表所示的引物对以及延伸引物,所述引物对的上游引物的序列如seqidno.1-39所示,所述引物对的下游引物的序列如seqidno.40-78所示,所述延伸引物的序列如seqidno.79-117所示。引物制备成引物储备液,然后还包括pcr反应试剂、sap酶消化试剂以及单碱基延伸反应试剂。
实施例2:
一种用于检测药物反应相关基因的方法,采用实施例1所述的试剂盒来操作,包括步骤:
1、dna提取:使用商品化试剂盒,提取待测样本中的dna。使用nanodrop2000仪器进行od值检测,1.25%琼脂糖凝胶电泳检测,dna质检合格,转移至96孔板,-20度储存备用。
2、合成引物和质检
按照实施例1的引物序列合成引物,通过基质辅助激光解吸电离飞行时间质谱仪(maldi-tof)进行质检,检测实际分子量与理论分子量是否一致,引物纯度是否达到实验要求。
3、引物稀释
3.1pcr引物储备液制备:配置pcr引物储备液,终浓度为100p(μmol/l);
3.2单碱基延伸引物储备液制备:配置单碱基引物储备液,终浓度为500p(μmol/l);
4、pcr反应
4.1总反应体系如下:
dddh2o:1.8μl;10*buffer:0.5μl;mg2+:0.4μl;dntp:0.1μl;hotstar:0.2μl;fprimer/rprimer:1μl;待检测dna样品:1μl(20ng-50ng);total:5μl。
4.2pcr扩增反应:按照下述的程序对pcr仪进行设置:预变性95℃2min;95℃30s、56℃30s、72℃60s,共45个循环;72℃5min;25℃∞。
5、sap酶消化反应
5.1按照下述顺序配制sap酶mix:dddh2o:1.53*460μl;sapbuffer:0.17*460μl;sapenzyme:0.3*460μl;total:2*460μl。
5.2按照下述程序,在pcr仪中进行sap酶消化:37℃40min;85℃5min;25℃∞。
6、单碱基延伸反应
6.1按照下述顺序配制单碱基延伸反应mix:三蒸水:0.619*460μl;10*iplexbuffer:0.2*460μl;terminatormix:0.2*460μl;引物:0.94*460μl;单碱基延伸酶:0.041*460μl;total:2*460μl。
6.2按照下述程序,在pcr仪中进行单碱基延伸反应:预变性94℃30s;94℃5s、52℃5s、80℃5s(5个内部循环),共40个外部循环;72℃3min;25℃∞。
7、树脂纯化
7.1将反应产物的384孔板中加入16μl三蒸水,在离心机中离心2000转离心3min;加入树脂,在反转摇匀仪上做树脂纯化反应35min,脱盐;反应完成后在离心机中离心2000转离心3min;
7.2将脱盐处理后的样品点在样品靶上,自然结晶。
8、质谱检测及数据分析
8.1maldi-tof-ms(基质辅助激光解析电离飞行时间质谱)反应;
8.2typer4.0软件检测质谱峰,并根据质谱峰图判读各样本目标位点基因型。最终得到每个样本的39个snp位点的基因型。
实施例3:样本检测
按照实施例2的操作步骤,随机抽取一份dna样本进行检测,可以得到所述snp位点的基因型,选取其中几个snp位点的检测峰图为例,详见图1-8,可以看出,本发明的方法可以快速准确的检测出所述位点的基因型。检测准确率可达99.9%,准确性高、灵活性强、通量大、检测周期短。然后根据检测结果,可进行遗传因素解读,本发明涉及的39个snp位点的药物敏感解读结果列表如下:
根据上述解读,就可以判断样本来源的临床用药风险,从而提高药物合理使用率和药物治疗率。
sequencelisting
<110>北京青航基因科技有限公司
<120>snp组合及在制备检测药物反应相关基因的产品中的应用
<130>p1810189
<160>117
<170>patentinversion3.5
<210>1
<211>30
<212>dna
<213>人工序列
<400>1
acgttggatggtgttttcggaacccatcac30
<210>2
<211>30
<212>dna
<213>人工序列
<400>2
acgttggatgtggaaccgcatcatcgtgga30
<210>3
<211>30
<212>dna
<213>人工序列
<400>3
acgttggatgaacatcaggattgtaagcac30
<210>4
<211>30
<212>dna
<213>人工序列
<400>4
acgttggatgtgctaaggtttcacctcctc30
<210>5
<211>29
<212>dna
<213>人工序列
<400>5
acgttggatgcaaagggtacctggacgac29
<210>6
<211>30
<212>dna
<213>人工序列
<400>6
acgttggatgctttttccatgtggagcagg30
<210>7
<211>30
<212>dna
<213>人工序列
<400>7
acgttggatgtcaatctggtgcagcagtgg30
<210>8
<211>30
<212>dna
<213>人工序列
<400>8
acgttggatgacagggtaactgcttaggac30
<210>9
<211>30
<212>dna
<213>人工序列
<400>9
acgttggatgaaatctcgtgcccaaacctg30
<210>10
<211>30
<212>dna
<213>人工序列
<400>10
acgttggatgttcagtctgtgtccttgacc30
<210>11
<211>30
<212>dna
<213>人工序列
<400>11
acgttggatgagtaagcagtagggagtaac30
<210>12
<211>29
<212>dna
<213>人工序列
<400>12
acgttggatgggtctccgtgggtcgcgtg29
<210>13
<211>30
<212>dna
<213>人工序列
<400>13
acgttggatggcagaaaatccatgtaccac30
<210>14
<211>30
<212>dna
<213>人工序列
<400>14
acgttggatgacgtctgcaggtatgtattc30
<210>15
<211>30
<212>dna
<213>人工序列
<400>15
acgttggatgcattctctcccttggctttc30
<210>16
<211>30
<212>dna
<213>人工序列
<400>16
acgttggatgggcacttcctggcttttaag30
<210>17
<211>30
<212>dna
<213>人工序列
<400>17
acgttggatgcgattgtctagagccttctc30
<210>18
<211>30
<212>dna
<213>人工序列
<400>18
acgttggatgcacagccactgtttccaacc30
<210>19
<211>30
<212>dna
<213>人工序列
<400>19
acgttggatgcactttccataaaagcaagg30
<210>20
<211>30
<212>dna
<213>人工序列
<400>20
acgttggatgaaggcatgtatgttggcctc30
<210>21
<211>30
<212>dna
<213>人工序列
<400>21
acgttggatgccgagaggtaaagaacgaag30
<210>22
<211>30
<212>dna
<213>人工序列
<400>22
acgttggatgctacacagatgctgtggtgc30
<210>23
<211>30
<212>dna
<213>人工序列
<400>23
acgttggatgtgatagtggccatcttcctg30
<210>24
<211>30
<212>dna
<213>人工序列
<400>24
acgttggatgagggcttctccacgatgatg30
<210>25
<211>30
<212>dna
<213>人工序列
<400>25
acgttggatgaccacatctcctccctgttc30
<210>26
<211>30
<212>dna
<213>人工序列
<400>26
acgttggatgtcaacagccacatgaatgcc30
<210>27
<211>30
<212>dna
<213>人工序列
<400>27
acgttggatggggtcaacttgtcccactta30
<210>28
<211>30
<212>dna
<213>人工序列
<400>28
acgttggatgcttgaaggagaaggtgtctg30
<210>29
<211>30
<212>dna
<213>人工序列
<400>29
acgttggatgaccctcctcaagtatacttc30
<210>30
<211>30
<212>dna
<213>人工序列
<400>30
acgttggatgccatgttacgacttgtctcc30
<210>31
<211>30
<212>dna
<213>人工序列
<400>31
acgttggatgtgatgccctacattgatctg30
<210>32
<211>31
<212>dna
<213>人工序列
<400>32
acgttggatgcaccaacttatgccaattctc31
<210>33
<211>30
<212>dna
<213>人工序列
<400>33
acgttggatgaatctgggtcatacatgtgg30
<210>34
<211>30
<212>dna
<213>人工序列
<400>34
acgttggatgcctaacgagatagtgaggag30
<210>35
<211>30
<212>dna
<213>人工序列
<400>35
acgttggatgttggtggctacaagatgtcg30
<210>36
<211>30
<212>dna
<213>人工序列
<400>36
acgttggatgtcccaagccataccatgtcc30
<210>37
<211>30
<212>dna
<213>人工序列
<400>37
acgttggatgcagctggataccagaaagac30
<210>38
<211>30
<212>dna
<213>人工序列
<400>38
acgttggatggtaatgtggtccaaacaggg30
<210>39
<211>30
<212>dna
<213>人工序列
<400>39
acgttggatggctaggattataggcgtgag30
<210>40
<211>30
<212>dna
<213>人工序列
<400>40
acgttggatggccttggaatggacaaaaac30
<210>41
<211>30
<212>dna
<213>人工序列
<400>41
acgttggatgacagggaggagatgtggttg30
<210>42
<211>30
<212>dna
<213>人工序列
<400>42
acgttggatggactgtaagtggtttctcag30
<210>43
<211>30
<212>dna
<213>人工序列
<400>43
acgttggatggcaggaggaaattgatgcag30
<210>44
<211>30
<212>dna
<213>人工序列
<400>44
acgttggatgtaccatcccacctctcattc30
<210>45
<211>30
<212>dna
<213>人工序列
<400>45
acgttggatggtggagaagcaccgtgaaac30
<210>46
<211>30
<212>dna
<213>人工序列
<400>46
acgttggatgtcccaagccatactatgtcc30
<210>47
<211>30
<212>dna
<213>人工序列
<400>47
acgttggatgactgttctccgtaccttcac30
<210>48
<211>30
<212>dna
<213>人工序列
<400>48
acgttggatggggtgatacatacacaaggg30
<210>49
<211>30
<212>dna
<213>人工序列
<400>49
acgttggatgatggagcagatgatgttggc30
<210>50
<211>30
<212>dna
<213>人工序列
<400>50
acgttggatggtctggacaagcactgaaag30
<210>51
<211>30
<212>dna
<213>人工序列
<400>51
acgttggatgtcaaccccatcatctactgc30
<210>52
<211>30
<212>dna
<213>人工序列
<400>52
acgttggatgctatatgctacacagcaggg30
<210>53
<211>30
<212>dna
<213>人工序列
<400>53
acgttggatgctatatgctacacagcaggg30
<210>54
<211>30
<212>dna
<213>人工序列
<400>54
acgttggatgattgttggcctgcacctctg30
<210>55
<211>29
<212>dna
<213>人工序列
<400>55
acgttggatggatgccttccatcagtcgg29
<210>56
<211>30
<212>dna
<213>人工序列
<400>56
acgttggatgagcagcatacccctagggac30
<210>57
<211>30
<212>dna
<213>人工序列
<400>57
acgttggatgtttctcactcgtcctggtag30
<210>58
<211>30
<212>dna
<213>人工序列
<400>58
acgttggatggcaataattttcccactatc30
<210>59
<211>30
<212>dna
<213>人工序列
<400>59
acgttggatgttgcctatggagacaacagc30
<210>60
<211>30
<212>dna
<213>人工序列
<400>60
acgttggatgtctacctgaagagcaagtcc30
<210>61
<211>30
<212>dna
<213>人工序列
<400>61
acgttggatgtgtcacaggtcactgcatgg30
<210>62
<211>30
<212>dna
<213>人工序列
<400>62
acgttggatgagtccacatgcagcaggttg30
<210>63
<211>30
<212>dna
<213>人工序列
<400>63
acgttggatgatgatgcagctctgatcctc30
<210>64
<211>30
<212>dna
<213>人工序列
<400>64
acgttggatgtgaggttctgcaccatctcc30
<210>65
<211>30
<212>dna
<213>人工序列
<400>65
acgttggatgtgaatgttcttggtgacggc30
<210>66
<211>30
<212>dna
<213>人工序列
<400>66
acgttggatgtgatggccgtgatcatggag30
<210>67
<211>30
<212>dna
<213>人工序列
<400>67
acgttggatgcttcacaaagcggaagaatg30
<210>68
<211>30
<212>dna
<213>人工序列
<400>68
acgttggatgcactttccagtacacttacc30
<210>69
<211>30
<212>dna
<213>人工序列
<400>69
acgttggatgagttgaacagggccctgaag30
<210>70
<211>30
<212>dna
<213>人工序列
<400>70
acgttggatgagataattgattgggccacg30
<210>71
<211>30
<212>dna
<213>人工序列
<400>71
acgttggatgtcactgaactaaaggctgac30
<210>72
<211>30
<212>dna
<213>人工序列
<400>72
acgttggatgccaatggtactatgggagtc30
<210>73
<211>30
<212>dna
<213>人工序列
<400>73
acgttggatgtatctggcaaccctaacctc30
<210>74
<211>30
<212>dna
<213>人工序列
<400>74
acgttggatgaggtcccacactcacagttt30
<210>75
<211>30
<212>dna
<213>人工序列
<400>75
acgttggatgtcaatctggtgcagcagtgg30
<210>76
<211>30
<212>dna
<213>人工序列
<400>76
acgttggatgtctgtgatgctcaaagggtg30
<210>77
<211>30
<212>dna
<213>人工序列
<400>77
acgttggatgacccagcttaacgaatgctc30
<210>78
<211>30
<212>dna
<213>人工序列
<400>78
acgttggatgtctgggaagtcaagcaagag30
<210>79
<211>15
<212>dna
<213>人工序列
<400>79
catcacaacccagct15
<210>80
<211>15
<212>dna
<213>人工序列
<400>80
atcgtggagaagccc15
<210>81
<211>16
<212>dna
<213>人工序列
<400>81
ttgtaagcaccccctg16
<210>82
<211>17
<212>dna
<213>人工序列
<400>82
ccctccttctccatgta17
<210>83
<211>17
<212>dna
<213>人工序列
<400>83
cgtttgcagccgtcgtc17
<210>84
<211>17
<212>dna
<213>人工序列
<400>84
ccgtgtcgatgaggtcc17
<210>85
<211>17
<212>dna
<213>人工序列
<400>85
gcagtggggtgaaaata17
<210>86
<211>19
<212>dna
<213>人工序列
<400>86
aggagaatttacctttccc19
<210>87
<211>19
<212>dna
<213>人工序列
<400>87
gggcccaaacctggtgatg19
<210>88
<211>20
<212>dna
<213>人工序列
<400>88
ggaaccaccttcctcttcca20
<210>89
<211>20
<212>dna
<213>人工序列
<400>89
ggggtgactcaccttcccag20
<210>90
<211>20
<212>dna
<213>人工序列
<400>90
gggagcgcagcagagcagtc20
<210>91
<211>22
<212>dna
<213>人工序列
<400>91
cccttaccacagcttgctcaca22
<210>92
<211>22
<212>dna
<213>人工序列
<400>92
agactcaaaatcttcaattgtt22
<210>93
<211>22
<212>dna
<213>人工序列
<400>93
aattgctttctctcaggtcaag22
<210>94
<211>23
<212>dna
<213>人工序列
<400>94
atgcacccatgatgatgaatatg23
<210>95
<211>24
<212>dna
<213>人工序列
<400>95
aaggtctgcctctctccctccctg24
<210>96
<211>24
<212>dna
<213>人工序列
<400>96
ccccctctgcaccttcaggttcag24
<210>97
<211>24
<212>dna
<213>人工序列
<400>97
tttcagtaatttgttatgggttcc24
<210>98
<211>25
<212>dna
<213>人工序列
<400>98
ttggggcctcctttgctgccctcac25
<210>99
<211>26
<212>dna
<213>人工序列
<400>99
aaataacgaagacttcaaagacactt26
<210>100
<211>22
<212>dna
<213>人工序列
<400>100
gttgcacgaggtccagagatac22
<210>101
<211>17
<212>dna
<213>人工序列
<400>101
gctgggctgcacgctac17
<210>102
<211>21
<212>dna
<213>人工序列
<400>102
gaaagatgatgcggttccagc21
<210>103
<211>17
<212>dna
<213>人工序列
<400>103
gtgaggaccagatctac17
<210>104
<211>16
<212>dna
<213>人工序列
<400>104
tatgccctccacctgg16
<210>105
<211>21
<212>dna
<213>人工序列
<400>105
aaattgtcccacttagatggc21
<210>106
<211>15
<212>dna
<213>人工序列
<400>106
aggtgtctgcgggag15
<210>107
<211>25
<212>dna
<213>人工序列
<400>107
aacactacgcatttatatagaggag25
<210>108
<211>23
<212>dna
<213>人工序列
<400>108
gactttgaagtatacttgaggag23
<210>109
<211>15
<212>dna
<213>人工序列
<400>109
cagctctgtccgatc15
<210>110
<211>24
<212>dna
<213>人工序列
<400>110
gttttagatgttaaatcacactta24
<210>111
<211>23
<212>dna
<213>人工序列
<400>111
tgggtcatacatgtggatatatg23
<210>112
<211>18
<212>dna
<213>人工序列
<400>112
ggagttattggaccaaga18
<210>113
<211>20
<212>dna
<213>人工序列
<400>113
gagggctgcaggcatacact20
<210>114
<211>20
<212>dna
<213>人工序列
<400>114
gccactcagcgacgagctcc20
<210>115
<211>22
<212>dna
<213>人工序列
<400>115
tctcaatctaccatgcgtcctg22
<210>116
<211>19
<212>dna
<213>人工序列
<400>116
tccaaacagggaagagata19
<210>117
<211>18
<212>dna
<213>人工序列
<400>117
cccgtgagccaccgcacc18
- 还没有人留言评论。精彩留言会获得点赞!