一种获得非转基因耐储存鲜食枸杞的方法与流程
本发明属于生物技术育种领域,涉及一种获得非转基因耐储存鲜食枸杞的方法,特别涉及一种利用基因组编辑技术定点突变宁夏枸杞果胶裂解酶相关基因,获得枸杞耐储存育种材料的方法。
背景技术:
枸杞(lyciumbarbaruml.)是我国重要的药用植物资源。含枸杞多糖、甜菜碱、枸杞色素等,现代医学证明枸杞具有广泛的药用价值。此外枸杞耐干旱,适宜沙地生长,因此可作为水土保持的灌木,又由于其耐盐碱,成为盐碱地开树先锋。
枸杞鲜果的各种营养成分均比干果高,已经逐渐成为受消费者喜爱的时令果品。鲜食枸杞具备果粒大、皮薄肉厚、籽少味甘。但枸杞鲜果为浆果,水分含量高达80%,且富含糖分,果实采收后变质的速度快,极易霉烂,果实供应期及货架期很短,难以满足市场需求。皮薄肉厚虽改良了口感,却增加了贮藏及运输过程的困难。使得枸杞鲜果的销售成本很高,风险极大。
技术实现要素:
为了获得非转基因耐储存鲜食枸杞,本发明提供了如下技术方案:
本发明提供了一种培育耐储存鲜食枸杞的方法,包括如下步骤:
抑制目的枸杞中果胶裂解酶基因的表达,从而使枸杞耐储存;
或抑制目的枸杞中果胶裂解酶的含量或活性,从而使枸杞耐储存。
上述方法中,所述果胶裂解酶为如下(a1)或(a2)或(a3):
(a1)序列表的序列3所示的蛋白质;
(a2)将(a1)经过一个或几个氨基酸残基的取代和/或缺失和/或添加且具有相同功能的蛋白质;
(a3)与(a1)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性且具有相同功能的蛋白质。
上述方法中,所述果胶裂解酶基因为如下(b1)-(b4)中任一种:
(b1)编码区如序列表的序列1所示的dna分子;
(b2)编码区如序列表的序列2所示的dna分子;
(b3)在严格条件下与(b1)或(b2)限定的dna分子杂交且编码果胶裂解酶的dna分子;
(b4)与(b1)或(b2)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性且编码果胶裂解酶的dna分子。
上述方法中,所述抑制枸杞中果胶裂解酶基因的表达或抑制枸杞中果胶裂解酶的含量或活性均是通过crispr/cas9系统实现。
上述方法中,所述crispr/cas9系统包括sgrna和cas9蛋白;
或所述crispr/cas9系统包括表达sgrna和cas9蛋白的质粒;
所述sgrna的靶序列为如下(c1)或(c2):
(c1)序列表的序列7所示的核酸分子;
(c2)与(c1)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性的核酸分子。
或所述通过crispr/cas9系统实现为将所述sgrna和所述cas9蛋白导入目的枸杞中。
向所述枸杞的细胞或组织中导入所述遗传物质或所述非遗传物质的方法为基因枪法、农杆菌侵染法、peg诱导原生质体法或其他任何导入方法。
上述方法中,所述sgrna的核苷酸序列为序列表中序列4编码的rna或序列10;
所述sgrna为如下(d1)或(d2)或(d3):
(d1)序列表的序列4编码的rna;
(d2)序列表的序列10所示的核酸分子;
(d3)与(d1)或(d2)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性的核酸分子。
或所述cas9蛋白的氨基酸序列为序列11。
本发明另一个目的是提供一种制备耐储存鲜食枸杞的方法。
本发明提供的方法,包括如下步骤:在目的枸杞的细胞或组织中导入cas9蛋白和特异sgrna,得到耐储存的鲜食枸杞;
所述sgrna的靶序列为如下(c1)或(c2):
(c1)序列表的序列7所示的核酸分子;
(c2)与(c1)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性的核酸分子。
上述方法中,所述sgrna为如下(d1)或(d2)或(d3):
(d1)序列表的序列4编码的rna;
(d2)序列表的序列10所示的核酸分子;
(d3)与(d1)或(d2)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性的核酸分子。
本发明第3个目的是提供一种sgrna或表达其的质粒。
本发明提供的sgrna,其靶序列为如下(c1)或(c2):
(c1)序列表的序列7所示的核酸分子;
(c2)与(c1)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性的核酸分子。
本发明还提供了一种蛋白,为如下(a1)或(a2)或(a3):
(a1)序列表的序列3所示的蛋白质;
(a2)将(a1)经过一个或几个氨基酸残基的取代和/或缺失和/或添加且具有相同功能的蛋白质;
(a3)与(a1)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性且具有相同功能的蛋白质。
上述中,所述sgrna为如下(d1)或(d2)或(d3):
(d1)序列表的序列4编码的rna;
(d2)序列表的序列10所示的核酸分子;
(d3)与(d1)或(d2)具有99%以上、95%以上、90%以上、85%以上或者80%以上同一性的核酸分子。
所述细胞为任何能作为导入受体并能经过组织培养再生为完整植株的细胞;所述组织为任何能作为导入受体并能经过组织培养再生为完整植株的组织;具体的,所述细胞为原生质体细胞或悬浮细胞;所述组织为叶盘、茎段或子叶。
枸杞鲜果的营养价值显著高于干果,但货架期短是限制鲜果销售的主要原因。本方法相较于传统育种技术能够快速地获得耐储存的新品种。安全性上,该方法采用序列特异核酸酶的瞬时表达方法,可使人工核酸酶基因不整合到受体基因组,同时获得无转基因痕迹的定点敲除的遗传材料。定点敲除与传统转基因技术相比,极大地减少了由于随机插入和整合所带来的非预期效应产生的安全性风险。因此,从技术层面上该技术比传统的转基因技术具有较高的安全性。成本上,该方法极大地减少了需要筛选的群体样本数量,只需要2-3年的育种周期,比转基因8-10年的育种周期显著缩短。政策上,基于安全性上的考虑,该方法能够更快速也更容易地获得政策的审批。
附图说明
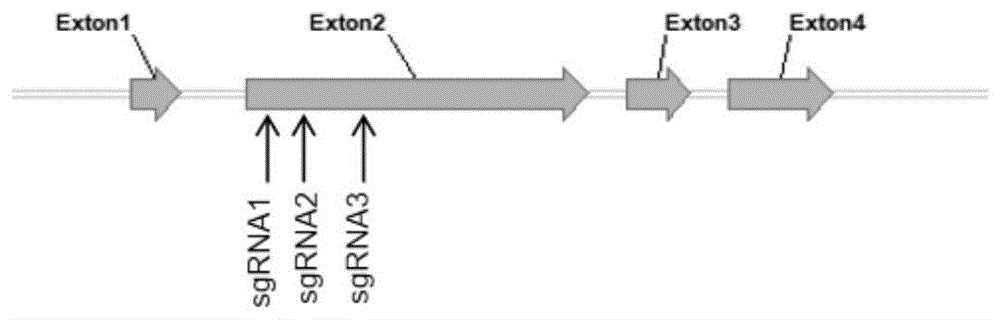
图1为枸杞lbpl基因待敲除靶位点的图。
图2为野生宁杞7号与pl突变体鲜果采摘后室温存放15天的对比效果图。
图3为phse401-lbpl1结构图。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1、枸杞lbpl基因的克隆及靶序列的确定
一、枸杞lbpl基因的克隆
以枸杞叶片总rna反转录得到的cdna为模板,采用rt-pcr方法扩增枸杞果胶裂解酶lbpl基因的全长cdna。基于茄科植物番茄pl基因的保守序列设计引物
f:atggg(g/c)cacttcctctgtttt
r:tt(t/c)agcaacgagaaccctttttac
以引物组合f/r进行扩增,rt-pcr结果经测序获得1212bp的片段(图2),为完整的开放读码框,编码403个氨基酸。通过blast比对,根据同源性将其代表的基因命名为lbpl。
lbpl基因的基因组dna的序列为序列1,cdna序列为序列2;该基因编码的蛋白lbpl的氨基酸序列为序列3。
二、枸杞lbpl基因待敲除靶位点的选择
lbpl基因座位号是solyc03g111690,位于枸杞第3条染色体,包含个4外显子,3个内含子,共编码403个氨基酸,构建敲除载体选择的靶位点位于第2外显子中(图1)。
靶位点序列及其sgrna的序列可以为如下3种:
sgrna-1的编码序列为:gggtatttatcgtgtggtaccgg(序列4),靶序列为5’-gggtatttatcgtgtggtaccgg-3’(序列7);
sgrna-2的编码序列为:cggttagctgactgtgcaattgg(序列5),靶序列为5’-cggttagctgactgtgcaattgg-3(序列8);
sgrna-3的编码序列为:actgtggattattttcgcgaggg(序列6),靶序列为5’-actgtggattattttcgcgaggg-3(序列9)。
分析sgrna-1的突变效率最高,故选择sgrna-1进行后续工作。
实施例2、lbpl基因的敲除
一、cas9蛋白的制备和体外转录的sgrna
1、cas9蛋白的制备
从中国科学院遗传与发育生物学研究所馈赠获得含有已经纯化好的cas9蛋白(氨基酸序列为序列11)。
2、靶位点sgrna的体外转录
sgrna-1的靶序列为target-lbpl1:5’-gggtatttatcgtgtggtaccgg-3’(序列7)
1)、重组质粒的构建
合成下述带有粘性末端(下划线部分)的单链引物:lbpl1f:5’-attggggtatttatcgtgtggtac-3’;lbpl1r:5’-aaacgtaccacacgataaataccc-3’,经过引物退火程序形成有粘性末端的双链dna;
再将有粘性末端的双链dna插入到phse401载体(记载在如下文献中:acrispr/cas9toolkitformultiplexgenomeeditinginplants.xinghl,dongl,wangzp,zhanghy,hancy,liub,wangxc,chenqj.bmcplantbiol.2014nov29;14(1):327)的两个bsai酶切位点之间,即得含有target-lbpl1的质粒。
质粒经测序验证阳性质粒,该阳性质粒具有序列4(sgrna对应的编码dna分子)所示的dna分子,表达序列4编码的sgrna,该阳性质粒命名为phse401-lbpl1(图3)。
2、含有lbpl的靶位点的sgrna的体外转录
合成引物:
t7-lbpl-sgrna-f:taatacgactcactatagggggtatttatcgtgtggtac,
grna-sc-r:aaaagcaccgactcggtgcca;
以phse401-lbpl1为模板进行pcr扩增,得到的pcr产物用pcr纯化试剂盒进行纯化(全式金ep101-02),pcr产物为含有t7启动子和lbpl靶点sgrna1的dna分子,利用全式金的t7体外转录试剂盒将其体外合成含有t7启动子和lbpl靶点sgrna1的rna(sgrna-lbpl1):ggguauuuaucgugugguacguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugcuuuuuuu(序列10),并在sgrna的3’末端加polya尾巴,以提高mrna的稳定性。
sgrna-lbpl1以溶液的形式存在,溶质为sgrna-lbpl1,溶剂为水,浓度为250ng/μl。
二、利用基因枪法转化纯化的cas9蛋白和体外转录的sgrna对枸杞lbpl基因进行定点编辑
将上述一得到的cas9蛋白和sgrna-lbpl1通过基因枪转化的方法导入宁夏枸杞宁杞7号中。以宁杞7号的叶盘为转化受体,转化后经过组织培养获得完整再生植株,进行分子检测,筛选出突变体,具体如下:
1、二氧化硅装载cas9蛋白和sgrna-lbpl1
选取孔径为10nm的二氧化硅au-msn为介质,将20mg的au-msn加入5mlph7.4的磷酸盐缓冲液(pbs)中进行超声处理,随后再加入7mg纯化好的cas9蛋白。将此混合体在22℃搅拌24小时,再用12000rpm离心,弃上清,将沉淀再用pbs缓冲液旋起,得到cas9蛋白-au-msn(10μg/μl)介质。将4μl的体外转录的sgrna-lbpl1(250ng/μl)加入到10μlcas9蛋白-au-msn(10μg/μl)介质中,再加入12.5μl2.5m的cacl2和5μl0.1m的亚精胺,再5000rpm离心15s,弃上清用,再用100%的酒精洗涤沉淀两次,再用5μl的100%酒精悬起mrna包裹的载有cas9蛋白的au-msn,形成sgrna-cas9-au-msn复合体。
2、基因枪转化枸杞叶盘
1)取宁杞7号的种子经3%naclo灭菌后放在ms培养基上发芽,而后每隔两周剪取2cm的茎段,扦插至1/2ms培养基上扩繁。
2)在诱导培养基(ms+1.0mg/lnaa+0.5mg/l6-ba)上覆盖两层中性滤纸,剪取枸杞无菌叶片,远轴端朝上,放置于诱导培养基上,预培养1天。
3)用bio-radpds-1000/he基因枪对叶片进行轰击,将5μl的sgrna-cas9-au-msn复合体装载在载样膜上;利用基因枪进行轰击,每次所述轰击的轰击距离为6cm,轰击压力为1100psi,轰击直径为2cm。
4)轰击后的叶片放置到25℃光照培养2天,然后切成9mm2的方块,分别接种在诱导培养基上,25℃光照培养2-4周,每两周继代一次。
5)将诱导出的愈伤组织继代至分化培养基(ms+0.1mg/lnaa+0.5mg/l6-ba)上,诱导成苗。
6)将2cm以上的小苗继代至生根培养基(ms+0.2mg/lnaa+0.01mg/l6-ba)上。14-28天后,获得t0代基因编辑后枸杞植株。
三、分子检测
将t0代基因编辑后枸杞植株幼苗提取基因组dna,以该dna为模板,进行pcr/re(polymerasechainreaction/restrictiondigestion)实验分析。同时设置野生型枸杞的dna作为对照。pcr/re分析方法参考了文献shan,qi.etal.rapidandefficientgenemodificationinriceandbrachypodiumusingtalens.molecularplant(2013),由于枸杞内源基因lbpl的靶标片段上存在限制性内切酶的识别序列(5’-ggtacc-3’),因而实验中采用限制性内切酶(psti)进行聚合酶链反应-限制性内切酶分析(pcr/re)实验。
其中,pcr扩增所用引物为:
lbpl-f:5’-cctccttgcctcctctaatcc-3’;
lbpl-r:5’-acagttctccacccgtaatgc-3’。
因为靶点sgrna-1选择在了psti酶切位点处,故如果跑胶条带为两条,显示pcr扩增条带被切开,暗示植物基因组在该靶点处没有发生变异,植物就不是敲除突变体;如果跑胶条带为一条,显示pcr扩增条带没有被切开,暗示植物基因组植物基因组在该靶点处发生变异,植物为纯合突变体;如果跑胶条带为三条,显示pcr扩增条带一条被切开,一条没被切开,暗示植物基因组在该靶点处一条链发生变异,一条链没有发生变异,植物是杂合突变体。
pcr/re实验分析结果显示,基因编辑后枸杞植株幼苗中的lbpl基因靶位点发生了突变。回收、测序图中的条带,测序结果表明在lbpl基因的靶位点都发生了碱基插入/删除类型的突变;
下面是一个突变的结果,按照第一个位点删除一个碱基:
aatttagggtatttatcgtgtggtaccggaaatccwt
aatttagggtatttatcgtgtg.taccggaaatccpl。
将具有该突变的植株命名为t0代枸杞突变体植株pl。
轰击了506个叶盘,获得了3662个枸杞植株,全部进行pcr/re实验检测,发现其中52个突变体,其中36个为纯合突变体。
四、pl纯合突变体的果实成熟期鉴定
将上述三筛选得到的t0代枸杞突变体植株pl中挑选纯合突变体,得到t0代枸杞纯合突变体植株pl。
将t0代枸杞纯合突变体植株pl与相同大小野生型宁杞7号种植于宁夏中宁新堡镇宋营村大田中,所有植物被分为两组,每组各100株。t0代枸杞纯合突变体植株pl与野生型7号在施肥、浇水、修剪及其它农田管理方式一致,第3年,在同一时间收获鲜果。通过比较鲜果单果重量、单株产量、颜色、可溶性固形物、枸杞多糖、枸杞甜菜碱,发现均无显著差异。
但将t0代枸杞纯合突变体植株pl与野生型7号(wt)的鲜果采摘后于相同环境条件:24度、相对湿度为20%的培养箱中放置15d后,野生型明显比突变体的腐坏程度大(如图2)。
序列表
<110>天津吉诺沃生物科技有限公司
<120>一种获得非转基因耐储存鲜食枸杞的方法
<160>11
<170>siposequencelisting1.0
<210>1
<211>2152
<212>dna
<213>artificialsequence
<400>1
tatataacttaaacgcgtttaattggacgtatgctagagtagtaatcatatgctaacaag60
agatagatactgttacttgaacagacaaaaactaccgtagtcttcttctctctatcatct120
aggttagaggaatctccgtaaaacatgtaatcacgaaggaacaagggacccatccctttt180
gtttaattacgctaatttctctactttttccttctttataaactcctccatttccctctc240
ttttcttcataacccaaaatcctctgtttttaaagctaatttataaaacaaacaatgggc300
acttcctctgtttttctactattccttctttcttttcttctccttctcccgtccctcctt360
gcctcctctaatcctcaacaagttgtcgatgaagtacacaggtacgtgcatttttaattt420
tattttaattcaacattgcatatttgaaaagggacagagtcagataggaacaaggagttc480
attcgtattaaaataattgcatatggtaatactcatgtgtacattattattataataatg540
caggagtataaatggttcaaggaggaatttagggtatttatcgtgtggtaccggaaatcc600
aatcgacgattgttggcgttgtgatccgaattgggagaaaaaccgtcagcggttagctga660
ctgtgcaattggatttgggaaaaacgctattggtggtagagacggtaaaatctacgtggt720
gaccgattcgggtgacgacaatgccgtcactccgaagccggggactctccggcacgcagt780
gattcagactgagccactgtggattattttcgcgagggatatggttatacagttgaaaga840
ggaattaattatgaattcgtttaaaacgatagacggaaggggcgctagcgtacacatagc900
gggtggtccgtgtataacaatacagtacgtaacgaatattattatacacggaattcatat960
acatgactgtaaacaaggtggaaatgctatggtgcggagcagtccatcgcattacgggtg1020
gagaactgtttcggacggtgatggagtgtccatattcggtgggagccatgtttgggtgga1080
tcattgttctttgtccaactgtaaggacggtctgattgatgcgattatggggtctacagc1140
aataaccatttcaaataattacatgacacatcatgataaagttatgctcttgggacatag1200
tgatacatatactcaagataaaaacatgcaagtaactatagcttttaatcactttggtga1260
aggtcttgtccaaagaatgccaaggtatgagcctgagttatttaacttttatatactgac1320
aattttgacaatgtgaattttatgaattattatttggaataaatgcagatgtagacatgg1380
ttacttccatgtggtgaacaatgactacacacattgggaaatgtatgctattggtggaag1440
tgctgatcccactatcaatagtcaagggaaccgatttcttgcccctgatatcaggtttag1500
caaagaggttagttagttatttcacttacatttttctatctagtctgttaatgagttaac1560
gggtaattttattatatttatgtaggttacaaagcacgaggatgcaccggagagtgaatg1620
gaagaattggaattggaggaccgatggggacctaatgttgaatggtgcattttttacgcg1680
atcaggagttagaactggttcatcaagttatgctaaagcttcgagtttgagtgcaaggcc1740
gtcttcattagtagccaatcttgtgtctagttctggtgcactcaactgtaaaaagggttc1800
tcgttgctaattattattattatttcctaaaaaagacacaagaaccaaagcaaaagggga1860
ttaattatttaacttggttttgaaaaaagaaaaaggatgtaatttgaagaaaaaaaccat1920
ctagtgagggtgaaaaagtaagggacaagaagtttagggttgtgcttttaatatcatttt1980
ttcactccctttatttttatttttactttttaagttttttttgacttgattgtcaaatac2040
aacctcggcctgttattctcagccattgggacttttagttcttgttggttttagagagag2100
gcaagctaagaagtgttgatatatacatcaatatatatgtatttatttttga2152
<210>2
<211>1212
<212>dna
<213>artificialsequence
<400>2
atgggcacttcctctgtttttctactattccttctttcttttcttctccttctcccgtcc60
ctccttgcctcctctaatcctcaacaagttgtcgatgaagtacacaggagtataaatggt120
tcaaggaggaatttagggtatttatcgtgtggtaccggaaatccaatcgacgattgttgg180
cgttgtgatccgaattgggagaaaaaccgtcagcggttagctgactgtgcaattggattt240
gggaaaaacgctattggtggtagagacggtaaaatctacgtggtgaccgattcgggtgac300
gacaatgccgtcactccgaagccggggactctccggcacgcagtgattcagactgagcca360
ctgtggattattttcgcgagggatatggttatacagttgaaagaggaattaattatgaat420
tcgtttaaaacgatagacggaaggggcgctagcgtacacatagcgggtggtccgtgtata480
acaatacagtacgtaacgaatattattatacacggaattcatatacatgactgtaaacaa540
ggtggaaatgctatggtgcggagcagtccatcgcattacgggtggagaactgtttcggac600
ggtgatggagtgtccatattcggtgggagccatgtttgggtggatcattgttctttgtcc660
aactgtaaggacggtctgattgatgcgattatggggtctacagcaataaccatttcaaat720
aattacatgacacatcatgataaagttatgctcttgggacatagtgatacatatactcaa780
gataaaaacatgcaagtaactatagcttttaatcactttggtgaaggtcttgtccaaaga840
atgccaagatgtagacatggttacttccatgtggtgaacaatgactacacacattgggaa900
atgtatgctattggtggaagtgctgatcccactatcaatagtcaagggaaccgatttctt960
gcccctgatatcaggtttagcaaagaggttacaaagcacgaggatgcaccggagagtgaa1020
tggaagaattggaattggaggaccgatggggacctaatgttgaatggtgcattttttacg1080
cgatcaggagttagaactggttcatcaagttatgctaaagcttcgagtttgagtgcaagg1140
ccgtcttcattagtagccaatcttgtgtctagttctggtgcactcaactgtaaaaagggt1200
tctcgttgctaa1212
<210>3
<211>403
<212>prt
<213>artificialsequence
<400>3
metglythrserservalpheleuleupheleuleuserpheleuleu
151015
leuleuproserleuleualaserserasnproglnglnvalvalasp
202530
gluvalhisargserileasnglyserargargasnleuglytyrleu
354045
sercysglythrglyasnproileaspaspcystrpargcysasppro
505560
asntrpglulysasnargglnargleualaaspcysalaileglyphe
65707580
glylysasnalaileglyglyargaspglylysiletyrvalvalthr
859095
aspserglyaspaspasnalavalthrprolysproglythrleuarg
100105110
hisalavalileglnthrgluproleutrpileilephealaargasp
115120125
metvalileglnleulysglugluleuilemetasnserphelysthr
130135140
ileaspglyargglyalaservalhisilealaglyglyprocysile
145150155160
thrileglntyrvalthrasnileileilehisglyilehisilehis
165170175
aspcyslysglnglyglyasnalametvalargserserproserhis
180185190
tyrglytrpargthrvalseraspglyaspglyvalserilephegly
195200205
glyserhisvaltrpvalasphiscysserleuserasncyslysasp
210215220
glyleuileaspalailemetglyserthralailethrileserasn
225230235240
asntyrmetthrhishisasplysvalmetleuleuglyhisserasp
245250255
thrtyrthrglnasplysasnmetglnvalthrilealapheasnhis
260265270
pheglygluglyleuvalglnargmetproargcysarghisglytyr
275280285
phehisvalvalasnasnasptyrthrhistrpglumettyralaile
290295300
glyglyseralaaspprothrileasnserglnglyasnargpheleu
305310315320
alaproaspileargpheserlysgluvalthrlyshisgluaspala
325330335
progluserglutrplysasntrpasntrpargthraspglyaspleu
340345350
metleuasnglyalaphephethrargserglyvalargthrglyser
355360365
sersertyralalysalaserserleuseralaargproserserleu
370375380
valalaasnleuvalserserserglyalaleuasncyslyslysgly
385390395400
serargcys
<210>4
<211>23
<212>dna
<213>artificialsequence
<400>4
gggtatttatcgtgtggtaccgg23
<210>5
<211>23
<212>dna
<213>artificialsequence
<400>5
cggttagctgactgtgcaattgg23
<210>6
<211>23
<212>dna
<213>artificialsequence
<400>6
actgtggattattttcgcgaggg23
<210>7
<211>23
<212>dna
<213>artificialsequence
<400>7
gggtatttatcgtgtggtaccgg23
<210>8
<211>23
<212>dna
<213>artificialsequence
<400>8
cggttagctgactgtgcaattgg23
<210>9
<211>23
<212>dna
<213>artificialsequence
<400>9
actgtggattattttcgcgaggg23
<210>10
<211>103
<212>rna
<213>artificialsequence
<400>10
ggguauuuaucgugugguacguuuuagagcuagaaauagcaaguuaaaauaaggcuaguc60
cguuaucaacuugaaaaaguggcaccgagucggugcuuuuuuu103
<210>11
<211>1368
<212>prt
<213>artificialsequence
<400>11
metasplyslystyrserileglyleuaspileglythrasnserval
151015
glytrpalavalilethraspglutyrlysvalproserlyslysphe
202530
lysvalleuglyasnthrasparghisserilelyslysasnleuile
354045
glyalaleuleupheaspserglygluthralaglualathrargleu
505560
lysargthralaargargargtyrthrargarglysasnargilecys
65707580
tyrleuglngluilepheserasnglumetalalysvalaspaspser
859095
phephehisargleuglugluserpheleuvalglugluasplyslys
100105110
hisgluarghisproilepheglyasnilevalaspgluvalalatyr
115120125
hisglulystyrprothriletyrhisleuarglyslysleuvalasp
130135140
serthrasplysalaaspleuargleuiletyrleualaleualahis
145150155160
metilelyspheargglyhispheleuilegluglyaspleuasnpro
165170175
aspasnseraspvalasplysleupheileglnleuvalglnthrtyr
180185190
asnglnleupheglugluasnproileasnalaserglyvalaspala
195200205
lysalaileleuseralaargleuserlysserargargleugluasn
210215220
leuilealaglnleuproglyglulyslysasnglyleupheglyasn
225230235240
leuilealaleuserleuglyleuthrproasnphelysserasnphe
245250255
aspleualagluaspalalysleuglnleuserlysaspthrtyrasp
260265270
aspaspleuaspasnleuleualaglnileglyaspglntyralaasp
275280285
leupheleualaalalysasnleuseraspalaileleuleuserasp
290295300
ileleuargvalasnthrgluilethrlysalaproleuseralaser
305310315320
metilelysargtyraspgluhishisglnaspleuthrleuleulys
325330335
alaleuvalargglnglnleuproglulystyrlysgluilephephe
340345350
aspglnserlysasnglytyralaglytyrileaspglyglyalaser
355360365
glnglugluphetyrlyspheilelysproileleuglulysmetasp
370375380
glythrglugluleuleuvallysleuasnarggluaspleuleuarg
385390395400
lysglnargthrpheaspasnglyserileprohisglnilehisleu
405410415
glygluleuhisalaileleuargargglngluaspphetyrprophe
420425430
leulysaspasnargglulysileglulysileleuthrpheargile
435440445
protyrtyrvalglyproleualaargglyasnserargphealatrp
450455460
metthrarglysserglugluthrilethrprotrpasnphegluglu
465470475480
valvalasplysglyalaseralaglnserpheilegluargmetthr
485490495
asnpheasplysasnleuproasnglulysvalleuprolyshisser
500505510
leuleutyrglutyrphethrvaltyrasngluleuthrlysvallys
515520525
tyrvalthrgluglymetarglysproalapheleuserglyglugln
530535540
lyslysalailevalaspleuleuphelysthrasnarglysvalthr
545550555560
vallysglnleulysgluasptyrphelyslysileglucyspheasp
565570575
servalgluileserglyvalgluaspargpheasnalaserleugly
580585590
thrtyrhisaspleuleulysileilelysasplysasppheleuasp
595600605
asnglugluasngluaspileleugluaspilevalleuthrleuthr
610615620
leuphegluaspargglumetileglugluargleulysthrtyrala
625630635640
hisleupheaspasplysvalmetlysglnleulysargargargtyr
645650655
thrglytrpglyargleuserarglysleuileasnglyileargasp
660665670
lysglnserglylysthrileleuasppheleulysseraspglyphe
675680685
alaasnargasnphemetglnleuilehisaspaspserleuthrphe
690695700
lysgluaspileglnlysalaglnvalserglyglnglyaspserleu
705710715720
hisgluhisilealaasnleualaglyserproalailelyslysgly
725730735
ileleuglnthrvallysvalvalaspgluleuvallysvalmetgly
740745750
arghislysprogluasnilevalileglumetalaarggluasngln
755760765
thrthrglnlysglyglnlysasnserarggluargmetlysargile
770775780
glugluglyilelysgluleuglyserglnileleulysgluhispro
785790795800
valgluasnthrglnleuglnasnglulysleutyrleutyrtyrleu
805810815
glnasnglyargaspmettyrvalaspglngluleuaspileasnarg
820825830
leuserasptyraspvalasphisilevalproglnserpheleulys
835840845
aspaspserileaspasnlysvalleuthrargserasplysasnarg
850855860
glylysseraspasnvalproserglugluvalvallyslysmetlys
865870875880
asntyrtrpargglnleuleuasnalalysleuilethrglnarglys
885890895
pheaspasnleuthrlysalagluargglyglyleusergluleuasp
900905910
lysalaglypheilelysargglnleuvalgluthrargglnilethr
915920925
lyshisvalalaglnileleuaspserargmetasnthrlystyrasp
930935940
gluasnasplysleuilearggluvallysvalilethrleulysser
945950955960
lysleuvalseraspphearglysasppheglnphetyrlysvalarg
965970975
gluileasnasntyrhishisalahisaspalatyrleuasnalaval
980985990
valglythralaleuilelyslystyrprolysleuglusergluphe
99510001005
valtyrglyasptyrlysvaltyraspvalarglysmetilealalys
101010151020
sergluglngluileglylysalathralalystyrphephetyrser
1025103010351040
asnilemetasnphephelysthrgluilethrleualaasnglyglu
104510501055
ilearglysargproleuilegluthrasnglygluthrglygluile
106010651070
valtrpasplysglyargaspphealathrvalarglysvalleuser
107510801085
metproglnvalasnilevallyslysthrgluvalglnthrglygly
109010951100
pheserlysgluserileleuprolysargasnserasplysleuile
1105111011151120
alaarglyslysasptrpaspprolyslystyrglyglypheaspser
112511301135
prothrvalalatyrservalleuvalvalalalysvalglulysgly
114011451150
lysserlyslysleulysservallysgluleuleuglyilethrile
115511601165
metgluargserserpheglulysasnproileasppheleugluala
117011751180
lysglytyrlysgluvallyslysaspleuileilelysleuprolys
1185119011951200
tyrserleuphegluleugluasnglyarglysargmetleualaser
120512101215
alaglygluleuglnlysglyasngluleualaleuproserlystyr
122012251230
valasnpheleutyrleualaserhistyrglulysleulysglyser
123512401245
progluaspasngluglnlysglnleuphevalgluglnhislyshis
125012551260
tyrleuaspgluileilegluglnileserglupheserlysargval
1265127012751280
ileleualaaspalaasnleuasplysvalleuseralatyrasnlys
128512901295
hisargasplysproilearggluglnalagluasnileilehisleu
130013051310
phethrleuthrasnleuglyalaproalaalaphelystyrpheasp
131513201325
thrthrileasparglysargtyrthrserthrlysgluvalleuasp
133013351340
alathrleuilehisglnserilethrglyleutyrgluthrargile
1345135013551360
aspleuserglnleuglyglyasp
1365
- 还没有人留言评论。精彩留言会获得点赞!