一种PS转座子系统及其介导的基因转移方法与流程
本发明涉及建立一种PS转座子系统介导的基因转移方法,同时还公开了该方法涉及的转基因供体质粒和转座酶真核表达及体外转录辅助质粒的构建方法,及其在制备转基因动物、研究基因功能和人类基因治疗中的应用。本发明属于动物基因工程领域。
背景技术:
基因转移是当前一项重要的生物技术手段,可以介导外源基因稳定的整合到宿主染色体中,所以在研究基因功能、制备转基因生物和人类基因治疗等领域中都有重要的应用价值。
转座子是基因组上一段可以自由跳跃的DNA序列,首先是由Mc.Clintock于20世纪40年代在玉米染色体中发现,后又陆续在细菌、真菌及昆虫等各种生物中发现。经过对转座子的注解之后发现无论在原核生物还是真核生物中都存在相当数量的转座子。转座子在不同生物间分布存在较大差异,尤其是高等真核生物中含量更高,例如转座子是哺乳动物基因组中最大的组分,几乎占到人类基因组的一半,在玉米中几乎占到80%。DNA转座子的转座过程遵循“剪切-粘贴”的机制在基因组上进行移动,因此被作为有效的基因转移工具进行开发。近年来,转座子在人类基因治疗,转基因鼠和斑马鱼等模式生物制备和功能基因组学的研究上已经取得了重要的进展。除此之外,转座子作为基因转移的载体具有很多优势:1)结构简单,与外源基因共同整合入受体基因组的只是两侧的TIR序列,对外源基因的影响很小,在整合位点也没有发现大片段的丢失和染色体重排的现象;2)相较与病毒载体,DNA转座子载体对插入片段限制较小;3)外源基因可以稳定整合到染色体中,而且通过生殖细胞传代后也能够长期的表达;4)转座酶可以催化单拷贝的基因精确的插入手提序列中,不再依靠随机整合且插入片段的大小也不会发生变化;5)转座子系统可以全部以裸露的DNA形式给予,也可以DNA与RNA或者蛋白质的形式提供转座酶相结合进行转座,所以其免疫原性较低。其中研究最为广泛的DNA转座子Sleeping Beauty(SB)、PiggBac(PB)和Tol2,这三种转座子不仅发生起源不同,在转座活性、插入偏好性方面也存在差异,因此在应用上也有所区别。例如SB偏好插入位点是“TA”,但是对基因没有偏好性,所以SB转座子常被用于人类基因治疗;PB偏好插入位点是“TTAA”,而且偏好插入基因内部,所以常被用于功能基因研究、基因捕获等;Tol2则没有很明显的偏好插入位点,插入位置多为基因上游调控区,故常用来研究增强子捕获等。
显微注射已经被广泛应用,且技术成熟稳定,同时科学家们利用转座子介导基因转移的特点,进一步完善了转座子介导细胞质显微注射的方法。由于DNA转座系统中的转座酶含有核定位信号序列(NLS),可以顺利的介导目的基因进入细胞核,注射可以不入核,直接进行细胞质注射。所以这项技术正受到重视,并取得了令人鼓舞的进展,这可能是突破大型哺乳动物转基因技术瓶颈的新方法。
转座子用于人类基因治疗主要包括目的基因克隆、基因转移、靶细胞选择和临床试验观察等阶段,其中基因转移则是基因治疗的关键步骤,而DNA转座子介导的基因转移在基因治疗中的优点就是高效、安全。迄今为止,转座子已经称为基因治疗中最常用的非病毒载体。此外,转座子还可以制备突变体,开展基因捕获,应用与功能基因组学研究。
DNA转座子介导的基因转移系统是备受科学家们青睐的转基因技术,但是具有自主转座活性的DNA转座子在脊椎动物中很少见。1996年,首次在白化青鏘(Oryziaslatipes)中发现具有天然活性的脊椎动物转座子,即Tol2转座子;2008年,在金鱼中发现了第二例具有自主转座活性的Tgf2转座子,并且该转座子与青鏘Tol2转座子元件十分相似。发明人利用生物信息学的手段,对脊椎动物的Tc1/Mariner转座子超家族挖掘研究中发现了passer(PS)家族,通过插入年龄分析等表明其可能具有较高的活性。在系统进化比较研究基础上进行了分子重构,获得PS转座子的两个末端重复序列TIR关键元件和转座酶的序列,并构建成一套基因转移载体系统,经细胞、转基因鼠和斑马鱼等验证,该载体系统能够有效介导基因转移,在转基因动物制备和基因治疗中具有很大应用潜力。
技术实现要素:
为了克服上述缺陷,本发明提供一种PS转座子系统及其介导的基因转移方法,采用生物信息学方法,发现Tc1/Mariner超家族中PS家族,进行分子重构,获得转座子两个末端重复序列和转座酶序列,并构建PS转座子供体质粒和表达PS转座酶的辅助质粒,组装成一套基因转移系统,命名为PS转座子系统。本发明的目的在于提供一种PS转座系统介导的高效基因转移方法,提高鼠、斑马鱼等动物的转基因制备效率,并可以有效应用于细胞基因转染整合、人类基因治疗和基因捕获等研究领域。
本发明所述的PS转座子系统主要由PS转座子供体质粒和PS转座酶辅助质粒构成。
本发明所述的转基因供体质粒(pLB-PS)包括PS转座子两侧末端重复序列和多克隆插入位点,多克隆插入位点(NruI/NotI/EcoRI酶切位点)可以插入需要转移的目的基因盒,该供体质粒(pLB-PS)的序列如SEQ ID No.1所示。目的基因可通过上述的任意一个多克隆插入位点插入转基因供体质粒中。目的基因插入转基因供体质粒中是指:目的基因插入PS5’TIR和PS3’TIR之间。
本发明所述的PS转座子两侧末端重复序列来源于Tc1/Mariner超家族中PS家族的分子重构所得,两侧末端反向重复序列(TIR)分别长度为28个核苷酸序列(即PS5’TIR和PS3’TIR),序列分别如SEQ ID No.2和SEQ ID No.3所示。
本发明所述的目的基因盒可以是报告基因表达盒,或者其它外源基因表达盒,或者基因捕获元件。
本发明所述的PS转座酶(即PS CDS,又称为PSase CDS)序列为利用生物信息学分析手段,对PS转座子家族进行分子重构获得转座酶序列,经化学合成获得,如SEQ ID No.5所示。
本发明所述的PS转座酶辅助质粒pcDNA3.9-PSase有两种形式,一是可以在细胞和体内直接表达的真核表达质粒,另一种是经体外转录辅助质粒,可以进行体外转录形成5’加帽PS转座酶的mRNA。
本发明所述的pcDNA3.9-PSase真核表达质粒包括病毒启动子序列(CMV)和PS转座酶cDNA序列和bGH多聚腺苷酸(PolyA)组成;载体构架来自pcDNA3.9载体,所述病毒启动子序列(CMV)长度为584个核苷酸序列,能够指导下游基因的表达;所述PS转座酶长度为1275个核苷酸序列;所述bGH多聚腺苷酸(PolyA)长度为225个核苷酸序列,该载体能够自主表达转座酶,同时该载体可以作为体外转录辅助质粒包括T7启动子、PS转座酶cDNA序列和bGH PolyA序列,其中T7启动子的长度为19个核苷酸序列(bp)该载体的序列,bGH PolyA序列终止转录,载体能够利用mMESSAGE Mmachine T7kit(购自Invitrogen公司)试剂盒进行PS转座酶5’加帽mRNA的体外转录,该载体的序列如SEQ ID No.4所示。
本发明提供的基于PS转座子系统的基因转移方法,经过细胞和胚胎水平验证能够高效介导基因转移,可应用于多个生物技术领域:1)使用本发明中给出的方法可有效地将目的基因盒插入到宿主基因组中,提高基因转移效率;2)与基因捕获技术结合可以有效开展动物基因功能研究;3)可以介导进行人类基因治疗等。
附图说明
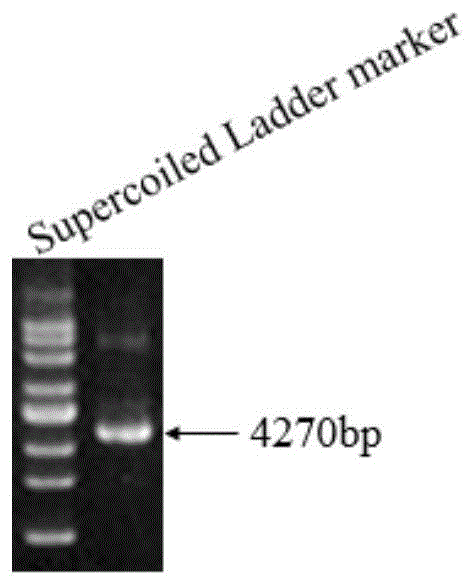
图1:pLB-PSase质粒电泳图;
图2:pcDNA3.9-PSase质粒电泳图;
图3:pcDNA3.9-PSase质粒图谱;
图4:pLB-PS质粒示意图;
图5:PS-TIR退火产物电泳图;
图6:PS-TIR纯化产物电泳图;
图7:pLB-PS质粒电泳图;
图8:pLB-PS质粒酶切产物电泳图;
图9:pPS-PGK-NEO质粒电泳图;
图10:pPS-PGK-NEO质粒酶切产物电泳图;
图11:pPS-PGK-NEO质粒图谱;
图12:PS-FAG-GFP PCR切回产物;
图13:pPS-FAG-GFP质粒电泳图;
图14:pPS-FAG-GFP酶切电泳图,1:pPS-FAG-GFP酶切;2:pPS-FAG-GFP质粒;
图15:pPS-FAG-GFP质粒图谱;
图16:pZB-RT-bGH酶切电泳图;
图17:pPS-PGK-NEO酶切电泳图;
图18:PS框架、Tyr-TYR-bGHpA片段切回电泳图,1:PS框架切回电泳图;2:Tyr-TYR-bGHpA片段切回电泳图;
图19:pPS-Tyr质粒电泳图;
图20:pPS-Tyr酶切电泳图,1-6:pPS-Tyr酶切;7:pPS-Tyr质粒;
图21:pPS-Tyr质粒图谱;
图22:Hela细胞G418抗性筛选阳性克隆图;
图23:PS与SB转座活性比较图;
图24:PS转座系统制备转基因小鼠图;
图25:不同时期显微注射斑马鱼胚胎荧光图;
图26:PS转座子显微注射斑马鱼阳性率比较图。
具体实施方式
为了更好地说明本发明,便于理解本发明的技术方案,本发明的典型但非限制的实施例如下:
下述实施例中提到的实验方法,如无特别说明均为常规方法。
实施例I、转座酶表达载体pcDNA3.9-PSase的构建
1、转座酶PSase载体合成。
PSase转座酶序列经化学合成后,克隆至LB载体上,构建p LB-PSase载体,质粒DNA用1%琼脂糖凝胶电泳检测(如图1)。
2、转座酶真核表达质粒及体外转录辅助质粒pcDNA3.9-PSase的构建
用限制性内切酶BamHI和XhoI将转座酶CDS从pLB-PSase载体中切出,琼脂糖凝胶回收1296bp的CDS序列。同时用BamHI和XhoI双酶切pcDNA3.9载体,切胶回收3.9kb片段作为载体框架。将转座酶CDS和pcDNA3.9载体框架连接后,转化感受态细胞Top10,挑单菌落至液体培养基LA培养,提质粒进行电泳鉴定(如图2),阳性克隆者命名为pcDNA3.9-PSase,质粒图谱如图3所示。
pcDNA3.9-PSase载体序列如SEQ ID No.4所示,包括CMV Enhancer promoter 235-818、T7promoter 863-881、PSase CDS 919-2193、Sp6promoter 2224-2242、bGH poly(A)signal 2268-2492、F1Origin 2538-2896、SV40poly(A)signal 2897-2998、Ampicillin Resistance gene 4208-5068。
实施例II、转座子表达载体的构建
1、转基因供体质粒pLB-PS载体构建
1.1PS两个末端反向重复元件(PS 3’和5’)的合成
对多物种进行Tc1/Marinier家族中PS转座子进行插入年龄等分析,并在系统进化比较研究基础上进行了分子重构,确定了高度保守的5’和3’TIR,分别为28bp的反向互补序列。并将此序列加上限制性内切酶位点,送华大基因公司合成,序列见表1。
1.2PS转座元件的聚合酶扩增
将上述化学合成的单链核苷酸链PS5’t(包括PS5’末端的反向重复元件和酶切位点,酶切位点用于插入目的基因)和PS3’t(包括PS3’末端的反向重复元件和酶切位点,酶切位点用于插入目的基因)先退火,再用Klenow聚合酶延伸,形成双链。反应体系为50μL,包括1ul PS5’t(100uM)、1ul PS3’t(100uM)、23ul超纯水,95℃变性5min,再以每秒0.1℃的温度降至25℃,保持5min。再加入5μL Klenow Buffer,2μL Klenow polymerase,5μL 4dNTP,13μL超纯水,37℃ 1.5h,80℃ 20min,降至常温,获得目标序列(PS TIR)。随后用Qiagen PCR纯化试剂盒纯化(如图5,6),经核酸浓度测定仪测定浓度后,-20℃保存备用。质粒示意图如图4所示,图4中基因插入位点即多克隆位点(酶切位点)。
1.3 p LB-PS载体构建
p LB-PS载体结构示意图如图4所示,将纯化的PS转座子片段(即上述1.2得到的目标序列PS TIR)与天根零背景载体pLB用T4连接酶连接,转化感受态细胞Top10,挑单菌落至含Amp的液体培养基LB培养,提质粒进行电泳及酶切鉴定(如图7,8),阳性克隆者命名为pLB-PS。pLB-PS载体序列如SEQ ID No.1所示,包括T7promoter 305-323;PS 5’TIR 380-407(SEQ ID No.2);PS 3’TIR 428-455(SEQ ID No.3);Ori 1257-1845;Ampicillin Resistance gene 2016-2876。SEQ ID No.1中,第408-427的碱基为多克隆位点(酶切位点),用于插入目的基因。
2、转座子载体pPS-PGK-NEO的构建
用限制性内切酶Nru1酶切PGK-NEO-bGHpA-TA克隆载体(PGK-NEO-bGHpA-TA克隆载体为本实验室保存载体),切胶回收1659bp的PGK-NEO-bGHpA表达框(其作为目的基因盒)。用Nru 1酶切LB-PS载体,回收3066bp的载体框架,将表达框与载体连接,转化感受态细胞Top10,挑单菌落至含Amp液体培养基LB培养,提质粒进行电泳及酶切鉴定,筛选大小及插入方向正确者(如图9,10),送华大基因测序,质粒图谱如图11所示。
3、转座子载体pPS-FAG-GFP的构建
以pZB-FAG-GFP质粒DNA为模板,按表2中列举的扩增PS-FAG-GFP的引物扩增FAG-GFP基因片段(扩增的FAG-GFP基因片段作为目的基因盒)。反应体系为50μl,10μL 5×SF Buffer、1μl dNTP、2μL 10μM引物PS-FAG-GFP-F、2μl 10μM引物PS-FAG-GFP-R(见引物序列表2)、1μl PhantaTM Super-Fidelity,1μl pZB-FAG-GFP质粒DNA模板,加超纯水至50μl(高保真酶购自诺唯赞公司)。PCR扩增程序:94℃ 40s,55℃ 40s,72℃ 1m30s,循环30次。PCR扩增产物用1%琼脂糖凝胶电泳检测。将PCR产物琼脂糖凝胶回收(如图12),与p LB载体连接,进行TA克隆,筛选出阳性克隆,提质粒及酶切鉴定(如图13,14),测序比对正确后保存供下一步实验用,命名为pPS-FAG-GFP,质粒图谱如图15。
4、转座子载体pPS-Tyr的构建
用限制性内切酶NheI酶切载体pZB-RT-bGH(如图16),回收Tyr-TYR-bGHpA片段(2555bp)(如图18),补平纯化后保存待用;用NruI酶切载体pPS-PGK-NEO(如图17),回收3070bp PS框架(如图18),与上述补平片段Tyr-TYR-bGHpA(其作为目的基因盒)连接,筛选出阳性克隆,提质粒及酶切鉴定(如图19,20),测序比对正确后保存待用,命名为pPS-Tyr,质粒图谱如图21。
表1 PS-TIR序列
表1中序列分别为SEQ ID No.6和SEQ ID No.7。表1中下换线处为酶切位点。
表2 PS-FAG-GFP引物序列
表2中序列分别为SEQ ID No.8和SEQ ID No.9。
实施例III、PS转座子介导目的基因在小鼠细胞水平表达检验
1、冻存细胞的复苏与培养
pPS-PGK-NEO和pcDNA3.9-PSase质粒用OMEGA无内毒素质粒提取试剂盒(购自OMEGA公司)提取,产物终浓度调整为500ng/ul用于细胞转染。
从液氮中取出装有人宫颈癌细胞(Hela)的冻存管(保存于本实验室的细胞),立即投入37-40℃的温水中快速晃动,直至冻存液完全融解;在1-2min内完成复温;将细胞悬液移入无菌的离心管,加入5mL培养液,轻轻吹匀;将细胞悬液800-1000rpm离心5min,弃上清;向含有细胞沉淀的离心管加入1mL完全培养基,轻轻吹匀,将细胞悬液转入细胞培养瓶,加入适量的完全培养基进行培养。
2、细胞转染与筛选
将人宫颈癌细胞Hela分成4组,各组分别转染pPS-PGK-NEO:pcDNA3.9-PSase和pPS-PGK-NEO:pcDNA3.9,pSB-PGK-NEO:pT2-CMV-SB100X和pSB-PGK-NEO:pT2-CMV每组3个重复。
转染前24h,向六孔板中各孔含10%胎牛血清(购自GIBCO公司)的2000μLDMEM高糖培养基(购自GIBCO公司)中按5×105个细胞/孔接种Hela细胞,使细胞在转染前达到约70-80%的汇合;将2种质粒按照1:1质量比混和稀释于用100μL的Opti-MEM(购自GIBCO公司)培养基中,并温和地混匀;将3μL FUGENE转染试剂(购自Promega公司)加入100μL的无血清、无抗生素的Opti-MEM培养基并轻轻混匀,于室温孵育5min;5min后,分别将100μL的转染试剂稀释液加入100μL的各组DNA稀释液中,轻轻混匀并于室温放置20min;将200μL混合物加入到准备好的孔内,轻轻来回晃动培养板,将其置于37℃、饱和湿度、5%CO2的培养箱中培养,于4h后用完全培养基代替转染培养基,完全培养24-48h后,将1%的细胞接种于6孔板中培养,当细胞达10-20%汇合时,加入浓度为600μg/mL的G418筛选培养液,筛选10-12天。用Giemsa染液(购自GIBCO公司)染色,统计阳性克隆数。
3、转染细胞阳性克隆鉴定
以pPS-PGK-NEO:pcDNA3.9-PSase(PS+/PSase+)与pSB-PGK-NEO:pT2-CMV-SB100X(SB+/SBase+)组为实验组,以pPS-PGK-NEO:pcDNA3.9(PS+/PSase-)与pSB-PGK-NEO:pT2-CMV(SB+/SBase-)组为对照组,含G418抗性培养液筛选10-12天后,Gimsa(吉姆萨)染色计数。
结果发现:PS实验组阳性细胞克隆数为560,PS对照组阳性细胞克隆数为18,SB实验组阳性细胞克隆数为427,SB对照组阳性细胞克隆数为18。PS实验组的细胞表达新霉素抗性基因的细胞数量显著高于SB实验组的细胞克隆数(如图22,23)。结果表明PS转座子系统能在Hela细胞中高效的介导新霉素抗性基因转染整合,并且高于SB转座子活性,即用抗性基因证实了PS转座子系统可以高效介导外源基因转移。
实施例IV、PS转座子介导目的基因在小鼠胚胎中的基因转移
1、小鼠受精卵的准备
成熟雌小鼠腹腔内注射5国际单位(IU)的孕马血清促性腺激素(PMSG)之后,约在48—54h后再注射2.5-5.0IU的人绒毛膜促性腺激素(hCG),约12h后即可诱发排卵。雌小鼠给予hCG后立刻与雄性小鼠合笼。交配后雌鼠的阴道栓易见,可用交配指示剂指示。受精卵收集时期在合笼后的次日早晨即显微注射前几小时。将输卵管仔细切开,采用输卵管冲洗术或输卵管壶部切开术收集受精卵。
2、细胞质显微注射小鼠受精卵
pPS-Tyr和pcDNA3.9-PSase质粒用OMEGA无内毒素质粒提取试剂盒提取,以线性pcDNA3.9-PSase为模板,用mMessagemMachine kit试剂盒制备PSase-mRNA,产物终浓度调整为20ng/μL,pPS-CAG-GFP和PS-mRNA以1:1摩尔比混合备用。
将pPS-Tyr质粒和PSase mRNA混合注射为实验组,以FVB鼠为模式生物,细胞质注射小鼠受精卵,观察后期鼠毛色的变化,如图24,可明显观察到其中毛色的变化。
实施例V、PS转座子介导目的基因在斑马鱼胚胎中的基因转移
1、斑马鱼受精卵的准备
公母鱼各饲养若干,于注射前一天晚上,移取1尾母鱼,1尾公鱼于繁殖盒中,隔板隔开培养,光刺激1h后避光过夜培养,第二天将隔板拿开,公母鱼追逐即开始产卵,自产卵开始计时,约10min后收卵,将收集的受精卵置于体视显微镜下进行斑马鱼胚胎注射实验。
2、显微注射斑马鱼胚胎
pPS-FAG-GFP和pcDNA3.9-PSase质粒用无内毒素质粒提取试剂盒提取,用NanoPhotometer核酸浓度测定仪测定浓度后保存备用。pcDNA3.9-PSase质粒同上所述,制备PS转座酶mRNA,测定浓度后保存备用。
将pPS-FAG-GFP质粒和PS转座酶mRNA以不同浓度混合注射斑马鱼单细胞期胚胎。固定转座子质粒终浓度为25ng/μL,PS-mRNA(由pcDNA3.9-PSase载体体外转录后获得)的终浓度设2个梯度:5ng/μL和25ng/μL,同时以单注射转座子质粒组为对照,未处理组(即未注射组)为空白对照组。每组3个重复,每个重复注射胚胎数均在100枚以上。将注射后的胚胎于1×E3培养液中培养,12-24h换液一次,换液的同时去除死亡胚胎,对照组按照同样的方法培养。分别在培养24h、48h和5d后于荧光显微镜下观察绿色荧光表达情况,同时统计成活胚胎数和阳性胚胎数,计算阳性率。
3、不同浓度转座酶介导目的基因表达检测
在荧光显微镜下检测荧光表达情况,结果显示各时期的各组胚胎在显微注射后24h、48h和5d三个时间段都有荧光表达,单注射转座子质粒组也有荧光表达,空白对照组没有荧光表达(如图25)。注射PS转座酶与否,斑马鱼表达绿色荧光蛋白的阳性率不同;在24h,48h和5d时,含有转座酶组(PS+组)阳性率(87.14%、92.77%、92.77%)显著高于未注射转座酶组(PS-组)阳性率(48.73%、61.51%、61.51%)(如图26)。结果表明PS转座子系统同样能在斑马鱼胚胎高效的介导绿色荧光蛋白基因转移,即在斑马鱼胚胎上证实了PS转座子系统有介导基因转移的功能。
以上显示和描述了本发明的基本原理、主要特征和本发明的优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是为了说明本发明的原理,在不脱离本发明精神和范围的前提下本发明还会有各种变化和改进,这些变化和改进都落入要求保护的本发明范围内。本发明要求保护范围由所附的权利要求书及其等同物界定。
序列表
<110> 扬州大学
<120> 一种PS转座子系统及其介导的基因转移方法
<130> xhx2019050501
<141> 2019-05-05
<160> 9
<170> SIPOSequenceListing 1.0
<210> 1
<211> 3070
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
gcccctgcag ccgaattata ttatttttgc caaataattt ttaacaaaag ctctgaagtc 60
ttcttcattt aaattcttag atgatacttc atctggaaaa ttgtcccaat tagtagcatc 120
acgctgtgag taagttctaa accatttttt tattgttgta ttatctctaa tcttactact 180
cgatgagttt tcggtattat ctctattttt aacttggagc aggttccatt cattgttttt 240
ttcatcatag tgaataaaat caactgcttt aacacttgtg cctgaacacc atatccatcc 300
ggcgtaatac gactcactat agggagagcg gccgccagat cttccggatg gctcgagttt 360
ttcagcaaga tggatcctac cgtattttcc gcactataag gcgcacctcg cgagcggccg 420
cgaattcggt gcgccttata gtgcggaaaa tacggtagga tccccatctt tctagaagat 480
ctcctacaat attctcagct gccatggaaa atcgatgttc ttcttttatt ctctcaagat 540
tttcaggctg tatattaaaa cttatattaa gaactatgct aaccacctca tcaggaaccg 600
ttgtaggtgg cgtgggtttt cttggcaatc gactctcatg aaaactacga gctaaatatt 660
caatatgttc ctcttgacca actttattct gcattttttt tgaacgaggt ttagagcaag 720
cttcaggaaa ctgagacagg aattttatta aaaatttaaa ttttgaagaa agttcagggt 780
taatagcatc cattttttgc tttgcaagtt cctcagcatt cttaacaaaa gacgtctctt 840
ttgacatgtt taaagtttaa acctcctgtg tgaaattgtt atccgctcac aattccacac 900
attatacgag ccggaagcat aaagtgtaaa gcctggggtg cctaatgagt gagctaactc 960
acattaattg cgttgcgctc actgccaatt gctttccagt cgggaaacct gtcgtgccag 1020
ctgcattaat gaatcggcca acgcgcgggg agaggcggtt tgcgtattgg gcgctcttcc 1080
gcttcctcgc tcactgactc gctgcgctcg gtcgttcggc tgcggcgagc ggtatcagct 1140
cactcaaagg cggtaatacg gttatccaca gaatcagggg ataacgcagg aaagaacatg 1200
tgagcaaaag gccagcaaaa ggccaggaac cgtaaaaagg ccgcgttgct ggcgtttttc 1260
cataggctcc gcccccctga cgagcatcac aaaaatcgac gctcaagtca gaggtggcga 1320
aacccgacag gactataaag ataccaggcg tttccccctg gaagctccct cgtgcgctct 1380
cctgttccga ccctgccgct taccggatac ctgtccgcct ttctcccttc gggaagcgtg 1440
gcgctttctc atagctcacg ctgtaggtat ctcagttcgg tgtaggtcgt tcgctccaag 1500
ctgggctgtg tgcacgaacc ccccgttcag cccgaccgct gcgccttatc cggtaactat 1560
cgtcttgagt ccaacccggt aagacacgac ttatcgccac tggcagcagc cactggtaac 1620
aggattagca gagcgaggta tgtaggcggt gctacagagt tcttgaagtg gtggcctaac 1680
tacggctaca ctagaaggac agtatttggt atctgcgctc tgctgaagcc agttaccttc 1740
ggaaaaagag ttggtagctc ttgatccggc aaacaaacca ccgctggtag cggtggtttt 1800
tttgtttgca agcagcagat tacgcgcaga aaaaaaggat ctcaagaaga tcctttgatc 1860
ttttctacgg ggtctgacgc tcagtggaac gaaaactcac gttaagggat tttggtcatg 1920
agattatcaa aaaggatctt cacctagatc cttttaaatt aaaaatgaag ttttaaatca 1980
atctaaagta tatatgagta aacttggtct gacagttacc aatgcttaat cagtgaggca 2040
cctatctcag cgatctgtct atttcgttca tccatagttg cctgactccc cgtcgtgtag 2100
ataactacga tacgggaggg cttaccatct ggccccagtg ctgcaatgat accgcgagac 2160
ccacgctcac cggctccaga tttatcagca ataaaccagc cagccggaag ggccgagcgc 2220
agaagtggtc ctgcaacttt atccgcctcc atccagtcta ttaattgttg ccgggaagct 2280
agagtaagta gttcgccagt taatagtttg cgcaacgttg ttgccattgc tacaggcatc 2340
gtggtgtcac gctcgtcgtt tggtatggct tcattcagct ccggttccca acgatcaagg 2400
cgagttacat gatcccccat gttgtgcaaa aaagcggtta gctccttcgg tcctccgatc 2460
gttgtcagaa gtaagttggc cgcagtgtta tcactcatgg ttatggcagc actgcataat 2520
tctcttactg tcatgccatc cgtaagatgc ttttctgtga ctggtgagta ctcaaccaag 2580
tcattctgag aatagtgtat gcggcgaccg agttgctctt gcccggcgtc aatacgggat 2640
aataccgcgc cacatagcag aactttaaaa gtgctcatca ttggaaaacg ttcttcgggg 2700
cgaaaactct caaggatctt accgctgttg agatccagtt cgatgtaacc cactcgtgca 2760
cccaactgat cttcagcatc ttttactttc accagcgttt ctgggtgagc aaaaacagga 2820
aggcaaaatg ccgcaaaaaa gggaataagg gcgacacgga aatgttgaat actcatactc 2880
ttcctttttc aatattattg aagcatttat cagggttatt gtctcatgag cggatacata 2940
tttgaatgta tttagaaaaa taaacaaata ggggttccgc gcacatttcc ccgaaaagtg 3000
ccacctgacg tctaagaaac cattattatc atgacattaa cctataaaaa taggcgtatc 3060
acgaggcccc 3070
<210> 2
<211> 28
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
ccgtattttc cgcactataa ggcgcacc 28
<210> 3
<211> 28
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
ggtgcgcctt atagtgcgga aaatacgg 28
<210> 4
<211> 5206
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
gacggatcgg gagatctccc gatcccctat ggtcgactct cagtacaatc tgctctgatg 60
ccgcatagtt aagccagtat ctgctccctg cttgtgtgtt ggaggtcgct gagtagtgcg 120
cgagcaaaat ttaagctaca acaaggcaag gcttgaccga caattgcatg aagaatctgc 180
ttagggttag gcgttttgcg ctgcttcgcg atgtacgggc cagatatacg cgttgacatt 240
gattattgac tagttattaa tagtaatcaa ttacggggtc attagttcat agcccatata 300
tggagttccg cgttacataa cttacggtaa atggcccgcc tggctgaccg cccaacgacc 360
cccgcccatt gacgtcaata atgacgtatg ttcccatagt aacgccaata gggactttcc 420
attgacgtca atgggtggac tatttacggt aaactgccca cttggcagta catcaagtgt 480
atcatatgcc aagtacgccc cctattgacg tcaatgacgg taaatggccc gcctggcatt 540
atgcccagta catgacctta tgggactttc ctacttggca gtacatctac gtattagtca 600
tcgctattac catggtgatg cggttttggc agtacatcaa tgggcgtgga tagcggtttg 660
actcacgggg atttccaagt ctccacccca ttgacgtcaa tgggagtttg ttttggcacc 720
aaaatcaacg ggactttcca aaatgtcgta acaactccgc cccattgacg caaatgggcg 780
gtaggcgtgt acggtgggag gtctatataa gcagagctct ctggctaact agagaaccca 840
ctgcttactg gcttatcgaa attaatacga ctcactatag ggagacccaa gcttggtacc 900
gagctcggat ccgccaccat ggctcctacc aaaagacacg cgtacaacgc tgagtttaaa 960
ctcaaggcga taagccacgc acaagaacac ggcaatagag cagcagcgag agaatttaat 1020
atcaatgaat caatggtgag gaagtggagg aagtatgagg atgagctccg ccaagtaaag 1080
aagacaacac agagtttccg cgggaacaaa gcgagatggc cacagttaga ggacaaagtt 1140
gaacagtggg ttgctgaaca aagagcagca agcagaagtg ttagtacagt cacaattcgt 1200
atgaaggcaa tagcgctagc tcgcgaacat aacatcagtg aattcagagg cggtccttct 1260
tggtgcttcc gttttatgaa acgacgtcat ctctccatcc gtacgcgcac tactgtgtca 1320
caacaactac cagctgatta tcaggaaaag ttggccactt tccgcacata ctgcagaaac 1380
aagataactg aaaaaaagat ccagccagag catatcatca atatggacga ggttccactc 1440
accttcgata tccctgtaaa ccgcactgtg gataaaacag gagcacgtac ggtgaatatt 1500
cgcaccacag ggaatgagaa aacgtccttc actgtagttc tcgcctgcca ggctaatggc 1560
cacaaacttc cacccatggt tattttcaag aggaagacct tgccgaaaga aaactttcca 1620
gctggcattg tcataaaagc taactcgaag ggatggatgg atgaagaaaa gatgagtgag 1680
tggttgagag aaatttatgt gaagagaccg ggtggttttt ttcacacagc tccgtcccta 1740
ttgatctatg actccatgcg cgcacatatc accgagcatg tcaaaaaaca agtgaagcac 1800
actaattcag tgctcgccgt cattccgggt ggattaacaa aagaactcca gccgctcgat 1860
gttggcgtca acagagcatt caaagctcga ctgcgaactg cgtgggagca gtggatgacc 1920
gaaggcgaac acacgttcac caagacgggg agacagcgcc ggacgacata tgctaatatc 1980
tgcaagtgga tagtaaatgc ctgggctggt atatcagtca caactgtggt ccgagctttt 2040
aggaaggcag gaattgtcac cgaactgcca gacaacagca gcgacactga ctcggttaat 2100
gatgactttg ataagacgga gccaggcgtt ttggatgccg caatagccca gctgttcaat 2160
tcggacacgg aagaagaagt tttcgaggga ttttagctcg agcatgcatc tagagggccc 2220
tattctatag tgtcacctaa atgctagagc tcgctgatca gcctcgactg tgccttctag 2280
ttgccagcca tctgttgttt gcccctcccc cgtgccttcc ttgaccctgg aaggtgccac 2340
tcccactgtc ctttcctaat aaaatgagga aattgcatcg cattgtctga gtaggtgtca 2400
ttctattctg gggggtgggg tggggcagga cagcaagggg gaggattggg aagacaatag 2460
caggcatgct ggggatgcgg tgggctctat ggcttctgag gcggaaagaa ccagctgggg 2520
ctctaggggg tatccccacg cgccctgtag cggcgcatta agcgcggcgg gtgtggtggt 2580
tacgcgcagc gtgaccgcta cacttgccag cgccctagcg cccgctcctt tcgctttctt 2640
cccttccttt ctcgccacgt tcgccggctt tccccgtcaa gctctaaatc ggggcatccc 2700
tttagggttc cgatttagtg ctttacggca cctcgacccc aaaaaacttg attagggtga 2760
tggttcacgt agtgggccat cgccctgata gacggttttt cgccctttga cgttggagtc 2820
cacgttcttt aatagtggac tcttgttcca aactggaaca acactcaacc ctatctcggt 2880
ctattctttt gatttataat ggttacaaat aaagcaatag catcacaaat ttcacaaata 2940
aagcattttt ttcactgcat tctagttgtg gtttgtccaa actcatcaat gtatcttatc 3000
atgtctgtat accgtcgacc tctagctaga gcttggcgta atcatggtca tagctgtttc 3060
ctgtgtgaaa ttgttatccg ctcacaattc cacacaacat acgagccgga agcataaagt 3120
gtaaagcctg gggtgcctaa tgagtgagct aactcacatt aattgcgttg cgctcactgc 3180
ccgctttcca gtcgggaaac ctgtcgtgcc agctgcatta atgaatcggc caacgcgcgg 3240
ggagaggcgg tttgcgtatt gggcgctctt ccgcttcctc gctcactgac tcgctgcgct 3300
cggtcgttcg gctgcggcga gcggtatcag ctcactcaaa ggcggtaata cggttatcca 3360
cagaatcagg ggataacgca ggaaagaaca tgtgagcaaa aggccagcaa aaggccagga 3420
accgtaaaaa ggccgcgttg ctggcgtttt tccataggct ccgcccccct gacgagcatc 3480
acaaaaatcg acgctcaagt cagaggtggc gaaacccgac aggactataa agataccagg 3540
cgtttccccc tggaagctcc ctcgtgcgct ctcctgttcc gaccctgccg cttaccggat 3600
acctgtccgc ctttctccct tcgggaagcg tggcgctttc tcaatgctca cgctgtaggt 3660
atctcagttc ggtgtaggtc gttcgctcca agctgggctg tgtgcacgaa ccccccgttc 3720
agcccgaccg ctgcgcctta tccggtaact atcgtcttga gtccaacccg gtaagacacg 3780
acttatcgcc actggcagca gccactggta acaggattag cagagcgagg tatgtaggcg 3840
gtgctacaga gttcttgaag tggtggccta actacggcta cactagaagg acagtatttg 3900
gtatctgcgc tctgctgaag ccagttacct tcggaaaaag agttggtagc tcttgatccg 3960
gcaaacaaac caccgctggt agcggtggtt tttttgtttg caagcagcag attacgcgca 4020
gaaaaaaagg atctcaagaa gatcctttga tcttttctac ggggtctgac gctcagtgga 4080
acgaaaactc acgttaaggg attttggtca tgagattatc aaaaaggatc ttcacctaga 4140
tccttttaaa ttaaaaatga agttttaaat caatctaaag tatatatgag taaacttggt 4200
ctgacagtta ccaatgctta atcagtgagg cacctatctc agcgatctgt ctatttcgtt 4260
catccatagt tgcctgactc cccgtcgtgt agataactac gatacgggag ggcttaccat 4320
ctggccccag tgctgcaatg ataccgcgag acccacgctc accggctcca gatttatcag 4380
caataaacca gccagccgga agggccgagc gcagaagtgg tcctgcaact ttatccgcct 4440
ccatccagtc tattaattgt tgccgggaag ctagagtaag tagttcgcca gttaatagtt 4500
tgcgcaacgt tgttgccatt gctacaggca tcgtggtgtc acgctcgtcg tttggtatgg 4560
cttcattcag ctccggttcc caacgatcaa ggcgagttac atgatccccc atgttgtgca 4620
aaaaagcggt tagctccttc ggtcctccga tcgttgtcag aagtaagttg gccgcagtgt 4680
tatcactcat ggttatggca gcactgcata attctcttac tgtcatgcca tccgtaagat 4740
gcttttctgt gactggtgag tactcaacca agtcattctg agaatagtgt atgcggcgac 4800
cgagttgctc ttgcccggcg tcaatacggg ataataccgc gccacatagc agaactttaa 4860
aagtgctcat cattggaaaa cgttcttcgg ggcgaaaact ctcaaggatc ttaccgctgt 4920
tgagatccag ttcgatgtaa cccactcgtg cacccaactg atcttcagca tcttttactt 4980
tcaccagcgt ttctgggtga gcaaaaacag gaaggcaaaa tgccgcaaaa aagggaataa 5040
gggcgacacg gaaatgttga atactcatac tcttcctttt tcaatattat tgaagcattt 5100
atcagggtta ttgtctcatg agcggataca tatttgaatg tatttagaaa aataaacaaa 5160
taggggttcc gcgcacattt ccccgaaaag tgccacctga cgtctc 5206
<210> 5
<211> 1275
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
atggctccta ccaaaagaca cgcgtacaac gctgagttta aactcaaggc gataagccac 60
gcacaagaac acggcaatag agcagcagcg agagaattta atatcaatga atcaatggtg 120
aggaagtgga ggaagtatga ggatgagctc cgccaagtaa agaagacaac acagagtttc 180
cgcgggaaca aagcgagatg gccacagtta gaggacaaag ttgaacagtg ggttgctgaa 240
caaagagcag caagcagaag tgttagtaca gtcacaattc gtatgaaggc aatagcgcta 300
gctcgcgaac ataacatcag tgaattcaga ggcggtcctt cttggtgctt ccgttttatg 360
aaacgacgtc atctctccat ccgtacgcgc actactgtgt cacaacaact accagctgat 420
tatcaggaaa agttggccac tttccgcaca tactgcagaa acaagataac tgaaaaaaag 480
atccagccag agcatatcat caatatggac gaggttccac tcaccttcga tatccctgta 540
aaccgcactg tggataaaac aggagcacgt acggtgaata ttcgcaccac agggaatgag 600
aaaacgtcct tcactgtagt tctcgcctgc caggctaatg gccacaaact tccacccatg 660
gttattttca agaggaagac cttgccgaaa gaaaactttc cagctggcat tgtcataaaa 720
gctaactcga agggatggat ggatgaagaa aagatgagtg agtggttgag agaaatttat 780
gtgaagagac cgggtggttt ttttcacaca gctccgtccc tattgatcta tgactccatg 840
cgcgcacata tcaccgagca tgtcaaaaaa caagtgaagc acactaattc agtgctcgcc 900
gtcattccgg gtggattaac aaaagaactc cagccgctcg atgttggcgt caacagagca 960
ttcaaagctc gactgcgaac tgcgtgggag cagtggatga ccgaaggcga acacacgttc 1020
accaagacgg ggagacagcg ccggacgaca tatgctaata tctgcaagtg gatagtaaat 1080
gcctgggctg gtatatcagt cacaactgtg gtccgagctt ttaggaaggc aggaattgtc 1140
accgaactgc cagacaacag cagcgacact gactcggtta atgatgactt tgataagacg 1200
gagccaggcg ttttggatgc cgcaatagcc cagctgttca attcggacac ggaagaagaa 1260
gttttcgagg gattt 1275
<210> 6
<211> 56
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
ggatcctacc gtattttccg cactataagg cgcacctcgc gagcggccgc gaattc 56
<210> 7
<211> 56
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
ggatcctacc gtattttccg cactataagg cgcaccgaat tcgcggccgc tcgcga 56
<210> 8
<211> 58
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
ccgtattttc cgcactataa ggcgcaccac tagtgctttt agaccttctt acttttgg 58
<210> 9
<211> 56
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
ccgtattttc cgcactataa ggcgcacctt aattaaagct tgggctgcag gtcgag 56
- 还没有人留言评论。精彩留言会获得点赞!