一种基于Y-STR基因座和Y-indel基因座的复合扩增体系及其使用的引物组合的制作方法
本发明属于法医学技术领域,具体涉及一种基于y-str基因座和y-indel基因座的复合扩增体系及其使用的引物组合,尤其涉及基于38个y-str基因座和3个y-indel基因座的复合扩增体系及其使用的引物组合。
背景技术:
短串联重复序列(shorttandemrepeat,str)属于第二代遗传标记,由于其重复单元小、突变速率高、在基因组中分布广等特点,现已广泛应用于群体遗传学及法医学领域。在人类基因组中,大约每15-20kb就有一个str位点,它们占人类基因组总量的10%。这些str通常由2-6个碱基组成串联重复单元,由于重复单元及重复次数的不同,它们在不同种族、地域的人群中显示出较大的差异性,表现为遗传多态性。
y-str遗传标记是分布于人类y染色体上的str遗传标记,是目前法庭科学领域检验鉴定的有效手段,是常染色体str及线粒体遗传标记等的重要补充。由于y染色体为男性个体所特有,其上绝大部分(95%以上)为非重组区,y-str呈现父系遗传特点,在父系遗传关系鉴定和混合斑男性个体检验等方面发挥着重要作用。同时,利用y-str在一个家系中相对保守的特点,可对重点地区的家系进行y-str分型检验,开展家系排查,从而辅助侦查人员快速锁定嫌疑人所在家系,加快案件的进程。近年来,y-str技术已帮助破获了大量命案积案。
随着y-str技术的发展,国内外y-str相关的扩增检验体系不断完善,试剂产品也不断丰富。最初,y-str扩增检测体系仅可检验几个y-str基因座。随后,试剂体系中基因座数量不断增加,检验效能不断增强,如promega公司首先推出了包含12个y-str基因座的powerplexysystem,然后研制开发了可检验23个y-str基因座的powerplexy23system;abi公司推出了包含17个y-str基因座的yfiler试剂盒,然后研制开发了可检验27个y-str基因座的yfilerplus;在国内,公安部物证鉴定中心自主研发了dnatypery系列试剂,无锡中德美联生物技术有限公司、苏州阅微基因技术有限公司等也不断推出y-str检验试剂,目前已有包含30余个y-str基因座的六色荧光扩增检测产品。上述产品可在男女成分混合或者多个男性成分混合的检材中发挥一定的鉴定作用,而且可以协助寻找犯罪嫌疑人所在家系,大大地缩小侦查范围。然而,已有试剂盒产品在基因座数量和基因座突变率方面均显示出不足,从而在一些案例应用中无法有效地区分男性个体。基于此,y-str检验试剂类产品应包含更多的基因座,尤其是应该包含公安部关于y数据库建设选用的20个y-str核心基因座(如dys19、dys385a/b、dys389i/ii、dys390、dys391、dys392、dys393、dys437、dys448、dys456、dys458、dys635、ygatah4、dys438、dys439、dys460、dys533及dys576)和15个优选基因座(如dys449、dys518、dyf387s1、dys627、dys570、dys447、dys444、dys549、dys557、dys643、dys481、dys596、dys527a/b)以及山东省公安机关关于y数据库建设选用的dys593和dys645,以获得更强的系统检验效能,在实际应用中发挥更大的作用。
技术实现要素:
本发明的目的是对男性个体进行str分型。
本发明首先保护引物组合,可包括引物1、引物2、引物3、引物4、引物5、引物6、引物7、引物8、引物9、引物10、引物11、引物12、引物13、引物14、引物15、引物16、引物17、引物18、引物19、引物20、引物21、引物22、引物23、引物24、引物25、引物26、引物27、引物28、引物29、引物30、引物31、引物32、引物33、引物34、引物35、引物36、引物37、引物38、引物39、引物40、引物41、引物42、引物43、引物44、引物45、引物46、引物47、引物48、引物49、引物50、引物51、引物52、引物53、引物54、引物55、引物56、引物57、引物58、引物59、引物60、引物61、引物62、引物63、引物64、引物65、引物66、引物67、引物68、引物69、引物70、引物71、引物72、引物73和引物74;
引物1可为seqidno:1所示的单链dna分子;
引物2可为seqidno:2所示的单链dna分子;
引物3可为seqidno:3所示的单链dna分子;
引物4可为seqidno:4所示的单链dna分子;
引物5可为seqidno:5所示的单链dna分子;
引物6可为seqidno:6所示的单链dna分子;
引物7可为seqidno:7所示的单链dna分子;
引物8可为seqidno:8所示的单链dna分子;
引物9可为seqidno:9所示的单链dna分子;
引物10可为seqidno:10所示的单链dna分子;
引物11可为seqidno:11所示的单链dna分子;
引物12可为seqidno:12所示的单链dna分子;
引物13可为seqidno:13所示的单链dna分子;
引物14可为seqidno:14所示的单链dna分子;
引物15可为seqidno:15所示的单链dna分子;
引物16可为seqidno:16所示的单链dna分子;
引物17可为seqidno:17所示的单链dna分子;
引物18可为seqidno:18所示的单链dna分子;
引物19可为seqidno:19所示的单链dna分子;
引物20可为seqidno:20所示的单链dna分子;
引物21可为seqidno:21所示的单链dna分子;
引物22可为seqidno:22所示的单链dna分子;
引物23可为seqidno:23所示的单链dna分子;
引物24可为seqidno:24所示的单链dna分子;
引物25可为seqidno:25所示的单链dna分子;
引物26可为seqidno:26所示的单链dna分子;
引物27可为seqidno:27所示的单链dna分子;
引物28可为seqidno:28所示的单链dna分子;
引物29可为seqidno:29所示的单链dna分子;
引物30可为seqidno:30所示的单链dna分子;
引物31可为seqidno:31所示的单链dna分子;
引物32可为seqidno:32所示的单链dna分子;
引物33可为seqidno:33所示的单链dna分子;
引物34可为seqidno:34所示的单链dna分子;
引物35可为seqidno:35所示的单链dna分子;
引物36可为seqidno:36所示的单链dna分子;
引物37可为seqidno:37所示的单链dna分子;
引物38可为seqidno:38所示的单链dna分子;
引物39可为seqidno:39所示的单链dna分子;
引物40可为seqidno:40所示的单链dna分子;
引物41可为seqidno:41所示的单链dna分子;
引物42可为seqidno:42所示的单链dna分子;
引物43可为seqidno:43所示的单链dna分子;
引物44可为seqidno:44所示的单链dna分子;
引物45可为seqidno:45所示的单链dna分子;
引物46可为seqidno:46所示的单链dna分子;
引物47可为seqidno:47所示的单链dna分子;
引物48可为seqidno:48所示的单链dna分子;
引物49可为seqidno:49所示的单链dna分子;
引物50可为seqidno:50所示的单链dna分子;
引物51可为seqidno:51所示的单链dna分子;
引物52可为seqidno:52所示的单链dna分子;
引物53可为seqidno:53所示的单链dna分子;
引物54可为seqidno:54所示的单链dna分子;
引物55可为seqidno:55所示的单链dna分子;
引物56可为seqidno:56所示的单链dna分子;
引物57可为seqidno:57所示的单链dna分子;
引物58可为seqidno:58所示的单链dna分子;
引物59可为seqidno:59所示的单链dna分子;
引物60可为seqidno:60所示的单链dna分子;
引物61可为seqidno:61所示的单链dna分子;
引物62可为seqidno:62所示的单链dna分子;
引物63可为seqidno:63所示的单链dna分子;
引物64可为seqidno:64所示的单链dna分子;
引物65可为seqidno:65所示的单链dna分子;
引物66可为seqidno:66所示的单链dna分子;
引物67可为seqidno:67所示的单链dna分子;
引物68可为seqidno:68所示的单链dna分子;
引物69可为seqidno:69所示的单链dna分子;
引物70可为seqidno:70所示的单链dna分子;
引物71可为seqidno:71所示的单链dna分子;
引物72可为seqidno:72所示的单链dna分子;
引物73可为seqidno:73所示的单链dna分子;
引物74可为seqidno:74所示的单链dna分子。
所述引物组合具体可由引物1、引物2、引物3、引物4、引物5、引物6、引物7、引物8、引物9、引物10、引物11、引物12、引物13、引物14、引物15、引物16、引物17、引物18、引物19、引物20、引物21、引物22、引物23、引物24、引物25、引物26、引物27、引物28、引物29、引物30、引物31、引物32、引物33、引物34、引物35、引物36、引物37、引物38、引物39、引物40、引物41、引物42、引物43、引物44、引物45、引物46、引物47、引物48、引物49、引物50、引物51、引物52、引物53、引物54、引物55、引物56、引物57、引物58、引物59、引物60、引物61、引物62、引物63、引物64、引物65、引物66、引物67、引物68、引物69、引物70、引物71、引物72、引物73和引物74组成。
上述任一所述的引物组合中,引物1、引物2、引物3、引物4、引物5、引物6、引物7、引物8、引物9、引物10、引物11、引物12、引物13、引物14、引物15、引物16、引物17、引物18、引物19、引物20、引物21、引物22、引物23、引物24、引物25、引物26、引物27、引物28、引物29、引物30、引物31、引物32、引物33、引物34、引物35、引物36、引物37、引物38、引物39、引物40、引物41、引物42、引物43、引物44、引物45、引物46、引物47、引物48、引物49、引物50、引物51、引物52、引物53、引物54、引物55、引物56、引物57、引物58、引物59、引物60、引物61、引物62、引物63、引物64、引物65、引物66、引物67、引物68、引物69、引物70、引物71、引物72、引物73和引物74的摩尔比可为150:150:70:70:140:140:180:180:170:170:300:300:160:160:130:130:130:130:120:120:80:80:90:90:140:140:180:180:100:100:150:150:180:180:210:210:120:120:200:200:210:210:160:160:400:400:180:180:120:120:120:120:200:200:200:200:250:250:400:400:150:150:230:230:130:130:190:190:210:210:140:140:300:300。
上述任一所述的引物组合中,引物1、引物3、引物5、引物7、引物9、引物11、引物13、引物15、引物17、引物19、引物21、引物23、引物25、引物27、引物29、引物31、引物33、引物35、引物37、引物39、引物41、引物43、引物45、引物47、引物49、引物51、引物53、引物55、引物57、引物59、引物61、引物63、引物65、引物67、引物69、引物71和引物73均可用荧光标记。
上述任一所述的引物组合中,引物1、引物3、引物5、引物7、引物9、引物11、引物13、引物15和引物17可用fam标记。引物19、引物21、引物23、引物25、引物27、引物29和引物31可用hex标记。引物33、引物35、引物37、引物39、引物41、引物43和引物45可用tamra标记。引物47、引物49、引物51、引物53、引物55、引物57和引物59可用rox标记。引物61、引物63、引物65、引物67、引物69、引物71和引物73可用sid标记。引物1、引物3、引物5、引物7、引物9、引物11、引物13、引物15和引物17的5’末端可用fam标记。引物19、引物21、引物23、引物25、引物27、引物29和引物31的5’末端可用hex标记。引物33、引物35、引物37、引物39、引物41、引物43和引物45的5’末端可用tamra标记。引物47、引物49、引物51、引物53、引物55、引物57和引物59的5’末端可用rox标记。引物61、引物63、引物65、引物67、引物69、引物71和引物73的5’末端可用sid标记。
本发明还保护一种基于y-str基因座和y-indels基因座的复合扩增体系,可包括上述任一所述引物组合;
所述y-str基因座可包括dys19、dys385a/b、dys389i/ii、dys390、dys391、dys392、dys393、dys437、dys448、dys456、dys458、dys635、y_gata_h4、dys438、dys439、dys460、dys533、dys576、dys449、dys518、dyf387s1、dys627、dys570、dys447、dys444、dys549、dys557、dys643、dys481、dys596、dys527a/b、dys508、dys593和dys645;
所述y-indels基因座可包括rs771783753、rs199815934和rs759551978。
所述y-str基因座具体可由dys19、dys385a/b、dys389i/ii、dys390、dys391、dys392、dys393、dys437、dys448、dys456、dys458、dys635、y_gata_h4、dys438、dys439、dys460、dys533、dys576、dys449、dys518、dyf387s1、dys627、dys570、dys447、dys444、dys549、dys557、dys643、dys481、dys596、dys527a/b、dys508、dys593和dys645组成。
所述y-indels基因座具体可由rs771783753、rs199815934和rs759551978组成。
dys576、dys449、dys518、dyf387s1、dys627和dys570为快速突变y-str基因座。
上述41个基因座(即38个y-str基因座和3个y-indels基因座)中包含了公安部关于y数据库建设选用的20个y-str核心基因座和15个优选基因座,还包括了山东省公安机关关于y数据库建设选用的dys593和dys645。
所述复合扩增体系具体可由上述任一所述引物组合组成。
上述任一所述复合扩增体系中,引物1、引物2、引物31、引物32、引物61和引物62在所述复合扩增体系中的浓度均可为150mm。引物3和引物4在所述复合扩增体系中的浓度均可为70mm。引物5、引物6、引物25、引物26、引物71和引物72在所述复合扩增体系中的浓度均可为140mm。引物7、引物8、引物27、引物28、引物33、引物34、引物47和引物48在所述复合扩增体系中的浓度均可为180mm。引物9和引物10在所述复合扩增体系中的浓度均可为170mm。引物11、引物12、引物73和引物74在所述复合扩增体系中的浓度均可为300mm。引物13、引物14、引物43和引物44在所述复合扩增体系中的浓度均可为160mm。引物15、引物16、引物17、引物18、引物65和引物66在所述复合扩增体系中的浓度均可为130mm。引物19、引物20、引物37、引物38、引物49、引物50、引物51和引物52在所述复合扩增体系中的浓度均可为120mm。引物21和引物22在所述复合扩增体系中的浓度均可为80mm。引物23和引物24在所述复合扩增体系中的浓度均可为90mm。引物29和引物30在所述复合扩增体系中的浓度均可为100mm。引物35、引物36、引物41、引物42、引物69和引物70在所述复合扩增体系中的浓度均可为210mm。引物39、引物40、引物53、引物54、引物55和引物56在所述复合扩增体系中的浓度均可为200mm。引物45、引物46、引物59和引物60在所述复合扩增体系中的浓度均可为400mm。引物57和引物58在所述复合扩增体系中的浓度均可为250mm。引物63和引物64在所述复合扩增体系中的浓度均可为230mm。引物67和引物68在所述复合扩增体系中的浓度均可为190mm。
上述复合扩增体系中,所述复合扩增体系还可包括进行pcr扩增反应所需的试剂;所述“进行pcr扩增反应所需的试剂”不包括pcr扩增反应所需的引物。
所述“进行pcr扩增反应所需的试剂”可包括dna聚合酶、dntp、mg2+、bsa、kcl、tris中的至少一种。
所述dna聚合酶在所述复合扩增体系中的浓度可为0.1u/μl。所述dntp在所述复合扩增体系中的浓度可为200μm(即datp、dttp、dctp和dgtp的浓度为200μm)。所述mg2+在所述复合扩增体系中的浓度可为1.5mm。所述bsa在所述复合扩增体系中的浓度可为0.8mg/ml。所述kcl在所述复合扩增体系中的浓度可为50mm。所述tris在所述复合扩增体系中的浓度可为10mm。
所述复合扩增体系具体可由上述任一所述引物组合和进行pcr扩增反应所需的试剂组成。
含有上述任一所述引物组合的试剂盒也属于本发明的保护范围。所述试剂盒用于男性个体的str分型。
上述任一所述复合扩增体系或含有上述任一所述引物组合的试剂盒的制备方法也属于本发明的保护范围。
上述任一所述复合扩增体系或含有上述任一所述引物组合的试剂盒的制备方法可包括将上述任一所述引物组合中的各条引物单独包装的步骤。
下述x1)或x2)也属于本发明的保护范围。
x1)上述任一所述引物组合或上述任一所述复合扩增体系,在制备用于男性个体的str分型的试剂盒中的应用。
x2)上述任一所述引物组合或上述任一所述复合扩增体系,在男性个体的str分型中的应用。
上述任一所述str分型具体可为y-str分型。
上述任一所述男性个体可为中国男性个体。
采用本发明提供的复合扩增体系对样本一(9948人类基因组dna标准品)或样本二(人(已知为男性)的血液采集卡)进行str分型。结果表明,样本一和样本二均得到了完整的str分型,并且峰型尖锐清晰,平衡性好,无pull-up峰、stutter带,无非特异性扩增产物出现。9948人类基因组dna标准品的结果还表明,该复合扩增体系对表1所示的38个y-str基因座和3个y-indels基因座的分型结果正确,完全能够满足法医y-str检验的要求。由此可见,本发明提供的复合扩增体系可用于扩增38个y-str基因座(其中7个为快速突变y-str基因座)和3个y-indels基因座,该复合扩增体系一次扩增可最多获得41个不同的等位基因片段,扩增效率非常高。同时,该复合扩增体系中包含7个快速突变的y-str基因座,检验效能也非常强。本发明提供的复合扩增体系在父系亲缘关系鉴定、家系排查等方面具有重要的应用价值。
附图说明
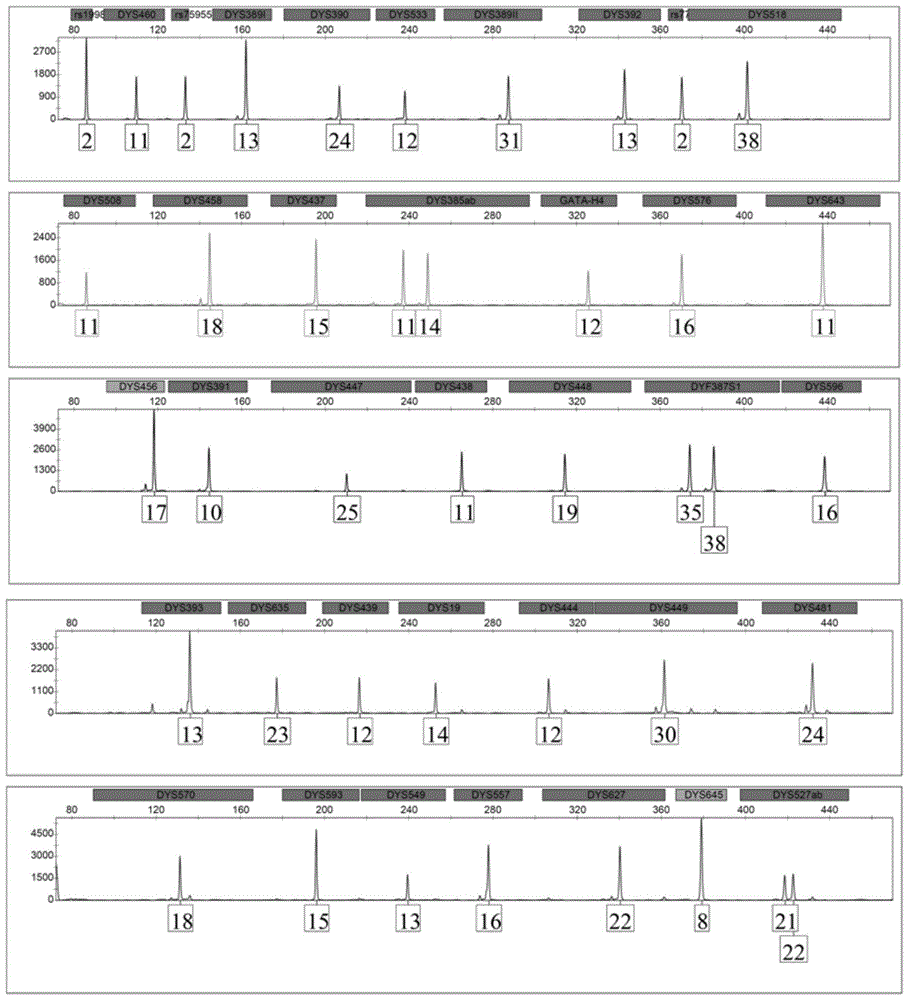
图1为样本一的dna检测图谱。
图2为样本二的dna检测图谱。
具体实施方式
下面结合具体实施方式对本发明进行进一步的详细描述,给出的实施例仅为了阐明本发明,而不是为了限制本发明的范围。
下述实施例中的实验方法,如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1、基于38个y-str基因座和3个y-indel基因座的复合扩增体系的制备
一、y-str基因座和y-indel基因座的筛选
本发明的发明人通过查阅文献获得大量候选的y-str基因座和y-indel基因座;然后应用人类基因组计划的研究成果,在ucsc网站(网址为:http://genome.ucsc.edu/)利用已报道的各基因座引物信息进行检索,获得相应的序列;将序列通过blast(网址为:https://blast.ncbi.nlm.nih.gov/blast.cgi)在genbank中进行检索查询,然后对现有的y-str基因座和y-indel基因座的核心及侧翼区序列信息、等位基因长度及分布等进行整理和系统研究,重点关注核心序列为4-5碱基重复的y-str基因座和插入片段不小于3bp的y-indel基因座。综合考虑各基因座的拷贝数、突变率、重复单元特征、序列结构复杂程度等技术指标,以及与其它商品试剂盒的兼容性等,最终筛选获得38个y-str基因座和3个y-indel基因座。
38个y-str基因座分别为:dys19、dys385a/b、dys389i/ii、dys390、dys391、dys392、dys393、dys437、dys448、dys456、dys458、dys635、y_gata_h4、dys438、dys439、dys460、dys533、dys576、dys449、dys518、dyf387s1、dys627、dys570、dys447、dys444、dys549、dys557、dys643、dys481、dys596、dys527a/b、dys508、dys593、dys645。其中,dys576、dys449、dys518、dyf387s1、dys627和dys570为快速突变y-str基因座。
3个y-indel基因座分别为:rs771783753、rs199815934和rs759551978。
需要说明的是,dys385a/b、dys527a/b和dyf387s1的扩增片段长度没有明显差异,习惯上以a/b加以区分,即dys385a/b分为dys385a和dys385b,dys527a/b分为dys527a和dys527b,dyf387s1分为dyf387s1a和dyf387s1b;dys389i/ii的扩增片段长度有明显差异,习惯上以i/ii加以区分,即dys389i/ii分为dys389i和dys389ii。
上述41个基因座中包含了公安部关于y数据库建设选用的20个y-str核心基因座和15个优选基因座,还包括了山东省公安机关关于y数据库建设选用的dys593和dys645。
二、基于38个y-str基因座和3个y-indels基因座的复合扩增体系的制备
1、根据各荧光染料发射波长的范围、荧光信号强弱等初步筛选出适合的荧光染料,而后通过大量实验,测试荧光染料的稳定性,最终获得互不干扰、品质稳定的六色荧光染料,实现六色荧光检测。
2、依据各基因座等位基因组成、范围及扩增片段长度,合理选取不同颜色的荧光标记,以区分位于片段重叠区域的各个基因座,然后制备基于38个y-str基因座和3个y-indels基因座的复合扩增体系。具体步骤如下:
(1)人工合成用于扩增每个基因座的引物,然后进行pcr扩增,获得每个基因座的特异性引物。
(2)综合单个基因座的扩增条件,选择适宜扩增程序,先将同一个颜色荧光标记的引物复合扩增,确认之后,再将不同颜色荧光标记的引物复合扩增。复合扩增时,观察各个基因座是否均有特异性产物和非特异产物的出现。如果不同基因座引物之间产生出现非特异的杂峰,则需要一一排除,找出这些基因座,重新设计并合成引物。此外,不同基因座的引物之间还会形成引物二聚体,降低引物的扩增效率,也需要重新设计并合成引物。
(3)反复测试、修改复合扩增体系,用复合扩增体系对男性的基因组dna进行扩增,观察各基因座之间是否会有重叠,因为扩增产物是根据理论大小设计的,实际过程中会因为扩增产物的序列结构不一致而造成迁移速率偏快或偏慢,导致理论大小与实际大小不符,经常导致相邻基因座之间发生重叠或者距离过于接近,影响后续分析。一旦发生此类现象,则需要重新设计该基因座引物,再经历步骤(1)和(2),最终获得满足条件的复合扩增体系。
(4)复合扩增体系的调平。具体为:初次复合扩增时,各个引物在反应体系中的浓度均为100mm;根据反应结果调整各个引物对的浓度。
(5)优化复合扩增反应参数。用9948人类基因组dna标准品对反应体系中的关键组分和环节进行调整,如扩增循环数、退火温度、延伸时间、热启动dna聚合酶用量、引物量、缓冲液浓度等等。
经过上述步骤,获得基于38个y-str基因座和3个y-indels基因座的复合扩增体系。该复合扩增体系由dna聚合酶、dntp、mg2+、bsa、kcl、tris和引物混合物组成;引物混合物由74条引物混合而成;74条引物的名称、核苷酸序列、等位基因和扩增的基因座等信息详见表1中第1-5列。该复合扩增体系中,dna聚合酶的浓度为0.1u/μl,dntp的浓度为200μm(即datp、dttp、dctp和dgtp的浓度均为200μm),mg2+的浓度为1.5mm,bsa的浓度为0.8mg/ml,kcl的浓度为50mm,tris的浓度为10mm,74条引物的浓度见表1中第6列。
表1
注:fam表示5’末端进行fam标记;hex表示5’末端进行hex标记;tamra表示5’末端进行tamra标记;rox表示5’末端进行rox标记,sid表示5’末端进行sid标记。
3、制备上述基于38个y-str基因座和3个y-indels基因座的复合扩增体系。
实施例2、基于38个y-str基因座和3个y-indel基因座的复合扩增体系的应用
typer500内标为公安部物证鉴定中心的产品。去离子甲酰胺为abi公司的产品,产品目录号为4311320。abi3130xl遗传分析仪为abi公司的产品。人(已知为男性)的血液采集卡由公安部物证鉴定中心日常dna数据库建设所提供。
待测样本为样本一或样本二:
样本一、1ng9948人类基因组dna标准品;
样本二:取人(已知为男性)的血液采集卡,用0.5mm手持式打孔器打下一片圆片。
1、取10μl实施例1步骤二中3制备的复合扩增体系,加入待测样本进行pcr扩增,得到pcr扩增产物。
反应程序:95℃预变性11min;94℃变性30s,59℃退火2min,72℃延伸1min,28次循环;60℃延伸60min;4℃保存。
2、完成步骤1后,将10μl上样混合物和1μlpcr扩增产物混合均匀,得到反应液。
上样混合物由10μltyper500内标和1000μl去离子甲酰胺混合而成。
3、完成步骤2后,取反应液,95℃变性5min,迅速转移至-20℃放置5min,然后用abi3130xl遗传分析仪进行毛细管电泳检测,得到dna检测图谱。电泳条件为:进样电压1.2kv,进样时间为30s。
样本一的实验结果见图1(rs77为rs771783753,rs1998为rs199815934,rs75955为rs759551978)。
样本二的实验结果见图2(rs77为rs771783753,rs1998为rs199815934,rs75955为rs759551978)。
结果表明,样本一和样本二均得到了完整的str分型,并且峰型尖锐清晰,平衡性好,无pull-up峰、stutter带,无非特异性扩增产物出现。9948人类基因组dna标准品的结果还表明,实施例1制备的复合扩增体系对表1所示的38个y-str基因座和3个y-indels基因座的分型结果正确,完全能够满足法医y-str检验的要求。
本发明提供的复合扩增体系可用于扩增表1所示的38个y-str基因座(其中7个为快速突变y-str基因座)和3个y-indel基因座,该复合扩增体系一次扩增可最多获得41个不同的等位基因片段,扩增效率非常高。同时,该复合扩增体系中包含7个快速突变的y-str基因座,检验效能也非常强。本发明提供的复合扩增体系将进一步推动y-str在父系亲缘关系鉴定、家系排查等方面的应用。
<110>公安部物证鉴定中心
<120>一种基于y-str基因座和y-indel基因座的复合扩增体系及其使用的引物组合
<160>74
<170>patentinversion3.5
<210>1
<211>21
<212>dna
<213>artificialsequence
<400>1
aatcaactcaactccagtgat21
<210>2
<211>23
<212>dna
<213>artificialsequence
<400>2
ctgatacctttgtttctgttcat23
<210>3
<211>20
<212>dna
<213>artificialsequence
<400>3
agcaagcacaagaataccag20
<210>4
<211>22
<212>dna
<213>artificialsequence
<400>4
cctctgcctatcatttattatg22
<210>5
<211>23
<212>dna
<213>artificialsequence
<400>5
gaaattattttttcagaaagcag23
<210>6
<211>24
<212>dna
<213>artificialsequence
<400>6
atcaaggctattaatcaaaaatac24
<210>7
<211>27
<212>dna
<213>artificialsequence
<400>7
tgtatccaactctcatctgtattatct27
<210>8
<211>23
<212>dna
<213>artificialsequence
<400>8
cgaattatccctgagtagcagaa23
<210>9
<211>22
<212>dna
<213>artificialsequence
<400>9
ccctgcattttggtaccccata22
<210>10
<211>22
<212>dna
<213>artificialsequence
<400>10
gagtgggagaaatggatgacag22
<210>11
<211>24
<212>dna
<213>artificialsequence
<400>11
tctatatctgtctattcatctaac24
<210>12
<211>22
<212>dna
<213>artificialsequence
<400>12
tgtatttattcatgatcagttc22
<210>13
<211>24
<212>dna
<213>artificialsequence
<400>13
ttgatgcaatgtaaattcctacag24
<210>14
<211>22
<212>dna
<213>artificialsequence
<400>14
catccatgttgctccaaaggac22
<210>15
<211>22
<212>dna
<213>artificialsequence
<400>15
ggaggcttgtcactaatttttg22
<210>16
<211>22
<212>dna
<213>artificialsequence
<400>16
ggatatctctcattagatattc22
<210>17
<211>24
<212>dna
<213>artificialsequence
<400>17
actccagcctgggcaacacaagtg24
<210>18
<211>22
<212>dna
<213>artificialsequence
<400>18
tccaaagtgctttgtggtgtgc22
<210>19
<211>21
<212>dna
<213>artificialsequence
<400>19
gccagcttattattccttctc21
<210>20
<211>22
<212>dna
<213>artificialsequence
<400>20
gatatgcagagagtagatatag22
<210>21
<211>22
<212>dna
<213>artificialsequence
<400>21
gactgagcaacaggaatgaaac22
<210>22
<211>21
<212>dna
<213>artificialsequence
<400>22
cattacaagcatgagccacca21
<210>23
<211>21
<212>dna
<213>artificialsequence
<400>23
ttgagtgcatgcccatccggt21
<210>24
<211>23
<212>dna
<213>artificialsequence
<400>24
cagcctgaggaacagaggaagac23
<210>25
<211>21
<212>dna
<213>artificialsequence
<400>25
gacaccatgccaaacaacaac21
<210>26
<211>22
<212>dna
<213>artificialsequence
<400>26
gtatctattccaattacatagt22
<210>27
<211>22
<212>dna
<213>artificialsequence
<400>27
cagcacatcacttgtatcctag22
<210>28
<211>22
<212>dna
<213>artificialsequence
<400>28
gaccatttcctctgatggtgaa22
<210>29
<211>22
<212>dna
<213>artificialsequence
<400>29
ggtcatgccagtaatcccagta22
<210>30
<211>21
<212>dna
<213>artificialsequence
<400>30
ctatcataagaacagcatggg21
<210>31
<211>23
<212>dna
<213>artificialsequence
<400>31
gctgcaccagtcactgtatttgc23
<210>32
<211>23
<212>dna
<213>artificialsequence
<400>32
ggcaaaagcataggaatgacaca23
<210>33
<211>22
<212>dna
<213>artificialsequence
<400>33
ggtctgttgtgggaccttgtga22
<210>34
<211>21
<212>dna
<213>artificialsequence
<400>34
ggacagaactaatggaatatc21
<210>35
<211>23
<212>dna
<213>artificialsequence
<400>35
cttgtgtatctattcattcaatc23
<210>36
<211>21
<212>dna
<213>artificialsequence
<400>36
cctggttgcatgcaattgcca21
<210>37
<211>23
<212>dna
<213>artificialsequence
<400>37
gcattgcgttatctctgcctttc23
<210>38
<211>22
<212>dna
<213>artificialsequence
<400>38
gcatggcttggttttatacatt22
<210>39
<211>22
<212>dna
<213>artificialsequence
<400>39
gagtaacaccattagagcatca22
<210>40
<211>23
<212>dna
<213>artificialsequence
<400>40
taggctgatgcaggagaatcgct23
<210>41
<211>21
<212>dna
<213>artificialsequence
<400>41
tatgggagaggcaaggatcca21
<210>42
<211>21
<212>dna
<213>artificialsequence
<400>42
ctcatatttctggccggtctg21
<210>43
<211>22
<212>dna
<213>artificialsequence
<400>43
gtgaagtttgcagtgagctgag22
<210>44
<211>22
<212>dna
<213>artificialsequence
<400>44
actggagtctgtaagtggctca22
<210>45
<211>22
<212>dna
<213>artificialsequence
<400>45
gtctgttccaagtggttcacag22
<210>46
<211>23
<212>dna
<213>artificialsequence
<400>46
gctagtgagagaaagtctccaat23
<210>47
<211>22
<212>dna
<213>artificialsequence
<400>47
ggttgtcattcctaatgtggtc22
<210>48
<211>22
<212>dna
<213>artificialsequence
<400>48
ccaaaaaatgaggtatgtctca22
<210>49
<211>23
<212>dna
<213>artificialsequence
<400>49
tcttggcttctcactttgcatag23
<210>50
<211>24
<212>dna
<213>artificialsequence
<400>50
tgtggaaccagcccaaatatccat24
<210>51
<211>23
<212>dna
<213>artificialsequence
<400>51
ggtgttatggttttaggtctaac23
<210>52
<211>22
<212>dna
<213>artificialsequence
<400>52
cctggcttggaattcttttacc22
<210>53
<211>21
<212>dna
<213>artificialsequence
<400>53
ctgcacctggaaatagtggct21
<210>54
<211>23
<212>dna
<213>artificialsequence
<400>54
tcactatgactactgagtttctg23
<210>55
<211>22
<212>dna
<213>artificialsequence
<400>55
ggtgtgaaccatttggcatgtt22
<210>56
<211>22
<212>dna
<213>artificialsequence
<400>56
ctggctgtctaagggatccaaa22
<210>57
<211>22
<212>dna
<213>artificialsequence
<400>57
ggctagagattcttggagtctc22
<210>58
<211>23
<212>dna
<213>artificialsequence
<400>58
gctgtaagctaagattgcaccat23
<210>59
<211>22
<212>dna
<213>artificialsequence
<400>59
gggtgagtcttttggaaacttg22
<210>60
<211>21
<212>dna
<213>artificialsequence
<400>60
cattgcagattcttggtccac21
<210>61
<211>21
<212>dna
<213>artificialsequence
<400>61
atcctggctgtgtcctccaag21
<210>62
<211>22
<212>dna
<213>artificialsequence
<400>62
aatgcagatattccctatctac22
<210>63
<211>23
<212>dna
<213>artificialsequence
<400>63
cacttgaacccaggaagcagacc23
<210>64
<211>23
<212>dna
<213>artificialsequence
<400>64
aaccacttgatttaaggcatcct23
<210>65
<211>23
<212>dna
<213>artificialsequence
<400>65
gtactgaatgacattaagatgta23
<210>66
<211>22
<212>dna
<213>artificialsequence
<400>66
gcataagtggtaatgtcccctt22
<210>67
<211>23
<212>dna
<213>artificialsequence
<400>67
cctactggcaggtgtgattgtga23
<210>68
<211>23
<212>dna
<213>artificialsequence
<400>68
gtgtttgacccagtgatatgttc23
<210>69
<211>21
<212>dna
<213>artificialsequence
<400>69
gacagcgcaggattccatcta21
<210>70
<211>23
<212>dna
<213>artificialsequence
<400>70
accatgtggataatgagcaaatg23
<210>71
<211>21
<212>dna
<213>artificialsequence
<400>71
ggtggcaatcatagtccatat21
<210>72
<211>22
<212>dna
<213>artificialsequence
<400>72
cccataaattccatccaagctc22
<210>73
<211>22
<212>dna
<213>artificialsequence
<400>73
agccacaacataagtaaggtag22
<210>74
<211>22
<212>dna
<213>artificialsequence
<400>74
cagctacttgggaggataaggc22
- 还没有人留言评论。精彩留言会获得点赞!