针对特异表位的抗基孔肯雅热的全人源单克隆抗体及应用的制作方法
本发明公开了一种抗体,属于微生物学和免疫学领域。
背景技术:
基孔肯雅热(chikungunyafever)是由基孔肯雅病毒(chikungunyavirus)引起的一种蚊媒传染病,最初流行于非洲的热带和亚热带地区,并不断扩展到南亚、东南亚、印度洋岛屿及美洲地区。基孔肯雅热的主要临床体征是发热、头痛、肌肉痛、皮疹和关节疼痛。在过去的十几年中,基孔肯雅热的爆发次数增多,流行范围不断扩大,已经在全球100多个国家和地区发生,造成全球范围内每年大约100万病例。2015年12月20日,世界卫生组织基于埃博拉疫情应对的教训,确定了可能导致严重疫情暴发的八种危险病原体(埃博拉、马尔堡病毒等)及三种次危险病原体(基孔肯雅热、血小板减少综合症伴严重发热和寨卡黄病毒),基孔肯雅病毒是三种次危险病原体之一。同时基孔肯雅病毒被美国过敏与感染性疾病研究所列为c类生物防御病原体。美国陆军认为基孔肯雅病毒是一种潜在的生物战剂/生物恐怖剂,由于其可以潜在的通过气溶胶感染。
目前,基孔肯雅热的治疗主要是退烧、镇痛等对症治疗,临床上没有特异性的治疗药物。埃及伊蚊和白纹伊蚊在中国广泛分布,且中国与一些基孔肯雅热流行国家人员往来密切,存在基孔肯雅病毒传播的风险。
文献报道抗体具有治疗效果:(1)多抗:法国巴斯德研究所从基孔肯雅病毒感染恢复期患者分离到多价抗体,该抗体表现出体外很强的中和活性,在动物模型具有有效的保护作用。(2)鼠单克隆抗体:美国圣路易斯华盛顿大学研发的四种鼠抗体chk-102,chk-152,chk-166,chk-263在小鼠试验中有效。抗体针对e1和e2糖蛋白,其中最有效的抗体chk-152进行了人源化,并且表现出与鼠抗体相似的中和特性。(3)人单克隆抗体:新加坡科技研究局从感染基孔肯雅病毒的患者分离出两种针对病毒包膜糖蛋白的人单克隆抗体5f10和8b10,对不同的基孔肯雅病毒株具有中和作用,预防性实验中在试验小鼠可100%发挥保护作用。美国加利福尼亚大学研发的一种单克隆抗体mabc9同样分离于感染基孔肯雅病毒的患者,其针对基孔肯雅病毒e2糖蛋白,在感染1天前小鼠给药可预防病毒血症和关节痛的发生。
多克隆抗体存在抗体增强的毒性效应,鼠单抗应用于人体,具有免疫原性且产生抗鼠抗体,需要进行免疫原性改造,人源单抗是最优选的治疗策略。目前获取人源单抗的技术包括噬菌体文库技术、单细胞pcr技术,相比噬菌体文库技术,单细胞pcr技术获得的单抗是天然配对抗体,稳定性更好。单细胞pcr技术可用于从灭活疫苗、减毒活疫苗免疫后的志愿者和天然感染者中筛选获得,天然感染者是更优选策略,因为病毒在体内持续复制和刺激,生成的抗体是天然的。us9738704曾公开了一株抗基孔肯雅病毒的单克隆抗体,所述抗体的ic50达到了2.17ug/ml。我们采用单细胞pcr技术从基孔肯雅病毒感染者恢复后的外周血中获得具有显著中和保护活性的单抗。
本发明拟采用单细胞pcr技术从基孔肯雅热感染者恢复后的外周血中获得具有更为优异的中和保护活性的单抗,用于基孔肯雅热的紧急治疗和预防。
技术实现要素:
本发明通过流式分选-单细胞pcr技术筛选到了一种针对特异表位的抗基孔肯雅病毒的单克隆抗体,所述单克隆抗体重链可变区cdr1、cdr2和cdr3区的氨基酸序列分别如seqidno:1的第26-33、51-58、97-109位序列所示;所述单克隆抗体轻链可变区cdr1、cdr2和cdr3区氨基酸序列分别如seqidno:5第27-32、50-52、88-93位序列所示。
在一个优选的技术方案中,所述单克隆抗体重链可变区氨基酸序列如seqidno:1所示,所述单克隆抗体轻链可变区的氨基酸序列如seqidno:5所示。
在一个更为优选的技术方案中,所述单克隆抗体重链恒定区的氨基酸序列如seqidno:3所示,所述单克隆抗体轻链恒定区的氨基酸序列如seqidno:7或seqidno:9所示。本发明一个优选的单克隆抗体被命名为12h9。
第二,本发明还提供了一种编码上述单克隆抗体重链和/或轻链的多核苷酸,所述多核苷酸中编码抗体重链可变区的序列如seqidno:2所示,编码抗体轻链可变区的序列如seqidno:6所示。
在一个优选的技术方案中,编码所述单克隆抗体重链恒定区的多核苷酸的序列如seqidno:4所示,编码所述单克隆抗体轻链恒定区的多核苷酸的序列如seqidno:8或者seqidno:10所示。
第三,本发明还提供了一种表达上述编码单克隆抗体重链和/或轻链的多核苷酸的功能元件。这种功能元件可以是传统的表达载体。
在一个优选的技术方案中,所述功能元件为线性表达框。
在另一个优选的技术方案中,所述功能元件为哺乳动物表达载体。
第四,本发明还提供了一种含有上述功能元件的宿主细胞。
在一个优选的技术方案中,所述细胞为expi293f细胞。
在另一个优选的技术方案中,所述细胞为cho-s细胞。
最后,本发明提供了上述单克隆抗体在制备基孔肯雅热治疗药物中的应用。
本发明提供的单克隆抗体显示出对基孔肯雅病毒感染细胞良好的中和保护效果。在细胞模型上,中和活性是文献报道的对照抗体(参考美国专利us9738704公开的8b10f8重链和轻链可变区序列,化学合成可变区基因并利用融合pcr技术获得全长抗体基因,克隆到表达载体pmh载体上,转染cho-s细胞,通过proteina亲和层析纯化获得并保存,本发明命名为ab2)的8倍。研究结果显示,本发明提供的抗体具有在制备基孔肯雅热治疗药物中广泛应用的前景。本发明公开的单克隆抗体还具有以下的技术优势:(1)全人源,在临床应用上,不需要进行人源化改造以降低人抗鼠抗体反应(hama反应),即低免疫原性。(2)高表达,在瞬时表达系统中的表达量接近0.3g/l,方便以后进行产业化。(3)单抗识别的表位是构象表位,识别的靶蛋白是e2蛋白,可以与已报道的人源单抗联合应用,避免耐药毒株的产生和提高疗效。(4)稳定性好,因为抗体基因来自人体的同一个细胞,是天然配对的,已知人体内igg1抗体的半衰期是21-28天,理论上公开的单抗具有一致的人体内半衰期。
附图说明
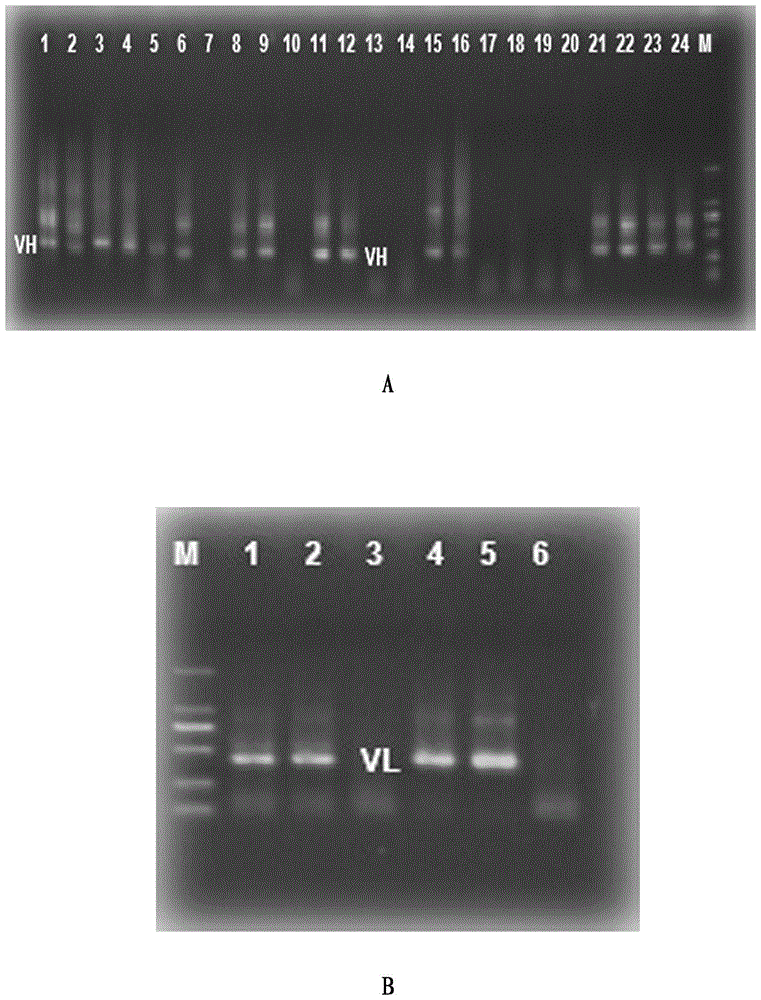
图1.重链和轻链可变区基因的pcr扩增核酸电泳检测;
图2.重链和轻链线性表达框的核酸电泳检测;
图3.表达载体的酶切鉴定核酸电泳检测;
图4.瞬时转染后的单抗表达量分析;
图5.亲和层析纯化后的单抗sds-page检测;
图6.单抗在细胞模型上的ic50测定;
图7.单抗在细胞模型上的保护活性检测;
图8.表位与基孔肯雅病毒结构蛋白和非结构蛋白的比对结果;
图9.镍柱亲和层析纯化后的表位融合蛋白sds-page检测;
图10.elisa检测表位融合蛋白与单抗的结合活性。
具体实施方式
下面结合具体实施例来进一步描述本发明,本发明的优点和特点将会随着描述而更为清楚。但这些实施例仅是范例性的,并不对本发明权利要求所限定的范围构成进一步的限定。
实施例1人源抗基孔肯雅病毒单克隆抗体的筛选和制备
1.1血液样品采集
在获得知情同意后,广州第八人民医院提供1名基孔肯雅热感染者恢复后11天的外周血,用于后续实验。
1.2流式细胞仪分选单细胞
将采集的血样利用ficoll密度梯度离心法分离pbmc,过程如下:
1.2.1取新鲜抗凝全血(edta抗凝)用等体积pbs稀释全血。
1.2.2在离心管中加入一定体积的分离液,将稀释后的血样平铺到分离液液面上方,保持两液面界面清晰。分离液、抗凝未经稀释全血、pbs(或生理盐水)体积为1:1:1。
1.2.3配平,室温,水平转子700-800g(2000-2500rpm),加速度3~4acc,离心20-30min。
1.2.4离心结束后,管底是红细胞,中间层是分离液,最上层是血浆/组织匀浆层,血浆层与分离液层之间是一层薄且较致密的白膜,即:单个核细胞(包括淋巴细胞和单核细胞)层。小心吸取白膜层到另一离心管中。
1.2.5用pbs/1640稀释到一定体积,颠倒混匀。室温,水平转子250-400g(1000-1500rpm),离心5-10min,弃上清。重复洗涤1-2次。
1.2.6用pbs或合适的培养基将淋巴细胞重悬备用。
将pbmc使用流式抗体染色:anti-cd3-fitc,anti-cd20-fitc,anti-cd19-pe-alexa610,anti-cd27-pe,anti-cd38-pe-cy5,每5×106个pbmc细胞各加入5μl上述抗体,孵育30分钟后,使用含2%fbs的pbs重复洗涤2-3次。利用mofloxdp流式分选细胞仪,选择浆细胞特异的细胞表面标记物(cd3neg/cd20low/cd19high/cd27high/cd38high)分选浆细胞,直接将单个浆细胞分选至96孔板中,每孔中预先加入含有5urna酶抑制剂及19.8μl去rna酶的水,-80℃冻存备用。
1.3利用单细胞-pcr技术扩增全人源单抗可变区基因
1.3.1反转录pcr
具体参考说明书(qiagen,210212),程序简单介绍如下:
通过流式细胞仪分选了1152(共12块细胞板,每块96个细胞)个单细胞,因此有1152个反转录反应体系。向每个反应体系中同时加入以下全部的针对重链(heavychain,h)、kappa轻链(kappachain,κ)、lamda轻链(lamdachain,λ)各亚型的特异引物(引物序列见表1)。
引物:
h:5′l-vh1、l-vh3、l-vh4/6,5′l-vh5、huigg-const-anti、3′cμch1;
κ:5′lvκ1/2、5′lvκ3、5′lvκ4、3′cκ543–566;
λ:5′lvλ1、5′lvλ2、5′lvλ3、5′lvλ4/5、5′lvλ6、5′lvλ7、5′lvλ8、3′cλ。
表1反转录pcr引物
pcr反应体系中包含:5×缓冲液6μl、dntp1.2μl、反转录酶(全式金生物技术有限公司,at311)1.2μl、引物如上、模板为单细胞,水补齐至30μl。
pcr反应条件为:50℃反转录30min,95℃预变性15min,接着95℃40s,55℃30s,72℃1min,40个循环,最后72℃延伸10min。
1.3.2巢式pcr
以反转录产物为模板,分别进行巢式pcr反应扩增h、κ、λ,过程如下:
引物:
h:vh3a-sense、vh3b-sense、mud、pw-cgamma;
λ:5′ageivλ1、5′ageivλ2、5′ageivλ3、5′ageivλ4/5、5′ageivλ6、5′ageivλ7/8、3′xhoicλ。
表2巢式pcr引物
pcr反应体系中包含:10×缓冲液2.5μl、10mmdntp0.5μl、dna聚合酶(全式金生物技术有限公司,ap141)0.25μl、引物如上、模板为反转录产物1μl、水补齐至25μl。
pcr反应条件为:94℃预变性4min,接着94℃30s,57℃30s,72℃45min,40个循环,最后72℃延伸10min。
1.3.3琼脂糖凝胶电泳
一个单细胞中重链和轻链基因均扩增成功的克隆,被认为是配对成功的克隆。取5μl巢式pcr扩增产物经1%琼脂糖凝胶电泳后,取配对的阳性克隆进行测序,测序获得的抗体可变区序列用vectornti软件及imgt网站进行分析,并进行抗体蛋白表达和功能验证。
1.3.4pcr扩增结果如图1-a和图1-b所示,图1-a显示重链可变区基因的扩增结果,其中泳道5是12h9重链可变区基因的扩增结果;图1-b是轻链可变区基因的扩增结果,其中泳道5是12h9轻链可变区基因的扩增结果。轻链和重链的可变区扩增片段均约400bp,符合预期。
1.4线性表达框的构建
扩增获得抗体基因,进行序列分析符合预期后需要对大量的配对基因进行表达和功能分析。相比传统的表达载体构建方法,构建线性表达框具有快速、高通量优点,因此首先构建了线性表达框。线性表达框含有在真核细胞内表达的所有原件,包括启动子、增强子、polya尾终止结构,参考商业化的表达载体pcdna3.4(thermofisherscientific,a14697),线性表达框从5’端依次含有cmv启动子序列(genbank登记号:x03922.1)、抗体可变区(从单细胞中扩增获得)、抗体恒定区(重链恒定区序列由seqidno:3所示,dna编码序列由seqidno:4所示,kappa型轻链恒定区序列由seqidno:7所示,dna编码序列由seqidno:8所示,lamda型轻链恒定区序列由seqidno:9所示,dna编码序列由seqidno:10所示)、bgh多聚a尾(genbank登记号:x03896.1)连接起来,将该线性形式的dna转染入细胞中进行抗体表达。
具体过程是通过体外重叠延伸pcr技术将各个pcr片段连接构建:
1.4.1以pcdna3.4(thermofisherscientific,a14697))为模板,扩增启动子-前导序列片段、多聚a尾片段:
扩增启动子-前导序列片段的pcr反应体系中包括:模板质粒pcdna3.4(thermofisherscientific,a14697)1ng,10×缓冲液5μl、10mmdntp1μl、dna聚合酶0.5μl、引物5'-cmv-forward(与cmv启动子上游序列匹配)(5'-cgatg-tacgggccagatatacgcgttg-3')、引物3'-leader-sequence(5'-acactggac-accttttaaaattag-3',用于重链的融合,信号肽序列的核苷酸序列5'-atgaacttcgg-gctcagcttgattttcct-tgtcctaattttaaaaggtgtc-3,编码的氨基酸序列为mnfglsliflvlilkgv;用于轻链的融合引物序列为5'-gtcaccagtggaacctggaacc-ca-3',全长信号肽序列核苷酸序列为5-atggattcacaggcccaggttcttatgttactg-ctgctatgggtatctggtacctgtggg,氨基酸序列为mdsqaqvlmllllwvsgtcg,信号肽序列来源鼠源单抗可变区)、水补齐至50μl。
扩增多聚a尾片段的pcr反应体系中包括:模板质粒psectag2(invitrogen,v90020)1ng,10×缓冲液5μl、10mmdntp1μl、dna聚合酶0.5μl、引物5'-bghpoly-(a)(5'-gcctcgactgtgccttctagttgc-3')、引物3'-bgh-poly(a)(5'-tccccagcatgcctgctattgtct-3')、水补齐至50μl。扩增片段长度215bp。
pcr反应条件:94℃预变性4min,接着94℃30s,60℃30s,72℃1min,30个循环,最后72℃延伸10min。
1.4.2扩增抗体恒定区
h链恒定区pcr体系中包含:重链恒定区模板10ng、10×缓冲液5μl、10mmdntp1μl、dna聚合酶0.5μl、引物5'-ch1(5'-accaagggcccatcggtcttcccc-3')、引物
κ链恒定区pcr体系中包含:κ链恒定区模板10ng、10×缓冲液5μl、10mmdntp1μl、dna聚合酶0.5μl、引物5'-cκ(5'-actgtggctgcaccatctgtcttc-3')、引物
λ链恒定区pcr体系中包含:λ链恒定区模板10ng、10×缓冲液5μl、10mmdntp1μl、dna聚合酶0.5μl、引物
以上黑体字序列部分
pcr反应条件为:94℃预变性4min,接着94℃30s,60℃60s,72℃3min,30个循环,最后72℃延伸10min。
1.4.3扩增抗体可变区
pcr体系中包含:模板为反转录pcr产物10ng,10×缓冲液5μl、10mmdntp1μl、dna聚合酶0.5μl、引物如表3中所示(将重链和轻链引物分别混合后加入体系中)、水补齐至50μl。
pcr反应条件:94℃预变性4min,接着94℃30s,60℃30s,72℃3min,30个循环,最后72℃延伸10min。
表3.线性表达框构建pcr引物
划线部分分别用于与上游片段和下游片段的融合。
pcr产物回收纯化:将以上pcr产物经1%的琼脂糖凝胶电泳后,切胶并使用omega公司回收试剂盒回收。
1.4.4分别扩增重链和轻链的线性表达框。
pcr反应体系中包括:
模板:纯化后的启动子-前导序列片段10ng、重链/轻链可变区片段10ng、重链/轻链恒定区片段10ng、多聚a尾片段10ng,10×缓冲液2.5μl、10mmdntp0.5μl、dna聚合酶(全式金生物技术有限公司,ap151-13)0.25μl、引物5'-cmv-forward(5'-cgatgtacgggccagatatacgcgttg-3')和3'-poly(a)(5'-tccccagcatgcctgctattgtct-3'、水补齐至25μl。
pcr反应条件为:94℃预变性4min,接着94℃30s,60℃30s,72℃3min,30个循环,最后72℃延伸10min。
1.4.5pcr产物回收纯化和定量:
pcr反应产物直接用omega公司回收试剂盒回收。dna定量:用nano(gehealthcare)对pcr回收产物进行定量。
1.4.6细胞接种:将293t细胞以2×105/ml接种于24孔细胞培养板中,在含有5%co2的细胞温箱中,37℃培养过夜。
1.4.7细胞共转染:次日,向200μl无血清的mem培养基中,加入构建成功的重链和轻链线性表达框pcr产物各1μg,混匀后加入4μl转染试剂turbofect(thermoscientific,r0531),共同孵育15-20min后逐滴加至过夜培养的293t细胞培养孔中。在含有5%co2的细胞温箱中,37℃培养48h后收细胞培养上清备用。
线性表达框的pcr融合扩增核酸电泳检测结果见图2-a和图2-b,图2-a是重链表达框的核酸电泳结果,其中泳道23是12h9的重链表达框电泳结果,扩增片段3700左右,符合预期;图2-b是轻链表达框的核酸电泳结果,其中泳道15是12h9的轻链表达框电泳结果,扩增片段3000左右,符合预期。
1.5表达载体的构建和酶切鉴定
重链线性表达框用ecori/noti酶切切胶回收约1.4kb大小的重链片段,表达载体(thermofisherscientific,a14697)使用ecori/noti酶切后回收,将重链和载体片段连接,转化dh5a,挑取克隆进行pcr检测、双酶切鉴定和序列测定,构建成功重链的表达载体pcdna3.4-12h9-h。同样,轻链线性表达框用ecori/noti酶切切胶回收约0.7kb大小的轻链片段,表达载体pcdna3.4使用ecori/noti酶切后回收,将轻链和载体片段连接,转化dh5a,挑取克隆进行pcr检测、双酶切鉴定和序列测定,成功构建轻链的表达载体pcdna3.4-12h9-λ。酶切鉴定的结果如图3所示,其中泳道1是pcdna3.4-12h9-h双酶切结果,泳道2是重链表达载体未进行酶切的结果;泳道3是轻链载体pcdna3.4-12h9-λ双酶切结果,泳道4是轻链表达载体未酶切结果。
1.6单抗的瞬时表达和亲和层析纯化
使用expi293表达系统,此表达系统将高表达expi293细胞系、化学成分明确的无血清培养基和一种高效转染试剂与专用增强剂结合在一起。取15μg重链和15μg轻链混合后转染expi293f细胞,按照说明书(thermofisherscientific,a14635)进行操作,5-6天后收获培养液,离心后上清约30ml,使用体积为5ml的预装proteina亲和层析柱,上样前使用20mmpbs平衡,待电导显示到基线后进样,上样结束后,使用20mmpbs洗涤色谱柱至基线平稳,使用0.1mph3.0的甘氨酸缓冲液洗脱目的蛋白,待od280近基线后,停止收集,使用至少3个柱体积的20mm的pbs洗涤色谱柱,至基线平稳后,用20%的乙醇洗涤色谱柱。单抗蛋白含量的测定使用紫外法,公式:蛋白含量=1.54a280-1.38a260。瞬时转染后的单抗表达情况见图4(横坐标为30ml无血清悬浮培养体系下单抗的瞬时表达量:mg,纵坐标列出了28株不同抗体),亲和层析纯化后的单抗sds-page检测结果见图5(泳道1为12h9单抗非还原电泳结果,预期150kda;_泳道2为分子量标记,泳道3为12h9单抗的还原sds-page电泳结果,重链(heavychain,h)预期50kda,轻链(lightchain,l)预期25kda)。合计28株单抗获得表达,经过纯化并测定蛋白浓度,用excel进行统计分析,本发明公开的单抗具有很好的表达水平,收集近30ml培养上清,纯化后抗体的量是6mg,按比例计算达到200mg/l的水平,按纯化得率90%计算,上清中的表达量超过220mg/l,。这是瞬时转染的表达量,通过制备稳转的细胞株,再经过培养基和工艺的优化,能够满足产业化的需求。
实施例2细胞中和实验
2.1实验前一天,将vero细胞用培养基(mem+10%fbs)稀释至1.5×105cells/ml浓度,接种到96孔细胞培养板,接种体积200μl/孔,置37℃5%co2细胞培养箱培养;
2.2实验当天,将纯化单抗用培养基mem+2%fbs自初始浓度(12h9单抗初始浓度100μg/ml,ab2对照抗体200μg/ml,参考美国专利us9738704公开的8b10f8重链和轻链可变区序列,化学合成可变区基因并利用融合pcr技术获得全长抗体基因,克隆到表达载体pmh载体上,转染cho-s细胞,通过proteina亲和层析纯化获得并保存,研究室命名为ab2)4倍稀释,加入96孔培养板,体积120μl/孔;随即每孔加入120μl基孔肯雅病毒悬液(用mem+2%fbs按1:1000稀释),充分混匀,置细胞培养箱共孵育1h;
2.3弃去96孔板中细胞培养上清,每孔加入200μl共孵育后的病毒-抗体混合悬液;另设置存活对照(不加病毒和抗体)和死亡对照(只加病毒),置37℃5%co2细胞培养箱继续培养72h;
2.472h后弃去细胞培养上清,加入50μl结晶紫室温染色15min,弃去,加入200μl/孔纯水,重复洗涤5次;
2.5弃尽洗液,加入100μl脱色液充分溶解,以od620为参考,用酶标仪测od570值;用(od样本孔-od死亡对照)/(od存活对照-od死亡对照)计算细胞活率,细胞活率和抗体浓度用graphpadprism5拟合曲线,计算抗体ic50值;
2.6单抗在细胞模型上的保护效果和ic50的结果,见图6(横坐标表示测定抗体的对数浓度,单位log:ug/ml,纵坐标表示测定抗体的保护率)和图7(公开单抗12h9和对照单抗ab2在vero细胞模型上的保护效果,其中阴性表示未加病毒,细胞全部存活,阳性表示加病毒未加单抗,细胞死亡,ab2表示加病毒和单抗ab2,12h9表示加病毒和单抗12h9)。本发明公开的单抗12h9的ic50是0.28μg/ml,文献中的对照单抗ab2的ic50是2.17μg/ml,本发明公开的单抗活性是文献报道单抗的8倍。
实施例3单抗识别表位研究
3.1elisa筛选特异性克隆:于96孔酶联板包被抗基孔肯雅病毒的单克隆抗体(100μg/ml,150μl/孔),4℃过夜;弃掉包被液体,加入5mg/mlbsa封闭缓冲液(0.1mol/lnahco3,ph8.6)于4℃封闭2h;倾去液体,用tbst(1ml/ltween-20,tbs)洗6次;加入噬菌体环7肽文库100μl(原库用tbst按1∶10稀释,约含噬菌体2×1011pfu/100μl),室温轻摇孵育30min;弃去液体,用tbst洗10次,洗掉未结合的噬菌体;加入100μl洗脱液(1mg/mlbsa,0.2mol/lglycine-hcl,ph2.2)洗脱,室温轻摇10min,迅速吸出液体并加入15μl中和液(1mol/ltris-hcl,ph9.1)中和;取1μl洗脱下来的噬菌体测滴度,余下的液体感染大肠杆菌er2738,扩增,纯化,进行下一轮筛选。第2轮和第3轮筛选保持抗体包被浓度不变,加入上一轮扩增纯化的噬菌体(2×1011pfu/100μl),洗涤液第二轮为0.2%tbst,第三轮为0.5%tbst。3次筛选后随机挑选50个分隔良好的阳性克隆于大肠杆菌er2738中扩增,纯化,用于噬菌体elisa表位融合蛋白和elisa检测。
单克隆抗体包被96孔酶联板(每孔2μg/ml),4℃孵育过夜;弃掉液体,加入封闭液(5mg/mlbsa,0.1mol/lnahco3,ph8.6),4℃孵育2h;弃掉液体,分别加入筛选纯化的噬菌体克隆(1×109pfu/孔,每克隆设2复孔),室温孵育2h;用0.5%tbst洗6次后,每孔加入hrp标记的小鼠抗m13噬菌体抗体(0.82mg/ml,1∶2000稀释,按照说明书操作)100μl,室温孵育1h;用tbst洗6次,abts显色,测定a450nm值。设立2组阴性对照,一组为单抗包板加1%bsa,另一组为无关抗体包板加噬菌体克隆。阳性克隆结果标准为a450nm值高于阴性对照2倍以上,且阴性对照值低于0.1。
3.2测序和比对:通过噬菌体elisa的鉴定,挑取结合活性高的6个噬菌体克隆,经peg/nacl纯化后抽提ssdna,交由北京中美泰和公司进行序列测定。测序引物为噬菌体肽库自带的-96giiiprimer。6个克隆5个读通,含有2种序列,其中25和27、29号克隆序列是c-rlplshy-c(命名为12h09-c7-25),与基孔肯雅病毒标准株s27结构蛋白(s27-sp)进行比对,最大同源性区域位于e2蛋白(s27-e2)的150-156氨基酸上,比对结果同源性4/7(见图8-a),与非结构蛋白(s27-nsp)进行比对最大同源性为3/7(见图8-b);26和30号克隆序列是c-rpwtpdw-c(命名为12h09-c7-26),与基孔肯雅病毒s27结构蛋白(s27-sp)进行比对,最大同源性区域位于e2蛋白(s27-e2)的172-178氨基酸上,比对结果同源性4/7(见图8-c),与非结构蛋白(s27-nsp)进行最大同源性比对,比对结果同源性2/7(见图8-d)。综上,单抗12h9识别的表位位于结构蛋白e2上,为构象表位,elisa检测大肠杆菌表达的e2蛋白不与该单抗特异结合(大肠杆菌表达的e2蛋白不具有天然构象)。
3.3elisa检测单抗与重组融合表位多肽的结合活性
3.3.1思路:将表位融合到硫氧还蛋白trx的c端进行融合表达,表位的c末端融合6个组氨酸,进行镍柱亲和层析纯化,纯化的表位多肽用elisa检测与单抗的特异结合活性。
3.3.2实验设计:以pet32-a为模板(含有硫氧还蛋白基因),通过融合pcr技术获得含有trx-表位-his6的融合基因,经过ndei/xhoi酶切后克隆到的表达载体的ndei/xhoi位点,构建表达质粒pet-12h09-c25和pet-12h09-c26。融合基因扩增使用的上游引物是t7promoter:序列5-ccctatagtgagtcgtatta-3,下游引物trx-12h09-c25:序列
3.3.3表达载体的构建:以pet-32a为模板,上游引物是t7promoter,下游引物trx-12h09-c25进行常规pcr扩增,pcr反应产物直接用omega公司回收试剂盒回收。经过ndei/xhoi双酶切后克隆到pet-32a的ndei/xhoi位点,连接转化大肠杆菌后,挑选氨苄抗性克隆进行pcr和双酶切鉴定,阳性克隆提取表达质粒pet-trx-12h09-c25和pet-trx-12h09-c26。
3.3.4表达菌株的构建:将pet-trx-12h09-c25和pet-trx-12h09-c26转化bl21(de3),挑选氨苄抗性的克隆,挑取单克隆接种到6mllb培养基中,过夜培养,次日1:50转接到300mllb培养基中,待od650达到0.8时,使用终浓度为1mm的iptg进行诱导表达,诱导时间5小时,培养基经过12000rpm离心10分钟,收集菌体,10倍体积的pbs缓冲液重悬菌体,用超声破碎菌体(功率65w,超声5s,停5s,合计20min),12000rpm离心15分钟,收集上清。
3.3.5镍柱亲和层析纯化重组融合多肽:首先在平衡ni柱之前给ni柱挂ni(步骤为先走3-5个柱体积的纯水,然后走3-5个柱体积的100mmniso4,挂上ni后再走3-5个柱体积的纯水),然后用bindingbuffer(20mmpb,500mmnacl,20mm咪唑,ph8.0,)平衡ni柱,柱子平衡后低流速上样,整个上样过程中使样品始终处于冰上。上样后用3-5个柱体积的bindingbuffer平衡,再用elutionbuffer(20mmpb,500mmnacl,50mm咪唑,ph8.0)进行梯度洗脱,收集280nm吸收峰处的目的蛋白。收集蛋白后,用0.1medta洗脱niso4,再用1mnaoh清洗ni柱,pbs中和ph值,最后将柱子用20%乙醇保存,(以上几种液体均走5个柱体积)。结果如图9所示,c25表位融合蛋白和c26表位融合蛋白均获得纯化,大小符合预期,纯度高,满足试验要求。
3.3.6elsia检测结合活性:将纯化的多肽包被96孔酶联板(每孔2μg/ml,100ul/孔),4℃孵育过夜;弃掉液体,加入封闭液(5mg/mlbsa,ph7.2pbs),4℃孵育1h;弃掉液体,加入2倍系列稀释的纯化单抗(首孔8μg/ml,设3复孔),室温孵育1h;用0.5%pbst洗3次后,每孔加入hrp标记的羊抗人抗体(sigma,1∶20000稀释,按照说明书操作)100μl,室温孵育1h;用pbst洗3次,abts显色,测定a450nm值。设立2组阴性对照,一组为空对照,另一组为无关抗体对照。阳性克隆结果标准为a450nm值高于阴性对照2倍以上,且阴性对照值低于0.1。
结果如图10所示,c25表位融合蛋白与公开单抗具有特异的结合活性,其他对照单抗与表位融合蛋白无结合(图未显示),表位c26融合蛋白与公开单抗具有弱结合活性,结合3.2描述的结果,表位c25与结构蛋白e2最大同源性区域位于e2蛋白的150-156氨基酸上,推测e2蛋白的150-156是公开单抗识别的关键区域。
序列表
<110>中国人民解放军军事科学院军事医学研究院
<120>针对特异表位的抗基孔肯雅热全人源单克隆抗体及应用
<160>10
<170>patentinversion3.3
<210>1
<211>119
<212>prt
<213>homosapiens
<400>1
glnvalglnleuglnglntrpglyalaglyleuleulysproserglu
151015
thrleuserleuthrcysalavaltyrglyglyserpheserglytyr
202530
tyrtrpsertrpileargglnproproglylysglyleuglutrpile
354045
glygluileasnhisserglyserthrasntyrasnproserleulys
505560
serargvalthrileservalaspthrserlysasnglnpheserleu
65707580
lysleuserservalthralaalaaspthralavaltyrtyrcysala
859095
alailealaglualaglythrglytyrpheasptyrtrpglyglngly
100105110
thrleuvalthrvalserser
115
<210>2
<211>357
<212>dna
<213>homosapiens
<400>2
cagggacagctgcagcagtggggcgcaggactgttgaagccttcggagaccctgtccctc60
acctgcgctgtctatggtgggtccttcagtggttactactggagctggatccgccagccc120
ccagggaaggggctggagtggattggggaaatcaatcatagtggaagcaccaactacaac180
ccgtccctcaagagtcgagtcaccatatcagtagacacgtccaagaaccagttctccctg240
aagctgagctcagtgaccgccgcggacacggctgtgtattactgtgcggccatagcagaa300
gctggtacgggatactttgactactggggccagggaaccctggtcaccgtctcctca357
<210>3
<211>330
<212>prt
<213>homosapiens
<400>3
alaserthrlysglyproservalpheproleualaproserserlys
151015
serthrserglyglythralaalaleuglycysleuvallysasptyr
202530
pheprogluprovalthrvalsertrpasnserglyalaleuthrser
354045
glyvalhisthrpheproalavalleuglnserserglyleutyrser
505560
leuserservalvalthrvalproserserserleuglythrglnthr
65707580
tyrilecysasnvalasnhislysproserasnthrlysvalasplys
859095
lysvalgluprolyssercysasplysthrhisthrcysproprocys
100105110
proalaprogluleuleuglyglyproservalpheleuphepropro
115120125
lysprolysaspthrleumetileserargthrprogluvalthrcys
130135140
valvalvalaspvalserhisgluaspprogluvallyspheasntrp
145150155160
tyrvalaspglyvalgluvalhisasnalalysthrlysproargglu
165170175
gluglntyrasnserthrtyrargvalvalservalleuthrvalleu
180185190
hisglnasptrpleuasnglylysglutyrlyscyslysvalserasn
195200205
lysalaleuproalaproileglulysthrileserlysalalysgly
210215220
glnproarggluproglnvaltyrthrleuproproserargaspglu
225230235240
leuthrlysasnglnvalserleuthrcysleuvallysglyphetyr
245250255
proseraspilealavalglutrpgluserasnglyglnprogluasn
260265270
asntyrlysthrthrproprovalleuaspseraspglyserphephe
275280285
leutyrserlysleuthrvalasplysserargtrpglnglnglyasn
290295300
valphesercysservalmethisglualaleuhisasnhistyrthr
305310315320
glnlysserleuserleuserproglylys
325330
<210>4
<211>993
<212>dna
<213>homosapiens
<400>4
gctagcaccaagggcccatcggtcttccccctggcaccctcctccaagagcacctctggg60
ggcacagcggccctgggctgcctggtcaaggactacttccccgaaccggtgacggtgtcg120
tggaactcaggcgccctgaccagcggcgtgcacaccttcccggctgtcctacagtcctca180
ggactctactccctcagcagcgtggtgaccgtgccctccagcagcttgggcacccagacc240
tacatctgcaacgtgaatcacaagcccagcaacaccaaggtggacaagaaagttgagccc300
aaatcttgtgacaaaactcacacatgcccaccgtgcccagcacctgaactcctgggggga360
ccgtcagtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacccct420
gaggtcacatgcgtggtggtggacgtgagccacgaagaccctgaggtcaagttcaactgg480
tacgtggacggcgtggaggtgcataatgccaagacaaagccgcgggaggagcagtacaac540
agcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaatggcaag600
gagtacaagtgcaaggtctccaacaaagccctcccagcccccatcgagaaaaccatctcc660
aaagccaaagggcagccccgagaaccacaggtgtacaccctgcctccatctcgggatgag720
ctgaccaagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagcgacatc780
gccgtggagtgggagagcaatgggcagccggagaacaactacaagaccacgcctcccgtg840
ctggactccgacggctccttcttcctctatagcaagctcaccgtggacaagagcaggtgg900
cagcaggggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactacacg960
cagaagagcctctccctgtctccgggtaaatga993
<210>5
<211>110
<212>prt
<213>homosapiens
<400>5
sertyrgluleuthrglnproalaservalserglyserproglygln
151015
serilethrilesercysthrglythrserseraspvalglyglytyr
202530
asntyrvalsertrptyrglnglnhisproglylysalaprolysleu
354045
metiletyraspvalserasnargproserglyvalserasnargphe
505560
serglyserlysserglyasnthralaserleuthrileserglyleu
65707580
glnalagluaspglualaasptyrtyrcyssersertyralaserser
859095
serthrtrpvalpheglyglyglythrlysleuthrvalleu
100105110
<210>6
<211>330
<212>dna
<213>homosapiens
<400>6
tcctatgagctgactcagcctgcctccgtgtctgggtctcctggacagtcgatcaccatc60
tcctgcactggaaccagcagtgacgttggtggttataactatgtctcctggtaccaacag120
cacccaggcaaagcccccaaactcatgatttatgatgtcagtaatcggccctcaggggtt180
tctaatcgcttctctggctccaagtctggcaacacggcctccctgaccatctctgggctc240
caggctgaggacgaggctgattattactgcagctcatatgcaagcagcagcacttgggtg300
ttcggcggagggaccaagctgaccgtccta330
<210>7
<211>106
<212>prt
<213>homosapiens
<400>7
glyglnprolysalaalaproservalthrleupheproproserser
151015
glugluleuglnalaasnlysalathrleuvalcysleuileserasp
202530
phetyrproglyalavalthrvalalatrplysalaaspserserpro
354045
vallysalaglyvalgluthrthrthrproserlysglnserasnasn
505560
lystyralaalasersertyrleuserleuthrprogluglntrplys
65707580
serhislyssertyrsercysglnvalthrhisgluglyserthrval
859095
glulysthrvalalaprothrglucysser
100105
<210>8
<211>321
<212>dna
<213>homosapiens
<400>8
ggtcagcccaaggctgccccctcggtcactctgttcccaccctcgagtgaggagcttcaa60
gccaacaaggccacactggtgtgtctcataagtgacttctacccgggagccgtgacagtg120
gcctggaaggcagatagcagccccgtcaaggcgggagtggagaccaccacaccctccaaa180
caaagcaacaacaagtacgcggccagcagctacctgagcctgacgcctgagcagtggaag240
tcccacaaaagctacagctgccaggtcacgcatgaagggagcaccgtggagaagacagtg300
gcccctacagaatgttcataa321
<210>9
<211>107
<212>prt
<213>homosapiens
<400>9
argthrvalalaalaproservalpheilepheproproseraspglu
151015
glnleulysserglythralaservalvalcysleuleuasnasnphe
202530
tyrproargglualalysvalglntrplysvalaspasnalaleugln
354045
serglyasnserglngluservalthrgluglnaspserlysaspser
505560
thrtyrserleuserserthrleuthrleuserlysalaasptyrglu
65707580
lyshislysvaltyralacysgluvalthrhisglnglyleuserser
859095
provalthrlysserpheasnargglyglucys
100105
<210>10
<211>324
<212>dna
<213>homosapiens
<400>10
cggaccgtggcggcgccatctgtcttcatcttcccgccatctgatgagcagttgaaatct60
ggtaccgctagcgttgtgtgcctgctgaataacttctatcccagagaggccaaagtacag120
tggaaggtggataacgccctccaatcgggtaactcccaggagagtgtcacagagcaggac180
agcaaggacagcacctacagcctcagcagcaccctgacgctgagcaaagcagactacgag240
aaacacaaagtctacgcctgcgaagtcacccatcagggcctgagctcgcccgtcacaaag300
agcttcaacaggggagagtgttag324
- 还没有人留言评论。精彩留言会获得点赞!