一种检测茶卡羊PIGY基因CNV标记的方法及其应用与流程
本发明属于分子遗传育种领域,具体涉及一种检测茶卡羊pigy基因拷贝数变异的方法,该方法利用实时定量pcr技术,以茶卡羊基因组dna为模板,以单拷贝ankrd1基因为参照,根据2*2-δct值确定个体的pigy基因拷贝数变异类型。
背景技术:
随着对基因组研究的深入,有证据证明拷贝数变异(copynumbervariations,cnvs)影响基因网络并调节相关基因的表达,促成个体表型的可变性,因此利用与体尺性状相关的cnvs的候选标记可以加速绵羊遗传育种进程。拷贝数变异一般是指在全基因组范围大于50bp的结构变异,是基因组内发生重排导致的。
目前,人和动物全基因组范围内的cnvs检测方法主要有三种,分别是微阵列比较基因组杂交芯片(array-basedcomparativegenomichybridization,acgh)、二代测序技术(next-generationsequencing,ngs)和snp芯片技术。在比较基因组杂交芯片中寡核苷酸探针芯片被广泛使用,具有高灵敏度、高精度和样本量小的特点;而细菌人工染色体芯片制作成本高、分辨率低且耗时长,很少会使用。此外,现有的芯片平台对新的拷贝数变异的检测效率很低。随着二代测序技术的发展,当前最有效的检测手段是通过重测序来检测基因组结构变异,但这种方法相较其他方法成本较高。
目前,对基因组己知的cnv进行检测主要有两种方法,即基于pcr的检测技术和基于杂交的检测技术。pcr检测技术主要包括实时荧光定量pcr(real-timeqpcr)、依赖连接的多重扩增探针杂交技术(multiplexligation-dependentprobeamplification,mlpa)和短片段多重定量技术(quantitativemultiplexpcrofshortfluorescentfragments,qmpsf)。目前使用最广泛的是qpcr技术,该方法敏感性高、操作方法简单、速度快、重复性好且污染少。杂交技术主要包括southernblotting杂交、荧光原位杂交(fluorescenceinsituhybridization,fish)、多重扩增探针杂交技术(multiplexamplifiableprobehybridization,maph)等,但这些方法成本比较高,检测时间长而且不够准确,目前使用较少。
茶卡羊是产自青海省海西蒙古族藏族自治州乌兰县的一个经过长期的自然选择和人工选育而形成的优良绵羊品种。茶卡羊的进一步选育提高对于茶卡羊的产业化和经济效益具有重要作用。pigy(phosphatidylinositolglycananchorbiosynthesisclassy)基因是pig基因家族的成员之一,位于绵羊6号染色体上,其表达产物与piga、pigc、pigh、pigp、pigq和dpm2基因的表达蛋白共同组成了gpi-gnt复合物,该复合物启动了糖基磷脂酰肌醇(gpi)的生物合成。gpi在内质网上合成,用于许多表面蛋白的锚定。含有gpi锚的蛋白质在细胞与细胞的相互作用中起到重要作用。目前仅发现pigy基因和多个与肌肉密度、胴体重等相关的数量性状位点存在重叠,对绵羊pigy基因cnv的分析和应用还处于空白阶段。
技术实现要素:
本发明的目的在于提供一种检测茶卡羊pigy基因cnv标记的方法及其应用。
为达到上述目的,本发明采用了以下技术方案:
一种检测茶卡羊pigy基因cnv标记的方法,包括以下步骤:
以茶卡羊个体全基因组dna为模板,以引物对pigy-cnv及引物对ankrd1-cnv为引物,通过实时荧光定量pcr分别扩增pigy基因的拷贝数变异区域以及作为内参的ankrd1基因的部分片段,然后根据定量结果鉴定茶卡羊个体pigy基因的拷贝数变异类型;
所述的引物对pigy-cnv为:
上游引物f1:5’-agagtggcgggtgataagtg-3’
下游引物r1:5’-cagtcctgccaaagacacca-3’;
所述的引物对ankrd1-cnv为:
上游引物f2:5’-tgggcaccacgaaattctca-3’
下游引物r2:5’-tggcagaaatgtgcgaacg-3’。
优选的,所述的pigy基因的拷贝数变异区域位于绵羊参考基因组序列nc_019463.236121601bp-36125200bp。
优选的,所述的拷贝数变异类型是根据2*2-δct将定量结果分为的三类:插入型,2*2-δct>2.5;缺失型,2*2-δct<1.5;正常型,2*2-δct=1.5~2.5。
优选的,所述的实时荧光定量pcr所用的扩增体系包括:50ng/μl模板dna1μl、10pmol/l的引物对pigy-cnv或引物对ankrd1-cnv所对应的上、下游引物各0.5μl、2×sybrgreenqpcrmix5μl和ddh2o3μl。
优选的,所述的实时荧光定量pcr所用的反应程序包括以下步骤:95℃预变性1min;95℃变性15s,60℃退火15s,72℃延伸30s,读板(plateread),共进行39个循环。
优选的,基于引物对pigy-cnv扩增的片段大小为90bp,基于引物对ankrd1-cnv扩增的片段大小为143bp。
上述检测茶卡羊pigy基因cnv标记的方法在茶卡羊分子标记辅助选择育种中的应用。
优选的,所述茶卡羊中,具有插入型拷贝数变异类型的个体在体重、胸围等生长性状上优于具有缺失型和正常型拷贝数变异类型的个体。
本发明的有益效果体现在:
本发明以待测茶卡羊的基因组dna为模板,利用实时荧光定量pcr检测基因组中pigy基因的拷贝数变异类型,根据检测结果,以及茶卡羊群体中的拷贝数变异情况与其体重、胸围、体高、体长等重要经济性状关联分析的结果,本发明提出的检测pigy基因cnv标记的方法可以用于快速建立茶卡羊生长性状(例如,体重、胸围)优势种群。该方法简单、快速、便于推广应用。
与现有技术相比,本发明具有以下优点:
(1)本发明提供的茶卡羊pigy基因cnv标记检测方法,不受年龄的限制,可用于茶卡羊个体的早期选育,甚至在刚出生时就可进行选择;
(2)可以对茶卡羊pigy基因拷贝数变异进行准确可靠的检测,操作简便;
(3)茶卡羊pigy基因cnv标记的检出,为其生长性状的分子标记辅助选择提供科学依据。
附图说明
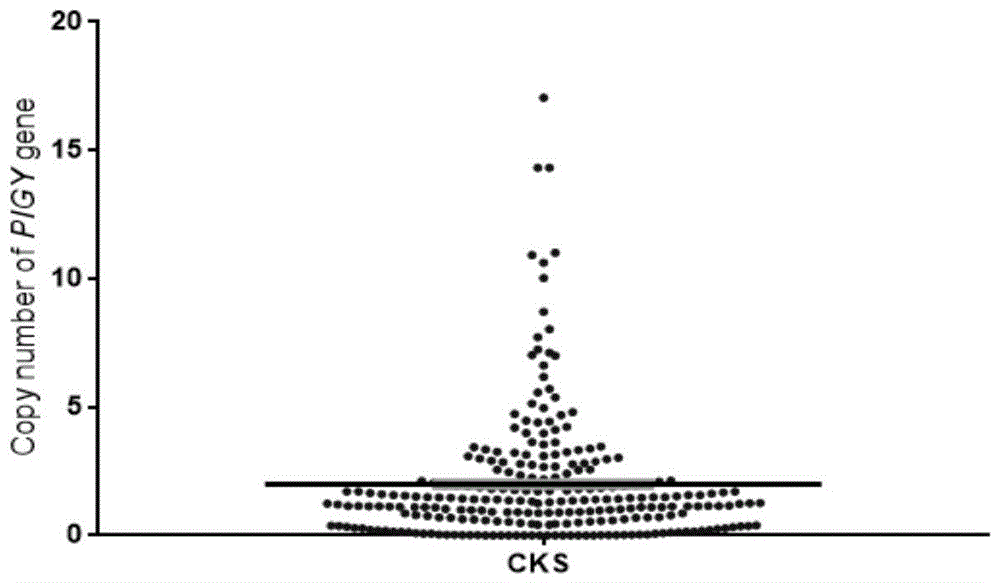
图1为312个茶卡羊pigy基因拷贝数变异分布图。
图2为本发明中进行qpcr(pigy基因)绘制的扩增曲线图。
图3是本发明中进行qpcr(pigy基因)绘制的溶解曲线图。
具体实施方式
下面结合附图和实施例对本发明做进一步详细说明。
本发明利用实时荧光定量pcr对茶卡羊pigy基因的拷贝数变异进行检测并用于分子育种,包括以下步骤:
(1)根据ncbi数据库的绵羊pigy基因序列,进行引物设计,并用普通pcr检测引物;
(2)采用实时荧光定量pcr(qpcr)技术检测候选位点在群体中的拷贝数变异情况;
(3)利用spss23.0软件将拷贝数变异类型与茶卡羊生长性状进行关联分析,筛选到与茶卡羊生长性状相关的cnv标记;
(4)根据拷贝数变异类型进行生长性状优异的茶卡羊选育。
利用实时荧光定量pcr对茶卡羊pigy基因的拷贝数变异进行检测的具体流程如下:
1、绵羊样本采集
本发明具体以312头茶卡羊作为检测对象,茶卡羊血液样本采集自青海省海西州乌兰县茶卡镇养殖场(2018年5月采集)。
2、血样基因组dna的提取
(1)冷冻血样(主要为血细胞)室温解冻,吸取500μl血液于1.5ml离心管中,加入等体积的磷酸缓冲液(pbs)混匀,温和摇动,4℃、12000r/min离心5min,弃去上清液;重复本步骤至上清液透明。
(2)在离心管中加入dna抽提缓冲液500μl,轻轻吹打,使血细胞沉淀脱离离心管壁,37℃水浴1h。
(3)加蛋白酶k至5μl(20mg/ml),并混匀,55℃水浴中消化过夜(16h左右)至絮状沉淀不见,溶液澄清,尚未澄清的,可补加10μl蛋白酶k混匀继续消化直至澄清。
(4)将反应液冷却至室温,加入500μltris饱和酚,温和摇动15min,使其充分混匀,4℃、12000r/min离心10min,将上层水相转入另一灭菌离心管;重复本步骤1次。
(5)加入氯仿500ml,温和摇动20min,使其充分混匀,4℃、12000r/min离心15min,将上层水相转入另一灭菌的1.5ml离心管。
(6)加入氯仿、异戊醇混合液(24:1)500ml,充分混合20min,4℃、12000r/min离心10min,将上清液转入另一1.5ml离心管中。
(7)加入0.1倍体积的naac缓冲液及2倍体积的冰冷的无水乙醇,混合转动离心管直至白色的絮状沉淀析出。
(8)4℃、12000r/min离心10min,弃去上清液,用70%的冰冷乙醇漂洗dna沉淀2次。
(9)4℃、12000r/min离心10min,弃上清液,室温下使乙醇挥发干净。
(10)干燥后的dna中加入80~100μl的te,4℃保存直至dna完全溶解,利用紫外分光光度仪和电泳检测其浓度和质量,-80℃保存。
3、目标序列及内参序列的扩增
以ncbi数据库(http://www.ncbi.nlm.nih.gov/)公布的绵羊pigy基因序列(genbankaccessionno.nc_019463.2)为参考序列(chr6:36120820bp-36123522bp),根据cnv区域在参考基因组中的位置(位于nc_019463.236121601bp-36125200bp),利用ncbi网站的primer-blast设计用于检测pigy基因拷贝数变异的实时荧光定量pcr引物,利用该引物在pigy基因候选区域内可以扩增大小为90bp的片段。内参序列为已知的不存在拷贝数变异的序列,即ankrd1基因中的一段143bp的序列,引物对序列信息如表1所示(设计完成时间2019年8月)。
表1.实时荧光定量pcr的引物信息
实时荧光定量pcr所用的扩增体系以10.0μl计为:50ng/μl模板dna(提取自血液样本的基因组dna)1μl、10pmol/l的上游引物0.5μl、10pmol/l的下游引物0.5μl、2×sybrgreenqpcrmix5.0μl,及ddh2o3.0μl。
qpcr扩增的反应程序为:95℃预变性1min;95℃变性15s,60℃退火15s,72℃延伸30s,读板(plateread),共进行39个循环;做溶解曲线是从65℃开始,每循环增加0.5℃,上升至95℃,进行5s,然后读板。
通过绘制扩增曲线(图2)和熔解峰确定引物适用于qpcr分析。根据绘制的熔解曲线,各样品曲线吻合在一起,且曲线走势平滑,峰高且尖,无引物二聚体或非特异扩增引起的杂峰(图3)。
4、拷贝数变异的判断
每个样品分别用目标序列和内参序列的引物进行扩增,并且每对引物进行3个重复。根据2*2-δct方法进行拷贝数变异的分析。其中δct=ct目的基因–ct内参基因。2*2-δct表示的是拷贝数。ct即cyclethreshold,为pcr扩增过程中扩增产物的荧光信号达到设定的阈值时所经过的扩增循环次数。
根据2*2-δct将定量结果分为三类:插入型,2*2-δct>2.5;缺失型,2*2-δct<1.5;正常型,2*2-δct=1.5~2.5,参见图1。
5、pigy基因cnv位点与生长性状的关联分析
生产数据:体高、体长、胸围、体重。
关联分析模型:先对数据进行描述分析,确定是否存在离群值,再利用最小二乘分析对数据校正;根据数据特征,应用spss23.0软件分析各基因型间的生产性状效应。在对基因型效应进行分析时采用了固定模型:
yijk=μ+ai+cnvj+eijk
其中:yijk为性状观察值,μ为总体均值,ai为第i头个体的年龄,cnvj为第j个拷贝数变异类型的固定效应,eijk为随机误差。各组数据间的差异性采用lsd多重比较进行检验,试验结果以mean±se形式表示。
关联分析结果表明(见表2):茶卡羊pigy基因上cnv类型为插入型的个体在生长性状(体重、胸围)上显著优于正常型和缺失型的个体;以上结果说明pigy基因cnv位点的插入型可以作为一个提高茶卡羊生长性状的候选分子遗传标记(cnv标记),用于茶卡羊的早期分子选育。
表2.茶卡羊pigy基因拷贝数变异与生长性状的关联性分析
注:平均值肩标上具有相同字母表示差异不显著(p>0.05),平均值肩标上字母不同表示差异显著(abc:p<0.05;abc:p<0.01)。*p<0.05;**p<0.01。
6、上述cnv标记在茶卡羊选育中的应用
获得的cnv候选分子遗传标记,可以用于寻找与其相关或紧密连锁的影响茶卡羊生长性状的数量性状基因座。还可以用于茶卡羊的分子标记辅助选择,即通过对茶卡羊pigy基因cnv位点拷贝数变异类型的检测,选择插入型的茶卡羊个体留种并扩繁,从而能够加快茶卡羊品种改良的选育进程。
<110>西北农林科技大学
<120>一种检测茶卡羊pigy基因cnv标记的方法及其应用
<160>4
<210>1
<211>20
<212>dna
<213>人工合成
<400>1
agagtggcgggtgataagtg20
<210>2
<211>20
<212>dna
<213>人工合成
<400>2
cagtcctgccaaagacacca20
<210>3
<211>20
<212>dna
<213>人工合成
<400>3
tgggcaccacgaaattctca20
<210>4
<211>19
<212>dna
<213>人工合成
<400>4
tggcagaaatgtgcgaacg19
- 还没有人留言评论。精彩留言会获得点赞!