一种用于评估幼年型粒单核细胞白血病预后的全基因组DNA甲基化检测方法与流程
本发明属于生物技术领域,具体涉及一种用于评估幼年型粒单核细胞白血病预后的全基因组dna甲基化检测方法。
背景技术:
幼年型粒单核细胞白血病(juvenilemyelomonocyticleukemia,jmml)是一类发病年龄小,预后差异显著的疾病。明确预后对于疾病治疗选择至关重要。目前此类疾病预后指标有限,jmml突变基因对于预后一定的指示意义,但是同种基因预后差异明显,如nras基因突变患者部分疾病进展迅速,病情凶险,只有通过造血干细胞移植(hematopoieticstemcelltransplantation,hsct)方有治愈可能;部分患者病情平稳,可获得自发缓解。
近年来研究表明表观遗传学在肿瘤发病机制及预后评估中起着举足轻重的地位。dna甲基化是一种被广泛研究的表观遗传修饰方式,与组蛋白修饰等方式一起,在调控基因表达和染色质构象等方面发挥了重要作用。通常地,甲基化dna指5-甲基胞嘧啶(5mc),它是在dna甲基转移酶(dnmt)的作用下将甲基基团添加到胞嘧啶的5’c位置上形成的结构(vertinopm,1996)。哺乳动物细胞中甲基化主要发生在cg双核苷酸的胞嘧啶上(goldbergad,2007),植物细胞中则存在很大比例的non-cg(chh、chg,h代表a、c、t)甲基化(jacksonjp,2002)。一般来说,高dna甲基化水平会抑制基因表达,去甲基化则可以使得基因重新表达。dna甲基化参与了众多的细胞生命活动,包括细胞分化、组织特异性基因表达、基因组印记、x染色体失活等(birda,2002;jonespa,2001;reikw,2003)。异常的dna甲基化会导致发育异常、肿瘤等疾病的发生。因此,dna甲基化的研究对于深入理解基因表达、个体发育以及疾病的发生、发展机制都具有重要意义。研究发现,在启动子cpg二核苷酸胞核嘧啶甲基化是人类基因组基因表达一种重要的负调节因素。肿瘤抑制基因的异常甲基化在恶性肿瘤的发病机制中起着决定性作用,包括髓系肿瘤和骨髓增生异常综合症。
技术实现要素:
本发明就是为了解决上述技术问题,所提供一种用于评估幼年型粒单核细胞白血病预后的全基因组dna甲基化检测方法。
本发明是通过以下技术方案实现的:
一种用于评估幼年型粒单核细胞白血病预后的全基因组dna甲基化检测方法,包括以下步骤:
a.文库构建
ⅰ.dna提取:使用基因组dna提取试剂盒对样本dna进行核酸提取;
ⅱ.基因组文库构建
ⅰ)样品打断:样品打断方法是covaris打断法,均可将样品dna打碎至100-300bp范围的片段;
ⅱ)dna片段末端修复、3’端加a碱基,连接测序接头;
ⅲ)采用zymoezdnamethylation-goldkit进行bisulfite处理;
ⅳ)处理后切胶回收,并进行文库片段大小选择;
ⅴ)合格的文库用于上机测序;
b.生物信息分析
ⅰ.标准信息分析;
ⅱ.数据统计与质控;
ⅲ.c碱基的甲基化水平分析;
ⅳ.全基因组甲基化水平分布趋势;
ⅰ)甲基化c碱基中分布比例;
ⅱ)所有c的甲基化水平;
ⅲ)各条染色体中c的甲基化水平;
ⅳ)统计不同基因区域内cg、chg和chh中c的甲基化水平;
ⅴ)不同基因元件区域中cg、chg和chh中c的甲基化水平;
ⅵ)chg,chh中甲基化c附近的序列特征分析;
c.构建全基因组dna甲基化图谱
ⅰ.染色体水平的甲基化c碱基的密度分布;
ⅱ.scaffold的甲基化c碱基密度分布;
ⅲ.不同基因组区域的甲基化分布特征;
ⅳ基因组不同转录元件中的dna甲基化水平差异性甲基化区域分析。
进一步的,在所述文库构建后,进行数据的过滤。
本发明的有益效果是:
本发明公开了一种临床评估幼年型粒单核细胞白血病预后的检测方法,本发明通过检测dna甲基化水平预测jmml预后,为jmml预后评估及甲基化药物的治疗提供理论基础和试验基础,本研究专利在jmml的预后及治疗研究中具有广阔的应用前景。
附图说明
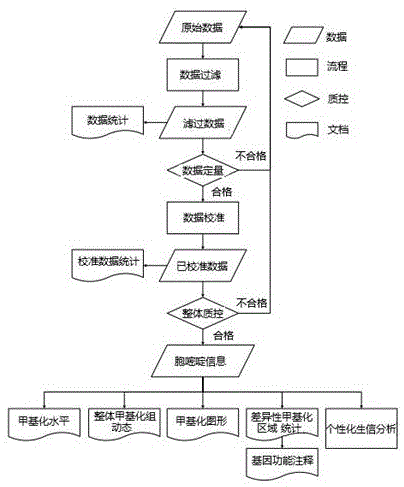
图1是本发明的信息分析流程图。
具体实施方式
为了使本技术领域的人员更好地理解本发明技术方案,下面结合说明书附图并通过具体实施方式来进一步说明本发明的技术方案。
一、文库构建策略:小片段文库。
1.1测序策略:测序平台为hiseqxten
1.2实验流程:
dna提取及质检
1)dna提取
使用基因组dna提取试剂盒对样本dna进行核酸提取,若所送样本为提取后的dna,则跳过此步。
基因组文库构建
1)样品打断
目前样品打断方法是covaris打断法,均可将样品dna打碎至100~300bp范围的片段。若所送样品为已打断至合适的大小的dna,则可以跳过此步。
2)dna片段末端修复、3’端加a碱基,连接测序接头;
3)采用zymoezdnamethylation-goldkit进行bisulfite处理;
4)处理后切胶回收,并进行文库片段大小选择;
5)合格的文库用于上机测序,若样本等级为不合格样本,不能进行下一步实验。
二、信息分析流程
2.1bs分析策略:比对
2.2信息分析流程图(图1)
2.3数据过滤
测序数据过滤:去除含adapter的reads;去除含n比例大于10%的reads;
去除低质量reads(质量值q≤5的碱基数占整条read的50%以上);去除duplication。
数据要求:q20%>85%。
三、生物信息分析内容
3.1标准信息分析
1.数据统计与质控
2.c碱基的甲基化水平分析
3.全基因组甲基化水平分布趋势
3.1甲基化c碱基中分布比例
3.2所有c的甲基化水平
3.3各条染色体中c的甲基化水平(该项分析只针对染色体信息齐全的物种)
3.4统计不同基因区域内cg、chg和chh中c的甲基化水平
3.5不同基因元件区域中cg、chg和chh中c的甲基化水平
3.6chg,chh中甲基化c附近的序列特征分析
四、全基因组dna甲基化图谱
4.1染色体水平的甲基化c碱基的密度分布(该项分析只针对染色体信息齐全的物种)
4.2scaffold的甲基化c碱基密度分布(该项分析只针对染色体信息不齐全的物种)
4.3不同基因组区域的甲基化分布特征
4.4基因组不同转录元件中的dna甲基化水平差异性甲基化区域(dmr)分析。
申请人声明,以上所述仅为本发明的具体实施方式,但本发明的保护范围并不局限于此,所属技术领域的技术人员应该明了,任何属于本技术领域的技术人员在本发明揭露的技术范围内,可轻易想到的变化或替换,均落在本发明的保护范围和公开范围之内。
- 还没有人留言评论。精彩留言会获得点赞!