用于血液肿瘤、淋巴瘤基因检测的探针组、试剂盒的制作方法
1.本发明涉及基因检测技术领域,尤其涉及一种用于血液肿瘤、淋巴瘤基因检测的探针组、包括该探针组的试剂盒,及采用该探针组、试剂盒提高血液肿瘤、淋巴瘤基因检测精确率的方法。
背景技术:
2.《2015年中国恶性肿瘤流行情况分析》数据显示,白血病和淋巴瘤居中国城乡肿瘤死亡的前十位,死亡率分别为53.4
‰
和52.1
‰
。血液肿瘤是一类具有高度异质性的疾病,目前国际上采用micm诊断体系即综合形态检测(morphology)、免疫检测(immunology)、细胞遗传学检测(cytogenetics)以及分子生物学分型(molecular biology)对患者进行临床诊断、分子分型及危险度分层。在此基础上选择最有效的治疗方案,从而实现血液肿瘤的精准诊疗。随着分子生物学及其相关技术的迅速发展,基于高通量测序技术的多基因组合检测在血液肿瘤的诊断分型、预后评估、治疗指导和微小残留监测(minimal residual disease,mrd)等方面的临床应用日益增多,已成为指导血液肿瘤临床诊疗和探索其分子机制的重要手段。2016年who造血与淋巴系统肿瘤分型指南、nccn指南等国内外权威指南共识都提出了与血液肿瘤诊断、鉴别诊断、预后判断、靶向治疗相关的基因突变。《二代测序技术在血液肿瘤中的应用中国专家共识(2018年版)》指出,基因突变检测是多种血液系统肿瘤诊断和辅助诊断、以及预后判断的重要依据。
3.采用高通量测序技术(ngs)对不同类型的血液肿瘤基因突变进行全面检测,能够为临床提供更为全面的分子遗传学信息,不仅有助于血液肿瘤患者的临床诊断和预后评估,同时还能为患者的个体化治疗方案提供指导。
4.目标区域是否能够100%覆盖,决定了血液肿瘤和淋巴瘤基于ngs的检测方法是否完备,即是否需要进行补齐。原因是血液肿瘤中的基因突变,除了目前已经报道的致病性明确的位点,还有一部分数量未知的新的致病位点,其位置可分布在相关基因整个编码区。目前国内市场检测血液肿瘤和淋巴瘤的ngs方法主要有两种建库方法:多重引物pcr方法,杂交捕获探针方法,不论哪种方法,罕有能够实现100%覆盖目标区域的富集建库方法。
技术实现要素:
5.为了解决现有针对血液肿瘤和淋巴瘤检测方法中,无法实现100%覆盖目标区域的问题,本发明的目的是提供一种用于血液肿瘤、淋巴瘤基因检测的探针组,采用该探针组,目标区域的覆盖度提升至100%,提升了检测的召回率;本发明还提供了一种包括该探针组的试剂盒;另外,本发明还将提供采用该探针组、试剂盒提高血液肿瘤、淋巴瘤基因检测精确率的方法,采用该方法进一步筛选去掉假阳性结果,精确率达到99%以上。
6.为了实现上述目的,本发明采用如下技术方案:
7.本发明的第一方面,提供一种用于血液肿瘤、淋巴瘤基因检测的探针组,基于血液肿瘤和淋巴瘤相关的163个基因组成的目标区域设计获得,其中捕获arid2、brca2、atg2b、
b2m、brca1、bcl2、apc、arid1b、ikzf1区域的探针序列如:seq id no:1-120所示。
8.本发明的第二方面,提供采用上述探针组的试剂盒。
9.其中,所述的试剂盒还包括文库构建试剂盒、杂交捕获试剂盒和pcr反应液。本发明的第三方面,提供一种提高血液肿瘤、淋巴瘤基因检测精确率的生物信息分析方法,包括如下步骤:
10.s1、核酸提取并构建样本库;
11.s2、将上述的探针组与样本库杂交,实现目标区域的富集;
12.s3、pcr反应、纯化及测序;
13.s4、变异位点检测,获得变异位点的合集;
14.s5、通过构建ml模型,去掉假阳性位点::
15.s51、随机获得多例血液肿瘤、淋巴瘤患者,采用步骤s1-s4获得变异位点合集,并统计与每个变异位点相关的多个参数;
16.s52、找出基因数据库中人群频率与变异位点合集中人群频率不在同一个数量级的位点,分为假阳性候选位点和假阴性候选位点,其中,假阳性候选位点为变异位点合集中人群频率大于基因数据库中人群频率的位点,且该位点不是已报道致病或可能致病的位点;其中,假阴性候选位点为变异位点合集中人群频率小于基因数据库中人群频率的位点;
17.s53、基于与每个变异位点相关的多个参数,采用支持向量机找出疑似假阳性候选位点和疑似假阴性候选位点,并进一步验证确认,得到假阳性位点,获得假阳位点的合集;
18.s54、将假阳性位点在待检样本的人群频率是否大于基因数据库中的人群频率,作为判断待检样品中假阳性位点的最终依据,以此制作pon文件,作为ml模型;
19.s6、将s4中变异位点的合集经过ml模型筛选,去掉假阳性位点,获得最终变异位点的合集,完成血液肿瘤、淋巴瘤基因检测。
20.其中,所述s51中的患者不少于1000例。
21.其中,所述s51中的参数包括覆盖深度,突变比例,正反向序列支持数,正反向变异序列支持数,基因型质量值,碱基质量值中位数,单倍型相位。
22.其中,所述s52中的基因数据库为gnomad-wes数据库。
23.其中,所述s53中与每个变异位点相关的参数不少于15个。
24.与现有技术相比,本发明实现的有益效果:本发明的探针组是基于血液肿瘤和淋巴瘤相关的163个基因组成的目标区域设计获得,通过对arid2、brca2、atg2b、b2m、brca1、bcl2、apc、arid1b、ikzf1共9个基因目标区域的捕获探针进行优化,获得120条探针序列,替换原有对应目标区域的探针序列,目标区域覆盖度由原来的98%提升至100%;本发明提高血液肿瘤、淋巴瘤基因检测精确率的方法是以上述设计的探针组捕获目标区域,经扩增、纯化、测序、常规变异检测后,引入ml模型,进一步筛选去掉假阳性位点,提高检测的精确率,尤其是血液肿瘤中低频(1%数量级)插入缺失变异的精确率,精确率可以达到99%以上。
附图说明
25.以下结合附图和具体实施方式来进一步详细说明本发明:
26.图1为191010-5重复4次实验对应的实验结果;
27.图2为191010-8重复4次实验对应的实验结果;
28.图3为191010-9重复4次实验对应的实验结果;
29.图4为191012-13重复4次实验对应的实验结果;
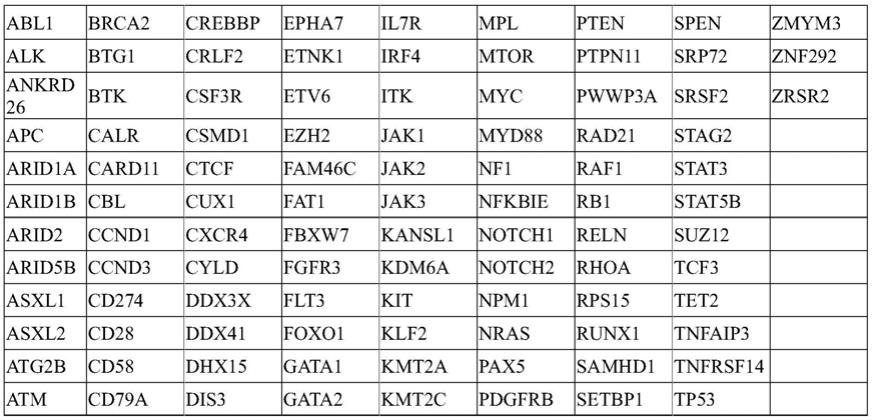
30.图5为191012-16重复4次实验对应的实验结果;
31.图6为验证191个插入缺失变异位点是否为假阳性位点的实验结果。
具体实施方式
32.在本文中所使用的术语“高通量测序技术”指的是第二代高通量测序技术及之后发展的更高通量的测序方法。第二代高通量测序平台包括但不限于illumina-solexa(miseq、hiseq-2000、hiseq-2500、hiseq x ten等)、abi-solid和roche-454测序平台等。随着测序技术的不断发展,本领域技术人员能够理解的其他测序平台也可采用。
33.精确率(precision)的公式是p=tp/(tp+fp),它计算的是所有"正确被检索的结果(tp)"占所有"实际被检索到的(tp+fp)"的比例.
34.召回率(recall)的公式是r=tp/(tp+fn),它计算的是所有"正确被检索的结果(tp)"占所有"应该检索到的结果(tp+fn)"的比例.
35.其中tp:真实阳性位点被正确检出;fp:非真实位点被错误检出;fn:真实位点未被正确检出。
36.本发明探针组的设计基于163个基因目标区域,包括与血液肿瘤和淋巴瘤相关的163个基因的全编码区和可变剪接区。
37.163个基因目标区域涉及与急性髓系白血病相关的58个基因,覆盖范围为58个基因的全编码区域和ankrd26、myc基因的utr区域,包括点突变、小片段插入缺失等变异类型;与急性淋巴细胞白血病相关的43个基因,覆盖范围为43个基因的全编码区域,包括点突变、小片段插入缺失等变异类型;与慢性髓性白血病相关的35个基因,覆盖范围为35个基因的全编码区域和myc基因的utr区域,包括点突变、小片段插入缺失等变异类型;与慢性淋巴细胞白血病相关的52个基因,覆盖范围为52个基因的全编码区域和bcl6、myc基因的utr区域,包括点突变、小片段插入缺失等变异类型;与骨髓增生异常综合征、骨髓增生异常综合征/骨髓增殖性肿瘤相关的46个基因,覆盖范围为46个基因的全编码区域,包括点突变、小片段插入缺失等变异类型;与骨髓增殖性肿瘤相关的37个基因,覆盖范围为37个基因的全编码区域,包括点突变、小片段插入缺失等变异类型;与造血系统早期克隆性造血病变相关的23个基因,覆盖范围为23个基因的全编码区域,包括点突变、小片段插入缺失等变异类型;与多发性骨髓瘤相关的55个基因,覆盖范围为55个基因的全编码区域,bcl6、ccnd1和myc基因的utr区域,包括点突变、小片段插入缺失等变异类型;与b细胞淋巴瘤相关的36个基因和t/nk细胞淋巴瘤相关的26个基因,覆盖范围为62个基因的全编码区域,bcl6、ccnd1基因的utr区域,包括点突变、小片段插入缺失等变异类型;与血液系统肿瘤分子分型、靶向药物、预后评估相关的163个基因,覆盖范围为163个基因的全编码区域,包括点突变、小片段插入缺失等变异类型。
38.163个基因目标区域对应的基因名称如表1所示:
39.表1
[0040][0041][0042]
本领域技术人员知晓:捕获的特异性受各种因素影响,如捕获探针的设计不佳,捕获条件不理想,基因组dna中重复序列的封闭不充分及基因组dna与捕获探针的比例不合适等因素都会影响捕获的特异性、敏感性、测序覆盖率等诸多结果。为了实现目标基因的高度富集和低脱靶率,本领域技术人员需要对探针的类型、长度、序列等进行大量实验摸索,需要通过创造性的探索工作才能够得到最佳的探针组合。基于已知目标区域设计的捕获探针,是否能够获得优异的特异性、敏感性和测序覆盖率,对于本领域技术人员来说是无法预期的。同时,对于产生突变的样本进行检测时,由于突变样本在组织样本中所占的比例会因个体而不同,因此,如果突变样本的丰度较低时,较容易导致的问题是探针往往无法准确地与突变的片段杂交,而导致检测的灵敏度低,这也需要对探针序列进行试验摸索。
[0043]
本发明采用现有技术共设计了8956条探针序列,但目标区域覆盖度无法达到100%,申请人通过反复试验发现,当对arid2、brca2、atg2b、b2m、brca1、bcl2、apc、arid1b、ikzf1共9个基因目标区域的捕获探针进行优化,获得120条探针序列,替换原有对应目标区域的探针序列,目标区域覆盖度由原来的98%提升至100%。
[0044]
使用在线设计工具https://sg.idtdna.com/site/order/ngs设计杂交捕获探针,探针的长度为120bp。考虑多态性位点snp,以及序列复杂度,如gc含量,重复序列,同源序列,以及血液肿瘤、淋巴瘤中已知变异位点,增加这些区域的探针密度,只需要完成注册,然后提交163个基因标志“gene symbol”,就可以得到8956条常规探针。
[0045]
以设计的8956条探针进行试验时,目标区域的覆盖度无法实现100%,本技术的发明人随机选择10个血液肿瘤样本,按照相同的实验和测序流程,得到10个样本的数据。统计其覆盖深度,均一性等质控指标,发现arid2、brca2、atg2b、b2m、brca1、bcl2、apc、arid1b、
ikzf1 9个基因10kbp区域没有足够的测序数据覆盖,分析其原因,主要是gc含量偏离目标区域,以及同源序列的存在。本技术的发明人反复调整这些区域探针的位置和长度,并修改探针上错配碱基的数量和位置,最后获得120条探针序列,实现了目标区域的100%覆盖。
[0046]
一、核酸提取
[0047]
1、样本获取来源:骨髓、外周血和淋巴瘤组织。
[0048]
2、核酸提取:采用商业化公司提取试剂盒,按说明书指示方法操作,环境温度:20~25℃,环境湿度:40%~60%。
[0049]
使用分光光度定量仪以及凝胶电泳系统检测提取的dna质量和浓度,经琼脂糖凝胶电泳检测无严重降解;ffpe dna,片段长度>500bp,od260/od280=1.8~2.0;od260/od230=2.0~2.5为合格。
[0050]
二、文库构建
[0051]
如下文库构建过程采用商业化的文库构建试剂盒,按照说明书指示方法进行如下操作。
[0052]
2.1、样本片段化
[0053]
1)打断仪准备
[0054]
①
提前30min打开打断仪,预冷到7℃。
[0055]
②
水槽内放超纯水,水位填充到12。
[0056]
③
依次打开制冷水浴电源、温度控制开关,covari、电脑上的软件sonolab,将水浴温度设定为5-8℃。
[0057]
④
按照表2进行设置打断条件(命名为163ngs),获得150-350bp片段大小的dna片段。
[0058]
表2
[0059]
目标峰值200峰值入射功率(w)175工作因数10%每次打断周期200时间(s)210
[0060]
2)样本准备
[0061]
①
1ug dna用te缓冲液稀释到130ul(0.2ml pcr管内),涡旋混匀,离心。
[0062]
②
转移至打断仪打断管内。
[0063]
3)进行打断
[0064]
按照设置好的163ngs条件进行打断。
[0065]
4)关打断仪
[0066]
抬起传感器,片刻后,依次关软件、打断仪、制冷水浴温度控制、制冷水浴。清空水浴,倒扣晾干。
[0067]
5)使用qubit荧光定量仪进行定量。
[0068]
2.2末端修复
[0069]
1)取出末端修复加a尾缓冲液(end repair&a-tailing buffer)和末端修复加a尾酶(end repair&a-tailing enzyme)置于冰上自然融解,混合均匀,瞬时离心备用。
[0070]
2)取打断后的dna 200ng进行建库。
[0071]
3)按照表3,在置于冰上的0.2ml pcr管中进行反应体系配制:
[0072]
表3
[0073]
组分体积已片段化的dna40μl末端修复加a尾缓冲液6μl末端修复加a尾酶4μl总计50μl
[0074]
注意:如片段化dna不足40μl,可用不含核酸酶的水补足至40μl。
[0075]
4)混合均匀,瞬时离心使全部反应液置于pcr管底部。
[0076]
5)在pcr仪上启动如表4的反应程序,等温度稳定至20℃时将反应管放进pcr仪:
[0077]
表4
[0078]
温度时间20℃30min65℃30min10℃hold
[0079]
2.3接头连接
[0080]
1)取出连接缓冲液(ligation buffer)和dna连接酶(dna ligase)置于冰上自然融解,混合均匀,瞬时离心备用。
[0081]
2)从pcr仪上取出末端修复后的pcr反应管,置于冰上,按照表5进行接头连接反应体系配制:
[0082]
表5
[0083]
组分体积步骤2.2反应产物50μl双标签接头(nanoprep
tm udi adapter,15μm)2μl连接缓冲液26μldna连接酶2μl总计80μl
[0084]
注意:按表5体系顺序依次加入试剂。
[0085]
3)混合均匀,瞬时离心使全部反应液置于pcr管底部。
[0086]
4)在pcr仪上启动如表6反应程序,等温度稳定至20℃时将反应管放进pcr仪:
[0087]
表6
[0088]
温度时间20℃15min4℃hold
[0089]
注意:此程序无需热盖。
[0090]
5)将未使用完的试剂放回-20℃保存。
[0091]
2.4片段筛选(使用片段单筛)
[0092]
1)提前将nanoprep
tm sp beads取出涡旋混匀,室温平衡30min后使用。
[0093]
2)向2.3连接体系pcr管中加入40μl nanoprep
tm sp beads,混合均匀,25℃孵育5~10min。
[0094]
3)将pcr管瞬时离心后放置于磁力架上5min至液体完全澄清,使用移液器吸取移弃上清。
[0095]
4)沿pcr管侧壁缓慢加入150μl 80%乙醇,注意勿扰动磁珠,静置30s,使用移液器吸取移弃上清。
[0096]
5)重复步骤4)1次。
[0097]
6)将pcr管瞬时离心后放置于磁力架上,使用10μl吸头移去少量残留乙醇,注意勿吸到磁珠。
[0098]
7)打开pcr管管盖,并于室温静置约5min,至乙醇挥发完全。
[0099]
注意:切勿过分干燥,否则会降低得率。
[0100]
8)移出pcr管,向pcr管中加入21μl nuclease free water(不含核酸酶的水),将磁珠悬浮均匀,25℃孵育2min。
[0101]
9)将pcr管瞬时离心后放置于磁力架上2min至液体完全澄清,使用移液器吸取20μl上清,并转移至1个新的0.2ml pcr管中,置于冰上备用。
[0102]
2.5 pcr扩增
[0103]
1)取出2
×
hifi pcr反应混合物和扩增引物混合物置于冰上自然融解,混合均匀,瞬时离心备用。
[0104]
2)按照表7在置于冰上的pcr管中进行反应体系配制:
[0105]
表7
[0106][0107][0108]
3)将pcr管放入pcr仪中启动表8程序:
[0109]
表8
[0110][0111]
4)将未使用完的试剂放回-20℃保存。
[0112]
2.6文库纯化、定量和质检
[0113]
1)向pcr管加入50μl的nanoprep
tm sp beads混合均匀,25℃孵育5~10min。
[0114]
2)将pcr管瞬时离心后放置于磁力架上5min,至液体完全澄清,使用移液器吸取移弃上清。
[0115]
3)沿pcr管侧壁缓慢加入150μl 80%乙醇,注意勿扰动磁珠,静置30s,使用移液器吸取移弃上清。
[0116]
4)重复步骤3)一次。
[0117]
5)pcr管瞬时离心,放置于磁力架上,使用10μl吸头移去少量残留乙醇,注意勿吸到磁珠。
[0118]
6)打开pcr管管盖,并于室温静置约5min,至乙醇挥发完全。
[0119]
注意:切勿过分干燥,否则会降低得率。
[0120]
7)向pcr管加入20μl te溶液使用移液器将磁珠悬浮均匀,25℃孵育2min。
[0121]
8)将pcr管瞬时离心后放置于磁力架上2min,至液体完全澄清,使用移液器小心将上清移取至一个新的0.2ml pcr管中进行保存,注意勿吸到磁珠。
[0122]
9)用qubit荧光定量仪对文库进行定量。
[0123]
10)使用2100片段分析仪器进行文库片段分布检测。
[0124]
三、探针捕获
[0125]
3.1 dna与捕获探针杂交
[0126]
3.1.1文库杂交
[0127]
1)准备工作
[0128]
a、提前取出杂交和清洗试剂盒(hybridization and wash kit)中的杂交试剂2
×
hybridization buffer(杂交缓冲液)和hybridization buffer enhancer(杂交缓冲增强剂)于室温融解。
[0129]
b、提前打开真空浓缩仪电源预热,调节温度为60℃。
[0130]
2)真空浓缩法
[0131]
a、进行多文库混合杂交捕获时,具体的杂交文库混合方法如下表9:
[0132]
表9
[0133][0134]
b、按照表9将各组分混合于一个0.2/1.5ml的低吸附离心管中,涡旋混匀,瞬时离心。
[0135]
c、将离心管放入提前预热至60℃的真空浓缩仪中干燥。
[0136]
d、待全部液体至完全干燥后,将离心管密封后备用。
[0137]
e、取出hm163 research panel v1.0(血液肿瘤淋巴瘤相关163个基因的杂交捕获探针)在冰上自然融解,使用后按需小量分装。
[0138]
f、根据表10配制杂交反应液,使用移液器混合均匀后加入到已经真空浓缩干燥的离心管底部,使用移液器轻柔的吹吸混匀15-20次,瞬时离心,25℃孵育5-10min。
[0139]
表10
[0140]
组分体积2
×
杂交缓冲液8.5μl杂交缓冲增强剂2.7μl无核酸酶的水1.8μlhm163 research panel v1.04μl总计17μl
[0141]
g、涡旋混匀杂交反应混合液,瞬时离心后,将离心管中的全部17μl杂交反应混合液转移至一个新的0.2mlpcr管中,瞬时离心,放在pcr仪中,启动如表11杂交程序:
[0142]
表11
[0143][0144][0145]
注意:洗脱程序中热盖温度务必设置为70℃。
[0146]
3.1.2文库洗脱
[0147]
1)准备工作
[0148]
a、取出杂交和清洗试剂盒(hybridization and wash kit)中的其他试剂室温自然融解,涡旋混合均匀。
[0149]
b、dynabeads
tm m-270链霉亲和素磁珠(dynabeads
tm m-270 streptavidin beads)涡旋混合均匀,室温平衡30min后方可进行链霉亲和素磁珠的清洗和捕获步骤。
[0150]
2)试剂配制
[0151]
a、洗脱缓冲液配制
[0152]
根据表12配制洗脱缓冲液的1
×
工作液:
[0153]
表12
[0154]
组分名称无核酸酶的水/μl洗脱缓冲液/μl总计/μl2
×
磁珠清洗缓冲液16016032010
×
清洗缓冲液i2522828010
×
清洗缓冲液ⅱ1441616010
×
清洗缓冲液ⅲ1441616010
×
加强清洗缓冲液28832320
[0155]
将配制好的以下两种缓冲液按以下体积分装至0.2mpcr管中:
[0156]
2份160μl 1
×
加强清洗缓冲液
[0157]
110μl 1
×
清洗缓冲液i
[0158]
注意:剩余的1
×
清洗缓冲液i不要丢弃,将用于后续室温洗脱实验操作中。
[0159]
上述两种热洗脱缓冲液至少要在65℃孵育15min。
[0160]
b、磁珠悬浮液配制
[0161]
表13
[0162][0163][0164]
3)链霉亲和素磁珠清洗
[0165]
a、将dynabeads
tm m-270链霉亲和素磁珠(dynabeads
tm m-270 streptavidin beads)涡旋混匀15s,确保完全混匀。吸取50μl m 270磁珠至1个1.5ml低吸附离心管中。
[0166]
b、向离心管中加入100μl 1
×
清洗缓冲液i,轻柔吹吸混匀10次,瞬时离心,置于磁力架上数分钟,待液体完全澄清,使用移液器移弃上清。将离心管从磁力架上移出。
[0167]
c、重复步骤b两次。
[0168]
d、向离心管中加入17μl磁珠悬浮液,轻柔吹吸混匀,将全部磁珠悬浮液转移至1个新的0.2ml低吸附pcr管中。
[0169]
注意:将含有悬浮捕获磁珠的0.2ml pcr管放入pcr仪65℃孵育5min
[0170]
4)链霉亲和素磁珠捕获
[0171]
a、16h杂交反应后,调节pcr仪进入到洗脱程序。
[0172]
注意:如果pcr仪不能分步设置热盖,可提前打开另外一台pcr仪运行洗脱程序,将
杂交管转移至新的pcr仪上。
[0173]
b、将重悬的链霉亲和素磁珠加入到杂交体系中,并使用移液器轻柔吹吸混匀或涡旋混匀。注意:此步骤务必使用低吸附吸头,操作要迅速。
[0174]
c、65℃孵育45min,每10-12min轻柔涡旋一次,确保磁珠完全重悬。
[0175]
5)热洗脱
[0176]
注意:热洗脱过程操作要迅速;吹吸混匀过程中尽量避免产生气泡。
[0177]
a、孵育结束后从pcr仪上取下pcr管,并向其中加入100μl 65℃1
×
清洗缓冲液i,吹吸混匀含有磁珠的杂交体系。
[0178]
b、将pcr管置于磁力架上1min,待液体完全澄清后,使用移液器吸取移弃上清。
[0179]
c、将pcr管从磁力架上移出,加入150μl 65℃10
×
加强清洗缓冲液,轻柔吹吸10次,放进pcr仪中65℃孵育5min。
[0180]
d、重复步骤b和c各一次。
[0181]
6)室温洗脱
[0182]
a、将pcr管瞬时离心后置于磁力架上1min,待液体完全澄清后吸取移弃上清,加入150μl室温1
×
清洗缓冲液i。
[0183]
b、涡旋混匀,室温孵育2min,期间涡旋混匀30s后静置30s,交替进行,确保充分混匀。
[0184]
c、将pcr管瞬时离心后置于磁力架上1min,待液体完全澄清后吸取移弃上清,加入150μl室温1
×
清洗缓冲液ⅱ。
[0185]
d、涡旋混匀,室温孵育2min,期间涡旋混匀30s后静置30s,交替进行,确保充分混匀。
[0186]
e、将pcr管瞬时离心后置于磁力架上1min,待液体完全澄清后吸取移弃上清,加入150μl室温1
×
清洗缓冲液ⅲ。
[0187]
f、涡旋混匀,室温孵育2min,期间涡旋混匀30s后静置30s,交替进行,确保充分混匀。
[0188]
g、将pcr管瞬时离心后置于磁力架上1min,待液体完全澄清后吸取移弃上清,从磁力架上移下样本管,样本朝外瞬时离心,再次放到磁力架上,弃去残留液体。
[0189]
h、将离心管从磁力架上取下,加入20μl无核酸酶的水,吹吸10次重悬磁珠,确保混合均匀,转移全部液体至一个新的0.2ml pcr管中。
[0190]
3.1.3 pcr扩增
[0191]
1)取出2
×
hifi pcr反应混合物和扩增引物混合物(nanoprep
tm dna library preparation kit(for)e96,纳昂达科技)于冰上自然融解,使用移液器或旋涡混匀仪轻柔混合均匀,瞬时离心备用。
[0192]
2)按照表14在置于冰上的pcr管中进行反应体系配制:
[0193]
表14
[0194]
组分体积2
×
hifi pcr反应混合物25μl扩增引物混合物1.25μl不含核酸酶的水3.75μl
带捕获dna的磁珠20μl总体积50μl
[0195]
3)将pcr管放入pcr仪中启动表15程序,热盖温度设定为105℃:
[0196]
表15
[0197][0198][0199]
表16
[0200]
混杂文库数(文库总量)1杂(500ng)4杂(2ug)8杂(4ug)12杂(6ug)扩增循环数13111010
[0201]
3.1.4文库纯化和定量
[0202]
注:nanoprep
tm sp beads需要提前取出,涡旋混合均匀,室温平衡至少30min后方可使用。使用新鲜配制的80%乙醇。
[0203]
1)向pcr管中加入75ul的nanoprep
tm sp beads,使用移液器或旋涡混匀仪混合均匀,25℃孵育5-10min。
[0204]
2)将pcr管瞬时离心后置于磁力架上5min,待液体完全澄清后,使用移液器吸取移弃上清。
[0205]
3)沿pcr管侧壁缓慢加入125μl 80%乙醇,注意勿扰动磁珠,静置1min,使用移液器吸取移弃上清。
[0206]
4)将pcr管瞬时离心后置于磁力架上,使用10ul吸头移去少量残留乙醇,注意勿吸到磁珠。
[0207]
5)打开pcr管管盖,并于室温静置约1-3min,至乙醇挥发完全。
[0208]
注:切勿过分干燥,否则影响最终得率。
[0209]
6)将pcr管从磁力架上移出,加入22ul te溶液,使用移液器或涡旋混匀仪将磁珠悬浮均匀,25℃孵育5min。
[0210]
7)将pcr管从磁力架上移出,加入22ul te溶液,使用移液器或涡旋混匀仪将磁珠悬浮均匀,25℃孵育5min。
[0211]
8)将pcr管瞬时离心后置于磁力架上1-2min,待液体完全澄清后,使用移液器小心将上清转移至一个新的0.2ml pcr管或1.5ml离心管中进行保存,注意勿吸到磁珠。
[0212]
9)使用qubit荧光定量仪对文库进行定量。
[0213]
10)使用2100片段分析仪器进行文库片段分布检测。
[0214]
四、测序及变异位点检测
[0215]
采用高通量测序技术进行测序操作。
[0216]
采用常规变异检测方法gatk mutect2检测出所有变异位点,根据变异位点获知待检样品所属者是否患血液肿瘤、淋巴瘤。
[0217]
实施例1
[0218]
随机抽取2组血液肿瘤、淋巴瘤患者的骨髓作为实验样本,每组实验样本为8个,分为a组和b组,a组采用常规方法设计(8956条探针)的探针组进行测试,b组将arid2、brca2、atg2b、b2m、brca1、bcl2、apc、arid1b、ikzf1对应的探针替换为优化后的120条探针进行测试,测试结果如表17和18所示,其中表17为未替换的覆盖度数据,表18为替换后的覆盖度数据。
[0219]
表17
[0220]
a组样本覆盖度(%)test00197.96test00297.72test00397.6test00498.2test00597.67test00697.83test00797.61test00898.45
[0221]
表18
[0222][0223]
如表17和18可知,采用常规方法设计的8956条探针组成的探针组,其捕获目标区域的覆盖度为98%,而将arid2、brca2、atg2b、b2m、brca1、bcl2、apc、arid1b、ikzf1对应的探针替换为优化后的120条探针时,其捕获目标区域的覆盖度达到100%。
[0224]
实施例2血液肿瘤、淋巴瘤基因检测
[0225]
采用上述步骤,选择5个已知阳性突变样本(191010-5,191010-8,191010-9,191012-13,191012-16),3个阴性对照样本(ntc-1,ntc-2,ntc-3),分别重复4次实验,实验
结果如图1、图2、图3、图4、图5,其中图1中1-1、1-2、1-3、1-4为191010-5重复4次实验的结果;图2中2-1、2-2、2-3、2-4为191010-8重复4次实验的结果;图3中3-1、3-2、3-3、3-4为191010-9重复4次实验的结果;图4中4-1、4-2、4-3、4-4为191012-13重复4次实验的结果;图5中5-1、5-2、5-3、5-4为191012-16重复4次实验的结果。
[0226]
如图1、图2、图3、图4、图5可知,191010-5,191010-8,191010-9,191012-13,191012-16重复4次,检测结果与已知结果完全一致;而阴性对照样本ntc-1,ntc-2,ntc-3没有检测出阴性对照样本,重复4次,结果一致。
[0227]
可见,采用本发明设计的探针捕获目标基因区域,进行血液肿瘤、淋巴瘤基因检测,有效平均深度达到1500x,且结果稳定、可靠。
[0228]
实施例3
[0229]
为了提升测试结果的精确率,降低假阳性,在常规变异位点检测的基础上,通过建立ml模型,筛选去掉假阳性位点,获得最终的变异位点合集。
[0230]
具体建模方法如下:
[0231]
1)随机获得1000例血液肿瘤、淋巴瘤患者,通过上述方法获得变异位点合集,并统计与每个变异位点相关的多个参数,如覆盖深度,突变比例,正反向序列支持数,正反向变异序列支持数,基因型质量值,碱基质量值中位数,单倍型相位;
[0232]
2)找出gnomad-wes数据库人群频率与变异位点合集中人群频率不在同一个数量级的位点,如人群频率低于0.1%,但1000例数据中检测出频率高于1%,这些位点在去除已报道致病、可能致病位点后,是假阳性位点的候选位点;人群频率高于5%,但1000例数据中检测出频率低于0.5%,这些位点是假阴性位点的候选位点;
[0233]
3)基于与每个变异位点相关的多个参数,将假阳性和假阴性候选位点作为队列,并分别等分为两组,一组用于训练,一组用于验证;用于训练的向量为15个参数,验证结果与原来分组不一致的位点,挑选出来,找到原始样本,使用一代验证或者荧光定量的方法,确认位点是否是假阳性位点,获得假阳位点的合集;
[0234]
4)将假阳位点在待检样本的人群频率是否大于基因数据库中的人群频率,作为判断假阳位点的最终依据,以此制作pon文件,作为ml模型。
[0235]
其中,上述1000例样本是随机选择的血液肿瘤、淋巴瘤样本,在未通过ml模型训练前,平均每个样本得到的变异位点数目(突变比例大于等于2%)约500个位点。经过ml模型训练后,其中5%的变异位点被认定为假阳性位点而被去掉,这些位点的特征之一是1-3bp的插入或者缺失,变异比例不超过10%,存在严重的链偏好性,即只有一个方向的测序支持序列;另一个特征是,pon文件记录10%以上的样本都存在这些变异,而这些变异并非已知的致病热点,经过ml模型认定为假阳性位点。而未经ml模型剔除的95%的变异位点中,选取191个小插入缺失变异,来验证通过ml模型筛选后测试结果的精确率,测试结果如图6所示。
[0236]
如图6中所示,191个小插入缺失变异中,只有一个为假阳性位点,精确率达到99.47%。
[0237]
上述的具体实施方式只是示例性的,是为了更好地使本领域技术人员能够理解本专利,不能理解为是对本专利包括范围的限制;只要是根据本专利所揭示精神的所作的任何等同变更或修饰,均落入本专利包括的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1