一种在水稻基因打靶中识别特异位点的SpCas9-NRRH突变体及其应用
本发明涉及生物技术和植物基因工程技术领域。具体而言,本发明涉及一种在基因打靶中识别特异位点的spcas9-nrrh突变体及其在水稻基因打靶方面的应用。
背景技术:
crispr/cas(clusteredregularlyinterspacedshortpalindromicrepeats/crispr-associatedproteins)系统作为一种重要的基因编辑工具,自诞生以来被广泛应用于作物的性状改良。crispr/cas9基因编辑技术主要包括两大核心内容:1)构建cas9/sgrna表达载体,将载体导入受体细胞表达发挥编辑作用;2)将表达纯化的cas9蛋白与合成的sgrna导入受体细胞发挥编辑作用。来源于链球菌streptococcuspyogenes的cas9蛋白spcas9最先被应用于基因编辑,该蛋白含有一个ruvc-like结构域和一个hnh核酸酶结构域,两者分别在靶dna的pam(protospacerasjacentmotif)序列“ngg”上游3nt处对dna双链进行切割,形成平末端。在真核系统中,需要在cas9蛋白中添加一段核定位信号以保证该蛋白进入细胞核正常发挥功能。sgrna是一段具有特定结构的单链rna,其5'端约20个碱基与靶dna互补配对结合,引导cas9/sgrna复合物对相应位点进行切割,决定编辑位点特异性。
目前,常用的源自化脓链球菌的spcas9主要识别5'-ngg-3'pam,通过人工改造spcas9蛋白的pam-interacting(pi)结构域的序列,使其可以识别更多种类的pam类型,比如spcas9-vqr识别5'-nag-3'pam,spcas9-ng识别5'-ng-3'pam。现有的高突变效率的spcas9基因数量有限,大部分突变体还是依赖于含有g的pam序列。但是这些pam扩展的编辑工具仍无法完全满足在实际应用过程中遇到的各种编辑情况。因此,如果能开发出一种在植物,尤其是水稻中能识别不同pam序列特别是非g的pam序列且高效率的基因组编辑技术,扩宽现有基因组编辑技术的编辑范围,将对于植物功能基因组学研究和作物分子育种具有重要的促进作用。
技术实现要素:
针对上述问题,本发明提供了一种在水稻基因打靶中识别nrrh的pam序列,且具有高效切割的spcas9-nrrh突变体。其中,n为a、t、c或g;r为a或t;h为a,t或c。其能够识别不同pam序列,尤其是非g的pam序列,并且具有高剪切效率。
具体而言,在第一个方面,本发明提供一种在水稻基因打靶中识别特异位点的spcas9-nrrh突变体,其特征在于,所述spcas9-nrrh突变体为a1)、a2)或a3):
a1)其氨基酸序列是序列表中seqidno.1所示的蛋白质;
a2)在a1)所示蛋白质的n末端添加一个甲硫氨酸残基,得到的蛋白质;
a3)在a1)所示蛋白质的n端或/和c端连接标签得到的融合蛋白质。
另一方面,本发明提供一种在水稻基因打靶中识别特异位点的spcas9-nrrh突变体基因,其特征在于,所述突变体基因包括:
b1)序列表中seqidno.2所示的核苷酸序列;
b2)与b1)限定的核苷酸序列具有75%或75%以上同一性,且编码权利要求1中所述spcas9-nrrh突变体的核苷酸序列;
b3)与b1)或b2)限定的核苷酸序列杂交,且编码spcas9-nrrh突变体的核苷酸序列。
另一方面,本发明提供一种表达盒,其特征在于,所述表达盒中包含所述的spcas9-nrrh变体基因。
另一个方面,本发明提供一种spcas9-nrrh的表达盒,具有式i结构:p-a-b-c-d
(i);其中,
(a)p为启动子;
(b)a为无或核定位信号序列nls;
(c)b为spcas9-nrrh基因序列;
(d)c为无或核定位信号序列nls;
(e)d为终止子。
附加条件是a和c中至多1个为无。
启动子包括但不限于ubi、actin、35s启动子,优选为ubi启动子。
另一个方面,本发明提供一种表达载体,包含权利上述的spcas9-nrrh突变体的表达盒,还包含了一个sgrna转录单元,所述sgrna靶向所述靶点序列;sgrna识别靶点序列的pam序列为nrrh,n为a、t、c或g;r为a或t;h为a,t或c。
该植物表达载体的构建方法是利用noti/saci酶切位点,用noti/saci酶切phun600载体并回收,由于合成的spcas9-nrrh序列两端加有noti/saci酶切位点,可以利用t4连接酶将spcas9-nrrh连接到phun600载体,得到植物表达载体phunch。
另一方面,本发明在表达载体的基础上,根据实验的实际需要,构建相应的基因打靶载体。
另一方面,本发明提供一种上述基因、表达盒或载体的应用,其特征在于,所述应用包括利用所述spcas9-nrrh突变体基因完成水稻体内dna双链的剪切,并在自身修复系统的作用下,获得带有突变位点的转基因植物或植物部分。
在另一个方面,本发明提供一种利用spcas9-nrrh突变体(phunch)表达载体(其含有所述spcas9-nrrh突变体基因),在表达载体的基础上只需进行简单的退火、酶切连接作用即可获得特异基因的打靶载体,将打靶载体导入水稻细胞的方法,包括下述步骤:
(1)将水稻种子去壳、灭菌后将胚分离出来,置于愈伤组织诱导培养基上以产生次级愈伤组织;
(2)将次级愈伤组织转移至新的愈伤组织诱导培养基预培养;
(3)将步骤(2)中获得的愈伤组织与携带spcas9-nrrh突变体的打靶载体的农杆菌接触15分钟;
(4)将步骤(3)的愈伤组织转移到上垫上三张无菌滤纸(加入2.5-3.5ml农杆菌悬浮培养基)的培养皿中,21-23℃培养48小时;
(5)将步骤(4)的愈伤组织置于前筛选培养基上培养5-7天;
(6)将步骤(5)的愈伤组织转移筛选培养基上,以获得抗性愈伤组织;
(7)将抗性愈伤组织转移到分化再生培养基中分化成苗;以及
(8)将步骤(7)的苗转移到生根培养基中生根。
其中所述步骤(1)中的种子是成熟种子;所述步骤(1)、(2)中的诱导培养基是说明书表1所列出的诱导培养基;所述步骤(3)中的与农杆菌接触是将愈伤组织浸泡在所述农杆菌悬浮液中;所述步骤(4)中的农杆菌悬浮培养基是说明书表1所列出的悬浮培养基;所述步骤(5)中的前筛选培养基是说明书表1所列出的前筛选培养基;所述步骤(6)中的筛选培养基是说明书表1所列出的筛选培养基;所述步骤(7)中的分化再生培养基是说明书表1所列出的分化再生培养基;所述步骤(8)中的生根培养基是说明书表1所列出的生根培养基。
在优选的实施方案中,所述水稻是粳稻,更优选地,所述水稻是粳稻日本晴。
表1培养基的示例性配方
表格中所提到的“n6majors”指的是,该n6majors中[no3-]/[nh4+]=40mm/10mm。
在优选的实施方案中,所述spcas9-nrrh突变体基因的核苷酸序列为seqidno:2所示的核苷酸序列,具体如:
atggacaagaagtactccatcggcctcgacatcggcaccaattctgttggctgggccgtgatcaccgacgagtacaaggtgccgtccaagaagttcaaggtcctcggcaacaccgaccgccactccatcaagaagaatctcatcggcgccctgctgttcgactctggcgagacagccgaggctacaaggctcaagaggaccgctagacgcaggtacaccaggcgcaagaaccgcatctgctacctccaagagatcttctccaacgagatggccaaggtggacgacagcttcttccacaggctcgaggagagcttcctcgtcgaggaggacaagaagcacgagcgccatccgatcttcggcaacatcgtggatgaggtggcctaccacgagaagtacccgaccatctaccacctccgcaagaagctcgtcgactccaccgataaggccgacctcaggctcatctacctcgccctcgcccacatgatcaagttcaggggccacttcctcatcgagggcgacctcaacccggacaactccgatgtggacaagctgttcatccagctcgtgcagacctacaaccagctgttcgaggagaacccgatcaacgcctctggcgttgacgccaaggctattctctctgccaggctctctaagtcccgcaggctcgagaatctgatcgcccaacttccgggcgagaagaagaatggcctcttcggcaacctgatcgccctctctcttggcctcaccccgaacttcaagtccaacttcgacctcgccgaggacgccaagctccagctttccaaggacacctacgacgacgacctcgacaatctcctcgcccagattggcgatcagtacgccgatctgttcctcgccgccaagaatctctccgacgccatcctcctcagcgacatcctcagggtgaacaccgagatcaccaaggccccactctccgcctccatggtgaagaggtacgacgagcaccaccaggacctcacactcctcaaggccctcgtgagacagcagctcccagagaagtacaaggagatcttcttcgaccagtccaagaacggctacgccggctacatcgatggcggcgcttctcaagaggagttctacaagttcatcaagccgatcctcgagaagatggacggcaccgaggagctgctcgtgaagctcaatagagaggacctcctccgcaagcagcgcaccttcgataatggcattatcccgcaccagatccacctcggcgagcttcatgctatcctccgcaggcaaggcgacttctacccgttcctcaaggacaaccgcgagaagattgagaagatcctcaccttccgcatcccgtactacgtgggcccgctcgccaggggcaactccaggttcgcctggatgaccagaaagtccgaggagacaatcaccccctggaacttcgaggaggtggtggataagggcgcctctgcccagtctttcatcgagcgcatgaccaacttcgacaagaacctcccgaacgagaaggtgctcccgaagcactcactcctctacgagtacttcaccgtgtacaacgagctgaccaaggtgaagtacgtgaccgaggggatgaggaagccagctttccttagcggcgagcaaaagaaggccatcgtcgacctgctgttcaagaccaaccgcaaggtgaccgtgaagcagctcaaggaggactacttcaagaaaatcgagtgcttcgactccgtcgagatctccggcgtcgaggataggttcaatgcctccctcgggacctaccacgacctcctcaagattatcaaggacaaggacttcctcgacaacgaggagaacgaggacatcctcgaggacatcgtgctcaccctcaccctcttcgaggaccgcgagatgatcgaggagcgcctcaagacatacgcccacctcttcgacgacaaggtgatgaagcagctgaagcgcctgcgctataccggctggggcaggctctctaggaagctcatcaacggcatccgcgacaagcagtccggcaagacgatcctcgacttcctcaagtccgacggcttcgccaaccgcaacttcatgcagctcatccacgacgactccctcaccttcaaggaggacatccaaaaggcccaggtgtccggccaaggcgattccctccatgaacatatcgccaatctcgccggctccccggctatcaagaagggcattctccagaccgtgaaggtggtggacgagctggtgaaggtgatgggcggccacaagccagagaacatcgtgatcgagatggcccgcgagaaccagaccacacagaagggccaaaagaactcccgcgagcgcatgaagaggatcgaggagggcattaaggagctgggctcccagatcctcaaggagcacccagtcgagaacacccagctccagaacgagaagctctacctctactacctccagaacggccgcgacatgtacgtggaccaagagctggacatcaaccgcctctccgactacgacgtggaccatattgtgccgcagtccttcctgaaggacgactccatcgacaacaaggtgctcacccgctccgacaagaacaggggcaagtccgataacgtgccgtccgaagaggtcgtcaagaagatgaagaactactggcgccagctcctcaacgccaagctcatcacccagaggaagttcgacaacctcaccaaggccgagagaggcggcctttccgagcttgataaggccggcttcatcaagcgccagctcgtcgagacacgccagatcacaaagcacgtggcccagatcctcgactcccgcatgaacaccaagtacgacgagaacgacaagctcatccgcgaggtgaaggtcatcaccctcaagtccaagctcgtgtccgacttccgcaaggacttccagttctacaaggtgcgcgagatcaacaactaccaccacgcccacgacgcctacctcaatgccgtggtgggcacagccctcatcaagaagtacccaaagctcgagtccgagttcgtgtacggcgactacaaggtgtacgacgtgcgcaagatgatcgccaagtccgagcaagagatcggcaaggcgaccgccaagtacttcttctactccaacatcatgaatttcttcaagaccgagatcacgctcgccaacggcgagattaggaagaggccgctcatcgagacaaacggcgagacaggcgagatcgtgtgggacaagggcagggatttcgccacagtgcgcaaggtgctctccatgccgcaagtgaacatcgtgaagaagaccgaggttcagaccggcggcttctccaaggagtccatcctcccaaagggcaactccgacaagctgatcgcccgcaagaaggactgggacccgaagaagtatggcggcttcaactctccgaccgcggcctactctgtgctcgtggttgccaaggtcgagaagggcaagagcaagaagctcaagtccgtcaaggagctgctgggcatcacgatcatggagcgcagcagcttcgagaagaacccaatcggcttcctcgaggccaagggctacaaggaggtgaagaaggacctcatcatcaagctcccgaagtacagcctcttcgagcttgagaacggccgcaagagaatgctcgcctctgctggcgtgcttcataagggcaacgagcttgctctcccgtccaagtacgtgaacttcctctacctcgcctcccactacgagaagctcaagggctccccagaggacaacgagcaaaagcagctgttcgtcgagcagcacaagcactacctcgacgagatcatcgagcagatctccgagttctccaagcgcgtgatcctcgccgatgccaacctcgataaggtgctcagcgcctacaacaagcaccgcgataagccaattcgcgagcaggccgagaacatcatccacctcttcaccctcaccaacctcggcgtgccagccgccttcaagtacttcgacaccaccatcgacaaaaagcgctacacctctaccaaggaggttctcgacgccaccctcatccaccagtctatcacaggcctctacgagacacgcatcgacctctcacaactcggcggcgattga
本发明的发明人以水稻pds、badh2基因为靶基因,针对nrrh的pam序列的不同组合,选择了24个靶点,构建了24个系列靶向载体,并利用农杆菌转化方法将载体导入水稻愈伤中,利用spcas9-nrrh突变体成功获得了目的基因敲除的材料。由此可见,本发明提供的spcas9-nrrh突变体能够对nrrh的pam序列附近的靶标序列进行编辑,由于其扩充了spcas9识别的pam位点序列,所以扩大了crispr/cas9系统在水稻基因组中的编辑范围,具有重大的应用价值。
附图说明
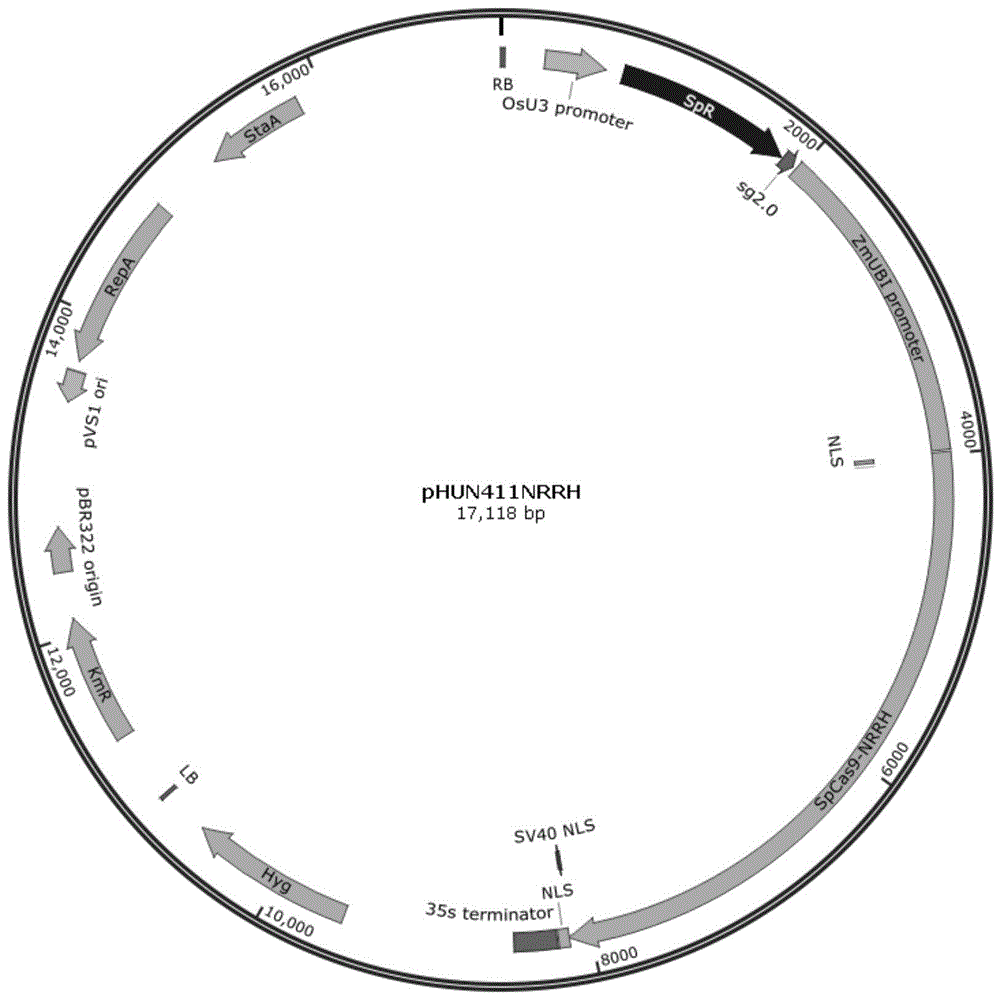
图1为phun411nrrh载体质粒示意图。
图2为spcas9-nrrh编辑系统的突变效率。
图3为转基因愈伤中部分位点的突变形式示例。
具体实施方式
以下结合附图叙述本发明的实施例。应该说明,下述实施例仅用于对本发明的示例性实现方式进行说明,而并非对本发明进行任何限制。本领域技术人员可以对本发明作出某些等同的改动和显而易见的改进。
在没有其他具体说明的情况下,下述具体实施方式中的操作均采用本领域通用的常规操作来进行。本领域技术人员可以很容易地从现有技术中获得关于这样的常规操作的教导,例如可以参照教科书sambrookanddavidrussell,molecularcloning:alaboratorymanual,3rded.,vols1,2;charlesnealstewart,alishertouraev,vitalycitovskyandtzvitzfira,planttransformationtechnologies等。下述实施例中所用的药材原料、试剂、材料等,如无特殊说明,均为市售购买产品。
实施例1—spcas9-nrrh突变体基因的获得
本申请的spcas9-nrrh突变体编码蛋白序列如seqidno:1所示。它与蛋白质spcas9的不同在于:第322位的氨基酸由i变为v,第409位的氨基酸由s变为i,第427位的氨基酸由e变为g,第654位的氨基酸由r变为l,第753位的氨基酸由r变为g,第1114位的氨基酸由r变为g,第1135位的氨基酸由d变为n,第1180位的氨基酸由d变为g,第1218位的氨基酸由g变为s,第1219位的氨基酸由e变为v,第1221位的氨基酸由q变为h,第1249位的氨基酸由p变为s,第1253位的氨基酸由e变为k,第1321位的氨基酸由p变为s,第1322位的氨基酸由d变为g,第1335位的氨基酸由r变为l。
本申请的发明人基于突变后的氨基酸序列进行对应的不同核苷酸序列的水稻应用尝试,
本申请的发明人通过尝试利用各种不同的方式对来自大肠杆菌的spcas9-nrrh基因进行改造,但是大部分序列在水稻应用过程中编辑效率低下,很多突变率不足5%,仅个别序列可以应用于水稻,申请人经过大量实验意外获得一条新的dna序列,发现其在22个pam靶标位点的编辑效率在7.3%~79.5%之间,极具应用价值。并给这个dna序列末端加上水稻偏好的终止密码子tga,形成一个新基因,该基因被命名为spcas9-nrrh,序列如seqidno:2所示。
将设计好的spcas9-nrrh基因送苏州金唯智生物科技有限公司合成后,连接于puc57-amp载体上,形成puc57-amp-spcas9-nrrh载体,并装载入大肠杆菌xl-blue菌株中。
实施例2——含有spcas9-nrrh基因植物打靶载体的构建
从上面含有puc57-amp-spcas9-nrrh载体的大肠杆菌xl-blue,用axygen质粒提取试剂盒中提取质粒,用noti/saci酶切,回收spcas9-nrrh片段。同时利用noti/saci酶对phun600进行线性化处理,回收phun600,将上述的spcas9-nrrh片段和phun600片段用t4连接酶(购于takara公司)进行连接,获得phun600-spcas9-nrrh,随后将spcas9匹配的sgrna表达框连在phun600-spcas9-nrrh载体上,得到植物表达载体,命名为phun411nrrh(图1)。
本发明的发明人以水稻pds、badh2基因为靶基因,针对nrrh的pam序列的不同组合,选择了24个靶点,靶点序列如表2所示。将靶点序列与phun411nrrh融合形成不同的phun411nrrh打靶载体共24个。利用冻融法将植物表达载体转入根癌农杆菌(agrobacteriumtumefaciens)eha105菌株中(安徽省农业科学院水稻研究所保存),用于遗传转化。
表2sgrna靶向序列及相应的pam序列
实施例3——phun411nrrh打靶载体的水稻遗传转化及突变体的获得
1、成熟胚愈伤组织的诱导和预培养
将日本晴(安徽省农业科学院水稻研究所保存)的成熟种子去壳,选取外观正常、洁净无霉斑的种子,用70%酒精,摇晃90sec,倒掉酒精;再用含tween20的50%次氯酸钠(原液有效氯浓度大于4%,每100毫升加入1滴tween20)溶液清洗种子,在摇床上晃动45min(180r/min)。倒掉次氯酸钠,无菌水洗5-10遍至无次氯酸钠气味,最后加入无菌水,30℃浸泡过夜。用手术刀片沿糊粉层分离胚,盾片朝上放置在诱导培养基(成分见表1)上,12粒/皿,30℃暗培养以诱导愈伤组织。
两周后出现球形、粗糙、浅黄色的次级愈伤组织,可以进行预培养操作,即将次级愈伤转至新的愈伤组织诱导培养基上,30℃暗培养预培养5天。预培养结束后,将状态良好、分裂旺盛的小颗粒用勺收集至50ml的无菌离心管中,用于农杆菌侵染。
2、农杆菌菌株的培养和悬浮液准备
将含有phun411nrrh打靶载体的农杆菌菌株eha105在含有50mg/l卡那霉素的lb平板上划线(成分见表1),28℃黑暗培养,24h后用无菌接种环将活化的农杆菌接种至新鲜的50mg/l卡那霉素的lb平板上,进行第二次活化,28℃黑暗培养过夜。在50ml的无菌离心管中加入20-30ml农杆菌悬浮培养基(成分见表1),用接种环将活化2次的农杆菌刮下,调整od660(opticaldensity660nm,660nm吸光值)至约0.10-0.25,室温静置30min以上。
3、侵染和共培养
向准备好的愈伤组织中(见步骤1),加农杆菌悬浮液,浸泡15min,其间不时轻轻晃动。浸泡结束后倒掉液体(尽量将液体滴净),用无菌滤纸吸去愈伤组织表面的多余的农杆菌菌液,并在超净台中用无菌风吹干。在100×25mm的一次性无菌培养皿垫上三张无菌滤纸,加入2.5ml农杆菌悬浮培养基,将吸干后的愈伤组织均匀分散在滤纸上,23℃黑暗培养48h。
4、前筛选和筛选培养
共培养结束后,将经共培养的愈伤组织均匀散布于前筛选培养基(成分见表1)中,30℃黑暗培养5天。前筛选培养结束后,将愈伤组织转至筛选培养基上(成分见表1),每个培养皿接25粒愈伤组织,30℃黑暗培养,2-3周后,抗性愈伤组织生长明显。
5、分子鉴定
以筛选15天新长出的一粒愈伤为检测样品。每个靶标载体取108粒愈伤,平均36粒愈伤为一个样品,一共三个重复。用ctab法进行dna小提。将所得到的基因组dna样品用于pcr分析。设计pcr引物用于扩增靶点附近的dna序列,长度约为180~500bp。引物序列如表3所示。将pcr组分首先在95℃保持5分钟,然后进行32个循环:94℃45秒、56℃45秒、72℃30秒,最后在72℃延伸10分钟。将pcr产物用于高通量的扩增子测序。所测结果与野生型序列进行比对。突变效率计算为:含有靶标突变的reads/总的reads数*100%。
表3用于扩增检测靶位点序列的pcr引物
在spcas9-nrrh和获得的转基因愈伤中的突变效率见图2(图中相邻两个指示柱中左侧为pds,右侧为badh2)。结果表明,除了spcas9-nrrh编辑系统在pam为taaa和caaa的编辑能力较低,突变效率低于5%外,而在其他的22个pam靶标位点的编辑效率在7.3%~79.5%。特别是在gaah的pam序列,编辑效率最高,平均编辑效率为50%以上。综上所述,本发明提供的spcas9-nrrh突变体能够对nrrh的pam序列附近的靶标序列进行编辑,由于其扩充了spcas9识别的pam位点序列,所以扩大了crispr/spcas9系统在植物基因组中的编辑范围,在水稻基因编辑过程中,将具有更广泛的应用。
序列表
<110>安徽省农业科学院水稻研究所
<120>一种在水稻基因打靶中识别特异位点的spcas9-nrrh突变体及其应用
<160>2
<170>siposequencelisting1.0
<210>4
<211>1367
<212>prt
<213>spcas9-nrrh
<400>4
asplyslystyrserileglyleuaspileglythrasnservalgly
151015
trpalavalilethraspglutyrlysvalproserlyslysphelys
202530
valleuglyasnthrasparghisserilelyslysasnleuilegly
354045
alaleuleupheaspserglygluthralaglualathrargleulys
505560
argthralaargargargtyrthrargarglysasnargilecystyr
65707580
leuglngluilepheserasnglumetalalysvalaspaspserphe
859095
phehisargleuglugluserpheleuvalglugluasplyslyshis
100105110
gluarghisproilepheglyasnilevalaspgluvalalatyrhis
115120125
glulystyrprothriletyrhisleuarglyslysleuvalaspser
130135140
thrasplysalaaspleuargleuiletyrleualaleualahismet
145150155160
ilelyspheargglyhispheleuilegluglyaspleuasnproasp
165170175
asnseraspvalasplysleupheileglnleuvalglnthrtyrasn
180185190
glnleupheglugluasnproileasnalaserglyvalaspalalys
195200205
alaileleuseralaargleuserlysserargargleugluasnleu
210215220
ilealaglnleuproglyglulyslysasnglyleupheglyasnleu
225230235240
ilealaleuserleuglyleuthrproasnphelysserasnpheasp
245250255
leualagluaspalalysleuglnleuserlysaspthrtyraspasp
260265270
aspleuaspasnleuleualaglnileglyaspglntyralaaspleu
275280285
pheleualaalalysasnleuseraspalaileleuleuseraspile
290295300
leuargvalasnthrgluilethrlysalaproleuseralasermet
305310315320
vallysargtyraspgluhishisglnaspleuthrleuleulysala
325330335
leuvalargglnglnleuproglulystyrlysgluilephepheasp
340345350
glnserlysasnglytyralaglytyrileaspglyglyalasergln
355360365
glugluphetyrlyspheilelysproileleuglulysmetaspgly
370375380
thrglugluleuleuvallysleuasnarggluaspleuleuarglys
385390395400
glnargthrpheaspasnglyileileprohisglnilehisleugly
405410415
gluleuhisalaileleuargargglnglyaspphetyrpropheleu
420425430
lysaspasnargglulysileglulysileleuthrpheargilepro
435440445
tyrtyrvalglyproleualaargglyasnserargphealatrpmet
450455460
thrarglysserglugluthrilethrprotrpasnpheglugluval
465470475480
valasplysglyalaseralaglnserpheilegluargmetthrasn
485490495
pheasplysasnleuproasnglulysvalleuprolyshisserleu
500505510
leutyrglutyrphethrvaltyrasngluleuthrlysvallystyr
515520525
valthrgluglymetarglysproalapheleuserglygluglnlys
530535540
lysalailevalaspleuleuphelysthrasnarglysvalthrval
545550555560
lysglnleulysgluasptyrphelyslysileglucyspheaspser
565570575
valgluileserglyvalgluaspargpheasnalaserleuglythr
580585590
tyrhisaspleuleulysileilelysasplysasppheleuaspasn
595600605
glugluasngluaspileleugluaspilevalleuthrleuthrleu
610615620
phegluaspargglumetileglugluargleulysthrtyralahis
625630635640
leupheaspasplysvalmetlysglnleulysargleuargtyrthr
645650655
glytrpglyargleuserarglysleuileasnglyileargasplys
660665670
glnserglylysthrileleuasppheleulysseraspglypheala
675680685
asnargasnphemetglnleuilehisaspaspserleuthrphelys
690695700
gluaspileglnlysalaglnvalserglyglnglyaspserleuhis
705710715720
gluhisilealaasnleualaglyserproalailelyslysglyile
725730735
leuglnthrvallysvalvalaspgluleuvallysvalmetglygly
740745750
hislysprogluasnilevalileglumetalaarggluasnglnthr
755760765
thrglnlysglyglnlysasnserarggluargmetlysargileglu
770775780
gluglyilelysgluleuglyserglnileleulysgluhisproval
785790795800
gluasnthrglnleuglnasnglulysleutyrleutyrtyrleugln
805810815
asnglyargaspmettyrvalaspglngluleuaspileasnargleu
820825830
serasptyraspvalasphisilevalproglnserpheleulysasp
835840845
aspserileaspasnlysvalleuthrargserasplysasnarggly
850855860
lysseraspasnvalproserglugluvalvallyslysmetlysasn
865870875880
tyrtrpargglnleuleuasnalalysleuilethrglnarglysphe
885890895
aspasnleuthrlysalagluargglyglyleusergluleuasplys
900905910
alaglypheilelysargglnleuvalgluthrargglnilethrlys
915920925
hisvalalaglnileleuaspserargmetasnthrlystyraspglu
930935940
asnasplysleuilearggluvallysvalilethrleulysserlys
945950955960
leuvalseraspphearglysasppheglnphetyrlysvalargglu
965970975
ileasnasntyrhishisalahisaspalatyrleuasnalavalval
980985990
glythralaleuilelyslystyrprolysleugluserglupheval
99510001005
tyrglyasptyrlysvaltyraspvalarglysmetilealalysser
101010151020
gluglngluileglylysalathralalystyrphephetyrserasn
1025103010351040
ilemetasnphephelysthrgluilethrleualaasnglygluile
104510501055
arglysargproleuilegluthrasnglygluthrglygluileval
106010651070
trpasplysglyargaspphealathrvalarglysvalleusermet
107510801085
proglnvalasnilevallyslysthrgluvalglnthrglyglyphe
109010951100
serlysgluserileleuprolysglyasnserasplysleuileala
1105111011151120
arglyslysasptrpaspprolyslystyrglyglypheasnserpro
112511301135
thralaalatyrservalleuvalvalalalysvalglulysglylys
114011451150
serlyslysleulysservallysgluleuleuglyilethrilemet
115511601165
gluargserserpheglulysasnproileglypheleuglualalys
117011751180
glytyrlysgluvallyslysaspleuileilelysleuprolystyr
1185119011951200
serleuphegluleugluasnglyarglysargmetleualaserala
120512101215
glyvalleuhislysglyasngluleualaleuproserlystyrval
122012251230
asnpheleutyrleualaserhistyrglulysleulysglyserpro
123512401245
gluaspasngluglnlysglnleuphevalgluglnhislyshistyr
125012551260
leuaspgluileilegluglnileserglupheserlysargvalile
1265127012751280
leualaaspalaasnleuasplysvalleuseralatyrasnlyshis
128512901295
argasplysproilearggluglnalagluasnileilehisleuphe
130013051310
thrleuthrasnleuglyvalproalaalaphelystyrpheaspthr
131513201325
thrileasplyslysargtyrthrserthrlysgluvalleuaspala
133013351340
thrleuilehisglnserilethrglyleutyrgluthrargileasp
1345135013551360
leuserglnleuglyglyasp
1365
<210>2
<211>4107
<212>dna
<213>spcas9-nrrh基因
<400>2
atggacaagaagtactccatcggcctcgacatcggcaccaattctgttggctgggccgtg60
atcaccgacgagtacaaggtgccgtccaagaagttcaaggtcctcggcaacaccgaccgc120
cactccatcaagaagaatctcatcggcgccctgctgttcgactctggcgagacagccgag180
gctacaaggctcaagaggaccgctagacgcaggtacaccaggcgcaagaaccgcatctgc240
tacctccaagagatcttctccaacgagatggccaaggtggacgacagcttcttccacagg300
ctcgaggagagcttcctcgtcgaggaggacaagaagcacgagcgccatccgatcttcggc360
aacatcgtggatgaggtggcctaccacgagaagtacccgaccatctaccacctccgcaag420
aagctcgtcgactccaccgataaggccgacctcaggctcatctacctcgccctcgcccac480
atgatcaagttcaggggccacttcctcatcgagggcgacctcaacccggacaactccgat540
gtggacaagctgttcatccagctcgtgcagacctacaaccagctgttcgaggagaacccg600
atcaacgcctctggcgttgacgccaaggctattctctctgccaggctctctaagtcccgc660
aggctcgagaatctgatcgcccaacttccgggcgagaagaagaatggcctcttcggcaac720
ctgatcgccctctctcttggcctcaccccgaacttcaagtccaacttcgacctcgccgag780
gacgccaagctccagctttccaaggacacctacgacgacgacctcgacaatctcctcgcc840
cagattggcgatcagtacgccgatctgttcctcgccgccaagaatctctccgacgccatc900
ctcctcagcgacatcctcagggtgaacaccgagatcaccaaggccccactctccgcctcc960
atggtgaagaggtacgacgagcaccaccaggacctcacactcctcaaggccctcgtgaga1020
cagcagctcccagagaagtacaaggagatcttcttcgaccagtccaagaacggctacgcc1080
ggctacatcgatggcggcgcttctcaagaggagttctacaagttcatcaagccgatcctc1140
gagaagatggacggcaccgaggagctgctcgtgaagctcaatagagaggacctcctccgc1200
aagcagcgcaccttcgataatggcattatcccgcaccagatccacctcggcgagcttcat1260
gctatcctccgcaggcaaggcgacttctacccgttcctcaaggacaaccgcgagaagatt1320
gagaagatcctcaccttccgcatcccgtactacgtgggcccgctcgccaggggcaactcc1380
aggttcgcctggatgaccagaaagtccgaggagacaatcaccccctggaacttcgaggag1440
gtggtggataagggcgcctctgcccagtctttcatcgagcgcatgaccaacttcgacaag1500
aacctcccgaacgagaaggtgctcccgaagcactcactcctctacgagtacttcaccgtg1560
tacaacgagctgaccaaggtgaagtacgtgaccgaggggatgaggaagccagctttcctt1620
agcggcgagcaaaagaaggccatcgtcgacctgctgttcaagaccaaccgcaaggtgacc1680
gtgaagcagctcaaggaggactacttcaagaaaatcgagtgcttcgactccgtcgagatc1740
tccggcgtcgaggataggttcaatgcctccctcgggacctaccacgacctcctcaagatt1800
atcaaggacaaggacttcctcgacaacgaggagaacgaggacatcctcgaggacatcgtg1860
ctcaccctcaccctcttcgaggaccgcgagatgatcgaggagcgcctcaagacatacgcc1920
cacctcttcgacgacaaggtgatgaagcagctgaagcgcctgcgctataccggctggggc1980
aggctctctaggaagctcatcaacggcatccgcgacaagcagtccggcaagacgatcctc2040
gacttcctcaagtccgacggcttcgccaaccgcaacttcatgcagctcatccacgacgac2100
tccctcaccttcaaggaggacatccaaaaggcccaggtgtccggccaaggcgattccctc2160
catgaacatatcgccaatctcgccggctccccggctatcaagaagggcattctccagacc2220
gtgaaggtggtggacgagctggtgaaggtgatgggcggccacaagccagagaacatcgtg2280
atcgagatggcccgcgagaaccagaccacacagaagggccaaaagaactcccgcgagcgc2340
atgaagaggatcgaggagggcattaaggagctgggctcccagatcctcaaggagcaccca2400
gtcgagaacacccagctccagaacgagaagctctacctctactacctccagaacggccgc2460
gacatgtacgtggaccaagagctggacatcaaccgcctctccgactacgacgtggaccat2520
attgtgccgcagtccttcctgaaggacgactccatcgacaacaaggtgctcacccgctcc2580
gacaagaacaggggcaagtccgataacgtgccgtccgaagaggtcgtcaagaagatgaag2640
aactactggcgccagctcctcaacgccaagctcatcacccagaggaagttcgacaacctc2700
accaaggccgagagaggcggcctttccgagcttgataaggccggcttcatcaagcgccag2760
ctcgtcgagacacgccagatcacaaagcacgtggcccagatcctcgactcccgcatgaac2820
accaagtacgacgagaacgacaagctcatccgcgaggtgaaggtcatcaccctcaagtcc2880
aagctcgtgtccgacttccgcaaggacttccagttctacaaggtgcgcgagatcaacaac2940
taccaccacgcccacgacgcctacctcaatgccgtggtgggcacagccctcatcaagaag3000
tacccaaagctcgagtccgagttcgtgtacggcgactacaaggtgtacgacgtgcgcaag3060
atgatcgccaagtccgagcaagagatcggcaaggcgaccgccaagtacttcttctactcc3120
aacatcatgaatttcttcaagaccgagatcacgctcgccaacggcgagattaggaagagg3180
ccgctcatcgagacaaacggcgagacaggcgagatcgtgtgggacaagggcagggatttc3240
gccacagtgcgcaaggtgctctccatgccgcaagtgaacatcgtgaagaagaccgaggtt3300
cagaccggcggcttctccaaggagtccatcctcccaaagggcaactccgacaagctgatc3360
gcccgcaagaaggactgggacccgaagaagtatggcggcttcaactctccgaccgcggcc3420
tactctgtgctcgtggttgccaaggtcgagaagggcaagagcaagaagctcaagtccgtc3480
aaggagctgctgggcatcacgatcatggagcgcagcagcttcgagaagaacccaatcggc3540
ttcctcgaggccaagggctacaaggaggtgaagaaggacctcatcatcaagctcccgaag3600
tacagcctcttcgagcttgagaacggccgcaagagaatgctcgcctctgctggcgtgctt3660
cataagggcaacgagcttgctctcccgtccaagtacgtgaacttcctctacctcgcctcc3720
cactacgagaagctcaagggctccccagaggacaacgagcaaaagcagctgttcgtcgag3780
cagcacaagcactacctcgacgagatcatcgagcagatctccgagttctccaagcgcgtg3840
atcctcgccgatgccaacctcgataaggtgctcagcgcctacaacaagcaccgcgataag3900
ccaattcgcgagcaggccgagaacatcatccacctcttcaccctcaccaacctcggcgtg3960
ccagccgccttcaagtacttcgacaccaccatcgacaaaaagcgctacacctctaccaag4020
gaggttctcgacgccaccctcatccaccagtctatcacaggcctctacgagacacgcatc4080
gacctctcacaactcggcggcgattga4107
- 还没有人留言评论。精彩留言会获得点赞!