一种五香粉中植物源性成分鉴别方法与流程
1.本发明属于食品质量安全检测技术领域,具体而言,涉及一种基于高通量测序技术的五香粉中植物源性物种成分鉴别方法。
背景技术:
2.五香粉,也称五香面,是一种传统的中式复合调味料,是将五种及五种以上香辛料干品粉碎后,按一定比例混合而成,用以平衡五种基本的口味(甜、酸、咸、苦、鲜)。五香粉广受人们喜爱,是中国烹饪中使用最广泛的食用香辛料。市售五香粉配方、口味均有较大差异,各生产厂家均有各自配方,且都保密,其主要调香原料大体有八角、桂皮、小茴香、砂仁、豆蔻、丁香、山奈、花椒、白芷、陈皮、草果、姜、高良姜、甘草等。五香粉以粉末状形式售卖,原始性状消失,且其原料多样并多有混杂,消费者难以辨别其是否掺假,这让不法分子有机可乘。一些生产经营者为了追求经济利益,多以廉价的、颜色相近的物质掺入其中,如廉价的农作物(谷糠、麸皮、玉米粉)和外部表形特征相似的物种。
3.由于五香粉中的物种原始特征已经消失,依据其外部形态和感官特征进行鉴别难度很大。加上五香粉成分复杂,导致掺假隐蔽,难以检测。最常用的理化分析方法,如色谱法和光谱法,主要是根据样品中的特异性化学组成来进行真伪鉴别,而生物体的成分容易受到季节、产地和环境等因素的影响,容易导致结果判定的不准确性。此外对于加工后的五香粉样品,由于其化学结构和组成发生了变化,并且产品中可能掺杂了其他配料,给化学组分的分离纯化和结构鉴定带来了困难,因此该方法的应用就存在一定的局限性。目前常用的基于dna的分子检测方法通常一次只能检测出一种或者少数几种物种,而不能快速准确分析出大量的物种信息。当样本数量很大时,需要花费很大的物力和人力。另外,当待测样品是混合物或者未知样品时,生物体的分离比较困难,加之物种的原始特征已经消失,这些现有的食品物种成分鉴别技术,无论从涉及的原料产品范围,还是从方法的适用性等方面,都远远无法满足现实的需求。
4.因此,本领域迫切需要开发一种高通量、高准确性、高质量的五香粉中植物源性成分鉴别方法,以实现非靶标多物种筛查的目的,破解目前的“黑箱”难题,来满足目前的市场需求。
技术实现要素:
5.本发明的目的在于提供一种基于高通量测序技术的五香粉中植物源性物种成分来源鉴别方法,该方法可以通过一次检测鉴定出五香粉产品中的所有植物源性成分,具有检测范围广、检测通量高、灵敏度高等优点。
6.为实现上述目的,本发明采取的技术方案是:
7.基于高通量测序技术的五香粉中植物源性成分鉴别方法,所述方法包括以下步骤:
8.(a)提供一待测样品;
9.(b)从所述样品中提取基因组dna;
10.(c)用带有碱基标签的its2和psba
‑
trnh基因通用引物对(b)中所述基因组dna进行多重pcr扩增,得到pcr扩增产物;
11.(d)对所述pcr扩增产物进行纯化、定量检测;
12.(e)用上一步骤中检测合格的pcr扩增产物构建测序文库并进行质量检验;
13.(f)对上一步测序文库进行高通量测序,获得its2和psba
‑
trnh基因片段序列;
14.(g)将上一步所得its2和psba
‑
trnh基因片段序列与本地its2和psba
‑
trnh基因序列数据库和/或公共数据库进行比对,根据测序序列与数据库中序列的同源性确定五香粉样品中的物种成分。
15.本发明所述五香粉样品为一类将多种香辛料按一定比例混合而成的复合粉状调味料。
16.步骤(c)中,所述its2基因扩增正反向引物分别为seq id no:1和seq id no:2所示,its2通用引物的具体反应条件为:94℃预变性5min,94℃变性30s,58℃退火30s,72℃延伸60s,35个循环,最后72℃延伸7min。
17.步骤(c)中,所述psba
‑
trnh基因扩增正反向引物分别如seq id no:3和seq id no:4所示,psba
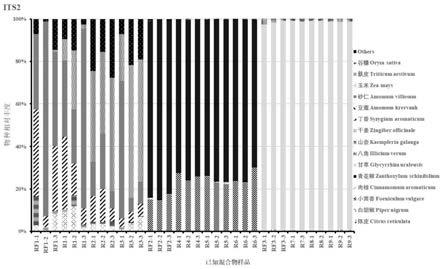
‑
trnh基因通用引物的具体反应条件为:94℃变性5min;94℃变性1min,57℃退火1min,72℃延伸1.5min,30个循环;72℃延伸7min。
18.步骤(d)中,对每个样本扩增产物进行纯化回收采用通用的pcr产物回收试剂盒。
19.所述(e)中包括以下步骤:pcr产物dna末端修复、dna片段选择、3’端加a尾、加接头桥式扩增和纯化,利用bioanalyser 2100毛细管电泳仪和进行测序文库的质量检验。
20.步骤(f)中,本发明所述高通量测序采用基于illumina第二代高通测序仪的测序平台进行。
21.步骤(g)中所述根据测序序列与数据库中序列的同源性确定五香粉样品中的物种成分为:
22.若待测五香粉扩增所得its2和/或psba
‑
trnh基因片段某条测序序列与本地its2和psba
‑
trnh基因序列数据库和/或公共数据库中某一植物的某条序列一致性大于等于97%,则所述待测五香粉中含有该条序列在数据库中所对应的植物物种;若待测五香粉扩增所得its2和/或psba
‑
trnh基因片段某条测序序列与本地its2和psba
‑
trnh基因序列数据库和/或公共数据库中某一植物的某条序列一致性小于97%,则所述待测五香粉中不含有或候选不含有该条序列在数据库中所对应的植物物种。
23.进一步的,上述(g)物种成分分析时,只有在至少两个重复中均检测到该物种时,才认为该物种存在于样品中。
24.进一步的,上述(g)中物种成分分析时,对于标签中未标注的物种,当物种相对丰度高于0.2%时,才认为存在于样品中。
25.进一步的,上述(g)中,结合its2和psba
‑
trnh基因序列的鉴别结果确定五香粉样品的成分。
26.与现有方法相比,本发明具有如下优点和有益效果:
27.五香粉成分多样且多有混杂,现行的分子生物学检测方法大多无法实现未知样品
中多种源性成分的同步快速鉴别,一个反应孔只能进行一种源性成分的检测。本方法基于高通量测序的研究思路,利用扩增子测序的方法,通过对组合基因片段进行扩增,可以非特异性地将五香粉中所有主要植物源性物种成分及杂质物种成分都检测出来,为食用香辛料行业的企业品控和市场监管提供技术支撑。
附图说明
28.图1是基于its2基因序列的已知混合物物种成分相对丰度柱形图。
29.图2是基于psba
‑
trnh基因序列的已知混合物物种成分相对丰度柱形图。
30.图3是基于its2基因序列的市售五香粉物种成分相对丰度柱形图。
31.图4是基于psba
‑
trnh基因序列的市售五香粉物种成分相对丰度柱形图。
具体实施方式
32.通过实施例的方式对本发明作进一步的说明,但本发明要求保护的范围并不局限于实施例表述的范围。
33.实施例1
34.本实施例为通过如下试验对基于高通量测序技术的五香粉物种成分分析方法进行准确度评价。
35.1.已知混合物的制备
36.选择五香粉传统的3种配方(表1),按照五香粉制备工艺,经粉碎、过筛、混合制备真实五香粉rf1,rf2,rf3。将粉碎后的麸皮、谷糠和玉米按照一定比例掺入五香粉中制备掺假五香粉,每个比例设有三个重复。
37.表1已知混合物制备方案
[0038][0039]
[0040]
2.样品基因组dna提取
[0041]
分别从每份已知混合物中随机称取三次,每次称取30mg。采用植物基因组dna提取试剂盒进行dna提取,将这三份dna混合用于后续研究。针对五香粉样品的特点,对dna提取步骤进行优化。为进一步破坏样品的细胞壁,向装有样品和裂解液的离心管中加入3.0mm研磨珠,随后将离心管置于组织研磨器中,在频率30hz下研磨90s。另外,为使dna裂解更充分,样品的裂解时间延长为120min。
[0042]
3.文库制备及高通量测序
[0043]
(1)pcr扩增及文库制备
[0044]
利用带有不同标记(表2)的通用引物ms2(seq id no:1所示的正向引物和seq id no:2所示的反向引物)和psba
‑
trnh(seq id no:3所示的正向引物和seq id no:4所示的反向引物)对已知混合物和市售五香粉dna进行pcr扩增。
[0045]
seq id no:1:5
’‑
gagtctttgaacgcaagttg
‑3’
[0046]
seq id no:2:5
’‑
tcctccgcttattgatatg
‑3’
[0047]
seq id no:3:5
’‑
gttatgcatgaacgtaatgctc
‑3’
[0048]
seq id no:4:5
’‑
cgcgcatggtggattcacaatcc
‑3’
[0049]
pcr产物使用agencourt ampure xp核酸纯化试剂盒进行纯化。随后,使用3.0核酸荧光蛋白定量仪测定纯化后pcr产物的浓度。对纯化后的pcr产物进行末端修复加a尾、连接测序接头、纯化回收、加入接头引物和pcr富集完成文库构建。构建完成的测序文库使用agencourt ampure xp核酸纯化试剂盒进行纯化。然后,使用nanodrop粗检文库浓度,使用agilent 2100检测文库片段,qpcr精确定量文库浓度。
[0050]
表2已知混合物样品标记
[0051][0052]
(2)高通量测序
[0053]
在本发明中,可用常规的测序技术和平台进行测序。
[0054]
在本发明中,将符合上机测序标准的文库片段,使用illumina miseq平台进行测
序。
[0055]
(3)数据分析
[0056]
在本发明的实施例中,数据分析包括以下步骤:使用blast算法进行比对,对每个otu进行物种注释,并分析物种丰度。测序数据经过拆分样本后,经去除标记和引物序列、过滤低质量序列、拼接、去除嵌合体,得到优质序列。利用参考序列作为数据库,以ncbi数据库中公布的植物its2和psba
‑
trnh序列为参考序列。以97%的相似度阈值将序列聚类成为otus(operational taxonomic units),然后对otus的代表序列进行物种注释,并分析物种丰度。
[0057]
具体包括:illumina miseq平台所得数据为pair
‑
end(pe)双端序列数据,经过qiime软件根据barcode序列拆分样本。去除barcode和引物序列后,使用pear软件过滤打分值低于20、含有模糊碱基、引物错配序列,并将其拼接得到raw tags。其中,在拼接时最小overlap为10bp,错配率为0.1。拼接后使用vsearch软件去除短序列,并根据ncbi nucleotide数据库用uchime方法比对去除嵌合体序列,得到clean tags。最后使用vsearch软件uparse算法对优质序列进行otu(operational taxonomic units)聚类,相似性阈值为97%。
[0058]
4.结果分析
[0059]
如图1所示,利用its2引物,在36份已知混合物中共检测出14种物种。其中,包括12种五香粉原材料(砂仁、豆蔻、干姜、山奈、小茴香、肉桂、陈皮、青花椒、丁香、八角、甘草和白胡椒)和2种掺假物(麸皮和玉米)。在已知混合物中包含的12种食用香辛料和3种掺假物中,利用psba
‑
trnh引物可鉴别出干姜、小茴香、肉桂、青花椒、陈皮、八角、甘草、白胡椒、麸皮和谷糠。总体而言,以its2基因为主,psba
‑
trnh基因为辅,可鉴别出已知混合物中的全部植物物种成分。
[0060]
实施例2
[0061]
本实施例为通过如下试验对市售五香粉进行分析。
[0062]
1.实验方法
[0063]
从市场上购买五香粉8份,每份样品中随机称取两次,每次称取30mg。随后进行dna提取,其余部分与实施例1一致。
[0064]
表4市售五香粉信息
[0065][0066]
2.结果分析
[0067]
从图3和图4中可以看出,在8份市售五香粉中,均存在不同程度的标签不符合的情况。6份五香粉中均检测到标签中标注的物种,而3号和8号样品仅检测到标签中标注的3种和6种成分。所有市售五香粉中存在标签中未标注的其他食用香辛料和野草。标签中未标注物种的存在可以用多种因素来解释,包括但不限于在五香粉供应链的早期(种植、收集、运输和储存)、加工过程、商业化产品中可能发生的故意掺假和无意替代。
[0068]
实验结果显示,本发明所提供的方法可以应用于市售五香粉中物种成分的检测。
[0069]
虽然已经对本发明的具体实施方案进行了描述,但是本领域技术人员应认识到,在不偏离本发明的范围或精神的前提下可以对本发明进行多种改变与修饰。因而,本发明意欲涵盖落在权利要求书及其同等物范围内的所有这些改变与修饰。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1