敲除cslA基因以提高谷氨酰胺转氨酶发酵水平的方法
本发明属于生物工程
技术领域:
,涉及一种敲除csla基因以提高谷氨酰胺转氨酶发酵水平的方法;具体说是一种利用crispr/cas9技术敲除类纤维素合成蛋白csla编码基因以提高谷氨酰胺转氨酶发酵水平的方法。
背景技术:
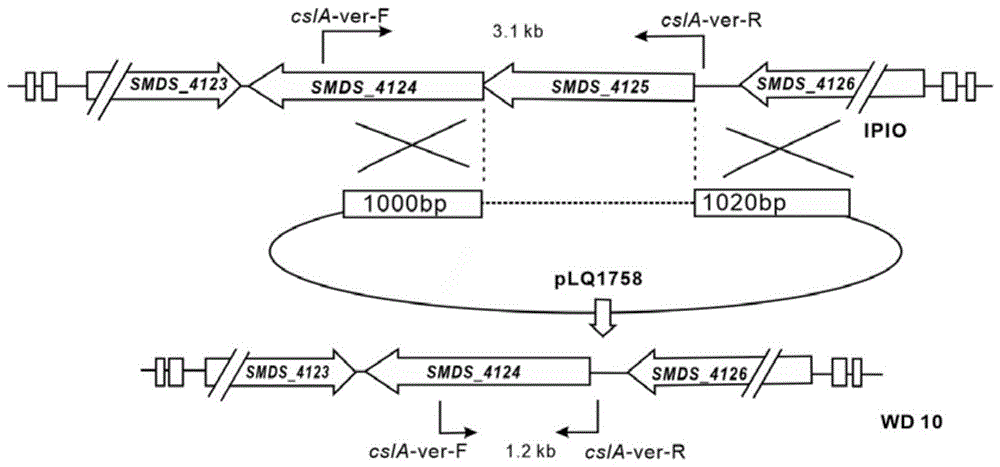
:谷氨酰胺转氨酶(tgase)是由茂源链霉菌(streptomycesmobaraensis)产生的一种单亚基蛋白,它能够催化蛋白质中谷氨酰胺残基的γ-酰胺基和赖氨酸的ε-氨基之间进行酰胺基转移反应,形成ε-(γ-谷酰胺)-赖氨酸的异型肽键,从而改变蛋白质的功能性质。tgase是外分泌蛋白,在胞内为pre-pro-mtgase初始形式,穿过细胞膜成为无活性的酶原pro-tgase,经过金属蛋白酶tamep切割信号肽成为frap-tgase,再经丝氨酸蛋白酶sm-tap切割成为最终成熟的tgase。tgase作为一种蛋白交联剂,因其稳定性好、使用安全等优点而被广泛应用,在食品领域通过交联谷氨酰胺残基和赖氨酸残基能够将小块肉类结合成大块,提高食品的美观度,通过整合氨基酸增加营养,生物合成tgase制作可降解塑料包装;在医药领域可用来交联抗体和药物分子生产抗体偶联药物,催化明胶和胶原蛋白形成支架植入人体使器官再生等等。本发明通过基因组学信息找到了类纤维素合成蛋白csla编码基因smds_4125。缺失csla编码基因可以使菌丝球变得小而疏松、有利于谷氨酰胺转氨酶酶原的分泌,最终可明显提高谷氨酰胺转氨酶产量。技术实现要素:本发明的目的在于提供一种利用crispr/cas9技术敲除csla基因以提高谷氨酰胺转氨酶发酵水平的方法;通过crispr/cas9技术敲除茂源链霉菌dsm40587基因组中csla编码基因smds_4125,可以使菌丝球变得小而疏松、有利于谷氨酰胺转氨酶酶原的分泌,最终可明显提高谷氨酰胺转氨酶产量。为实现上述目的,本发明提供了以下技术方案:第一方面,本发明涉及一种提高谷氨酰胺转氨酶发酵水平的方法,利用crispr/cas9技术敲除茂源链霉菌dsm40587基因组中的类纤维素合成蛋白csla编码基因获得突变株,发酵生产谷氨酰胺转氨酶。作为一个实施方案,所述csla编码基因为序列如seqidno.1所示的csla编码基因smds_4125。通过敲除茂源链霉菌dsm40587基因组中csla编码基因smds_4125,使菌丝球变得小而疏松,提高谷氨酰胺转氨酶酶原的分泌效率,进而提高谷氨酰胺转氨酶产量。作为一个实施方案,所述方法包括如下步骤:s1、设计并构建用于敲除smds_4125的crispr/cas9质粒载体plq1758;s2、将所述crispr/cas9质粒载体plq1758通过接合转移导入受体菌dsm40587中进行特异性切割和同源重组修复;s3、通过安普霉素抗性和pcr验证筛选,并通过37℃无抗松弛培养丢失质粒,得到smds_4125缺失的突变株。作为一个实施方案,所述质粒载体plq1758的构建方法包括:a1、通过crispy-web设计smds_4125的grna;a2、通过pcr扩增分别得到131bp含有目标grna的sgrna的pcr片段、smds_4125区域右侧1020bp上游同源臂的pcr片段和smds_4125区域左侧1000bp下游同源臂的pcr片段;a3、再通过overlappcr扩增得到2151bp包括sgrna和上下游同源臂的pcr片段;最终通过gibson组装的方法连入游离型质粒pkccasdo的spei/hindiii位点。作为一个实施方案,所述grna序列为:ccgccgcgcgtcggcaggcg。作为一个实施方案,步骤a2中,以茂源链霉菌dsm40587的基因组dna为模板,使用引物csla-up-f/r通过pcr扩增得到smds_4125区域右侧1020bp上游同源臂的pcr片段;所述引物csla-up-f/r的序列如seqidno.2、3所示。作为一个实施方案,步骤a2中,以pkccas9do质粒为模板,使用引物csla-sgrna-f/r通过pcr扩增得到131bp含有目标grna的sgrna的pcr片段;所述引物csla-sgrna-f/r序列如seqidno.6、7所示。作为一个实施方案,步骤a2中,以茂源链霉菌dsm40587的基因组dna为模板,使用引物csla-down-f/r通过pcr扩增得到smds_4125区域左侧1000bp下游同源臂的pcr片段;所述引物csla-down-f/r的序列如seqidno.4、5所示。作为一个实施方案,步骤a3中,overlappcr扩增使用的引物为如seqidno.2所示的csla-up-f和如seqidno.7所示的csla-sgrna-r。作为一个实施方案,所述发酵包括如下步骤:将活化后的所述突变株孢子接种于种子培养基中,30℃、200-220rpm条件下培养24h,按10%接种量转接至发酵培养基中,30℃、200-220rpm条件下发酵26-28h。还进一步包括收集发酵液并进行酶活检测和菌丝体形态观察的步骤。作为一个具体示例,所述发酵包括如下步骤:将活化后的所述突变株孢子接种于种子培养基中,30℃、200rpm条件下培养24h,按10%接种量转接至发酵培养基中,30℃、200rpm条件下发酵26h。作为一个实施方案,所述种子培养基包括甘油1-3w/v%,酵母提取物0.4-0.8w/v%,鱼粉蛋白胨1-3w/v%,mgso4·7h2o0.1-0.3w/v%,k2hpo4·3h2o0.1-0.3w/v%。作为一个具体示例,所述种子培养基包括甘油2w/v%,酵母提取物0.6w/v%,鱼粉蛋白胨2.5w/v%,mgso4·7h2o0.2w/v%,k2hpo4·3h2o0.2w/v%。作为一个实施方案,所述发酵培养基包括甘油1-3w/v%,酵母提取物0.4-0.8w/v%,鱼粉蛋白胨1-3w/v%,mgso4·7h2o0.1-0.3w/v%,k2hpo4·3h2o0.1-0.3w/v%,发酵促进剂0.1-0.4w/v%。作为一个具体示例,所述发酵培养基包括甘油2w/v%,酵母提取物0.6w/v%,鱼粉蛋白胨2.5w/v%,mgso4·7h2o0.2w/v%,k2hpo4·3h2o0.2w/v%,发酵促进剂0.1w/v%。第二方面,本发明涉及一种用于敲除类纤维素合成蛋白csla编码基因的crispr/cas9质粒载体,所述质粒载体包含csla编码基因smds_4125区域右侧(1020bp)的上游同源臂、smds_4125区域左侧(1000bp)下游同源臂,以及(131bp)含有靶向smds_4125的grna的sgrna。作为一个实施方案,csla编码基因smds_4125序列如seqidno.1所示。作为一个实施方案,所述grna序列为:ccgccgcgcgtcggcaggcg。第三方面,本发明还涉及一种前述的crispr/cas9质粒载体的构建方法,所述方法包括如下步骤:b1、设计smds_4125的grna;b2、通过pcr扩增分别得到(131bp)含有目标grna的sgrna的pcr片段、smds_4125区域右侧(1020bp)上游同源臂的pcr片段和smds_4125区域左侧(1000bp)下游同源臂的pcr片段;b3、再通过overlappcr扩增得到(2151bp)包括sgrna和上下游同源臂的pcr片段;最终通过gibson组装的方法连入游离型质粒pkccasdo的spei/hindiii位点。作为一个实施方案,步骤b1中,所述grna序列为:ccgccgcgcgtcggcaggcg。第四方面,本发明还涉及一种高产谷氨酰胺转氨酶的基因工程菌,敲除茂源链霉菌dsm40587基因组中csla编码基因smds_4125,即得所述基因工程菌。第五方面,本发明还涉及一种高产谷氨酰胺转氨酶的基因工程菌,所述基因工程菌为将前述的crispr/cas9质粒载体导入宿主菌株,筛选得到csla编码基因缺失突变株。作为一个实施方案,所述宿主菌株为茂源链霉菌dsm40587。作为一个实施方案,所述crispr/cas9质粒载体通过接合转移导入宿主菌株中进行特异性切割和同源重组修复。第六方面,本发明还涉及一种前述的高产谷氨酰胺转氨酶的基因工程菌的构建方法,所述方法包括:将所述crispr/cas9质粒载体通过接合转移导入宿主菌株中进行特异性切割和同源重组修复;通过安普霉素抗性和pcr验证筛选,并通过37℃无抗松弛培养丢失质粒,得到smds_4125缺失的突变株。与现有技术相比,本发明具有如下有益效果:1)在茂源链霉菌dsm40587中,利用crispr/cas9技术在染色体上切割和同源重组修复,导致类纤维素合成蛋白csla编码基因smds_4125缺失,本发明所得到的工程菌株的tgase产量大幅提高;2)本发明通过使菌丝球变得小而疏松,提高tgase酶原的分泌效率来提高tgase发酵水平,最终tgase发酵产量提高了2.5倍;通过本发明可显著提tgase的发酵产量,同时使发酵成本大幅降低。附图说明通过阅读参照以下附图对非限制性实施例所作的详细描述,本发明的其它特征、目的和优点将会变得更明显:图1为基因smds_4125敲除过程示意图;图2为基因smds_4125缺失突变株与野生型菌株的tgase发酵产量示意图;图3为基因smds_4125缺失突变株与野生型菌株的菌丝体形态(160倍相差)显微镜示意图。具体实施方式下面结合实施例对本发明进行详细说明。以下实施例将有助于本领域的技术人员进一步理解本发明,但不以任何形式限制本发明。应当指出的是,对本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干调整和改进。这些都属于本发明的保护范围。下列实施例中未注明具体条件的实验方法的,按照常规条件或制造厂商的建议条件。本发明所涉及的质粒pkccas9do已经在sci数据库文献《hehuang,guosongzheng,weihongjiang,haifenghu,andyinhualu.one-stephigh-efficiencycrispr/cas9-mediatedgenomeeditinginstreptomyces.actabiochimicaetbiophysicasinica(4),231-43.》中记载。本发明所涉及的菌株茂源链霉菌dsm40587已经在sci数据库文献《zhang,l,zhang,l,yi,h,du,m,ma,c,&han,x,etal.(2012).enzymaticcharacterizationoftransglutaminasefromstreptomycesmobaraensisdsm40587inhighsaltandeffectofenzymaticcross-linkingofyakmilkproteinsonfunctionalpropertiesofstirredyogurt.journalofdairyscience,95(7),3559-3568.》中记载。本发明提供一种利用crispr/cas9技术敲除类纤维素合成蛋白csla编码基因以提高谷氨酰胺转氨酶发酵水平的方法,是通过敲除茂源链霉菌dsm40587基因组中csla编码基因smds_4125,使菌丝球变得小而疏松、有利于谷氨酰胺转氨酶酶原的分泌,最终可明显提高谷氨酰胺转氨酶产量。所述csla编码基因smds_4125的序列如seqidno.1所示。构建步骤如下:第一步:设计并构建用于敲除smds_4125的crispr/cas9质粒载体plq1758;第二步:将第一步构建得到的crispr/cas9质粒载体plq1758通过接合转移导入受体菌dsm40587中进行特异性切割和同源重组修复;第三步:通过安普霉素抗性和pcr验证筛选,并通过37℃无抗松弛培养丢失质粒,得到smds_4125缺失的突变株,记为wd10。所述质粒载体plq1758的构建步骤是:首先,通过crispy-web设计smds_4125的grna;其次,通过pcr扩增分别得到131bp含有目标grna的sgrna的pcr片段、smds_4125区域右侧1020bp上游同源臂的pcr片段和smds_4125区域左侧1000bp下游同源臂的pcr片段,再通过overlappcr扩增得到2151bp包括sgrna和上下游同源臂的pcr片段;最终通过酶切连接的方法连入游离型质粒pkccasdo的spei/hindiii位点。所述发酵包括以下步骤:将活化后的csla编码基因缺失突变株孢子接种于种子培养基中,30℃、200rpm条件下培养24h,按10%接种量转接至发酵培养基中,30℃、200rpm的条件下发酵26h,收集发酵液并进行酶活检测和菌丝体形态观察。所述种子培养基包括甘油1-3w/v%,酵母提取物0.4-0.8w/v%,鱼粉蛋白胨1-3w/v%,mgso4·7h2o0.1-0.3w/v%,k2hpo4·3h2o0.1-0.3w/v%;所述发酵培养基包括甘油1-3w/v%,酵母提取物0.4-0.8w/v%,鱼粉蛋白胨1-3w/v%,mgso4·7h2o0.1-0.3w/v%,k2hpo4·3h2o0.1-0.3w/v%,发酵促进剂0.1-0.4w/v%。实施例1本实施例为制备csla编码基因缺失突变株wd10的具体过程。具体包括以下步骤:步骤一:质粒plq1758的构建。具体操作如下:首先,通过crispy-web设计smds_4125的grna,序列为:ccgccgcgcgtcggcaggcgseqidno.12;其次,以pkccas9do质粒为模板,使用引入overlap的引物csla-sgrna-f/r通过pcr扩增得到131bp含有上述grna的sgrna的pcr片段,同时以茂源链霉菌dsm40587的基因组dna为模板,分别使用两组引入overlap的引物csla-up-f/r、csla-down-f/r通过pcr扩增得到smds_4125区域右侧1020bp上游同源臂的pcr片段和smds_4125区域左侧1000bp下游同源臂的pcr片段,再以以上三个pcr片段为模板,使用引物csla-up-f/csla-sgrna-r通过overlappcr扩增得到2151bp包括sgrna和上下游同源臂的pcr片段;通过基因测序确认grna和同源臂的正确性;最终通过gibson组装的方法连入游离型质粒pkccasdo的spei/hindiii位点。通过上述方法得到用于敲除csla编码基因smds_4125的质粒plq1758。在37℃水浴条件下,采用spei/hindiii两种限制性内切酶进行酶切处理,可以观察到2.1kb左右的目标条带,表明质粒构建正确。步骤二:将用于crispr/cas9的质粒plq1758导入野生型菌株dsm40587,并筛选得到smds_4125缺失突变株,此突变株表征茂源链霉菌dsm40587的类纤维素合成蛋白的失活。图1示意了基因smds_4125敲除的过程。具体操作如下:将crispr/cas9质粒载体plq1758转化进入宿主et12567(puz8002)中。将对应et12567(puz8002)接种于含有apr(终浓度50μg/ml)、kan(终浓度50μg/ml)和chl(终浓度25μg/ml)三种抗生素的lb中,37℃培养20h,然后用新鲜的lb溶液漂洗菌体以除去培养物中的抗生素。同时制备的dsm40587新鲜菌丝体(约24h培养物),用2×孢子预萌发液溶液漂洗2~3次之后,将其与之前制备的宿主菌et12567(puz8002)混合(受体菌细胞和供体菌的比例约为1:10)均匀后涂布于含有10mm镁离子的isp4mym固体培养基上,于37℃培养箱倒置培养。12h后取出平板,分别将安普霉素(终浓度50μg/ml)和甲氧苄啶(终浓度50μg/ml)两种抗生素加入1ml无菌水中混匀后覆盖在isp4mym固体培养基上,将固体培养基晾干后转移至30℃培养箱中倒置培养。一般3~5d后可见平板上有接合子长出,将其转接于含有安普霉素(终浓度50μg/ml)和甲氧苄啶(终浓度50μg/ml)两种抗生素的isp4mym固体培养基上扩大培养得到单菌落。采用csla-ver-f/r引物,通过菌丝体pcr的方法对单菌落进行筛选验证,验证接合子正确后转接至isp4mym平板,37℃培养传代三次丢失质粒,采用tipa-yz-f/r为引物,通过菌丝体pcr的方法对单菌落筛选得到不含质粒且不混有野生型的smds_4125缺失突变株,记为wd10。*上述步骤一中涉及的内切酶识别位点(酶切位点)如下:spei识别位点:hindiii识别位点:5'...a^ctagt...3'5'...a^agctt...3'3'...tgatc^a...5'3'...ttcga^a...5'*上述步骤中所用到的引物序列如表1所示:表1引物序列表引物名称碱基序列csla-up-fcgacggccagtgccaagcttgcgcacagacagccgagaggseqidno.2csla-up-rgtcggtcaggtgccggagtccagtcattccccccacacgcseqidno.3csla-down-fgactccggcacctgaccgacseqidno.4csla-down-rgtcggtcaggtgccggagtccagtcattccccccacacgcseqidno.5csla-sgrna-fcccaagaccaagaagtgggcgcaccgactcggtgccacttseqidno.6csla-sgrna-rgtcctaggtataatactagtccgccgcgcgtcggcaggcgseqidno.7csla-ver-fctgatgccggggaccgggatgaacgagttgseqidno.8csla-ver-rggccggggttcatgtaccggcgtaccacgcseqidno.9tipa-yz-fcaaccgagcgttctgaacaaaseqidno.10tipa-yz-rcgctcccttctctgacgccgseqidno.11*步骤一中基因片段制备所采用的pcr体系及条件:pcr反应体系:dna模板30ng,引物20pmol,50%dmso5μl,dntp10nmol,缓冲液25μl,taqdna聚合酶1个单位,加纯水补齐至50μl;pcr条件:95℃5min;95℃15s;65℃15s;72℃30s-2min;循环30次;72℃10min。*步骤二中通过pcr验证筛选突变株时所采用的pcr体系及条件:pcr体系:dna模板10~100ng,引物10pmol,50%dmso2μl,2×mix缓冲液10μl,加纯水补齐至20μl;pcr条件:95℃10min;95℃15s;60℃15s;72℃30s-2min;循环30次;72℃10min。实施例2本实施例为利用csla编码基因缺失突变株wd10发酵产生tgase的过程。具体步骤如下:将csla编码基因缺失突变株wd10涂布于固体isp4mym培养基上活化,30℃培养5-7d后,刮取一平板孢子接种至种子培养基中,30℃、200rpm条件下培养24h,按10%的接种量转接至发酵培养基,30℃、200rpm条件下发酵26h,收集发酵液进行酶活检测。表2种子培养基及发酵培养基的成分构成实施例3本实施例为利用比色法检测tgase的酶活的方法。具体为:取100μl发酵液上清于试管中,其中一管加入100μl水作为对照,加入1ml37℃预热好的a液,37℃反应10min后,加入1mlb液终止反应。用1cm的石英比色皿,在紫外分光光度计525nm处测定反应液的吸光度。最终将od525带入由标准曲线换算得到的公式,计算出tgase的酶活。其中,溶液配制方法如下:a液:称取9.688g三羟甲基氨基甲烷,2.780g盐酸羟胺,1.229g还原型谷胱甘肽,4.048g底物n-苄氧羰基-l-谷氨酰甘氨酸(na-cbz-gln-gly)于烧杯中,加350ml水,调节ph为6.0,加水定容至400ml。b液:3mol/l盐酸,12%三氯乙酸,5%fecl3溶于0.1mol/lhcl中,将三种溶液等量混合均匀。图2为基因smds_4125缺失突变株与野生型菌株的tgase发酵产量示意图。结果表明,在实验室摇瓶水平下突变株的产量对比野生型菌株提高2.5倍。图3为基因smds_4125缺失突变株与野生型菌株在相差显微镜下的菌丝体形态照片。结果表明,突变株的菌丝球较野生型菌株变得小而疏松。以上对本发明的具体实施例进行了描述。需要理解的是,本发明并不局限于上述特定实施方式,本领域技术人员可以在权利要求的范围内做出各种变形或修改,这并不影响本发明的实质内容。序列表<110>上海交通大学江苏东汇生物科技有限公司泰兴市东圣生物科技有限公司<120>敲除csla基因以提高谷氨酰胺转氨酶发酵水平的方法<130>kag45790<160>12<170>siposequencelisting1.0<210>1<211>1857<212>dna<213>茂源链霉菌dsm40587(streptomycesmobaraensisdsm40587)<400>1atgacgtccacgcctgccgacgcgcggcggcagatcccgtacgatccgccccgcgacccg60tcggtcaccacccggccgcgcatccccgcgcccccggggccggacgacgcgcggcgcaga120cccgcccctccgcgctcggcccgcccgcgtgcccccagatccttccatccccgctacgac180tacgagcactacagccggctcgccggcccgctcacccgcccgggggacggcccctacacc240gtccgctaccgcagcctgctggccgacgagccgcaccgggtccgggccgcgctcctgctg300ggcgcggcccccgtcgtctccgtcgtcctcctcctctggctgctccagcccgcccactgg360gtgcagcgcgaccacgtcggaccgctgatgcgcgtcctcgacctggtcatgctggtctcc420atcgggctgatcgagctcttccggacgctgaacgtcatctccaacgcccatgccacgctg480gtcgcccgcgacccgatacccgtcgtccccgagaccggcacccgggtcgcgttcctcacc540tcgttcgtgcccggcaaggagccgatcgcgatggtcacccgcaccctggaggccgccgtg600cgggtccgccaccgcggccctgtggacgtctggctcctggacgagggcgacgaccccgag660gtcaaggcggtctgcgcccggctgggggtgcgccacttcacccgcaaggggatcgagagg720tggaaccgccccaagggcccgcaccgggcgcggaccaagcacggcaactacaacgcctgg780ctggacgcgcacggcgacgactacgacttcttcgcctccgtggacaccgaccacgtgccg840ctgcccaacttcctggagcgcatgctcggttacttccgtgaccccaatgtcgcctttgtc900gttggacctcaggtgtacgggaactacgacgcgacggtcaccaaggccgccgagagccag960cagttcctcttccatgccctcatccagcgcgcgggcaaccgctacggcgcccccatgttc1020gtcggcaccaacaacgcggtgcggatcagcgccctcaagggcgtcggcggtctctacgac1080tcgatcaccgaggacatggccaccggcttcgagctccaccgccaccgcaatccggccacc1140ggccgccgctggcgctccgtctacacgcccgacgtcctcgccgtcggggagggccccaac1200gcctggacggacttcttcacccagcagctgcgctggtcccggggcacctacgagacgatc1260atcaagcagtactggaagggccccttcaccctcgcgcccggccctttcctcaactacacg1320ctcatggtcatctactatccgatgaccgcggtcaactggatgctgggggccctcagttgc1380accctcttcctgggcctgggcgcggccggcatccagatcgactcacaggtctggatgatg1440ctctacagcgacgccgccgcactccagatcggcctctacatctggaaccgccggcacaac1500gtctcccctcacgagcccgagggctccggcggggtcgccggcatggtgatgtccgctctc1560tccgcgcccgtctacgcccgctcgctgctcgacgccgtcctgcgccgcaagagccgcttc1620gtggtcacccccaagggtgactccgccagcccggacaccctcttcggcaccttccgcatc1680cacctcttcttcctactgatcttcggcggttcgctggtggcctcgttcttcctcggccac1740agccatgcggcgatgcgcacctgggcggccctggccgtcgtcatcacggtcgcgccgatc1800gtcgcctggtggtggaccgcgcgccacggcaagcagcccgagggagcccagccatga1857<210>2<211>40<212>dna<213>人工序列(artificialsequence)<400>2cgacggccagtgccaagcttgcgcacagacagccgagagg40<210>3<211>40<212>dna<213>人工序列(artificialsequence)<400>3gtcggtcaggtgccggagtccagtcattccccccacacgc40<210>4<211>20<212>dna<213>人工序列(artificialsequence)<400>4gactccggcacctgaccgac20<210>5<211>40<212>dna<213>人工序列(artificialsequence)<400>5gtcggtcaggtgccggagtccagtcattccccccacacgc40<210>6<211>40<212>dna<213>人工序列(artificialsequence)<400>6cccaagaccaagaagtgggcgcaccgactcggtgccactt40<210>7<211>40<212>dna<213>人工序列(artificialsequence)<400>7gtcctaggtataatactagtccgccgcgcgtcggcaggcg40<210>8<211>30<212>dna<213>人工序列(artificialsequence)<400>8ctgatgccggggaccgggatgaacgagttg30<210>9<211>30<212>dna<213>人工序列(artificialsequence)<400>9ggccggggttcatgtaccggcgtaccacgc30<210>10<211>21<212>dna<213>人工序列(artificialsequence)<400>10caaccgagcgttctgaacaaa21<210>11<211>20<212>dna<213>人工序列(artificialsequence)<400>11cgctcccttctctgacgccg20<210>12<211>20<212>dna<213>人工序列(artificialsequence)<400>12ccgccgcgcgtcggcaggcg20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1