一种提高NMN生物合成酶Nampt的酶活的创新方法与流程
本发明属于基因工程技术领域,具体涉及一种提高nmn生物合成酶nampt的酶活的创新方法。
背景技术:
烟酰胺单核苷酸(nicotinamidemononucleotide,nmn)是一种有机分子,也是一种核苷酸,具有逆转衰老和延长寿命的作用。
目前,多采用酶促反应来实现烟酰胺单核苷酸的合成,天然烟酰胺磷酸核糖转移酶(niacinamidephosphoribosyltransferase,nampt)存在酶活性较低的问题,导致传统的酶促反应成本高昂、反应条件苛刻、生产工艺不稳定、每个批次产品指标相差大、反应产能低,难以实现工厂化大规模生产,限制了nmn的大规模应用。
技术实现要素:
有鉴于此,本发明的目的在于提供一种提高nmn生物合成酶nampt的酶活的创新方法,具体在于提供一种编码nampt的突变蛋白的重组表达载体及重组菌和nampt突变蛋白,利用重组菌表达所述nampt突变蛋白后,酶活显著提高,可实现工厂化大规模生产。
为了实现上述发明目的,本发明提供以下技术方案:
本发明提供了一种烟酰胺磷酸核糖转移酶nampt的突变蛋白,对烟酰胺磷酸核糖转移酶nampt的氨基酸序列进行点突变,所述烟酰胺磷酸核糖转移酶nampt的氨基酸序列包括seqidno.1所示的序列;
所述点突变的位点包括:n67k,n164l,r166w,a208g,a245t,s248a,v365l或s382m。
本发明还提供了一种包括编码上述突变蛋白的核苷酸序列的重组表达载体。
优选的,所述重组表达载体的基础载体包括ppsumo载体。
本发明还提供了一种表达上述突变蛋白或包括上述重组表达载体的重组菌。
优选的,所述重组菌的基础菌株包括大肠杆菌。
本发明还提供了上述重组菌的构建方法,包括以下步骤:(1)对seqidno.1所示氨基酸序列的编码基因进行密码子优化,得优化基因;
(2)以所述优化基因为模板,利用定点突变引物和高保真酶分别进行pcr扩增,得扩增产物;
(3)利用dpni酶分别消化所述扩增产物后分别连接ppsumo载体,得重组表达载体;
(4)将所述重组表达载体分别转化大肠杆菌感受态细胞,挑选阳性克隆子,得所述重组菌。
优选的,步骤(1)所述优化基因的核苷酸序列包括seqidno.2所示的序列。
优选的,步骤(2)所述定点突变引物包括:针对n67k点突变的定点突变引物n67k-f和n67k-r,所述n67k-f的核苷酸序列如seqidno.3所示,所述n67k-r的核苷酸序列如seqidno.4所示;
针对n164l点突变的定点突变引物n164l-f和n164l-r,所述n164l-f的核苷酸序列如seqidno.5所示,所述n164l-r的核苷酸序列如seqidno.6所示;
针对r166w点突变的定点突变引物r166w-f和r166w-r,所述r166w-f的核苷酸序列如seqidno.7所示,所述r166w-r的核苷酸序列如seqidno.8所示;
针对a208g点突变的定点突变引物a208g-f和a208g-r,所述a208g-f的核苷酸序列如seqidno.9所示,所述a208g-r的核苷酸序列如seqidno.10所示;
针对a245t点突变的定点突变引物a245t-f和a245t-r,所述a245t-f的核苷酸序列如seqidno.11所示,所述a245t-r的核苷酸序列如seqidno.12所示;
针对s248a点突变的定点突变引物s248a-f和s248a-r,所述s248a-f的核苷酸序列如seqidno.13所示,所述s248a-r的核苷酸序列如seqidno.14所示;
针对v365l点突变的定点突变引物v365l-f和v365l-r,所述v365l-f的核苷酸序列如seqidno.15所示,所述v365l-r的核苷酸序列如seqidno.16所示;
针对s382m点突变的定点突变引物s382m-f和s382m-r,所述s382mk-f的核苷酸序列如seqidno.17所示,所述s382m-r的核苷酸序列如seqidno.18所示。
优选的,步骤(2)所述pcr扩增的程序包括:94℃预变性2min;98℃变性10s,55~65℃退火30s,68℃延伸4min,30循环;68℃延伸4min。
优选的,步骤(4)所述挑选阳性克隆子,包括利用nampt-f和nampt-r进行菌液pcr,所述nampt-f的核苷酸序列如seqidno.23所示,所述nampt-r的核苷酸序列如seqidno.24所示。
本发明提供了一种烟酰胺磷酸核糖转移酶nampt的突变蛋白,所述突变蛋白为利用foldx和deepddg两款软件分析目标蛋白nampt的结构,预测出的多个影响酶功能的关键位点,进行酶的半理性设计。在本发明实施例中,根据点突变原理,设计引物构建的10个突变菌株,经测序验证显示在设定的位点均成功突变,故10个突变菌株成功克隆。克隆的10个突变体中,有8个突变体的活性高于野生型(e.colidh5α-ppsumo-nampt)菌株,其中nampt-v365l突变体的活性最高,nmn的产量为45.42mg/l,相对于野生型(28.11mg/l)提高了62%。突变株nampt-s248a,nampt-n164l,nampt-s382m,nampt-a245t,nampt-a208g的nmn产量相对于野生型提高了34%,27%,27%,22%和17%,而nampt-v467l和s155i的nmn产量分别下降了53%和31%。
附图说明
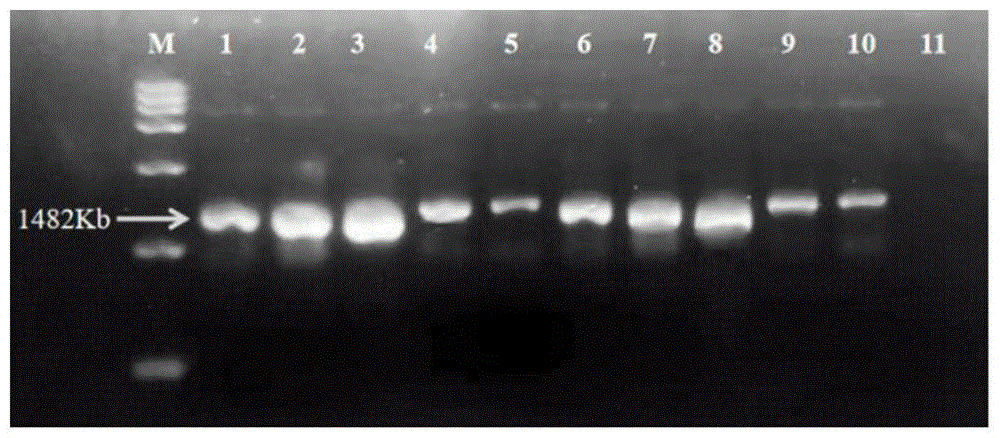
图1为菌落pcr验证结果,其中m:15000dlmarker;1:n67k,2:s155i;3:n164l;4:r166w;5:a208g;6:a245t;7:s248a;8:v365l;9:s382m;10:v467l;11:阴性对照;
图2为突变菌株测序结果比对图,图中n为nampt原始序列,1-10为突变菌株,其中1:n67k,2:s155i;3:n164l;4:r166w;5:a208g;6:a245t;7:s248a;8:v365l;9:s382m;10:v467l;红色序列中的白色缺失部位为突变位点的位置;
图3为nampt定点突变的菌株对转化反应生成nmn的影响。
具体实施方式
本发明提供了一种烟酰胺磷酸核糖转移酶nampt的突变蛋白,对烟酰胺磷酸核糖转移酶nampt的氨基酸序列进行点突变,所述烟酰胺磷酸核糖转移酶nampt的氨基酸序列包括seqidno.1所示的序列:gnaaaeaefnillatdsykvthykqyppntskvysyfecrekktenskvrkvkyeetvfyglqyilnkylkgkvvtkekiqeakevyrehfqddvfnergwnyilekydghlpievkavpegsviprgnvlftventdpecywltnwietilvqswypitvatnsreqkkilakylletsgnldgleyklhdfgyrgvssqetagigasahlvnfkgtdtvagialikkyygtkdpvpgysvpaaehstitawgkdhekdafehivtqfssvpvsvvsdsydiynacekiwgedlrhlivsrsteapliirpdsgnpldtvlkvldilgkkfpvtenskgykllppylrviqgdgvdintlqeivegmkqkkwsienvsfgsggallqkltrdllncsfkcsyvvtnglgvnvfkdpvadpnkrskkgrlslhrtpagnfvtleegkgdleeyghdllhtvfkngkvtksysfdevrknaqlnieqdvaph;
所述点突变的位点包括:n67k,n164l,r166w,a208g,a245t,s248a,v365l或s382m。
本发明优选通过综合使用基于序列保守性分析和基于结构吉布斯自由能变分析的方法,利用foldx和deepddg两款软件,预测高质量突变位点;其中foldx软件使用生物信息学方法来模拟突变位点对蛋白质解折叠自由能(δg)的影响,如果突变体δg(mutant)小于野生型δg(wild),则说明该突变对蛋白质的热稳定性具有积极作用。如果突变后δg增加,则表明该突变位点不利于蛋白质的稳定性。蛋白质工程中deepddg软件可以准确预测由于点突变引起的蛋白质稳定性变化情况。deepddg分析是一种基于神经网络的方法,该神经网络已在5700多个手动计划的实验数据点上进行了测试。对于三个独立的测试集,皮尔逊相关系数为0.48~0.56。软件分析结果表明,突变残基溶解性,接触面积是最重要的特征,这表明掩埋的疏水面积是蛋白质稳定性的主要决定因素。利用上述方法,本申请实施例中共选定了10个突变位点,分别为n67k,s155i,n164l,r166w,a208g,a245t,s248a,v365l,s382m和v467l,且上述10个突变位点的氨基酸和核苷酸序列变化如表1所示。
表1点突变的氨基酸和核苷酸序列变化
本发明还提供了一种包括编码上述突变蛋白的核苷酸序列的重组表达载体。
本发明所述重组表达载体的基础载体优选包括ppsumo载体,且编码所述突变蛋白的核苷酸序列优选连接入ppsumo载体的hindiii和ndei酶切位点之间。
本发明还提供了一种表达上述突变蛋白或包括上述重组表达载体的重组菌。
本发明所述重组菌的基础菌株优选包括大肠杆菌。
本发明还提供了上述重组菌的构建方法,包括以下步骤:(1)对seqidno.1所示氨基酸序列的编码基因进行密码子优化,得优化基因;
(2)以所述优化基因为模板,分别利用定点突变引物和高保真酶进行pcr扩增,得扩增产物;
(3)利用dpni酶消化分别所述扩增产物后分别连接ppsumo载体,得重组表达载体;
(4)将所述重组表达载体分别转化大肠杆菌感受态细胞,挑选阳性克隆子,得所述重组菌。
本发明对seqidno.1所示氨基酸序列的编码基因进行密码子优化,得优化基因。本发明优选遵循大肠杆菌偏好的密码子进行密码子优化,所得优化基因的核苷酸序列包括seqidno.2所示的序列(1482bp):catatgaacgctgctgctgaggccgagttcaatatattgttagcgaccgactcgtacaaggtcacgcattataaacagtatcctcctaacacatcaaaggtctactcatatttcgagtgccgcgagaagaagacggagaactcgaaagtccgaaaggtgaagtatgaagaaacagtgttctacgggcttcagtatattcttaacaaatatcttaaaggcaaagttgttacaaaggagaagatccaggaagctaaagaagtttatcgcgaacatttccaagacgatgtcttcaatgagcgcggctggaactatattcttgagaagtacgacggccatcttcctattgaagttaaagctgttcctgaaggctcagttattcctcgcggcaacgtcctgtttaccgtcgagaatacggatcctgaatgttattggcttacaaactggattgaaacaattcttgttcagtcatggtatcctattacagttgctacaaactcacgcgaacagaagaagatcctagctaaatatcttcttgaaacatcaggcaaccttgatggccttgaatataaacttcatgatttcgggtaccgcggcgtttcatcacaggaaacagctggcattggcgcttcagctcatcttgttaactttaaaggcacagatacagttgctggcattgctcttattaagaagtactacggcacaaaggacccagttcctggttattcagttcctgctgctgaacattcaacaattacagcttggggaaaggatcatgagaaggacgcgttcgagcacattgttacacagttcagtagtgttcctgtttcagttgtttcagattcttatgatatttataacgcttgtgagaagatctggggagaggaccttcgccatcttattgtttcacgctcaacagaagctcctcttattattcgccctgattcaggcaaccctcttgatacagttcttaaagttcttgatattcttggcaagaagttcccggttaccgagaattccaagggttataaacttcttcctccttatcttcgcgttattcagggcgatggcgttgatattaacacacttcaggaaattgttgaaggcatgaaacagaagaagtggtccattgagaatgtctcatttggctcaggcggcgctcttcttcagaaacttacacgcgatcttcttaactgttcatttaaatgttcttatgttgttacaaacggccttggcgttaacgtgttcaaagatcccgtagcagaccctaacaaacgctcaaagaagggtcgactttcacttcatcgcacacctgctggcaactttgttacacttgaagaaggcaaaggcgatcttgaagaatatggccatgatcttcttcatacagtgttcaagaatggcaaggtaacgaagtcctactcatttgatgaagttcgcaagaatgcgcagcttaacattgaacaggatgttgctcctcataagctt。
得优化基因后,本发明以所述优化基因为模板,利用定点突变引物和高保真酶进行pcr扩增,得扩增产物。本发明所述高保真酶优选包括kod-plus-neo酶,购自东洋纺(上海)生物科技有限公司。
本发明所述定点突变引物的信息优选如表2所示:
表2定点突变的引物信息
本发明所述引物在设计时,优选的设置突变位点位于两条引物上,分别位于正向突变引物重叠区下游,紧邻重叠区,反向突变引物5’端,引物包括5’端重叠区和3’端延伸区,除了突变位点之外,每条引物的长度大约为25~30bp,5’端的重叠区包含15~20个碱基,3’端延伸区至少含有10个碱基。
本发明所述pcr扩增的体系以50μl计,优选包括突变引物f/r(10μm)各1.5μl,10×pcrbufferforkod-plus-neo5μl,2mmdntps5μl,25mmmgso43μl,dna模板<1ng,kod-plus-neo(1u/μl)1μl,ddh2o补足50μl。本发明在配制所述体系时,优选在冰上操作,且kod-plus-neo酶最后加入,以保证酶的活性。本发明所述pcr扩增的程序优选包括:94℃预变性2min;98℃变性10s,55~65℃退火30s,68℃延伸4min,30循环;68℃延伸4min。
得扩增产物后,本发明利用dpni酶分别消化所述扩增产物后分别连接ppsumo载体,得重组表达载体。本发明优选使用takara公司的dpni快切酶去除模板dna(未突变)中的甲基化,且酶切体系以50μl计,优选包括:dpni酶1μl,10×quickcutbuffer5μl,扩增产物<1μl,ddh2o补足50μl。本发明所述消化优选包括将所述酶切体系放置37℃恒温培养箱中静置酶切3h,于85℃金属浴中加热5min,使酶失活后,4℃短暂储存。
本发明将dpni酶消化后的扩增产物连接入ppsumo载体,得所述重组表达载体,所述连接优选包括连接入ppsumo载体的hindiii和ndei酶切位点之间。本发明对所述连接的方法并没有特殊限定,利用本领域的常规方法进行连接即可。
得重组表达载体后,本发明将所述重组表达载体转化大肠杆菌感受态细胞,挑选阳性克隆子,得所述重组菌。
本发明对所述转化的方法并没有特殊限定,利用本领域的常规转化方法进行转化即可。本发明在筛选阳性克隆子时,优选用无菌牙签挑取平板中的单菌落于20μl无菌的ddh2o中,于95℃金属浴中加热5min,13000rmp高速离心2min,上清即可作为验证pcr模板,而后利用引物nampt-f(seqidno.23:taatccttattcagtggtggtggtggtggtgctc)和nampt-r(seqidno.24:aggaagcttgcatatgaacgctgctgctg)进行菌液pcr。
本发明所述菌液pcr的体系以50μl计,优选包括:premixtaq25μl,nampt-f/r各1μl,模板<1ng,ddh2o补足50μl。本发明所述菌液pcr的程序优选包括:94℃预变性2min;98℃变性10s,55℃退火15s,72℃延伸30s,30循环;72℃延伸2min。本发明对利用所述菌液pcr挑选出来的阳性克隆子优选再进行验证测序,测序正确的即为所述重组菌。
本发明在得到所述重组菌后,优选以烟酰胺单核苷酸(nmn)的产量作为筛选烟酰胺磷酸核糖转移酶(nampt)正向突变菌株的依据,且产生nmn的转化体系以25μl计,优选包括:12.5μl粗酶液、12.5μl母液(1mmnam,1mmprpp,1mmmncl2,1mmmgcl2)。本发明将上述转化体系混合均匀,于37℃,180rmp摇床中反应15min后,在95℃的金属浴中加热1min,使酶失活从而终止反应;然后用ph为6.0的pbs缓冲液稀释到500μl,10000rmp,离心5min,0.22μm的微孔滤膜过滤除菌,转移至液相小瓶中采用hplc法测量nmn的产量。经验证,本发明获得的10个突变体中,具有比野生型nampt菌株(e.colidh5α-ppsumo-nampt)更高催化活性的突变体为n67k,n164l,r166w,a208g,a245t,s248a,v365l和s382m;催化活性降低的菌株为s155i和v476l。
本发明对所述野生型nampt菌株(e.colidh5α-ppsumo-nampt)的构建方法并没有特殊限定,优选以双酶切的方法进行构建,更优选由苏州金唯智公司合成的来源于小鼠的烟酰胺磷酸转移酶(mnampt)序列(氨基酸序列如seqidno.1所示),并且将其构建到载体pet-30a中克隆到细胞e.coli-dh5α和e.colibl21(de3)中,目的基因两端含有酶切位点hindiii和ndei,并在末尾处加上6×his标签,即构建得到e.colidh5α-ppsumo-nampt。
下面结合实施例对本发明提供的一种提高nmn生物合成酶nampt的酶活的创新方法进行详细的说明,但是不能把它们理解为对本发明保护范围的限定。
实施例1
(1)用移液枪吸取200μl甘油保藏的菌株e.colidh5α-ppsumo-nampt接种于20mllb培养基(含卡那霉素50μg/ml),在37℃,200rpm的条件下恒温摇床振荡过夜培养;利用plasimidminikiti(100)试剂盒(购自omega)抽提质粒;
(2)在冰上配制pcr扩增反应体系(50μl),最后加入kod-plus-neo酶,以保证酶的活性:突变引物f/r(10μm)各1.5μl,10×pcrbufferforkod-plus-neo5μl,2mmdntps5μl,25mmmgso43μl,dna模板<1ng,kod-plus-neo(1u/μl)1μl,ddh2o补足50μl;
所涉及的突变引物为表2所示的引物,且由艾基生物技术有限公司合成;
pcr扩增程序:94℃预变性2min;98℃变性10s,55~65℃退火30s,68℃延伸4min,30循环;68℃延伸4min;4℃保存。
(3)取5μl上述pcr反应产物,加1μl6×loadingbuffer,用点样吸头转入琼脂糖凝胶胶孔中电泳中,于120v电压,室温下运行25min;
(4)使用takara公司的dpni快切酶去除模板dna(未突变)中的甲基化,酶切体系(50μl):dpni酶1μl,10×quickcutbuffer5μl,(2)中扩增产物<1μl,ddh2o补足50μl。本发明所述消化优选包括将所述酶切体系放置37℃恒温培养箱中静置酶切3h,于85℃金属浴中加热5min,使酶失活后,4℃短暂储存,利用双酶切的方法连接入ppsumo载体的hindiii和ndei多酶切位点之间,而后转化到e.colibl21(de3)感受态细胞;
(5)将上述重组菌导入具有卡那霉素(50μg/ml)抗性的固体琼脂培养基中,用无菌牙签挑取平板中的单菌落于20μl无菌的ddh2o中,于95℃金属浴中加热5min,13000rmp高速离心2min,上清即可作为验证pcr模板:premixtaq25μl,nampt-f/r各1μl,模板<1ng,ddh2o补足50μl;
程序优选包括:94℃预变性2min;98℃变性10s,55℃退火15s,72℃延伸30s,30循环;72℃延伸2min;4℃保存。
(6)将验证出来条带的阳性克隆子(至少3个/平板,图1),1%接种于含有lb卡那霉素(50μg/ml)抗性液体培养基的锥形瓶(20ml/50ml)中,37℃,180rmp的恒温摇床中振荡培养12h,次日,每个瓶子取1ml送于艾基生物技术有限公司测序,测序结果(图2)与原始nampt酶进行比对,将成功突变成与设计一致的菌株进行甘油管保菌。
测序所用的引物列于表3。
表3各突变位点的测序引物
实施例2
对实施例1中得到的10个突变体进行sds-page电泳表达验证
1、诱导表达
(1)种子培养:按1%的接种量,从甘油管中取100μl菌液接种到含有kana(50μg/ml)的lb液体培养基的锥形瓶(10ml/50ml)中,于条件设置为37℃,200rmp的恒温摇床中振荡培养12h。
(2)发酵培养:按2%的接种量,将种子液接种到含有kana抗性的lb液体培养基的锥形瓶(100ml/500ml)中,37℃,180rmp恒温摇床中培养2~3h至光密度值od600为0.5~0.6时,加入0.25mm的iptg,在30℃,180rmp的条件下进行诱导表达12h。
2、样品前处理
(1)取1ml菌液于1.5mlep管中,6000rmp,25℃,离心3min,弃上清。
(2)加入1mlph值为7.4的pbs缓冲溶液重悬菌体。
(3)调光密度值od600为1.0,取10μl的4×proteinloadingbuffer加入30μl的(2)中菌溶液,振荡混合均匀。
(4)沸水煮10min。
(5)瞬时离心3min,取上清20μl上样,sds-page凝胶电泳:90v运行30min,改调电压为120v运行1.5h。
3、突变后nampt酶活测试
(1)粗酶液的准备:1)取10ml菌液于50ml离心管中,4℃,4000rpm,离心20min,弃上清。
2)加入10mlph为7.4的pbs缓冲溶液吹打重悬菌体,放入冰盒内。
3)吸取200μl2)中的细胞溶液至96孔酶标板中,调节细胞光密度值od600至1.0。
4)取10ml上述3)进行4℃,4000rpm离心20min,用2mlpbs缓冲液重悬,将其浓缩5倍。
5)准备一个洁净的25ml的小烧杯,将细胞液倒入烧杯中,置于冰水浴中。
6)将超声细胞破碎仪参数设定为功率30%,工作5s,暂停5s,进行细胞超声破碎10min。
7)破碎完毕后,4℃,4000rpm的条件下,离心20min上清液即为粗酶液,可放置-20℃冰箱保存。
(2)转化反应:转化体系:12.5μl粗酶液、12.5μl母液(1mmnam,1mmprpp,1mmmncl2,1mmmgcl2),混合均匀,于37℃,180rmp摇床中反应15min后,在95℃的金属浴中加热1min,使酶失活从而终止反应。用ph为6.0的pbs缓冲液稀释到500μl,10000rmp,离心5min,0.22μm的微孔滤膜过滤除菌,转移至液相小瓶中采用hplc法测量nmn的产量。
hplc法测量条件:
①色谱柱:chromcorec18反相色谱柱5μm,4.6×250mm。
流动相:a=磷酸盐缓冲液(ph3.5),b=100%甲醇。
柱温:25℃。
流速:1.0ml/min,波长260nm处紫外检测,进样量20μl。
②标准曲线的测定
称量10~15mg的烟酰胺单核苷酸标准品,用无菌的ddh2o定容至25ml,用ddh2o稀释10倍,作为100%nmn标准液,然后将100%nmn标准液稀释成浓度为1%,10%,20%,40%,60%,80%的nmn标准液,用0.22μm的微孔滤膜进行过滤除菌,利用hplc测定不用浓度nmn的出峰面积,以nmn浓度为横坐标,峰面积为纵坐标绘制nmn标准曲线。
③hplc测定反应液中的nmn
(1)上样前处理:反应后的转化液于1.5mlep管中,稀释20倍至500μl,室温下,10000rpm离心3min,用无菌的1ml针头吸取离心后的上清液,通过一次性无菌的0.22μm的微孔滤膜过滤除菌并转移至hplc专用的液相小瓶中,得到待测样品。
(2)将液相小瓶放入hplc的自动加样器中,按照方法①设定hplc的参数条件,测定酶转化反应后的nmn的产量。
结果如图3所示,设计的10个突变体中,具有比野生型nampt菌株更高催化活性的突变体为n67k,n164l,r166w,a208g,a245t,s248a,v365l和s382m。催化活性降低的菌株为s155i和v476l。在突变体中,nampt-v356l具有最高的活性。当将突变体nampt-v356l用于催化时,所获得产物nmn的浓度为45.42mg/l,与野生型nampt(约28.11mg/l)相比,增加了62%,该位点从中性非极性的缬氨酸突变为中性非极性亮氨酸。因此,在基因序列356的位置,亮氨酸对酶活性有正向作用。其次,突变体nampt-s248a的催化活性比野生型nampt菌株高34%,表明疏水性氨基酸(丝氨酸:中性极性亲水氨基酸,丙氨酸:中性非极性疏水氨基酸)改善该区域的环境以增加nampt的稳定性是有益的。此外,突变体n164l,s382m,a245t和a208g的活性均略有提高,nmn产量分别提高到35.82mg/l,35.75mg/l,34.35mg/l和33.03mg/l。酶活性比原始菌株分别高27%,27%,22%,17%。研究结果也表明其他突变体具有不同程度降低活性的副作用。例如,nampt-v467l从缬氨酸突变为酸性亮氨酸,酶活性大大降低,该菌株nmn产量仅为9.31mg/l;nampt-v467l和nampt-s155i的nmn产量分别下降了53%和31%。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110>泓博元生命科技(深圳)有限公司
<120>一种提高nmn生物合成酶nampt的酶活的创新方法
<160>34
<170>siposequencelisting1.0
<210>1
<211>491
<212>prt
<213>人工序列(artificialsequence)
<400>1
glyasnalaalaalaglualaglupheasnileleuleualathrasp
151015
sertyrlysvalthrhistyrlysglntyrproproasnthrserlys
202530
valtyrsertyrpheglucysargglulyslysthrgluasnserlys
354045
valarglysvallystyrglugluthrvalphetyrglyleuglntyr
505560
ileleuasnlystyrleulysglylysvalvalthrlysglulysile
65707580
glnglualalysgluvaltyrarggluhispheglnaspaspvalphe
859095
asngluargglytrpasntyrileleuglulystyraspglyhisleu
100105110
proilegluvallysalavalprogluglyservalileproarggly
115120125
asnvalleuphethrvalgluasnthraspproglucystyrtrpleu
130135140
thrasntrpilegluthrileleuvalglnsertrptyrproilethr
145150155160
valalathrasnserarggluglnlyslysileleualalystyrleu
165170175
leugluthrserglyasnleuaspglyleuglutyrlysleuhisasp
180185190
pheglytyrargglyvalserserglngluthralaglyileglyala
195200205
seralahisleuvalasnphelysglythraspthrvalalaglyile
210215220
alaleuilelyslystyrtyrglythrlysaspprovalproglytyr
225230235240
servalproalaalagluhisserthrilethralatrpglylysasp
245250255
hisglulysaspalaphegluhisilevalthrglnpheserserval
260265270
provalservalvalseraspsertyraspiletyrasnalacysglu
275280285
lysiletrpglygluaspleuarghisleuilevalserargserthr
290295300
glualaproleuileileargproaspserglyasnproleuaspthr
305310315320
valleulysvalleuaspileleuglylyslyspheprovalthrglu
325330335
asnserlysglytyrlysleuleuproprotyrleuargvalilegln
340345350
glyaspglyvalaspileasnthrleuglngluilevalgluglymet
355360365
lysglnlyslystrpserilegluasnvalserpheglyserglygly
370375380
alaleuleuglnlysleuthrargaspleuleuasncysserphelys
385390395400
cyssertyrvalvalthrasnglyleuglyvalasnvalphelysasp
405410415
provalalaaspproasnlysargserlyslysglyargleuserleu
420425430
hisargthrproalaglyasnphevalthrleuglugluglylysgly
435440445
aspleugluglutyrglyhisaspleuleuhisthrvalphelysasn
450455460
glylysvalthrlyssertyrserpheaspgluvalarglysasnala
465470475480
glnleuasnilegluglnaspvalalaprohis
485490
<210>2
<211>1482
<212>dna
<213>人工序列(artificialsequence)
<400>2
catatgaacgctgctgctgaggccgagttcaatatattgttagcgaccgactcgtacaag60
gtcacgcattataaacagtatcctcctaacacatcaaaggtctactcatatttcgagtgc120
cgcgagaagaagacggagaactcgaaagtccgaaaggtgaagtatgaagaaacagtgttc180
tacgggcttcagtatattcttaacaaatatcttaaaggcaaagttgttacaaaggagaag240
atccaggaagctaaagaagtttatcgcgaacatttccaagacgatgtcttcaatgagcgc300
ggctggaactatattcttgagaagtacgacggccatcttcctattgaagttaaagctgtt360
cctgaaggctcagttattcctcgcggcaacgtcctgtttaccgtcgagaatacggatcct420
gaatgttattggcttacaaactggattgaaacaattcttgttcagtcatggtatcctatt480
acagttgctacaaactcacgcgaacagaagaagatcctagctaaatatcttcttgaaaca540
tcaggcaaccttgatggccttgaatataaacttcatgatttcgggtaccgcggcgtttca600
tcacaggaaacagctggcattggcgcttcagctcatcttgttaactttaaaggcacagat660
acagttgctggcattgctcttattaagaagtactacggcacaaaggacccagttcctggt720
tattcagttcctgctgctgaacattcaacaattacagcttggggaaaggatcatgagaag780
gacgcgttcgagcacattgttacacagttcagtagtgttcctgtttcagttgtttcagat840
tcttatgatatttataacgcttgtgagaagatctggggagaggaccttcgccatcttatt900
gtttcacgctcaacagaagctcctcttattattcgccctgattcaggcaaccctcttgat960
acagttcttaaagttcttgatattcttggcaagaagttcccggttaccgagaattccaag1020
ggttataaacttcttcctccttatcttcgcgttattcagggcgatggcgttgatattaac1080
acacttcaggaaattgttgaaggcatgaaacagaagaagtggtccattgagaatgtctca1140
tttggctcaggcggcgctcttcttcagaaacttacacgcgatcttcttaactgttcattt1200
aaatgttcttatgttgttacaaacggccttggcgttaacgtgttcaaagatcccgtagca1260
gaccctaacaaacgctcaaagaagggtcgactttcacttcatcgcacacctgctggcaac1320
tttgttacacttgaagaaggcaaaggcgatcttgaagaatatggccatgatcttcttcat1380
acagtgttcaagaatggcaaggtaacgaagtcctactcatttgatgaagttcgcaagaat1440
gcgcagcttaacattgaacaggatgttgctcctcataagctt1482
<210>3
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>3
cgggcttcagtatattcttaaaaaatatcttaaagg36
<210>4
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>4
tttaagaatatactgaagcccgtagaacactgt33
<210>5
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>5
cctattacagttgctacactgtcacgcgaac31
<210>6
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>6
cagtgtagcaactgtaataggataccat28
<210>7
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>7
gttgctacaaactcatgggaacagaagaag30
<210>8
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>8
ccatgagtttgtagcaactgtaataggatacc32
<210>9
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>9
ggaaacagctggcattggcggctcagctcatct33
<210>10
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>10
gccgccaatgccagctgtttcctgtgatgaaac33
<210>11
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>11
cctggttattcagttcctgctaccgaacattcaac35
<210>12
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>12
ggtagcaggaactgaataaccaggaactg29
<210>13
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>13
tcctgctgctgaacatgcgacaattacag29
<210>14
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>14
actatgttcagcagcaggaactgaataac29
<210>15
<211>37
<212>dna
<213>人工序列(artificialsequence)
<400>15
attaacacacttcaggaaattctggaaggcatgaaac37
<210>16
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>16
cagaatttcctgaagtgtgttaatatcaacg31
<210>17
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>17
attgagaatgtctcatttggcatgggcggcgctc34
<210>18
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>18
catgccaaatgagacattctcaatggacc29
<210>19
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>19
ggattgaaacaattcttgttcagatctggtatccta36
<210>20
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>20
gatctgaacaagaattgtttcaatccagtttg32
<210>21
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>21
agtgttcaagaatggcaagctgacgaagtcctactc36
<210>22
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>22
cagcttgccattcttgaacactgtatgaagaag33
<210>23
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>23
taatccttattcagtggtggtggtggtggtgctc34
<210>24
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>24
aggaagcttgcatatgaacgctgctgctg29
<210>25
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>25
aaaggtctactcatatttcgagtgccg27
<210>26
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>26
tctgttcgcgtgagtttgtagca23
<210>27
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>27
gtacgacggccatcttcctattga24
<210>28
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>28
gaactgggtcctttgtgccgtag23
<210>29
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>29
ggcgcttcagctcatcttgttaa23
<210>30
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>30
tgaataacgcgaagataaggaggaag26
<210>31
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>31
agaggaccttcgccatcttattg23
<210>32
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>32
aggacttcgttaccttgccattc23
<210>33
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>33
gttcaaagatcccgtagcagacc23
<210>34
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>34
gctagttattgctcagcggtggc23
- 还没有人留言评论。精彩留言会获得点赞!