基于KASP技术开发的不结球白菜核心SNP分子标记集及其应用
基于kasp技术开发的不结球白菜核心snp分子标记集及其应用
技术领域
1.本发明属于分子生物学及植物分子育种技术领域,具体涉及基于kasp技术开发的不结球白菜核心snp分子标记集及其应用。
背景技术:
2.不结球白菜(brassica rapa ssp.chinensis makino)为十字花科芸薹属芸薹种白菜亚种,包括小白菜、菜薹、薹菜、乌塌菜、分蘖菜等多个变种。不结球白菜原产中国,具有生长周期短、营养丰富、适应性强等特点,在全世界范围内栽培广泛,是一种具有重要经济价值的叶菜类蔬菜。
3.分子标记(rapd、ssr、indel、snp等)被广泛地应用于不结球白菜种质资源遗传多样性评价、遗传图谱构建与基因定位、分子标记辅助育种等方面。其中,rapd技术较易受到各种因素的影响。无论是模板的质量和浓度,短的引物序列,pcr的循环次数,基因组dna的复杂性,技术设备等,都有可能导致rapd结果的不稳定和难重复。ssr标记因具有多态性高、操作简单等优点,在我国蔬菜作物遗传育种中曾经被普遍应用。但是其无法满足大规模、高通量、自动化的检测需求。snp标记作为第三代分子标记,具有以下优势:一是在基因组中密度高分布更均匀;二是高通量,检测位点可以达到几百万个;三是容易实现数据统计的自动化和数据整合比较。目前,snp已经被应用到蔬菜作物如辣椒、甜瓜、西瓜、黄瓜的品种鉴定以及种子纯度检测等方面。随着高通量基因型分型技术的发展,具有较高snp使用效率和分析通量的竞争性等位基因特异性pcr(competitive allele
‑
specific pcr,kasp)技术问世。kasp是一种基于荧光检测的基因分型技术,即基于引物末端碱基的特异匹配对snp进行分型,并通过两个位点特异探针以及2个荧光探针来检测多个snp位点,从而对目标snp进行精准的双等位基因分型,具有高度稳定性与准确性。该技术目前主要被应用于snp或indel基因的分型研究中,并正逐渐成为遗传图谱构建、基因定位、分子辅助育种技种质资源鉴评的主要技术手段。
4.传统对不结球白菜种质和品种的鉴定评价主要依靠表型。随着我国不结球白菜种质资源收集保护工作的不断推进,以及育种杂交品种数量的逐年增多,传统的表型鉴定方法,因耗时耗力、周期长且准确性不高,成为种质资源高效利用的瓶颈。
5.因此,有必要开发一种快速、准确、有效的不结球白菜种质和品种鉴定评价方法。
技术实现要素:
6.为了克服上述现有技术的不足,本发明提出了基于kasp技术开发的不结球白菜核心snp分子标记集,基于上述snp分子标记集可以实现对不结球白菜种质和品种的鉴定评价。
7.为了实现上述目的,本发明所采用的技术方案是:
8.本发明提供了基于kasp技术开发的不结球白菜核心snp分子标记集,所述不结球
白菜核心snp分子标记集包括42个snp标记,所述42个snp标记的编号为snp1~42(snp1、snp2、snp3、snp4、snp5、snp6、snp7、snp8、snp9、snp10、snp11、snp12、snp13、snp14、snp15、snp16、snp17、snp18、snp19、snp20、snp21、snp22、snp23、snp24、snp25、snp26、snp27、snp28、snp29、snp30、snp31、snp32、snp33、snp34、snp35、snp36、snp37、snp38、snp39、snp40、snp41、snp42);
9.所述snp1标记为碱基c/t,位于不结球白菜1号染色体上,位置为不结球白菜全基因组序列的第21612761位;
10.所述snp2标记为碱基c/t,位于不结球白菜1号染色体上,位置为不结球白菜全基因组序列的第21624075位;
11.所述snp3标记为碱基g/a,位于不结球白菜1号染色体上,位置为不结球白菜全基因组序列的第27291001位;
12.所述snp4标记为碱基c/t,位于不结球白菜1号染色体上,位置为不结球白菜全基因组序列的第29042081位;
13.所述snp5标记为碱基c/t,位于不结球白菜2号染色体上,位置为不结球白菜全基因组序列的第12579271位;
14.所述snp6标记为碱基t/a,位于不结球白菜2号染色体上,位置为不结球白菜全基因组序列的第14670199位;
15.所述snp7标记为碱基t/c,位于不结球白菜2号染色体上,位置为不结球白菜全基因组序列的第6287090位;
16.所述snp8标记为碱基g/c,位于不结球白菜2号染色体上,位置为不结球白菜全基因组序列的第17101169位;
17.所述snp9标记为碱基c/a,位于不结球白菜3号染色体上,位置为不结球白菜全基因组序列的第7872365位;
18.所述snp10标记为碱基t/g,位于不结球白菜3号染色体上,位置为不结球白菜全基因组序列的第27336042位;
19.所述snp11标记为碱基t/c,位于不结球白菜3号染色体上,位置为不结球白菜全基因组序列的第10862382位;
20.所述snp12标记为碱基t/a,位于不结球白菜3号染色体上,位置为不结球白菜全基因组序列的第10870310位;
21.所述snp13标记为碱基c/t,位于不结球白菜3号染色体上,位置为不结球白菜全基因组序列的第11407563位;
22.所述snp14标记为碱基t/c,位于不结球白菜3号染色体上,位置为不结球白菜全基因组序列的第14544571位;
23.所述snp15标记为碱基c/t,位于不结球白菜4号染色体上,位置为不结球白菜全基因组序列的第10563214位;
24.所述snp16标记为碱基c/g,位于不结球白菜4号染色体上,位置为不结球白菜全基因组序列的第19380396位;
25.所述snp17标记为碱基c/t,位于不结球白菜4号染色体上,位置为不结球白菜全基因组序列的第11760387位;
26.所述snp18标记为碱基c/g,位于不结球白菜4号染色体上,位置为不结球白菜全基因组序列的第21624754位;
27.所述snp19标记为碱基g/c,位于不结球白菜5号染色体上,位置为不结球白菜全基因组序列的第1066885位;
28.所述snp20标记为碱基g/t,位于不结球白菜5号染色体上,位置为不结球白菜全基因组序列的第17807854位;
29.所述snp21标记为碱基c/a,位于不结球白菜5号染色体上,位置为不结球白菜全基因组序列的第1889510位;
30.所述snp22标记为碱基c/t,位于不结球白菜5号染色体上,位置为不结球白菜全基因组序列的第1581635位;
31.所述snp23标记为碱基g/a,位于不结球白菜6号染色体上,位置为不结球白菜全基因组序列的第23758128位;
32.所述snp24标记为碱基g/c,位于不结球白菜6号染色体上,位置为不结球白菜全基因组序列的第25457983位;
33.所述snp25标记为碱基t/c,位于不结球白菜6号染色体上,位置为不结球白菜全基因组序列的第18142570位;
34.所述snp26标记为碱基g/c,位于不结球白菜6号染色体上,位置为不结球白菜全基因组序列的第19301511位;
35.所述snp27标记为碱基g/c,位于不结球白菜7号染色体上,位置为不结球白菜全基因组序列的第15969630位;
36.所述snp28标记为碱基t/c,位于不结球白菜7号染色体上,位置为不结球白菜全基因组序列的第14610710位;
37.所述snp29标记为碱基g/t,位于不结球白菜7号染色体上,位置为不结球白菜全基因组序列的第15631302位;
38.所述snp30标记为碱基a/g,位于不结球白菜7号染色体上,位置为不结球白菜全基因组序列的第1750463位;
39.所述snp31标记为碱基g/c,位于不结球白菜8号染色体上,位置为不结球白菜全基因组序列的第18036348位;
40.所述snp32标记为碱基t/c,位于不结球白菜8号染色体上,位置为不结球白菜全基因组序列的第19882040位;
41.所述snp33标记为碱基c/t,位于不结球白菜8号染色体上,位置为不结球白菜全基因组序列的第1929087位;
42.所述snp34标记为碱基t/a,位于不结球白菜8号染色体上,位置为不结球白菜全基因组序列的第14605882位;
43.所述snp35标记为碱基c/g,位于不结球白菜9号染色体上,位置为不结球白菜全基因组序列的第37635371位;
44.所述snp36标记为碱基c/a,位于不结球白菜9号染色体上,位置为不结球白菜全基因组序列的第3011899位;
45.所述snp37标记为碱基c/a,位于不结球白菜9号染色体上,位置为不结球白菜全基
因组序列的第34173525位;
46.所述snp38标记为碱基g/t,位于不结球白菜9号染色体上,位置为不结球白菜全基因组序列的第39079259位;
47.所述snp39标记为碱基g/a,位于不结球白菜10号染色体上,位置为不结球白菜全基因组序列的第9999223位;
48.所述snp40标记为碱基c/a,位于不结球白菜10号染色体上,位置为不结球白菜全基因组序列的第11493584位;
49.所述snp41标记为碱基a/t,位于不结球白菜10号染色体上,位置为不结球白菜全基因组序列的第16824105位;
50.所述snp42标记为碱基t/a,位于不结球白菜10号染色体上,位置为不结球白菜全基因组序列的第19172885位。
51.本发明还提供了用于扩增上述不结球白菜核心snp分子标记集的kasp引物,所述kasp引物包括42个kasp引物组合,每个kasp引物组合用于扩增对应的snp标记。
52.进一步地,每个kasp引物组合由两条末端碱基不同的等位基因正向引物f1和f2以及一条反向引物r构成。
53.更进一步地,引物f1的5’端添加fam荧光标签序列,所述fam荧光标签序列如seq id no:127所示;引物f2的5’端添加hex荧光标签序列,所述hex荧光标签序列如seq id no:128所示.
54.具体地,所述kasp引物由seq id no:1~126所示的核苷酸序列组成。具体地,42个snp标记(snp1、snp2、snp3、snp4、snp5、snp6、snp7、snp8、snp9、snp10、snp11、snp12、snp13、snp14、snp15、snp16、snp17、snp18、snp19、snp20、snp21、snp22、snp23、snp24、snp25、snp26、snp27、snp28、snp29、snp30、snp31、snp32、snp33、snp34、snp35、snp36、snp37、snp38、snp39、snp40、snp41、snp42)对应的kasp引物组合分别为:seq id no:1~3、seq id no:4~6、seq id no:7~9、seq id no:10~12、seq id no:13~15、seq id no:16~18、seq id no:19~21、seq id no:22~24、seq id no:25~27、seq id no:28~30、seq id no:31~33、seq id no:34~36、seq id no:37~39、seq id no:40~42、seq id no:43~45、seq id no:46~48、seq id no:49~51、seq id no:52~54、seq id no:55~57、seq id no:58~60、seq id no:61~63、seq id no:64~66、seq id no:67~69、seq id no:70~72、seq id no:73~75、seq id no:76~78、seq id no:79~81、seq id no:82~84、seq id no:85~87、seq id no:88~90、seq id no:91~93、seq id no:94~96、seq id no:97~99、seq id no:100~102、seq id no:103~105、seq id no:105~108、seq id no:109~111、seq id no:112~114、seq id no:115~1117、seq id no:118~120、seq id no:121~123、seq id no:124~126。
55.本发明还提供了含有上述kasp引物的检测产品,所述检测产品包括检测试剂、试剂盒和芯片。
56.本发明还提供了上述不结球白菜核心snp分子标记集或上述kasp引物或上述检测产品在以下任一方面中的应用:
57.(1)不结球白菜种质资源或者品种的鉴定;
58.(2)不结球白菜snp指纹图库的构建;
59.(3)不结球白菜的遗传多样性分析;
60.(4)不结球白菜的遗传图谱构建及基因分型;
61.(5)不结球白菜的分子标记辅助育种;
62.(6)不结球白菜种质资源或者品种的纯度检测;
63.(7)不结球白菜的分子基因定位。
64.优选地,上述应用的具体方法包括以下步骤:
65.s1、提取不结球白菜样品的基因组dna;
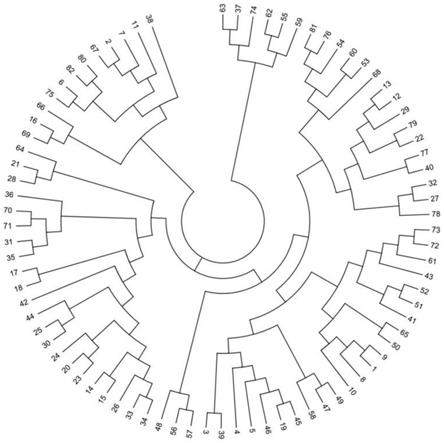
66.s2、以步骤s1中的dna为模板,采用权利要求2
‑
5任一项所述的kasp引物进行pcr扩增,并将pcr扩增产物放置在bio
‑
rad cfx connect实时荧光定量pcr仪上获取对应的产物荧光信号值,完成基因分型;
67.s3、采用数据分析软件bio
‑
rad cfx manager 3.1对步骤s2的实验结果进行分析。
68.进一步地,pcr扩增的反应体系为10μl,包括20ng/μl~30ng/μl的dna2μl,每个kasp引物组合0.28μl(其中两条f引物各0.07μl,r引物0.14μl),以及kasp master mixture 5μl。
69.进一步地,pcr扩增的反应条件为:94℃,15min;94℃,20sec,63℃~55℃,1min,每个循环降0.8℃,共10个循环;94℃,20sec,55℃,1min,共26个循环;荧光信号值的读取条件为16℃,20s。
70.与现有技术相比,本发明的有益效果是:
71.本发明基于kasp技术开发了不结球白菜的核心snp分子标记集,该不结球白菜核心snp分子标记集能够快速、准确、有效地鉴定评价种质资源材料或者育成品种,可以解决因大量种质积累而带来的资源冗余、保存管理不便以及知识产权纠纷等问题。为遗传图谱构建、基因定位、分子辅助育种等提供分子标记。
附图说明
72.图1为a06:19301511标记在82份资源中的多态性图谱;
73.图2为a03:7876365标记在82份资源中的多态性图谱;
74.图3为基于kasp标记的不结球白菜遗传距离聚类图;
75.图4为25个广东省不结球白菜地方种质资源的指纹图谱。
具体实施方式
76.下面对本发明的具体实施方式作进一步说明。在此需要说明的是,对于这些实施方式的说明用于帮助理解本发明,但并不构成对本发明的限定。此外,下面所描述的本发明各个实施方式中所涉及的技术特征只要彼此之间未构成冲突就可以相互组合。
77.下述实施例中的实验方法,如无特殊说明,均为常规方法,下述实施例中所用的试验材料,如无特殊说明,均为可通过常规的商业途径购买得到。
78.实施例1基于kasp技术开发不结球白菜核心snp分子标记
79.1、kasp引物设计
80.提取尖叶菜心“70天”(广东省农作物种质资源保护库gdⅱ2d00187)和圆叶菜心“c40”(广东省农作物种质资源保护库gdⅱ2d00114)的基因组dna。
81.利用illuminahiseqtm技术对“70天”和“c40”的基因组dna进行基因重测序,将重测序结果与白菜基因组(http://brassicadb.cn/)进行比对分析,选择有多态性且在染色体上均匀分布,位点前后各50bp不存在其他变异的snp位点150个,然后设计引物将该150个snp位点转化成kasp标记。
82.引物设计的方法为:根据snp位点和侧翼序列,利用软件primer premier 5.0按awais等(awais r,wen w e,gao f m,zhai s n,jin h,liu j d,guo q,zhangy j,dreisigacker s,xia x c,he z h.development and validation ofkasp assays for genes underpinning key economic traits inbread wheat.theoretical andapplied genetics,2016,129(10):1843
‑
1860)介绍的引物设计方法对差异位点设计kasp标记引物,开发kasp分子标记。每个标记设计2条snp特异性引物(f1/f2)和一条通用引物(r),其中,f1尾部添加能够与fam荧光结合的特异性序列5
’‑
gaaggtgaccaagttcatgct
‑3’
,f2尾部添加能够与hex荧光结合的特异性序列5
’‑
gaaggtcggagtcaacggatt
‑3’
。引物由生工生物工程(上海)股份有限公司合成。
83.2、kasp引物筛选
84.(1)收集82份不结球白菜种质资源组成不结球白菜自然群体,该自然群体包括菜心种质资源53份,小白菜种质资源29份(表1)。
85.表1 82份不结球白菜种质资源的详情表
86.[0087][0088]
(2)不结球白菜dna的提取:
[0089]
①
取不结球白菜的嫩叶约2g,用液氮研磨,在液氮快蒸发干时迅速加入1000μl 2%ctab提取缓冲液,混匀后置于65℃水浴中温浴50min,每隔10min摇动一次;
[0090]
②
静置至室温后在4℃下12000rpm离心10min,将上清约800μl转移到新的2ml离心管;
[0091]
③
加入等体积的氯仿/异戊醇(氯仿:异戊醇=24:1),颠倒混匀,静置3
‑
5分钟,在4℃下12000rpm离心10min,将上清液约600μl转入新的1.5ml离心管中;
[0092]
④
加等体积
‑
20℃下预冷的异丙醇,缓慢混匀,缓慢颠倒20次,置于
‑
20℃下培养30min;
[0093]
⑤
在4℃下12000rpm离心10min,看到底部白色沉淀后,弃上清,先后分别加入800μl 75%和95%酒精洗涤两次沉淀,弃上清,室温下于通风橱中晾干;
[0094]
⑥
加入100μl无菌水溶解,得到不结球白菜种质资源的dna。
[0095]
利用“70天”和“c40”为实验材料,对开发设计的kasp引物进行筛选。以“70天”和“c40”的dna为模板,加入kasp引物和通用的kaspmaster mix(购自广州固德生物技术有限公司)进行pcr(聚合链式反应)扩增。pcr反应体系为:10μl包括20ng/μl~30ng/μl的dna2μl,所述的分子标记引物共0.28μl,其中两条f引物各0.07μl,r引物0.14μl,以及5μl的2
×
kaspmaster mixture。反应的条件为:94℃,15min;94℃,20sec,63℃~55℃,1min,每个循环降0.8℃,共10个循环;94℃,20sec,55℃,1min,共26个循环。待反应完成后,将pcr扩增产物放置bio
‑
rad cfx connect实时荧光定量pcr仪上获取对应的产物荧光信号值,完成基因分型。荧光信号值的读取条件为16℃,20s。
[0096]
利用上述分型成功的kasp标记再对82份不结球白菜种质资源进行基因型检测,最终获得42个分型清晰的高质量kasp标记,这42个kasp标记均匀分布于不结球白菜10条染色体上,构成本发明的不结球白菜核心snp分子标记集。各引物对名称和核苷酸序列如表2所示。
[0097]
利用power marker v3.25软件计算各标记的等位基因频率、多态性信息含量,42个kasp标记在82份不结球白菜种质资源中多态性信息含量(pic)变化范围为0.1700
‑
0.4944,平均值为0.4032,pic值大于0.4032的位点占所有位点的61.9%;主要等位基因频率(maf)变化范围为0.4817
‑
0.9024,平均值为0.6289(部分标记的多态性图谱如图1和图2所示)。
[0098]
表2基于kasp技术开发的不结球白菜核心snp分子标记的引物信息
[0099]
[0100]
[0101]
[0102]
[0103]
[0104][0105]
实施例2不结球白菜kasp分子标记在不结球白菜遗传多样性分析中的应用
[0106]
将实施例1中的42个kasp标记的不结球白菜自然群体的基因分型数据形成a
‑
b矩阵,利用power marker v3.25软件计算各种质资源间的nei’s遗传距离。结果表明,82份种质资源的nei’s遗传距离变化范围为0.2352
‑
0.9594,平均值为0.5025。同时,根据距离矩阵构建系统发生树,进行neighbor
‑
joining聚类分析,生成结果如图3所示,可见82份不结球白菜种质资源可以划分三大类群。
[0107]
实施例3基于kasp技术开发不结球白菜核心snp分子标记应用于不结球白菜种质资源指纹图谱构建
[0108]
从实施例1中42个kasp标记中筛选pic值大于平均值0.4032,缺失率小于0.1,且均匀分布在10条染色体上的标记作为构建不结球白菜种质资源指纹图谱的核心标记,最终获得21个kasp标记用于构建不结球白菜种质资源的指纹图谱。
[0109]
利用21个核心kasp标记对26份广东省不结球白菜地方种质资源(表3)进行基因型检测,构建不结球白菜种质资源的dna指纹图谱。将26份广东省不结球白菜地方种质资源的核心位点分型结果转化为二元编码数据,得到主要广东省不结球白菜地方种质资源的指纹图谱(图4)。利用该dna指纹图谱可对广东省不结球白菜地方种质资源进行有效区分。
[0110]
表3 26份广东省不结球地方种质资源的详情表
[0111]
[0112][0113]
以上对本发明的实施方式作了详细说明,但本发明不限于所描述的实施方式。对于本领域的技术人员而言,在不脱离本发明原理和精神的情况下,对这些实施方式进行多种变化、修改、替换和变型,仍落入本发明的保护范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1