睡莲的SSR引物对及合成方法和应用
睡莲的ssr引物对及合成方法和应用
技术领域
1.本发明属于生物技术领域,特别是涉及一种睡莲的ssr引物对及合成方法和应用。
背景技术:
2.睡莲是睡莲科(nymphaeaceae)睡莲属(nymphaea l.)植物的统称,为多年生水生宿根花卉,世界名花,根据生态学特征,可分为耐寒睡莲和热带睡莲,睡莲花叶俱美,香气浓郁芬芳,花朵可泡茶,叶柄和花柄可食用,部分热带睡莲花梗粗壮挺拔,高出水面,是优良的鲜切花材料,具有很高的食用价值和观赏旅游、水景造景价值。
3.种质资源是植物遗传改良的物质基础,睡莲种质资源是发展睡莲鲜切花、食用生产的物质基础,也是睡莲研究领域尤其是睡莲新品种选育和生物技术研究的基础,经过长期的自然变异和人工选择,市场上存在大量的睡莲品种资源,但是不同品种间遗传背景不明,且同物异名和同名异物的现象较为普遍,造成种质资源冗余度上升,给资源圃保存和遗传改良带来繁缛的工作、低下的效率,另外在新品种不断更新的趋势下,部分传统代表品种因价格低廉而逐渐退出市场,造成资源遗失,因此,构建睡莲品种的核心种质,即采用一定的方法选取最小数量的种质资源以最大程度地代表种质资源的遗传多样性,对其保护和利用具有重要的意义。
4.目前主要基于形态标记和分子标记两类数据构建核心种质,对于睡莲等水生花卉来说,资源的收集和保存较为困难,表型极易受到季节和环境变化的影响,但这种影响是不可遗传的,其基因层面的遗传物质并未改变,因此形态标记不可取,使用分子标记能更准确地对多种花卉等进行鉴别、分类,然而日前构建核心种质的方法工作量大,在工作中操作人员会接触到有毒有害化学用品,如巯基乙醇、硝酸银等,危害其身体健康,且构建的引物特异性不高,用于种类鉴别等易受环境的影响。
技术实现要素:
5.本发明的目的在于提供一种睡莲的ssr引物对及合成方法和应用,提供的ssr引物对稳定性好、多态性高、特异性强,能用于对睡莲进行特异性扩增,基于该扩增产物能准确划分睡莲的种类,用于系统发育树构建和dna指纹图谱构建时更加准确。
6.本发明所采用的技术方案是,睡莲的ssr引物对,序列如seq id no:1
‑
2所示、如seq id no:3
‑
4所示、如seq id no:5
‑
6所示、如seq id no:7
‑
8所示、如seq id no:9
‑
10所示、如seq id no:11
‑
12所示、如seq id no:13
‑
14所示、如seq id no:15
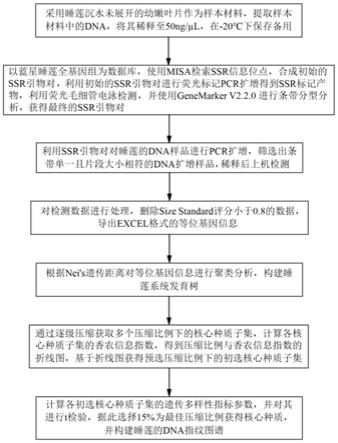
‑
16所示、如seq id no:17
‑
18所示、如seq id no:19
‑
20所示、如seq id no:21
‑
22所示、如seq id no:23
‑
24所示、如seq id no:25
‑
26所示、如seq id no:27
‑
28所示、如seq id no:29
‑
30所示、如seq id no:31
‑
32所示。
7.睡莲的ssr引物对的合成方法,按照以下过程进行:以蓝星睡莲全基因组为数据库,使用misa检索ssr信息位点,合成初始的ssr引物对;
8.misa检索的条件为:二核苷酸至少重复6次,三核苷酸、四核甘酸、五核苷酸和六核
甘酸均至少重复5次;
9.利用初始的ssr引物对进行荧光标记pcr扩增得到ssr标记产物,利用荧光毛细管电泳检测,并使用genemarker v2.2.0进行条带分型分析,获得最终的ssr引物对。
10.进一步的,所述pcr扩增系统如下:7.5μl 2
×
taq pcr master mix,2.0μl mix primer,2μl(50
‑
200ng)dna template(dna模板),3.5μl ddh2o;
11.pcr扩增体系如下:96℃3min、96℃30sec、(59℃~61℃)30sec、72℃1min,30个循环,72℃10min,12℃下保存。
12.睡莲的ssr引物对在睡莲系统发育树和dna指纹图谱构建上的应用。
13.睡莲的ssr引物对的应用,包括以下步骤:
14.s1,采用多种睡莲沉水未展开的幼嫩叶片作为样本材料,提取dna样品,将其稀释至50ng/μl,在
‑
20℃下保存备用;
15.s2,利用ssr引物对对睡莲的dna样品进行pcr扩增,筛选出条带单一且片段大小相符的dna扩增样品,稀释后进行上机检测;
16.s3,对检测数据进行处理,删除size standard评分小于0.8的数据,导出excel格式的等位基因信息;
17.s4,根据nei's遗传距离对等位基因信息进行聚类分析,构建睡莲的系统发育树。
18.进一步的,还包括以下步骤:
19.s5,通过逐级压缩获取多个压缩比例下的核心种质子集,计算各核心种质子集的香农信息指数,得到压缩比例与香农信息指数的折线图,基于折线图获得预选压缩比例下的初选核心种质子集;
20.s6,计算各初选核心种质子集的遗传多样性指标参数,并对其进行t检验,据此选择15%为最佳压缩比例获得核心种质,并构建睡莲属的dna指纹图谱。
21.本发明的有益效果是:1、本发明根据蓝星睡莲全基因组数据开发了16对睡莲属的ssr分子标记引物对,所述引物对的稳定性好、多态性高、特异性强,能够参与多种睡莲属植物的pcr扩增,使其扩增效果好,获得的扩增产物特异性较高,用于系统发育树构建和dna指纹图谱构建时能够对各睡莲种类进行准确划分;
22.2、本发明实施例基于开发的16对ssr引物对构建ssr
‑
pcr扩增反应体系,并确定每对ssr引物对的最佳退火温度,使引物对与dna模板的结合效率较高,进而获得更高的pcr扩增效率,同时体系中的非特异性扩增较少,特异性扩增效率提高;
23.3、本发明实施例以15%为最佳压缩比例,构成的核心种质品种量少,方便进行保存,且能覆盖睡莲属的全部遗传信息。
附图说明
24.为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
25.图1是本发明实施例的流程图。
26.图2是本发明实施例的效果图。
27.图3是睡莲种质资源的发育树图。
28.图4是不同压缩比例与香农信息指数的折线图。
29.图5是基于种质资源构建的dna指纹图谱。
具体实施方式
30.下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
31.睡莲的16对ssr引物对序列为:ntg006的序列如seq id no:1
‑
2所示,ntg011的序列如seq id no:3
‑
4所示,ntg012的序列如seq id no:5
‑
6所示,ntg014的序列如seq id no:7
‑
8所示,ntg036的序列如seq id no:9
‑
10所示,ntg042的序列如seq id no:11
‑
12所示,ntg051的序列如seq id no:13
‑
14所示,ntg071的序列如seq id no:15
‑
16所示,ntg106的序列如seq id no:17
‑
18所示,ntg107的序列如seq id no:19
‑
20所示,ntg111的序列如seq id no:21
‑
22所示,ntg115的序列如seq id no:23
‑
24所示,ntg146的序列如seq id no:25
‑
26所示,ntg152的序列如seq id no:27
‑
28所示,ntg156的序列如seq id no:29
‑
30所示,ntg188的序列如seq id no:31
‑
32所示。
32.睡莲的16对ssr引物对对应的退火温度及目的片段大小如下:
[0033][0034]
实施例
[0035]
如图1所示,使用以下步骤利用蓝星睡莲全基因组数据合成ssr引物对,并基于该引物对240份睡莲资源进行扩增,获得睡莲的系统发育树和dna指纹图谱:
[0036]
步骤s1,采用多地保存和种子保存进行睡莲资源的繁育和保存,选取不同睡莲资源中沉水还未展开的幼嫩叶片作为样本材料;
[0037]
使用植物基因组试剂盒提取样本材料中的dna,使用紫外分光光度计检测dna样本的浓度和纯度,用1%的琼脂糖凝胶电泳检测dna样本的完整性,选择紫外分光光度计检测结果
‑
光密度od值在1.8~2.1之间,跑胶结果显示为单一、清晰条带的dna样品,将dna样品稀释到50ng/μl,在
‑
20℃下保存备用;
[0038]
步骤s2,选择蓝星睡莲全基因组作为数据库,使用misa检索ssr信息位,合成睡莲属的初始ssr引物对;
[0039]
使用初始ssr引物对进行荧光标记pcr扩增,得到带有荧光基团的ssr标记产物,对目标ssr标记产物进行筛选,获得多态性、特异性和稳定性良好的ssr引物对;
[0040]
蓝星睡莲是目前睡莲属中唯一一个公布的全基因组数据库,用其作为同属基因组,开发睡莲属的ssr引物对,引物的开发效率较高、特异性较好;使用misa软件时选择默认参数,检索条件为二核苷酸至少重复6次,三核苷酸~六核苷酸至少重复5次;
[0041]
所述荧光标记pcr扩增采用15μl pcr反应体系,成分包括:7.5μl 2
×
taq pcr master mix,2.0μl mix primer,2μl(50
‑
200ng)dna template(dna模板),3.5μl ddh2o;具体扩增程序如下:96℃3min、96℃30sec、(59℃~61℃)30sec、72℃1min,30个循环,72℃10min,12℃下保存;该过程中设置ssr引物对的退火温度为59℃~61℃,使引物对与dna模板的结合效率较高,pcr扩增效率较好,同时扩增体系中的非特异性扩增较少,特异性扩增较多,基于该扩增产物进行ssr引物对筛选,得到的引物对稳定性好、多态性高、特异性强;
[0042]
根据引物合成时预估的目的片段大小区间,进行荧光毛细管电泳,读取在预估目的片段区间范围内出现的峰值大小及峰状态,使用genemarker v2.2.0对目的片段的峰值进行条带分型分析,去除在目的片段区间没有峰、出现连续波浪形杂峰或在目的片段区间外有峰的扩增产物对应的ssr引物对,保留在目的片段区间内出现单个或多个孤峰、峰形较好的扩增产物对应的ssr引物对,将其作为最终的ssr引物对;
[0043]
如图2中a~h所示为使用引物对ntg006对8份睡莲dna样本进行扩增,获得的扩增产物的毛细管电泳图,在预设目的片段120~160bp间出现了1~2个较好的峰值,经过反复试验后,发现不同样本间的峰值稳定,说明引物对ntg006在睡莲属植物样本中有较好的扩增性和多态性,因此将其作为最终的ssr引物对,重复上述过程对合成的ssr引物对进行筛选,获得最终的16对ssr引物对;本发明实施例使用上述过程进行引物对筛选,相比传统的pcr扩增和凝胶电泳不仅数据精确度得到了显著提升,效率也大大提高;
[0044]
步骤s3,使用获得的睡莲的ssr引物对对240中睡莲的dna样本进行pcr扩增,采用15μl pcr反应体系,具体扩增程序如下:96℃3min、96℃30sec、(59℃~61℃)30sec、72℃1min,30个循环,72℃10min,12℃下保存;
[0045]
步骤s4,取3μl pcr扩增产物进行琼脂糖凝胶电泳鉴定,检测pcr条件是否单一、片段大小是否与预期一致,筛选出条带单一且片段大小相符的dna扩增样品,对照dna marker的浓度进行定量,将筛选出的dna扩增样品稀释至相同的浓度范围,进行上机检测;
[0046]
步骤s5,使用genemarker软件对基因分析仪的检测结果进行处理,根据位点信息设置分析panel;
[0047]
使用genescan 500liz size standard进行数据分析,核查size standard评分,剔除评分小于0.8的数据,导出excel格式的等位基因信息;
[0048]
步骤s6,根据样品间及群体间的nei's遗传距离,使用r包poppr的aboot方法对各睡莲资源进行聚类分析,选择nei距离,bootstrap1000次构建样本系统发育树,如图3所示;
[0049]
图3中从起始点按顺时针分布的各睡莲样本分别是:hh1、gn27、ln6、ln17、gn1、ln14、ln8、ln5、ln31、gn23、gn17、gn20、ln30、ln16、ln29、ln28、gn24、ln25、gn14、ln21、gn32、gn10、ln18、ln11、gn7、gn28、ln4、ln2、gn21、ln24、gn4、ln7、gn5、ln27、ln15、gn12、gn31、gn30、ln12、ln9、ln10、gn3、gn9、gn29、gn15、ln33、ln19、gn25、gn26、ln13、ln23、ln20、gn19、gn13、ln1、ln3、gn6、gn33、gn18、ln32、gn22、gn11、gn16、ln26、ln22、gn8、la6、hh2、la10、la9、ha2、la1、la4、la26、la8、la7、la5、la11、la24、la14、la28、la25、la13、la22、la18、la17、la15、la21、la2、la19、la23、ha1、la3、la12、la16、la27、la20、gh1、gl3、gl4、gl6、gl2、ll6、ll3、gl5、ll1、ll2、ll4、ll7、ll5、lb26、hh3、gl1、l10、l9、l8、g2、g1、l7、l13、l12、l11、l6、l4、l3、l5、l2、l1、gb24、lb8、gb59、gb47、lb21、gb58、gb60、gb50、lb20、gb12、gb8、lb19、gb10、gb61、gb17、gb63、gb57、hb1、gb55、gb22、gb79、gb84、gb2、gb45、gb74、gb72、
gb70、gb68、gn2、hb2、gb62、lb13、gb44、gb9、gb1、gb33、lb24、gb80、gb83、gb67、gb66、gb76、gb65、lb23、lb15、lb12、gb6、lb1、lb3、lb16、gb77、gb75、gb71、gb81、gb69、gb82、gb64、lb25、lb6、gb36、gb32、gb37、gb43、gb51、gb31、gb23、gb53、gb39、gb21、gb46、gb38、gb78、lb5、gb34、gb49、gb13、lb11、gb11、lb4、gb48、gb26、gb27、gb56、gb15、lb7、gb52、lb18、gb35、lb14、gb16、gb18、gb54、gb42、gb25、lb22、gb40、gb5、gb3、gb14、lb2、gb73、gb7、gb20、lb9、lb17、gb30、gb19、lb10、gb28、gb41、gb4、gb29;
[0050]
由图3可知,本发明实施例将240个睡莲资源聚类成6枝,其中新热带亚属的小雪夜睡莲(hh1)单独聚类为1枝,65个广温带睡莲(编号gn27~gn8)聚类为1枝,30个澳大利亚睡莲亚属(编号la6~la20)和1个新热带睡莲亚属(hh2)聚类为1枝,12个古热带睡莲亚属(编号gl3~ll5)和1个新热带睡莲亚属(gh1)聚类为1枝,15个跨亚属睡莲(广温带睡莲和广热带睡莲杂交后代,编号l10~l1)和一个广热带睡莲亚属lb26、1个新热带睡莲亚属(hh3)及1个古热带睡莲亚属(gl1)聚类为1枝,111个广热带睡莲亚属(编号gb29~gb24)和1个广温带睡莲亚属(gn2)聚为1枝;来源于同一地理位置的睡莲资源,其亲缘关系比较接近,而使用本发明实施例构建系统发育树,能将大部分地理来源相同的睡莲资源聚在一起,说明本发明实施例合成的引物对具有良好的稳定性、多态性和特异性,能够对来源不同的睡莲资源进行特异性扩增,进而对其进行准确的划分;
[0051]
计算观测平均等位基因数(na)、有效等位基因数(ne)、nei’s基因多样性指数(h)和香农信息指数(i)等遗传多样性指标参数,结果如表2所示:
[0052]
表2 240种睡莲种质资源的遗传多样性指标参数
[0053][0054]
由表2可知:使用16对新合成的睡莲ssr引物对对240种睡莲的dna样本进行pcr扩增得到205个多态性位点,表明睡莲种质资源具有较为丰富的遗传多样性,蕴涵着广泛的遗传变异;
[0055]
步骤s7,按逐级压缩的方式获取多个压缩比例下的核心种质,计算各核心种质子集的香农信息指数(i),获取压缩比例与香农信息指数的折线图,如图4所示,由图4可知压缩比例为15%、25%、35%时各核心种质子集的香农信息指数存在转折点,将其作为预选压缩比例获得各比例下的初选核心种质;
[0056]
步骤s8,计算各初选核心种质的观测等位基因数(na)、有效等位基因数(ne)、nei’s基因多样性指数(h)和香农信息指数(i),具体计算结果如表3所示,用以评价原始种质、核心种质和保留种质的遗传多样性;
[0057]
其中原始种质为全部样本种质,即240种睡莲的dna样品检测结果,保留种质是原始种质减去初选核心种质后剩余的样本种质;
[0058]
表3 15%、25%、35%三种压缩比例下睡莲样本的遗传信息参数
[0059][0060]
分别对核心种质、原始种质及保留种质的各遗传信息参数进行t检验,检验结果如表4所示,核心种质与原始种质无明显差异(p>0.05),评价核心种质的代表性;
[0061]
表4 t检验结果
[0062][0063][0064]
由于核心种质包含的样本数越少,其保存越容易,在15%的压缩比例下,核心种质与原始种质无明显差异(p>0.05),且核心种质与保留种植也无明显差异,说明以15%为压缩比例获得的核心种质,包含的遗传信息能最大限度的表征睡莲的遗传多样性信息,此外在15%、25%、35%压缩比例下各核心种质之间亦并无明显的差异(p>0.05),所以选择15%作为核心种质的最佳压缩比例,在最大限度保留睡莲遗传多样性信息的基础上,减少睡莲核心种质的数量,使其便于保存,减少了保存的难度和成本;
[0065]
步骤s9,将压缩比例为15%时获得的核心种质各位点的等位基因编码成如图5所示的01矩阵形式的指纹图谱,以直观的展示每个核心种质的差异。
[0066]
本说明书中的各个实施例均采用相关的方式描述,各个实施例之间相同相似的部分互相参见即可,每个实施例重点说明的都是与其他实施例的不同之处。尤其,对于系统实
施例而言,由于其基本相似于方法实施例,所以描述的比较简单,相关之处参见方法实施例的部分说明即可。
[0067]
以上所述仅为本发明的较佳实施例而已,并非用于限定本发明的保护范围。凡在本发明的精神和原则之内所作的任何修改、等同替换、改进等,均包含在本发明的保护范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1