龋齿标志基因及应用的制作方法
1.本发明涉及生物技术领域,具体的,本发明涉及龋齿标志基因及其应用,更具体的,本发明涉及一种试剂盒、试剂在制备试剂盒中的用途、用于预防或者治疗龋齿的药物组合物或者食品组合物、确定个体是否患有龋齿方法、确定个体是否患有龋齿的装置、一种装置、一种筛选药物的方法。
背景技术:
2.龋齿又称蛀牙、虫牙,是一种细菌性疾病,会引起继发性牙髓炎和根尖周炎,严重时会引起牙槽骨和颌骨炎症,更可能发生病变形成龋洞,使牙冠遭到破坏,最终导致牙齿丧失,龋病是一种非常普遍的口腔疾病,严重影响人们的生活质量,其发病率高,分布广,可出现在任何年龄段,尤其以青少年居多。 2018年中国口腔健康流行病学调查结果显示,儿童龋病患病率较10年前明显上升了5.8%。
3.由于龋齿早期症状不明显,可能会误认为牙齿敏感,从而不引起重视,且它常发于不容易保持清洁的牙面上,如牙齿的点隙、裂沟与邻接面等,不易被发现。龋齿可通过拍摄x线牙片进行确定,在龋坏处可见黑色阴影。或者可用光纤维透照、电阻抗、超声波、弹性模具分离、染色等技术,以提高龋病早期诊断的准确性和灵敏性,但检查过程较复杂。
4.随着人体基因组测序完成及高通量测序技术的高速发展,基因筛查成为龋齿诊断的又一方向,可以通过唾液的样本确定是否患有龋齿。目前已有大量关于龋病的探究,如链球菌、放线菌属以及氟化物的应用,仍然需要在基因水平进行研究,从而揭示龋齿基因标志物。
技术实现要素:
5.本发明旨在至少在一定程度上解决上述技术问题之一或至少提供一种商业选择。
6.依据本发明的第一方面,提供一种试剂盒,包括适于检测第一基因集中至少一种基因的试剂,所述第一基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、12_gene_id_64866、 13_gene_id_16066、14_gene_id_30522、17_gene_id_45526和19_gene_id_2042,其中,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq idno:1-7中的核酸序列具有不小于90%的同一性。根据本发明具体实施例的试剂盒,可以准确的检测第一基因集中的至少一种基因的试剂,从而准确区分或诊断龋齿患者和健康个体。
7.在本发明的第二方面,本发明提出了试剂在制备试剂盒中的用途,所述试剂适于检测第一基因集中的至少一种基因。根据本发明的实施例,所述试剂盒用于诊断龋齿或检测龋齿的治疗效果,所述第一基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、12_gene_id_64866、13_gene_id_16066、 14_gene_id_30522、17_gene_id_45526和19_gene_id_2042,其中,所述第一基因集中的基因与seq id no: 1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-7中的核酸序列具有不小于90%
的同一性。根据本发明具体实施例的试剂制备的试剂盒,可以准确的检测所述第一基因集中的至少一种基因,极准确的区分龋齿患者和健康个体,由此,可以有效的在早期进行龋齿诊断,或用于检测治疗过程中龋齿的变化。
8.在本发明的第三方面,本发明提出了一种用于预防或者治疗龋齿的药物组合物或者食品组合物。根据本发明的实施例,含有提高所述第二基因集中的至少一种基因丰度的物质,所述第二基因集由以下基因组成:4_gene_id_31452和ncbiaceo_c_2_630,其中,所述第二基因集中的基因与seq id no:8、 9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、9中的核酸序列具有不小于90%的同一性。根据本发明实施例的龋齿标志基因中第二基因集的基因可以非侵入性的在早期发现或辅助检测龋齿,确定个体患有龋齿的概率高低或者个体处于健康状态的概率高低;同时,提高龋齿高风险人群或已龋齿患者肠道内的所述第二基因集中的各种基因的丰度,可以降低患龋齿的概率或减缓、治愈龋齿,因此,所述包含提高所述第二基因集中的至少一种基因丰度的药物或者食品组合物能够有效预防或治疗龋齿。
9.在本发明的第四方面,本发明提出了一种确定个体是否患有龋齿的方法。根据本发明的实施例,包括:(1)确定所述个体的粪便样本中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因;(2)将步骤(1)中得到的所述丰度与预定的阈值进行比较,以便确定所述个体是否患有龋齿;其中,所述第一基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、 12_gene_id_64866、13_gene_id_16066、14_gene_id_30522、17_gene_id_45526和19_gene_id_2042,其中,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-7中的核酸序列具有不小于90%的同一性,所述第二基因集由以下基因组成: 4_gene_id_31452和ncbiaceo_c_2_630,其中,所述第二基因集中的基因与seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、9中的核酸序列具有不小于 90%的同一性。根据本发明实施例的方法可以依据个体的粪便样本中的所述标志基因的丰度确定个体是否患有龋齿,所述标志基因是发明人对大量已知状态的粪便样本进行验证,通过差异比较分析各种肠道微生物基因在龋齿组和健康组粪便样本中的丰度,而确定下来的。
10.在本发明的第五方面,本发明提出了一种确定个体是否患有龋齿的装置。根据本发明的实施例,包括:丰度确定单元,用于确定所述个体的粪便样本中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因;比较单元,用于将所得到的所述丰度与预定的阈值进行比较,以便确定所述个体是否患有龋齿;其中,所述第一基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、 12_gene_id_64866、13_gene_id_16066、14_gene_id_30522、17_gene_id_45526和19_gene_id_2042,其中,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-7中的核酸序列具有不小于90%的同一性,所述第二基因集由以下基因组成: 4_gene_id_31452和ncbiaceo_c_2_630,其中,所述第二基因集中的基因与seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、9中的核酸序列具有不小于 90%的同一性。所述标志基因是发明人通过差异比较分析各种肠道内的微生物基因在龋齿患者和健康人群的粪便样本中的丰度并经过分析,和对大量已知
状态的粪便样本的验证而确定下来的,根据本发明实施例的装置可以准确确定个体是否为龋齿的高风险人群或龋齿患者。
11.在本发明的第六方面,本发明提出了一种装置。根据本发明的实施例,包括:计算机可读存储介质,其上存储有计算机程序,所述程序用于执行第四方面所述的方法;以及一个或者多个处理器,用于执行所述计算机可读存储介质中的程序。根据本发明实施例的装置可以准确确定个体是否为龋齿的高风险人群或龋齿患者。
12.在本发明的第七方面,本发明提出了一种筛选药物的方法。根据本发明的实施例,所述药物用于治疗或者预防龋齿,所述方法包括:将候选药物施用于受试者,检测施用前后,所述受试者粪便中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因,其中,满足下列条件至少之一的候选药物适于用于治疗或者预防龋齿:(1)进行所述施用后,所述第一基因集中的至少一种基因的所述丰度降低;和(2)进行所述施用后,所述第二基因集中的至少一种基因的所述丰度升高;其中,所述第一基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、12_gene_id_64866、 13_gene_id_16066、14_gene_id_30522、17_gene_id_45526和19_gene_id_2042,其中,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq idno:1-7中的核酸序列具有不小于90%的同一性;所述第二基因集由以下基因组成:4_gene_id_31452 和ncbiaceo_c_2_630,其中,所述第二基因集中的基因与seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、9中的核酸序列具有不小于90%的同一性。根据本发明实施例的方法可以生产或筛选出降低所述标志基因中第一基因集中各种基因丰度,和/或升高肠道内微生物标志基因中第二基因集中的各种基因丰度的药物,对于辅助减轻龋齿的临床症状具有重要意义。
附图说明
13.本发明的上述和/或附加的方面和优点从结合下面附图对实施方式的描述中将变得明显和容易理解,其中:
14.图1是本发明的实施例中的筛选鉴定龋齿标志基因的试验分析流程示意图;以及
15.图2是本发明的实施例中的标志基因作为诊断指标的auc评价结果示意图,其中,specificity表示特异度,即预测为阳性且实际为阳性,真阳性,纵坐标sensitivity表示敏感度,即真阴性:
16.2-a为第一期29个样品数据roc曲线下auc值和置信区间结果图;
17.2-b为第二期15个样品数据roc曲线下auc值和置信区间结果图。
具体实施方式
18.下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中,自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,仅用于解释本发明,而不能理解为对本发明的限制。需要说明的,本文中所使用的术语“第一”或者“第二”等仅为方便描述,不能理解为指示或暗示相对重要性,也不能理解为之间有先后顺序关系。
19.在本发明的描述中,除非另有说明,“多个”的含义是两个或两个以上。在本文中,
除非另有明确的规定和限定,术语“相连”、“连接”等术语应做广义理解,例如,可以是固定连接,也可以是可拆卸连接,或一体地连接;可以是机械连接,也可以是电连接;可以是直接相连,也可以通过中间媒介间接相连,可以是两个元件内部的连通。
20.生物学标志物是从生物学介质中可以检测到的细胞、生物化学或分子改变。生物学介质包括各种组织、粪便、体液、细胞、呼气、头发等。
21.所称的丰度指在某一核酸序列群体中该种序列的丰富程度。例如在一组核酸序列中某种核酸序的丰富程度,可表示为该种核酸序列的数目占该组序列的总数的比例。
22.同一性,本发明,为了比较两个或更多个核苷酸序列,可以通过将[第一序列中与相应位置的核苷酸相同的核苷酸的数目相除]来计算第一序列和第二序列之间的“序列同一性”的百分比。第二个序列中的核苷酸]减去[第一个序列中核苷酸的总数],然后乘以[100%],其中第二个核苷酸序列中每个核苷酸的缺失,插入,取代或添加-相对于第一核苷酸序列-被认为是单个核苷酸(位置)上的差异。
[0023]
或者,可以使用标准设置,使用用于序列比对的已知计算机算法,例如ncbi blast v2.0,计算两个或多个核苷酸序列之间的序列同一性程度。
[0024]
用于确定序列同一性程度的一些其他技术,计算机算法和设置例如在wo 04/037999,ep 0 967 284, ep 1 085 089,wo 00/55318,wo 00/78972,wo 98/49185和gb 2357768-a。
[0025]
需要说明的是,本技术中所述的“标志基因”的长度不受特别限制,可以为完整基因,也可以为基因的编码区或非编码区,更进一步地,根据不同的实验目的,可以选择相关的任意的核酸片段作为标志基因。
[0026]
根据本发明的一个实施方式提供的一种试剂盒,包括适于检测第一基因集中的至少一个基因的试剂,所述第一基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、12_gene_id_64866、 13_gene_id_16066、14_gene_id_30522、17_gene_id_45526和19_gene_id_2042,其中,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq idno:1-7中的核酸序列具有不小于90%的同一性。
[0027]
根据本发明的一个具体的实施方案,所述试剂盒进一步包括适于检测第二基因集中的至少一个基因的试剂,所述第二基因集由以下基因组成:4_gene_id_31452和ncbiaceo_c_2_630,其中,所述第二基因集中的基因与seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的 seq id no:8、9中的核酸序列具有不小于90%的同一性。
[0028]
根据本发明的具体实施例,所述试剂盒包括适于检测所述第一基因集中全部所述基因的试剂。
[0029]
根据本发明的具体实施例,所述试剂盒包括适于检测所述第二基因集中全部所述基因的试剂。
[0030]
根据本发明的具体实施例,所述标志基因是发明人通过对大量患龋齿个体和大量健康对照个体的粪便样本中的微生物的丰度的差异比较分析、以及验证,而确定下来的,明确了肠道微生物基因中与龋齿相关的标志基因。利用包含检测所述标志基因的试剂的试剂盒能够确定个体处于患有龋齿状态的概率高低或者处于健康状态的概率高低,能够用于非
侵入性的早期发现或辅助检测龋齿。
[0031]
根据本发明的具体实施例,所述适于检测所述第一基因集中至少一个基因和第二基因集中至少一个基因的试剂不受特别限制,任何可以检测所述标志基因的试剂均包含在本发明的范围内,如:利用pcr 技术、高通量测序等方法进行检测时使用的试剂。
[0032]
根据本发明提供的试剂在制备试剂盒中的用途,所述试剂适于检测第一基因集中的至少一种基因,所述试剂盒用于诊断龋齿或者检测龋齿的治疗效果,所述第一基因集由以下基因组成: 10_gene_id_46693、11_gene_id_51306、12_gene_id_64866、13_gene_id_16066、14_gene_id_30522、 17_gene_id_45526和19_gene_id_2042,其中,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-7中的核酸序列具有不小于90%的同一性。
[0033]
根据本发明的具体实施例,所述标志基因是发明人通过对大量患龋齿个体和大量健康对照个体的粪便样本中的微生物的丰度进行差异比较分析、以及对大量已知状态的粪便样本中的微生物基因的丰度进行验证,而确定下来的,明确了肠道微生物基因中龋齿相关的基因标志物。利用检测所述标志基因的试剂能够确定个体患有龋齿的概率高低或者处于健康状态的概率高低,能够用于非侵入性的早期发现或辅助检测龋齿。
[0034]
根据本发明一些具体的实施例,所述试剂进一步适于检测第二基因集中的至少一种基因,所述第二基因集由以下基因组成:4_gene_id_31452和ncbiaceo_c_2_630,其中,所述第二基因集中的基因与 seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、9 中的核酸序列具有不小于90%的同一性。
[0035]
根据本发明的具体实施例,所述适于检测所述第一基因集中的至少一个基因和第二基因集中的至少一个基因的试剂不受特别限制,可以检测所述微生物基因的试剂均包含在本发明的范围内,如:利用pcr 技术、高通量测序等方法进行检测时使用的试剂。
[0036]
根据本发明提供的一种用于预防或者治疗龋齿的药物组合物或者食品组合物,含有提高第二基因集中的至少一种基因丰度的物质,所述第二基因集由以下基因组成:n20_gi_0009810和n48_gi_0125557,其中,所述第二基因集中的基因与seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、9中的核酸序列具有不小于90%的同一性。
[0037]
上述标志基因是发明人通过差异比较分析各种肠道微生物基因在龋齿疾病组和健康组的粪便样本中的丰度,以及经过大量已知状态的粪便样本的验证,而确定下来的。所述标志基因中的第二基因集中的基因相较于龋齿患者群体,在健康群体组中显著富集,所述显著富集是指与在龋齿患者组中的丰度相比,上述基因在健康组中的丰度均具有统计意义地高于或者明显地、实质性地高于在龋齿患者组中的丰度;能够使该部分基因丰度提高的物质能够用于治疗龋齿或者益于龋齿患者服用,能够使其丰度提高的物质不限于治疗龋齿的药物和功能性食品。该实施例提供的能够使所述第二基因集中的基因丰度提高的物质能够用于制备治疗龋齿的药物和/或用于制备功能性食品、保健药等,所述药物或食品可有效治疗或缓解龋齿。
[0038]
根据本发明提供的一种确定个体是否患有龋齿的方法,包括步骤(1)和(2)。
[0039]
(1)确定所述个体的粪便样本中的标志基因的丰度。
[0040]
所述标志基因包括第一基因集和第二基因集中的至少一种基因。其中,所述第一
基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、12_gene_id_64866、13_gene_id_16066、14_gene_id_30522、17_gene_id_45526和19_gene_id_2042,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-7中的核酸序列具有不小于90%的同一性;所述第二基因集由以下基因组成:4_gene_id_31452和ncbiaceo_c_2_630,所述第二基因集中的基因与seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、 9中的核酸序列具有不小于90%的同一性。
[0041]
根据本发明的一些具体的实施例,步骤(1)进一步包括:获得所述个体的粪便样本中的核酸测序数据;将所述测序数据与参考基因组进行比对;基于所述比对的结果,确定所述标志基因的丰度。
[0042]
根据本发明的具体实施例,在步骤(1)中,按照下列公式确定所述标志基因的丰度: ab(g)=ab(ug)+ab(mg),其中,g表示基因的编号,ab(g)表示基因g的丰度,ab(ug)表示所述测序数据与所述基因g的参考序列唯一比对的读段的丰度,ab(mg)表示所述测序数据与所述基因g 的参考序列非唯一比对读段的丰度;ab(ug)=ug/lg,其中,ug表示所述测序数据中与所述基因g 的参考基因唯一比对的读段数目,lg表示所述基因g的参考基因长度;其中,mg为所述测序数据中与所述基因g的参考基因非唯一比对的读段的数目,i表示所述非唯一比对读段的编号,coi为所述第i读段对应的丰度系数;其中,co
i,g
表示针对所述标志基因g,所述非唯一比对的读段i的丰度系数,n为所述非唯一比对的读段i能够比对的基因的总数,j 表示所述非唯一比对的读段i能够比对的基因的编号。上述丰度确定公式,基于比对结果中的唯一和非唯一比对上组装序列的读段对该组装序列的丰度的贡献情况,充分利用测序数据的同时确定的丰度十分准确。。
[0043]
所述的测序数据通过对样本中的核酸序列进行测序得来,测序依据所选的测序平台的不同,可选择但不限于半导体测序技术平台比如pgm、ion proton、bgiseq-100平台,合成边测序的技术平台,比如illumina公司的hiseq、miseq序列平台以及单分子实时测序平台,比如pacbio序列平台。测序方式可以选择单端测序,也可以选择双末端测序,获得的下机数据是测读出来的片段,称为读段(reads)。
[0044]
比对可以利用已知比对软件进行,例如soap、bwa和teramap等,在比对过程中,一般对比对参数进行设置,设置一个或者一对读段(reads)最多允许有s个碱基错配(mismatch),例如设置s≤2,若reads中有超过s个碱基发生错配,则视为该reads无法比对到(比对上)该组装片段上。所述的获得的比对结果包含各条读段与各参考基因的比对情况,包括读段是否能够比对上某个或某些基因的参考序列、只唯一比对到一种基因还是比对到多种基因的参考序列、比对到基因组的位置、比对到基因组的唯一位置还是多个位置等信息。
[0045]
reads与参考基因组比对,比对上的可以被分为两部分:a)unique reads(u):唯一比对上一个基因的序列;称这些reads为unique reads。即,如果reads比对上的序列均来自
同一基因,定义这些reads 为unique reads;b)multiple reads(m):比对上一个以上基因的序列,定义为multiple reads。即,如果reads比对上的序列来自至少两种基因,定义这些reads为multiple reads。
[0046]
(2)丰度比较,以确定个体是否患有龋齿。
[0047]
根据本发明的一个实施例,将步骤(1)中得到的所述丰度与预定的阈值进行比较,以便确定所述个体是否患有龋齿。
[0048]
根据本发明的一些具体实施例,所述阈值为预先设定的。将基因标志物中的各种基因在健康个体以及患病个体中的丰度进行预先测定并保存,用以作为设定阈值的依据。所述阈值可以为一数值或者数值范围,基于已知患病或健康状态个体中的标志基因的丰度均值,该基因对应的阈值可以设为该基因丰度均值的95%的置信区间(confidence interval)。
[0049]
所述的置信区间是指由样本统计量所构造的总体参数的估计区间。在统计学中,一个概率样本的置信区间是对这个样本的某个总体参数的区间估计。置信区间展现的是这个参数的真实值有一定概率落在测量结果的周围的程度。置信区间给出的是被测量参数的测量值的可信程度,即前面所要求的“一定概率”,这个概率被称为置信水平。
[0050]
根据本发明的一些具体实施例,当步骤(1)中确定的标志基因的丰度达到所述患龋齿丰度阈值,未达到所述不患龋齿丰度阈值时,确定所述个体患有龋齿,当(1)中确定的标志基因的丰度达到不患龋齿丰度阈值,未达到患龋齿丰度阈值时,确定所述个体不患龋齿。
[0051]
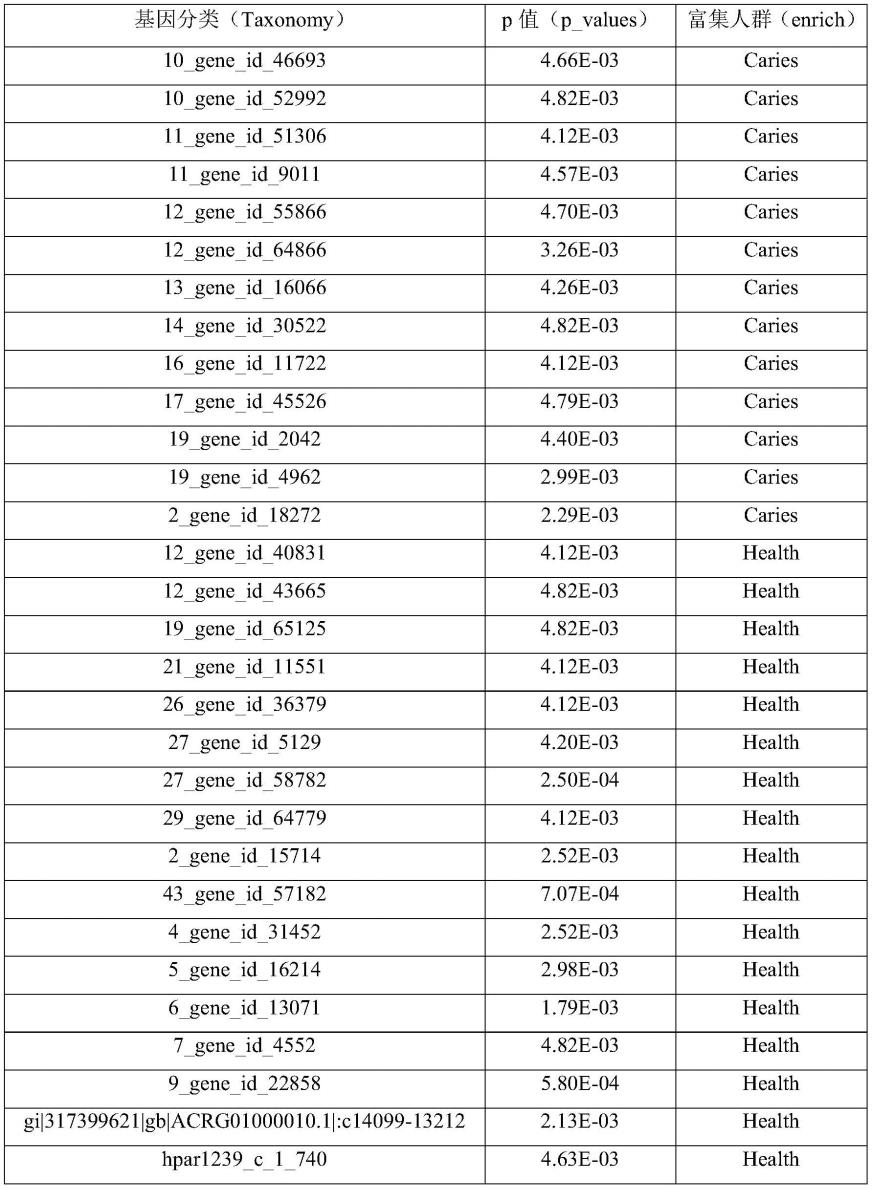
需要说明的是,根据目的或要求不同,可能对确定个体状态结果的可信程度有不同的要求,本领域技术人员可以选择不同的显著性水平或阈值。
[0052]
该方法基于检测个体的粪便样本中的标志基因中的各种基因的丰度,将分别将检测确定的各种基因的丰度与其阈值进行比较,依据获得的比较结果能够确定个体为龋齿个体或者为健康个体的概率。为早期发现龋齿提供一种非侵入性的辅助检测或者辅助干预治疗的方法。
[0053]
以上任一实施例中的利用标志基因确定个体是否患有龋齿的方法的全部或部分步骤,可以利用包含可拆分的相应单元功能模块的装置/系统来施行,或者将方法程序化、存储于机器可读介质,利用机器运行该可读介质来实现。
[0054]
根据本发明提供的一种确定个体是否患有龋齿的装置,该装置包括:丰度确定单元,用于确定所述个体的粪便样本中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因;比较单元,用于将所得到的所述丰度与预定的阈值进行比较,以便确定所述个体是否患有龋齿;其中,所述第一基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、12_gene_id_64866、 13_gene_id_16066、14_gene_id_30522、17_gene_id_45526和19_gene_id_2042,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-7 中的核酸序列具有不小于90%的同一性,所述第二基因集由以下基因组成:4_gene_id_31452和 ncbiaceo_c_2_630,所述第二基因集中的基因与seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、9中的核酸序列具有不小于90%的同一性。上述对本发明任一实施例的利用标志基因确定个体是否患有龋齿的方法的技术特征和优点的
描述,同样适用本发明这一方面的装置,在此不再赘述。
[0055]
根据本发明的实施例,所述丰度确定单元适于通过下列步骤确定所述丰度:获得所述个体的粪便样本中的核酸测序数据;将所述测序数据与参考基因组进行比对;基于所述比对的结果,确定所述标志基因的丰度。
[0056]
所述的测序数据通过对样本中的核酸序列进行测序得来,测序依据所选的测序平台的不同,可选择但不限于半导体测序技术平台比如pgm、ion proton、bgiseq-100平台,合成边测序的技术平台,比如illumina公司的hiseq、miseq序列平台以及单分子实时测序平台,比如pacbio序列平台。测序方式可以选择单端测序,也可以选择双末端测序,获得的下机数据是测读出来的片段,称为读段(reads)。
[0057]
比对可以利用已知比对软件进行,例如soap、bwa和teramap等,在比对过程中,一般对比对参数进行设置,设置一个或者一对读段(reads)最多允许有s个碱基错配(mismatch),例如设置s≤2,若reads中有超过s个碱基发生错配,则视为该reads无法比对到(比对上)该组装片段上。所述的获得的比对结果包含各条读段与各参考基因的比对情况,包括读段是否能够比对上某个或某些基因的参考序列、只唯一比对到一种基因还是比对到多种基因的参考序列、比对到基因组的位置、比对到基因组的唯一位置还是多个位置等信息。
[0058]
reads与参考基因组比对,比对上的可以被分为两部分:a)unique reads(u):唯一比对上一个基因的序列;称这些reads为unique reads。即,如果reads比对上的序列均来自同一基因,定义这些reads 为unique reads;b)multiple reads(m):比对上一个以上基因的序列,定义为multiple reads。即,如果 reads比对上的序列来自至少两种基因,定义这些reads为multiple reads。
[0059]
根据本发明的一个实施例,按照下列公式确定所述标志基因的丰度:ab(g)=ab(ug)+ab(mg),其中,g表示基因的编号,ab(g)表示基因g的丰度,ab(ug)表示所述测序数据与所述基因g的参考序列唯一比对的读段的丰度,ab(mg)表示所述测序数据与所述基因g的参考序列非唯一比对读段的丰度;ab(ug)=ug/lg,其中,ug表示所述测序数据中与所述基因g的参考基因唯一比对的读段数目, lg表示所述基因g的参考基因长度;其中,mg为所述测序数据中与所述基因g的参考基因非唯一比对的读段的数目,i表示所述非唯一比对读段的编号,coi为所述第i读段对应的丰度系数;其中,co
i,g
表示针对所述标志基因g,所述非唯一比对的读段i 的丰度系数,n为所述非唯一比对的读段i能够比对的基因的总数,j表示所述非唯一比对的读段i能够比对的基因的编号。上述丰度确定公式,基于比对结果中的唯一和非唯一比对上组装序列的读段对该组装序列的丰度的贡献情况,充分利用测序数据的同时确定的丰度十分准确。上述对本发明任一实施例的利用标志基因确定个体是否患有白塞病的方法的技术特征和优点的描述,同样适用本发明这一方面的装置,在此不再赘述。
[0060]
根据本发明的又一个实施例提供的一种装置,包括:计算机可读存储介质,其上存储有计算机程序,所述程序用于执行前面所述的一种确定个体是否患有龋齿方法;以及一
个或者多个处理器,用于执行所述计算机可读存储介质中的程序。
[0061]
根据本发明的又一个实施例提供的一种筛选药物的方法,所述药物用于治疗或者预防龋齿,所述方法包括:将候选药物施用于受试者,检测施用前后,所述受试者粪便中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因,其中,满足下列条件至少之一的候选药物适于用于治疗或者预防龋齿:(1)进行所述施用后,所述第一基因集中的至少一种基因的所述丰度降低;和(2) 进行所述施用后,所述第二基因集中的至少一种基因的所述丰度升高;其中,所述第一基因集由以下基因组成:10_gene_id_46693、11_gene_id_51306、12_gene_id_64866、13_gene_id_16066、14_gene_id_30522、 17_gene_id_45526和19_gene_id_2042,所述第一基因集中的基因与seq id no:1-7所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-7中的核酸序列具有不小于90%的同一性,所述第二基因集由以下基因组成:4_gene_id_31452和ncbiaceo_c_2_630,所述第二基因集中的基因与seq id no:8、9所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:8、 9中的核酸序列具有不小于90%的同一性。
[0062]
利用本发明这一方面的筛选治疗龋齿的药物的方法,通过合理有效地应用确定的龋齿标志基因进行筛选,能够获得提高肠道有益基因的丰度和/或降低肠道潜在致病基因的丰度的药物。
[0063]
以下结合具体实施例对本发明的方法和/或装置进行详细的描述。除另有交待,以下实施例中涉及的未特别交待的试剂、序列、软件及仪器,都是常规市售产品。
[0064]
以下实施例包括第一阶段和第二阶段,即对应发现阶段和验证阶段。
[0065]
实施例1生物标志物的鉴定
[0066]
该示例中,发明人通过对16个龋齿患者和13个健康对照的唾液样品进行研究,从而获得唾液中基因成分。发明人通过实验测序得到约37.3gb高质量健康人数据以及49.3gb高质量龋齿患者测序数据构建了龋齿参照基因集,并和homd基因集构建一个更加完整的非冗余基因集。宏基因组分析显示,35 个基因与龋齿密切相关,其中22个基因在健康人的口腔中富集,13个基因在龋齿病人的口腔中富集。
[0067]
1、测序数据的获取
[0068]
1.1样本收集和dna提取
[0069]
龋齿患者来自浙江大学医学院附属口腔医院,实验共采集了16个龋齿患者和13个健康人的唾液样品,唾液需要在口中保持3分钟。受试者流口水到无菌低温小瓶中,然后将每个唾液样品移入无菌的1.5 ml eppendorf管中,液氮速冻,80℃保存。
[0070]
将获得的29个人的唾液样本尽心总dna的提取。使用qiaamp dna mini kit试剂盒提取dna,具体操作步骤参考试剂盒中的说明书。
[0071]
1.2构建文库及测序
[0072]
dna建库按仪器制造商(illumina)的操作指南进行。对文库进行pe100 bp测序。illumina hiseq2000 (illumina,san diego,ca)平台对29个样品的文库进行测序。每个样本平均产生2.98gb(sd.
±
0.82gb) 高质量测序结果,总计86.6gb测序数据量。
[0073]
参照图1的实验流程,鉴定龋齿的相关基因标志物,其中省略的步骤或者细节为本领域技术人员所熟知,几个重要步骤介绍如下面步骤所述。
[0074]
2、生物标志物的鉴定
[0075]
2.1测序数据的基本处理
[0076]
1)测序数据经过质控:获得第一阶段的29个唾液样品的测序数据以后,对其进行过滤,质控按以下标准进行:a)去除低质量碱基(q20)大于50%的reads;b)移除大于5个n碱基的reads;c)移除尾部低质量(q20)和n碱基。丢失成对reads的序列被认为是单条reads用于组装,并进行基因预测。
[0077]
2)从http://www.homd.org/ftp/all_oral_genomes/20160329下载得到homd基因集。
[0078]
2.2基因丰度分析
[0079]
利用soapalign 2.21将经过处理的paired-end clean reads比对(匹配)到非冗余基因集,所称的非冗余基因集来自为利用样本数据及homd基因集构建的非冗余基因序列集。比对参数为
–
r 2
–
m 100
–
x 1000。reads与非冗余基因集的比对结果,可分为两部分:a)unique reads(u):reads只比对上一个基因的序列;这些reads被定义为unique reads。即,如果这些序列来自同一基因,发明人将这些reads 定义为unique reads。b)multiple reads(m):如果reads比对上两个及两个以上基因的序列,定义为multiplereads。即,如果比对上的序列来自不同基因,发明人定义这些reads为multiple reads。
[0080]
对于基因g,其丰度为ab(g),与特有的u reads和共享的m reads相关,丰度的计算方式如下:
[0081]
ab(g)=ab(ug)+ab(mg),
[0082]
其中,g表示基因的编号,ab(g)表示基因g的丰度,ab(ug)表示所述测序数据与所述基因g的参考序列唯一比对的读段的丰度,ab(mg)表示所述测序数据与所述基因g的参考序列非唯一比对读段的丰度;
[0083]
ab(ug)=ug/lg,
[0084]
其中,ug表示所述测序数据中与所述基因g的参考基因唯一比对的读段数目,lg表示所述基因g 的参考基因长度;
[0085][0086]
其中,mg为所述测序数据中与所述基因g的参考基因非唯一比对的读段的数目,i表示所述非唯一比对读段的编号,coi为所述第i读段对应的丰度系数;
[0087][0088]
其中,co
i,g
表示针对所述标志基因g,所述非唯一比对的读段i的丰度系数,n为所述非唯一比对的读段i能够比对的基因的总数,j表示所述非唯一比对的读段i能够比对的基因的编号。
[0089]
对于这些reads,发明人以加和的n个基因的unique reads的丰度作为标准。即对于multiple reads,发明人把其所比对上的n个基因的unique reads丰度之和作为分母。
[0090]
2.3关联分析/筛选基因标记物
[0091]
为了获得与龋齿密切相关的基因标记物,发明人利用龋齿患者组(16例)与健康人组(13例)两组唾液基因丰度数据,在基因层面进行龋齿相关性研究。
[0092]
基于上步骤得到的基因丰度表,发明人设置标准如下:(1)龋齿患者组或健康人组基因丰度的中位数必须大于0.00001;(2)通过wilcoxon秩和检验进行检验,得到每个物种和龋齿的相关性p值; (3)使用一个相对严格的阈值(p_values《0.005)。发明人得到35个与龋齿密切相关的基因标志物。其中在龋齿(caries)患者中富集的有13个基因,在健康人(health)中富集的有22个基因。这35个基因标记物如下表1所示。
[0093]
表1
[0094]
[0095][0096]
实施例2基因标记物的验证
[0097]
为了证实实施例1中的分析结果,进一步比较验证群体中的9个龋齿患者及6个健康人的唾液中基因丰度,并根据验证情况对最终基因进行筛选。验证群体测序数据的获得与处理,参照实施例1进行。
[0098]
验证结果如下:上述富集在健康人群中的22个基因中,2个在验证集中得到高质量的验证 (p_values《0.05),验证群体中在健康人富集的基因标志物的p值如下表2所示。
[0099]
表2
[0100]
基因分类(taxonomy)p值(p values)富集人群(enrich)4_gene_id_314522.80e-03healthncbiaceo_c_2_6304.96e-02health
[0101]
对于上述富集在龋齿患者中的13个基因,其中7个在验证集中得到高质量的验证(p_values《0.05),验证群体中龋齿患者富集的基因的p值情况如下表3所示,10_gene_id_46693、11_gene_id_51306、 12_gene_id_64866、13_gene_id_16066、14_gene_id_30522、17_gene_id_45526、19_gene_id_2042。
[0102]
表3
[0103]
基因分类(taxonomy)p值(p_values)富集人群(enrich)10_gene_id_466933.85e-02caries11_gene_id_513063.90e-02caries12_gene_id_648663.90e-02caries13_gene_id_160663.85e-02caries14_gene_id_305223.90e-02caries17_gene_id_455263.85e-02caries19_gene_id_20422.11e-02caries
[0104]
发明人认为,可以将从健康人富集的2个基因标记物,作为龋齿患病的反向指标,或作为研发治疗龋齿的基因制剂药物成分,或作为检测龋齿、监测龋齿进程的指标;将龋齿患者富集的7个基因标记物,作为龋齿患病的正向指标,用于龋齿简便、非创伤式的检测和诊断。
[0105]
发明人利用这9个微生物的基因标记物,构建一个综合指标,估计roc(receiver-operatingcharacteristic)曲线下面积auc,auc值越大,表示诊断能力越高,通过auc值反映龋齿的诊断能力。分别对一期(第一阶段)的29个样品和二期(第二阶段)的15个样品进行评测,具体情况如图2所示,两者均表现出了很好的诊断能力,在一期得到auc=95%,如图2-a所示,置信区间为81.1%-100%;二期得到auc=94.4%,如图2-b所示,置信区间为82.5%-100%。
[0106]
上述基因集中所述基因分别包括如下所示的核苷酸序列:
[0107]
10_gene_id_46693:
[0108]
gtgaccgcgttggtattttcaccggctcatatcaaacgatgcgccctgacctgggaagcg cccgactatagtgcttcctacccggccggatacaccgatcagggagttgccgtcattgcg gtagctcaggaacaccgccaggcgcagggtttttgctactcggcgctgtcgacggtggtt tttatttcgaagcaggacggcacccgcgccgtcattaatgaggagcccctgctctttcag cacactcattcgctcatggagtcctgcgatgaggtgcatccccgtctgctgggccgagcc gtgttggccgcctgggattacatcccggagcagtaccgttgcaacctggtgacgattgcg gccgatcgtaagcatctgaagcacctgttccgggattgcgaccacctgaagacgattatg gagtaccgcgctttcgagaaggtgctggacccctacgataatcactccctgggtgaggtg cacgtgaagctggccggcacccccgcctacgagcggatgcaggccgaggtcacccgtacg cgcgagaccgtggatgccatggtgcacgacgctcttgccccccgcccgcttgaacagctg gaggattacagtgtctttacggactgctcgtttcgttccacgcacaacaagaagtaccag cgcggtggccgcatgggcatcgccggagtgagcgaggacggcttttactttcactctcac tatgacggcgtgaatatcatgaccggagagctgtcggccgtgctggccgcatataccgtg tttcacagcggttcgcgccgcctgattatcaataccgattccttgggcgccatcaccttc gtgttgcgcctggcgcacagccgctggaccttcgagacctgggcctccgagggtgtgcag gatgaacgcgtggtcgcccagatgcgctccctcaccgaggcaatccgcgagaagcgcgtg ctggttaagcacgttccgggccataccggtcacggcctgcaggagagcagcgactccgtg tcgaagatgcaccggcactttcccggacaaattgttgataaacggcatcaaagcgaattt aaccagcgctgtgagtcgattattaccgctctggcggggtgcgcgaaggcggttcgcctg atggctcccgactgggtgcagattaaaccctcttcaattcacgcgggaaaccgtttgtgg cgttag(seq id no:1)。
[0109]
11_gene_id_51306:
[0110]
gtggcacctgaacagccccatgaatacggtgcgtccgacatcaccgtgctggaaggcttg gaggcggttcgtaaacgccccggcatgtacatcggctcgaccaccgagcgcggtctgcac cacctggtctacgaggtcgttgataactccgtggacgaagcgctcgccggttacgcctcc gaaatcgacgtgaccatccaggccgatggcgcggtgcgcgtcaaggatgacggccgcggt attcccgtggatgagcaccccaccgagcacaaatccaccgtcgaggtcgtcatgaccgtg ctgcacgccggcggtaagtttggcggcggcggctactcggtctccggtggtctgcacggc gtgggtatctccgtggtgaacgccctatcgacccgcgtgattaccgaggtgcaccgccag ggctacgcgtggcacatctccttctccaacggtggcgtgcctgacggtcccctgcgtcgc ggcgagccctccgataagaccggcacgattcagaccttctatccggatcccgagattttc gagactgtcgagttcgattttgagaccctgcgcgctcgcttccaacagatggccttcctg aataagggcctgaccatcaccctgaccgatgagcgcgagaagttcaccggtgacgaggtt gccgaagaggagaaggaaaacactctcaaccgcgtgcgtgcgaacgctgacggtttcatg accgtcacttaccgttacgacaacggcctgtttgactacgtgcacttcctgaatgccggc gcgaagaccatcgtgaacgaagagattgtctccttcgaatccgaggactctgaccgtaac atgagcctggaagtggccatgcagtggaccggtgcgtacaaggaatctgtctactcctac gcaaacacgatcaacacccatgagggcggcacccatgaagagggtttccgtgccgcgctg acctccctggtgaaccgttacgctcgcgacaacaagctgttgcgcgataaggacgataac ctcaccggtgaagacgtgcgtgagggcctgaccgcggtcatctcgattaagatttccgag ccgcagttcgagggtcagacgaagaccaagctgggtaactcggaggcgaagtccttcacc cagcgcgaggtgaacgatcacctgagcgactggttcgaccgtaaccccgccgtggccaag gacatcgtgcgtcgcgcgattaccgccgcccaggcgcgtatcgccgcccgcaaggcccgt gagacgacccgccgcaagggcctgctggagaccagctccatgcccggtaagctcaaggac tgctcctcgaaggacccgtcgatctctgagatttacctggtggagggtgactccgcgggt ggttcggcagttcagggtcgtaacccgaacact
caggctattctgccgctgcgcggtaag atcctgaacgttgagcgcgctcgcctggaccgcgccctgtcgaacgccgaaattcaggcg atgattaccgccttcggtaccggcatcggcgacgacttcaacatcgaaaaggcccgttac cacaagatcgtgctcatggccgatgcggacgtggatggccagcacattacgaccctgttg ctgaccctgctcttccgcttcatgcgcccgctgattgaggccggttacgtgtacctggct cagccgccgctgtaccgcatcaagtggtcgaacgccccgcatgattacgtctactccgat gcagagcgcgacgaggctctggcacgtggtttggcgaataaccagcgtctgcccaaggac aacggtattcagcgttacaagggtctgggcgagatggactacaccgagctgtgggacacc accatggatccggcacgccgtacccttctgcaggtgaagatggaggacgcggctgccgcg gaccagatcttctcggtgctgatgggtgaggacgttgaggctcgccgtgagtttatccag caaaacgcaaaggacatccgattcctcgatatctaa(seq id no:2)。
[0111]
12_gene_id_64866:
[0112]
atgccgaagcaaagccgcctggcgggtttcctcggcctggagccctacaacgccaaggac tctgacgcccaggactccaacaccgcggataccgcgcccgaggagccccgctcccacgag cagtcgggtcgcggcggggagcaccaccgcgcggaggcccaccgtgtagaggcccgcgtc gaagagcctcgagccgacgaccgcgccgcgcgagaccgctcgctgggggagcacaccgca tccaagcacggtgcgcccgcagtcgcacacccgtacacacccgaaaccacagacgaggcg gaggaaccccgcgaggatcgccgaggcgatgagccccggcacgagcccgcgagcgaaccc gcagaccattgggaggacgccccggtgccgaacaccgaatacaccgagcagtacgccgac acctactcgaacgacggctactacgaccagggctacgagcccgaagagaccaacggatac acctacggttacgaggaagaagaggacgaattgcgtcgtatcaccactatccacccccgc tcctacaatgacgcaaagattatcggtgaggcgttccgcgagaacatccccgtgattatg aacgtcgaggaaatgccggacgccgacgccaagcgcctggtggacttcgcatccggtctg gccttcgcactggagggtcgcatcgagcgcgtgacctcgaaggtcttcctgctgaccccc tccaacctcgaggtgctgggcgtcgccaactccgagaacaccgcagacttcaactccgag ggtgaattccccttcgaccagggctaa(seq id no:3)。
[0113]
13_gene_id_16066:
[0114]
atggactggaagcccgtagtggatatcgtggtgaccgcggttttcctgctgggtatcggc ctggccctgtggggtctgctgacggtgcaccgtagcccgtacttttccgaagtgcagaag cttaagtggcttctgctggtgctgttcctgccgtatcttggccccctggtttggattttg cgggttcgtaccgagcgcgcggaggccgcacagcgggagctggaggcggcgaagcgggac ggggctccgctcgaggaatccggcgaggttgcggctcgggccgagtag(seq id no:4)。
[0115]
14_gene_id_30522:
[0116]
atgacttacaaactagtactcctgcgccacggccagtccgagtggaacgagaagaacctg ttcaccggctgggtggatgtcaacctgaccgacaagggccgcgccgaggctaagcgcggc ggcgagctgctggcagagcgcaacatcctccccgatgttgtgcacacctccctgcagcgt cgcgctatcaacaccgcaaacatcgccctggatgcagcggaccgcctgtggattcccgtc aagcgcacctggcgcctgaacgagcgtcactacggcgccctgcagggtaagaacaagtcc gagattcgtgaggaatacggcgacgagaagttcatgacctggcgtcgttcctacgacgtt ccgccgcccccgctggacgacaacgatccctactcgcaggcacacgaccctcgttacgcc aacgttgagaacgctccccgcaccgagtgcctcaaggacgttctgggccgtatgctcccc tactgggagtcggacatcaagcccgacctggcagcaggcaagaccgttctggtggcagcg cacggtaactccctgcgctcgctggtgaagcacctcgagggcatttccgacgaggacatc gccggcctgaacatcccgaccggtatcccgctgtactacgagctggatgagaacttcaag cccattaaggctggcgagtacctcgatcccgaggccgctaaggacgccatcgcagccgtg gcaaaccagggcaagtaa
(seq id no:5)。
[0117]
17_gene_id_45526:
[0118]
atgtcttcgtcccgcacctccgcccgcaagggctcctcgcagcgcgccccgttcaccccc accgtgcgcttcgccatcgtctacgtgctggcgctcgtgatttcattggcggtttggttc ttctaccgcgccgtctacgaggacgacacctcgttcctggtgcccatcgtgatttccctg gcgacgtcctacgttgccgcgcacgcccttacgggcggccgttccccgcgcaactag(seq id no:6)。
[0119]
19_gene_id_2042:
[0120]
atgaagaagactgactacaacctcgccgtctactgcggcgcacgctccggcaacgacccg ctcttcaccgagcgcgcccgcgagctcggcaccctctgcgcccggaacaatattggcgtg gtctacggcggcggccgcaccggcctgatgggcaccgtcgcgggcgcgaccctggatgcc ggcggcgtggtctacggcgtggtcacccacaagctggtcggtctggagggcgtctacccc ggccagaccaccctcgacattgtggacaccatgcaccagcgcaaaatcgccatgatcgag aaggccgacgctttcgtcgccatgcccggcggccccggcaccctggaagagttcttcgag gtgttcagctggcagttcctcgacatccactccaagccggttgcgttgctgaacgtgagc ggctactgggataagctcatcgacgcgatgcactacatggcctccaccggcctgctgaac gagcgctacctggacgaactcattatcgcgaccactcccgaggagctgctggatgcgctc gccaaggacctagagcgccgcgccgccgagaagaacactgctgcaaagaacgctgctgaa taa(seq id no:7)。
[0121]
4_gene_id_31452:
[0122]
atgaccatgactaccctcccccgttacgccgttttcggcaatcccgttgcacacagtaaa tcgccgcaaatccatcagcagtttgcgctgcaggaaggcgcggccatcgaatatgggcgc atttgtgccgaaatcggcggttttgccgaggctgttgcggcgtttcgtgccgagggcggg caaggtacgaatgtaaccgtcccgttcaagcaggaagcttttgcgctggcggatgagcat tccgaacgcgctttggctgccggcgcggtcaatacgctagtgtggttggaagacggcagg atacgcggcgacaacaccgacggtatcggtttggccaacgacatcacgcaggtcaaaaat attgccatcgaaggcaaaaccattttgcttttgggtgcaggcggcgcggtgcgcggcgtg attccggtattgaaagaacaccgcccagcccgtatcgtcattgccaaccgtacccacgcc aaagccgaggaattggcgcagcttttcggcattgaagccgtcccgatggcggatttgaac ggcggttttgatatcatcatcaacggtacgtccggcggcttgagcggtcagcttcctgcc gtcaatcctaaaattttccgcggctgccgccttgcctacgatatggtttacggcgaagcg gcgcaggcgtttttgaactttgcccaaagcagcggtgcggccgaagtttcagacggcctg ggcatgctggtcggtcaggctgccgcctcctaccatatttggcgcggatttacgcccgat atccgccctgttatcgaatacatgaaagccctgtaa(seq id no:8)。
[0123]
ncbiaceo_c_2_630:
[0124]
ttgaattatgccctcgcgcatcaaagcgatttgggcgcacaccaaatttggctcacgccc gaaggccatcaggtcgataaagtcagcgtcctgctctatgccaaacccgcgcaggaaagc ctgattgcccaaaaagcgcgcctcgacggcatcgcgtccgaactggaaaacctcgccccc gaactctccgccgccgaagccgcgttcaaacaggcggaagccgcagtgcgctcgtccgaa gtgcagcacaaaaacctgattcagcagcaacagcaacacacgcgccaatacagccaagca cagcaacgcgccgccgaacttctggcgcgcaccaaccaagggcaaatccgccgcgaacat atcgagcgcgaactggcgcagttggcggaagaacaaaccgtgttgcaacatacttcagac ggcctcgcagacgacatcgctactttgcaggaagccgccgccgaactcgaacaccaacag cataccaccgcgcacagccgccaagagcagcaaggccgtctgaaacaggcgcagcttgcc ttgttggaagccaaccgccaatacgggctggccgaagtcgccgtccacaagctcaaccag caaaaacaaaactaccagcagcaaatcgcccggctcgaacagcaaaccctagactggcag gaacg
ccagcaagagcttgcccttgcctatgaaaccgagttccaaaacgacgagcagcac atcaagctcgacgagttgaccgaagccgtacacacgctggacgaagaatacatcgccgcg caggaaagactcgcgcagattcaggagcagggcagggagcaatacgcccgcgtacaaacc ctgcaaaccaagctgccgcagcttcaggctgccacccaaaccgccctgttgcagcagcag gaagccctgatcaatgctaaacgctaccatcaaaacctgaccgaacgcgccgccgatttg gacgcgctcgaagcgttggcaaaagaatcgccgaaagtattgaacagcagcatcggcagc ctcacccagcaaatcgaagccatcggcagcctcacccagcaaatcgaagccctcggcgcc gtcaacctcgccgccctgcaagaactcgaagaagcgcgcgaacgcgacggctactaccgc agccaaagcgaagacgtgcaggccgccatcacccttttggaagaagccatcgcccaaatc gacgacaaaaccaaagcgcgcttcaaagaaaccttcgatgccgtcaacggcaaagtccaa accttcttccccaccctgttcggcggcggcgaagccaccctcaaaatgataggcgacgac ctcctgaccgcaggcgtatccatcatggcgcgtccgcccggcaagaaaaacagcaccatc cacctcctctccggcggcgaaaaagccctcaccgccatgagcctcgtgttcgccctgttc agcctcaaccccgcccccttctgccttctggacgaagtcgacgccccgctggacgatgcc aacacctcgcgtttctgcaacctggtcaaagaaatgtcggcgcaaacccagttcctctac atctcccacaaccgcttgaccatggaaatggcagagcagctcgtcggcgtaaccatgcag gaaaaaggcgtatcacgcgtcgtcgccgtggacattaaacaggcgttagaaatggctgag ccgaattga(seq id no:9)。
[0125]
rmuc_c_2_613:
[0126]
atgtccgagcagaagatcctcgagatgcccctcgccgacctcgaccccgaggttgcagaa gccattgagctggagcgcaagcgccagcagaacaccctcgagatgattgcatccgagaac ttcgtcccccgtgccgtcctcgaaagccagggctcggtactgaccaacaagtacgctgag ggttaccccggccgccgctactacggcggctgcgaattcgttgacatcgttgagtccctc gctatctcccgtgtgaaggagctcttcggtgcggagcacgcgaacgttcagccgcactcc ggtgcgcaggcgaacaacgccgtcatgcacgcactgctgaatcccggcgacaccattatg ggtctgtccctggcacacggtggccacctgacccacggcatgaagctgaacgtctccggc aagctgtacaacgttgtggcgtacggcgtggacgagtccggccgcgttgacatggatcag gtgcgtgaactggctctggctgagcgccccaagctgattattgccggctggtccgcgtac ccccgtcagctggacttcgctgcgttccgcgcgattgctgacgaaatcggtgcatacctg tgggtggacatggctcacttcgcgggcctggtggctgcgggtctgcacccgaaccccgtc ccgcacgcacacgtggtctcctccaccgtgcacaagaccctgggcggcccccgttccggc ttcatcctctgcaccgaggagctgaagaagaagattgactccgcagtgttccccggccag cagggcggcccgctcatgcacgtgatcgctgcgaaggctaccgcgttcaaggtcgccgca tctgacgcgttcaaggaccgtcagaagcgcactgttgagggcgcacagatcctcgcgaac cgcctgctgcagcaggacgtgaaggacgccggcgtgtcgatcgcctccggtggtaccgac gttcacctggtgctcgttgacctgcgcgaccacgccctcaacggcaaggaagcagaggat ctgctgcacgccgctggcattaccgtcaaccgtaacgccgtgccgaacgacccgcgcccg ccgatggtgacctccggtctgcgtatcggtacccccgcgctagcgacccgcggcttcgac gctgagggcttcaccgaggtagccgacattatcgcttccgtactgctgccgaacccggac atcgctgcgctgcgtgcccgcgttgaggcactgtgcgccaagtaccccctctaccagggc agcgagaactggtag(seq id no:10)。
[0127]
2_gene_id_15714:
[0128]
atgactcccccttctctttccggagcacccactccccaggcgaccgagagcctcaccgta gtggcaccgaatatcccggcggcttctgcgcacctcgctgagcgtattgctgaagagcag cgagcctacctgcacgcttttgacactctcatggctgagttttttgcccgccagcgcgag ctgctcgagggcatttctgccgaaactctacccctgctggaggctattgagtccctctcc accggcggcaagcgcctgcgcgccctgctgagctactggggttggcgaggtgccggcggc gcacccgtcaccgaggaccgcgcgtcctggtctattgtgaaggctggcatggcggtggaa ctgttc
cagacctccgcgctgattcacgacgacattatcgaccgctccgatacccgccgc ggcgctccctccgtgcacaagcgtttcgaggctgcgcacgagcagaatagttggcgcggt gatgctttcaactacggtctgaccggcggtattcttgccggcgatctgaccctggcgtgg tctgccgaggttttcgcgtccctgggtgagggagcgatttatggcgccccggcgcgcact atttttgatcggatgcgcaccgaggttctggcgggtcagtacctggatgtctactccgag gttctcgagaccgaggacgctcaggcggctctgcagcgtgccctgaacgtgattcgtttc aagtccgccaagtactcgtgcgaacaccccttcacgattggtggcgctctggctctgcag gctaccgcgctggagaacggcgccggtgccgtgagcgagcagcacccggtacttgtcggt taccgcgcgttcggtctgccgctgggtgagggcttccagctgcgtgatgacgagctgggc gtcttcggtcagcccgaggtgaccggtaagcccgccggcgatgacctgcgtgagggtaag cgtaccgtcttggtggcgctcacctccgccgccctcgacgaaaaggatgcggcgcttcta cacaatagtttgggcgatcccgatctgagcaatgagcaggtggagcgcatccgcgagctg atggtttcttcgggggctttcgccaagcatgagcagctaatcgagcagaagtcacaggct gtgtttgaggcgctggaggcgatgaacctggacgagctggtgcatgcggcgctgaccgat attgtgggccgtgcactgcgtcgtaaggcatag(seq id no:11)。
[0129]
ngon1109_c_1_2082:
[0130]
atgaaagtaggtttcgtcggctggcgcggtatggtcggttcggttttgatgcagcgtatg aaagaagaaaacgacttcgcccacattcccgaagcgtttttctttacctcttccaacgtc ggcggcgcggcacctgatttcggtcaggcggctaaaacattattggacgcgaacgacgtt gccgagctggcgaaaatggacattatcgtcacctgccaaggcggcgattacaccaaatcc gtcttccaagccctgcgcgacagcggctggaacggctactggattgacgcggcgtcctca ctgcgcatgaaagacgacgcgattatcgccctcgaccccgtcaaccgcaacgtcatcgac aacggtctcaaaaacggcgtgaaaaactacatcggcggcaactgtaccgtctccctgatg ctgatggctttgggcggcctgttccaaaacgatttggtcgaatgggcgaccagcatgacc taccaagccgcttccggcgcgggcgcgaaaaatatgcgcgaactcatcagcggcatgggc gcgattcacgcccaagtggcggacgagcttgccgatccttccagcgcgattctcgatatc gaccgcaaagtgtccgatttcctccgcagcgaagactatccgaaagccaacttcggcgta ccgctcgccggcagcctgattccgtggattgacgtggatttgggcaacggccagtccaaa gaagaatggaagggcggcgtggaaaccaacaaaatcctcggacgcagcggcaatccgacc gtcatcgacggcctgtgcgtccgcatcggcgcgatgcgctgccacagccaagccatcact ctgaagttgaaaaaagacctgcctgtttccgaaatcgaagcgattttggcaggcgcgaac gactgggtgaaagtcgtccccaacgaaaaagaagccagcatccgcgagctgacgcccgcc aaagttaccggcacgctgtccgtccctgtcggacgcatccgcaaactgggcatgggcggc gaatacatcagcgcgttcaccgtcggcgaccaacttttgtggggtgcagccgaaccgatg cgccgcgtgttgcgtatcgtgttgggcagcctgtaa(seq id no:12)。
[0131]
10_gene_id_52992:
[0132]
atgacacttttacaaacgattattatctccattgtcgaaggactgacggagttccttcct gtgtcatctacgggtcacatgattattacacaaaatcttttaggtgtaccatcaggtgac gagtttgtacatgcctttaccttcatcatccagtttggtgctatcctctctgtcgtatgc ttatattggaagcgctttttccagatcgaccatacacctgtacctgctgataaaacttgt actttcaagaagaatattcatccaatccgcttctactggttattgttcattagtgtccta acttatactttcaagaagattattcatccaatccgcttctactggttattgttcattggt gtcctaccagctgttatcattggcttagcagctaagaaaagtggtttacttgactggctg ctcgactcagtttgggttgtagcaataatgctcgttgtgggtggtatcttcatgctttat tgtgataaattgttcaataagggaaaagaagagaatcaagtaacagaaaagcgtgccttt agcattggtcttttccagtgcctatcggttatccctggcacaagccgttctatggctact atcgttggcggtatggcaaacggattgacacgtaagcgtgcagcagagttctcattcttc cttgc
agtacctactatggctggtgcaacactgctcgacctacttgatttattaaaaggt gacagcacatgggcgtctgctcataaccttataatgctcgctgtaggctgtgttatatca tttatcgttgcgctattggcaatgaagtggtttgtaagtttccttgcaaaatatggtttc aagtggttcggttggtatcgtatcatcgttggcgttatcattattataatgctgctctgt gatattccactgaatatggttgactaa(seq id no:13)。
[0133]
2_gene_id_18272:
[0134]
atggttatgacgacagttcacccggctctttcttttgagctgttccctccgcgctcacac gcaagctgtgagtcgctgattcgcaccattgcggagctcgagggtaccaaccccgactac gtttcggtcacctattccggtgacgcgaagcgccgtcagaagacgctggcgctgctcgac tacctggtgcacgaaacctccctcacccccctggcacacctgatttgtgcgggccactcg gtgcacgagctggagcgtgcggtccgcctgattctgggtctgggcgtgcgcggtttcctg gcgctgcgcggtgacgtgcaggaaggtcaggtttctgagcttcccttcgcccgctacctg gtcgagctgattcgccgcgtggaacgcgaagatttcgcccacctggcggcgggtaaggtg tccatcggtgtggcggcgtacccgcagaagcaccccgagtccgcatccctgctgcaggat attgagattctgctgtcgaagcagcgtgccggcgcggatttcgctatcacccaggtgttc ttcgaggatgagcgctacagtgccctggtcgattcggctcgcctggcgggtgtggacatc ccgattattccgggcatcatgccggtgacctccctgaagcgcctgcagaacctctgccag atggcgggcatgtcggttccggcggatcttgcgtacgccctggagacggcaaccagcagc gcagagctgcaccgcatcggcgtggactacgcactgaaccagtgccgtaccgtgatggat gcgggcgcaccgggcctgcacttcttcaccttcaacgagcatgccgcggtgctggacgtt ctggatcagctggacttgccccgatacagcaaccgtttctcccaacccctgagcgagctg gaattcgcctaa(seq id no:14)。
[0135]
gi|317399621|gb|acrg01000010.1|:c14099-13212:
[0136]
atgcttgatagtatttactctgccatcgacgccctctccgttcttgcgccattcgttatt cctttagccgttattttgggattactgattggtagctttctgaatgtcgtcatttatcgc acgccgataatgatggagcgtgaatggacacaattttccaaagagcatttgggcatcgag ctggcagacgaagaaaaacagccatttaatctgcgtaaaccggattcacgctgcccaaaa tgcaaaagtccgatcagagtttggcaaaatatacccatcctcagctatgttttcctaggt gggaaatgccacgcctgcaaaacccatatcagcatacgctatccgttgattgagctgctg acaggtgtcttattcggtattgtcacgtggcaatacggctggtcatgggcaactattggc ggattgattctgactgctatattgattgcgctgacctttatcgatgccgatacgcaatat ttgcccgacagcctgactcagcccctaatttggatcggcctgctgtttaatctgaacgat acattcgtgcccctgcattcagccgtttggggcgcgattgcaggctatatgagcctttat actttatgcgccgtctataaacttctgaccggtaaaatcggcatgggcaatggcgatttc aaacttttggccgcactcggtgcatggctgggcgtaggaattttgcctgttctgatcttc atggctgccttagtcgggctgttaggcgcactcatcgcccgcgtcggtaaaggtcagtat tttgcttttggtcccagccttgctgtagcaggatggattattttggttgccaatgcacct attacccaactggtacaatggtggctcgtacaatcaggattccgataa(seq id no:15)。
[0137]
12_gene_id_40831:
[0138]
atgcgcattgcactcttctccacgtgcatcgttgacggcatgtacccccgcgtagcacgc gccaccgtcgaaattcttgagcgcctgggccacgaggttgtcttccccccgaaccagacc tgctgctcccagatgcacgtcaactccggctacttcgatgacggctacgaagtcatcaag aaccacgttgaagctttcgactcctgggactgggacgttgctgttgcaccctccggctct tgcgtcggctccctgcgccaccagcaggcaatggttgcacgtagccgcggcgacgaggca ctggctctgaaggcagagcagatcgcttcccgcaccttcgagctctcccagctgctgatt gacattctcggcatcaccaacgccgccgagcagctcggctcctacttccccgagcacgtc acctaccacagctcctgccac
ggtatgcgtatgctcggcctgggcacccgccagcaggac ctggtccgcaccgttgagggtatcaagtacacccagattgagggcatggaccagtgctgc ggcttcggcggtaccttctccttcaagaacgctgacgtctccggcgcaatggtcaacgac aaggcagacaacgttgaagcaaccggtgcttccgtctgcaccggcggcgactgctcctgc ctgatgaacatcggtggcgcactctcccgtcgcggctccaacgtgaagaccatccacttc gctgagatcctcgcctccaccaaggaaaacccgctcaagatcaactccgagcatgttgag ctctcgatcgcctaa(seq id no:16)。
[0139]
hpar1239_c_1_740:
[0140]
atgaaactcaaagcactttctcttgcattacttgccttacctatcgcttcttttgcagaa gaacccgtaccgaattatccagctgatgtaacctttactgttgaagcagaaaaagcagta gaacgtgatttattgcaagtctcccttttttaccaagcggaaggcaatgatttatccgcg ctcaataaaaccatggcagaaaaaatgaataaagccattgaattagcgaaagcacagagc tcggtcgaaattactgataattctcgtaataccatggttcgttacgataataaaggcaaa cagcaaggttggattgcgcatgcaggattaaccttagaaagtaaagactctcaagcatta tcaacattagtaaatgaactagatggcgtattggccattgctcaggtcaatgcctctgtt tctcgtgaaaaattaagtagtttagaaaatgaattaaccaaagaagctttagcaaaactt aaagataaagcactcttagttcaagaatccttgcaggtgaaaggttatcgcatacagaac cttgaaatttcctcagctaatgattcagtcgctgatttccgcccttatgctgcagccgaa ctaagctctaaaagtttatactcgagtgggaaagatgaaacttacactcaaagtggcaaa gagaaaattaaagtcagtgtgaatgcgcgaattgcattgcttaaagaataa(seq id no:17)。
[0141]
19_gene_id_4962:
[0142]
atgcacaccccgcagcgccccaacggcgacatcttcaaccgcatggtctccggcgccgcc tacatgcccgacgccgaatgtgcggcacagactaagcgcatccacgccgccaccgtcctc gcggaggagcactacgcccgaggcgagcacgcccaggccatgcacgtctaccgcgaaaac ctcggccacctgggcgagcactcccacatccgccccggtgcgcgcttcgactacggcgtg aacacctacattggcgacggttccttcttcaacttcgactgcgtattcctggacgtctgc ccgattcgcatcggatccaccgtgctcgtcggcaacaacgtgcagttcctcacccccacc cacccgctcaaccccgtcgaccgcgccgcgtactgggaaggcggcaagcccatcaccgtt gaggacaacgtctggatcggtggcggcgccatcatcctgggcggcgtgaccatcggcaag aactccgtcatcggtgcgggtaccgtcgtcaccaaggacgtacccgcaaactccgtcgtg gtcggcaaccccggacgcgtggttcgcaccctcgacgagaacgaacgacccgcatacccc cacccctactccgccgaatccctcgaagaggcccgggccttccagtccgagcacagcgag taa(seq id no:18)。
[0143]
9_gene_id_22858:
[0144]
ttgtctgaaaaaattgccgtcaaacgccgttgggtacaccctatttccgacaaaatccca ttggaccaagctgctttgattgagcctctgtctgtcggtcaccatgcttatgtacgcagt ggcgcgaaagaaggcgatgtcgcattggttggcggcgcaggtccaatcggtttgctgttg gctgccgtattgaaagccaaaggcatcaaagtcatcatcaccgaattgagcaaagcacgt aaagacaaagcacgcgaagccggcgtggccgactacatcctcgacccgtccgaagtcgac gtcgttgaagaagtgaaaaaactgaccaacggcgaaggcgtagatgtcgcattcgaatgc accagcgtcaacaaagtgctggacaccatggtcgaagcgtgccgtccgactgcgaaagtc gtcatcgtatccatttggagccaccccgctaccatcaacgtccacagcgttgtgatgaaa gagctggacgtgcgcggcaccatcgcctactgcaacgaccacgccgaaaccatcaaa(seq id no:19)。
[0145]
21_gene_id_11551:
[0146]
aaactcgtcaccctcaccgtcaccgcaatcggcctcatcaccctcatcaccgtcgcctgc gcatggcccatcatcagcatcatgggctcaggatggagccccgaacaacgacagctcggc ttcatcttctccctctggtgcc
taccccaaatcttcttctacggcctctacaccgtcatc ggacaggtcctcaacgccaaagacgcattcggcgcctacatgtggtcgcccgtactcaac aacgtcatcaccatcctgtcgctgttcctattcatcttcctcttcggcgcacaaaccccc aaccccaaccccatccacaccgtagcaagctggaccaacgcacaaaccctcgtgctcgca ggctcrcaaactcgtcaccctcaccgtcaccgcaatcggcctcatcaccctcatcaccgt cgcctgcgcctggcccatcatcagcatcatgggctcaggatggagccccgaacaacggca gctcggcttcatcttcgccctctggtgcctaccccaaatcttcttctacggcctctacac ggtcatcggacaggtcctcaacgccaaagacgcattcggcgcctatat(seq id no:20)。
[0147]
7_gene_id_4552:
[0148]
ccccagaataccgacaaaggagatcctttcgtgatcgaagaaatccttgccgaagccggc gagaagatggaaaagaccatcgaagcaaccaagactgagttcgctaaggtccgcaccggt cgtgtgaaccccgcaatcttcagcggcctgaccgccgagtactacggtgcaccgaccccg ctgcagcagctggtgtccttcaacgttgaggacgcacgtaccgtcaagatcgttcccttc gatgtgaccgcactggcagctatcgagaaggcgctgcgtgactccgacctgggtgttaac cccagcaacgacggtgcagctatccgcgttgtcatgcccgaaatgaccgctgagcgccgt aaggaatacaccaagctcgtcaaccacaaggctgaagaggctcgtatctccatccgcaac gttcgccgccacgcgaagaccgctattgacaagctggtcaaggacagcgaaatcggtgaa gacgagggcgcacgcgctgagaaggaactggacgcactgaccaagaagcacatcgaccag gtcgacgcactgtacaagcacaaggaagctgaactgctcgaggtctaa(seq id no:21)。
[0149]
29_gene_id_64779:
[0150]
atcatcgataccgcaggtgtgcgccgtcgcggcaaagtggatgaagcagtggaaaaattt tccgttatcaaagcgatgcaggcggttgaggcagcaaacgttgccgttttggtgttggac gctcaacaagatatcgccgaccaagatgcgacgattgcaggttttgctttggaagcaggc cgtgccttggtggtggcggtcaataaatgggacggcatcagcgaagagcgccgcgagcaa gtgaaacgcgatatcaaccgcaaactgtatttccttgattttgctaagttccactttatt tctgcgttgaaagagcgcggtatagacggtttgtttgaaagcattcaagctgcctacgac gcagcgatgattaagatgccgacgccgaaaatcacgcgcgtcctgcaaagcgcgatcgag cgtcagcaaccgccgcgtgctggcttggtgcgcccgaaaatgcgctatgctcaccaaggc ggcatgaatccgccggtgattgtggttcac(seq id no:22)。
[0151]
19_gene_id_65125:
[0152]
ttggcggcggtrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrrgcc ctgtatgcctgcccgccgccgcccgaaaacggttttgcagacagcctgaaacaagaaaca ggtcgtctgaaaatcgctttttggaagcagacatggtttggcggcggcaacgacgaaggc acggaagccgcgttcgcccatagtttgaaactgatgcaagatgcgggacacgaattggaa gaagccacgcccgattttgcgccacccgaacagctcaaccgcgccgcccgcatcatcgtc atgggcgaaacggcaaaactgttctaccaatatcaatacgaaaccggacaaaaactgccg caccgcctgcttgagccgacaacgtgggcaatgattgtgcaggggcagcaaatcagcgcg ggcgaaatggcatgggcgcgcgacgtgatgctggcgcaggaacgtgccgccaaagcattt ttcacacgctac(seq id no:23)。
[0153]
26_gene_id_36379:
[0154]
aggaatatttttatgaaacaggcggttgatccttatgcgagtttccgtgaactgacgctg cgggggatgatactcggtgcgttgattacggtgatttttacggcatccaatgtgtatttg ggcttgaaggtgggcttgacttttgcgtcttcgattccggctgcggtgatttcgatggcg gttttgaagttttttaaaggcagtaatattctggaaaataatatggtgcagactcaggct tcggccgcgggtacgctctccagtattatttttatcttgccgggtttgctga
tggccggt tattgggcgggcttcccgttttggcagacaacgctgctgtgtatgtccggcggtattttg ggcgtgattttcactrrrrrrrrrrrrgtaatattctggaaaataatatggtgcaaactc aggcttcggctgcgggcacgctctccagtattatttttatcttaccgggtttgctga(seq id no:24)。
[0155]
ncbiaceo_c_3_745:
[0156]
atgattgaatttcccaaccagcccgatttccctgagcaacacaccgccgtcgccgtcggt accgtcaacatgagccagcaggcgatacgggtgttgatagagaatgccaccgaaaaggca agtattctgaccatcgcccgagttgccgccattcagagcatcaaacagaccgccaacctg attccgctgcacctgcccggacgcatcatcggcgtgcaggttgatttcgatattaacgtt gagctggcctgcctgaaggccacattgaccgtccaagcgaacagcaacggcagcattgcc accgaagccctgactggcctaaatttggcgctcttgagtgtttacgacatgatgaaagac gttgaccgcaatatgatgctcagcggcatccgcctcgaatccgaaaccagcagtgaaggc cgtcctttcctgtttgacaacgcatacgaaaatattaatttctaa(seq id no:25)。
[0157]
16_gene_id_11722:
[0158]
cacgctaaggcaattctcggcatggacatcaagggccacaccgttcaccgcgtgctggtc gccgacggcgcagacatcgctgaagagtactacttctccctcctgctggaccgtgcaaac cgctcctacctggcaatgtgctcggtagaaggcggtatggaaattgagcagctggcagct gagcgtcccgaggcgctggccaaggttgaggttgacccgctggtcggcctggaccaggct aaggccgaggagattgtgaaggcagcaggcttctccgcagaactgcaggagaaggttgtt cccgtcctcgtcaagctgggtgaggtcttcagcaaggaagacgcaaccctggttgaggtt aaccccctcgttctgacccccgagggcaccatcctggccctggacggcaaggtctccctg gacgacaacgcagaattccgccagcccgagcacgaggct(seq id no:26)。
[0159]
27_gene_id_58782:
[0160]
caatccctctcccaccgttatcccaaagccgaaactgccgcgctggacaatgtgtcgttc gatattgccgacggcgaatgtttggggctgctcggccacaacggtgcggggaaaacgacg ctgatgtcgcttttggcaggcttgcaggaagtacggcagggcgaaattttgtttgacggc aagcctttgcacctgttaagccgtagcgagcggcagaaaatcggtttggtgccgcaggat tttgcgttttatccgcagctttcggtgtgggacaacctgctgtttttcgcctcgctttac aaagtacgcgacaaaggccgtctgaacgcgctgttggaacagaccgacctgaccgcccac aaaaacaaa(seq id no:27)。
[0161]
5_gene_id_16214:
[0162]
atgagccagaatccctatgagccgcagcccgcggcgtccctggtttccggcgggtatttt cccggtcaggtgcccggcactcaggcgtcttcagctcccgcccagcgcctggtggcgatg gaagaagagcctgaagagaagatgattgaggcggcactgcgccctaagtccctggatgac tttgtgggtcagcgccgtgtgcgtcagcagctgtccctggtgctggaggcttcgaagatg cgtggccgtagcgccgaccatgtgctgctgtcgggccctcccggtctgggtaagaccact ctcgcaatgattatrrrrrrrrrrrrrrrrrrrrrrrtagcgccgaccatgtgctactgt cgggccctcccggtctgggtaagaccactctcgcgatga(seq id no:28)。
[0163]
rmuc_c_3_792:
[0164]
atgagctcgatcgaaggcatcgaagacccctttggccccggaaccgcaggcggcaccgcc ctgctcgaacgcgaagaaacccagctttctgagcccggcgaccacgagcgcttctcccac tacgtacgcaaggaaaagatcctcgaatcggcgctgaccggtgaacccgtacgcgccctc tgcggcaaaatgtgggtacccggccgtgacccgcagaaattccccgtctgcccccagtgc aaggaaatctacgaaggcctcgcacccggcaatgacggcgacaacggctccggcgaataa (seq id no:29)。
[0165]
11_gene_id_9011:
[0166]
atggcatcgtctgctactgccactcaggctgctgagtctggcgcatcctcaaagagcgat gagaagcgcggtttcttcggccgtatttttgcattccttcgcgaagttatcgcggagttg aagaaggttaccactcccaccggacgcgagctgattggttacttcctcgcggttctgctg ttcgtcgtggtgatgctcctgctgatttccggcctggattatctgttcggccagggggca ttctggatctttggtaacggaacgattcagcgtaactag(seq id no:30)。
[0167]
》6_gene_id_13071
[0168]
ggcgcgcaccaagtcggcaacgggcgagttggcgctgggtttgtcgtcgtgaaacaagcg cacgcctttgccttcgaaaaagccgataccgaagccgtcggcgtggtggtcggtaatgcc gccgcgacggcggaagccttcaaaagaaaacatgatgtctgtgggagtgttgcaattcat gccgagcagttggcacatggtgttaacctttatttttggggtatttatagggttattatg gtgtatcgggcaggcagtttcaatatgtag(seq id no:31)。
[0169]
》12_gene_id_55866
[0170]
ggcgcaggtgagcgcggcaagctcacgcaaccggcgcttaaccaggttgcgggtcaccgc gttgcccaccgccttcgacacaatgaaacctacccgggttgtttttccgggatttttcgc cgtagatacaacgatgttccggcgtccactgcggatgccggaacggaaggcgtgggagaa ttcagcgccggtgcgcattcggtgccgttgaggcagcacgtctccccctttacatggtgg ggaggctcggcaggg(seq id no:32)。
[0171]
》43_gene_id_57182
[0172]
cattgcacgaaaaccaataactttatgaccgcacaagaatttccacaccagcataccgcc cattgcgaaagcggtgtgatgtccaccctgcttaaataccatggccacaatttagacgaa gccatgattttcggcattgcccacgcgctgacatttgtctggatgccgctgatcaaatta aacggcatgccgctggtt(seq id no:33)。
[0173]
》12_gene_id_43665:
[0174]
ggtggcatccaccaggtgacgcagagactgcttggtctcctcgtaatcacgagtcttcag accacagtccgggttcacccacaggtcagcaatggtgccgtaaccaatcgcagccttcag cagggactcaatctcctcctgaga(seq id no:34)。
[0175]
27_gene_id_5129:
[0176]
ttgaggntcgatgccgtcgcgaacgaagaacacctgaatcagctgcgcggggcgcagggt ggtttcagcgacgagggagcgcatggctgcggtggtgcgcaggcggcgcgggcggcgggt gaggttcatgatctctcctga(seq id no:35)。
[0177]
实施例3个体状态的检测
[0178]
利用12个唾液样本进行样本来源的个体状态的检测。
[0179]
参照实施例2的方法确定各唾液样本中的表3的10_gene_id_46693、11_gene_id_51306、 12_gene_id_64866、13_gene_id_16066、14_gene_id_30522、17_gene_id_45526、19_gene_id_2042的丰度,判断各样本中这7种基因的丰度是否落入各自在疾病对照组或者健康对照组的丰度的95%的置信区间, 7种基因的丰度均落入疾病组对应区间的样本对应的个体的状态判定为龋齿患者,均落入健康组对应区间的样本对应的个体的状态判定为非龋齿患者。
[0180]
结果显示,利用上述方法12个样本进行个体状态判断,其中对10个样本个体的状态的判断,与记录的该样本来源个体的状态一致。
[0181]
另外,发明人发现对表2和表3中的基因联合检测,例如检测表3中的基因标志物被
富集,同时表 2中基因标记物不被富集,能够更准确的判断发现龋齿患者或易感人群。
[0182]
在利用标志物治疗龋齿的方案中,发明人发现使表3中的基因标志物丰度得到抑制或者清除,同时使表2中基因标记物被富集,治疗效果极佳。
[0183]
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不一定指的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任何的一个或多个实施例或示例中以合适的方式结合。
[0184]
尽管已经示出和描述了本发明的实施例,本领域的普通技术人员可以理解:在不脱离本发明的原理和宗旨的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由权利要求及其等同物限定。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1