基于水解的探针和STR基因分型的方法
本发明涉及包含短串联重复序列(str)的基因分型样品。本发明公开了由3个dna区域组成的荧光标记的杂合dna:rna探针,其中一个区域包含至少1个rna残基,而另一个区域包含至少1个猝灭物(quencher)。本发明还涉及利用所述探针和rnase h2酶的方法,所述rnase h2酶识别当探针与含有str的dna样品杂交时形成的rna:dna双链体。该酶切割含有猝灭物的区域,导致荧光信号增加。当样品中的重复数与探针中的重复数完全对应时,杂交、随后的酶促识别和随后的探针切割在较高温度下发生。本发明的探针和方法在用于法医dna分析的便携式设备中特别有用。
背景技术:
1、脱氧核糖核酸(dna)用于个体的识别目的,包括亲属关系分析和法医dna基因分型。为此目的检查了dna中的多态性,例如短串联重复序列(str)和单核苷酸多态性(snp)。str仍然是许多应用选择的多态性。str基因座的特征是短的(通常为4个核苷酸)重复序列,这些重复序列在特定群体中的重复数量是多态性的。[1]
2、在人类基因组中,包含这种特定类型多态性的不同区域被识别。通过分析大量str基因座获得了统计上独特的概况(profile)以用于法医目的,这些str基因座大多位于人类基因组的非编码区。在欧洲,通常检查一组12个str,称为欧洲标准集(ess)。该组现在扩展了5个额外的基因座。[2]在美国,使用组合dna索引系统(codis),其包含13个核心基因座和7个额外的基因座。[3]
3、通常,这些基因座通过毛细管电泳(ce)进行分析,毛细管电泳是一种dna大小分离技术。ce是一个漫长的过程,其需要庞大的设备。此外,电泳所需的高电位意味着需要精确(accurate)的电源。总之,ce并不适合在便携式设备中实现。单机设备,例如rapidhit(applied biosystems)[4]己上市。这个特殊的设备质量为82kg,因此妨碍了常规dna痕迹的现场分析。
4、犯罪调查将从现场dna分析中受益匪浅,因为这可以加快调查速度。除此之外,在芯片上实施这些分析将降低污染风险,避免对训练有素的人员的需求,并降低社会成本。[5]
5、已经描述了可能集成在便携式设备中的str基因分型的替代检测方法。几乎所有这些方法都是基于杂交的方法,使用所谓的str探针。str基因座与snp基因座相比相当长,这意味着需要长探针。基于杂交的方法依赖于双链体稳定性。样品和探针之间的部分错配,在此称为异源双链体形成,将导致双链体不稳定,这反映在较低的解链温度上。然而,探针越长,错配对双链体稳定性的影响就越小。由于探针中存在重复单元,不仅str基因座根据定义很长,而且可能的等位基因具有高度的相似性:即使当探针和样品不共享相同数量的重复时,也存在大部分探针与样品完美匹配,只有一小部分显示与样品错配。
6、为了增加1个重复错配的不稳定效应(destabilising effect),us9404148b2[6,7]描述了在溶液中使用的hybeacon探针以及阻断剂寡核苷酸,从而缩短了探针长度。该测定在由lgc商业化的paradna设备中实施[8]。基因分型是通过传统的解链曲线分析完成的。该系统的缺点是探针设计限制,使得无法设计能够对完整dna概况所需的所有基因座进行基因分型的系统,并且需要第二个寡核苷酸作为阻断剂,从而显著增加了系统的复杂性。描述了使用多种合成寡核苷酸的其他系统,例如us9783842b[11]描述了基于差异杂交的方法,us7501253b2[12]描述了分支迁移测定,以及us6753148b2描述了基于探针和样品双链体稳定性的方法,即使用捕获探针和报告探针的“夹心杂交法”和“环出法”[13]。类似的缺点,例如对于hybeacon探针描述的增加的复杂性,在后面的系统中也会遇到。
7、us12/276849[9,10]描述了dpfret方法,其是一种基于解链曲线的方法,省略了阻断寡核苷酸的使用。该系统的缺点是使用了有毒的嵌入染料,其也改变了寡核苷酸的解链行为。
8、除了使用多种合成寡核苷酸外,酶促切割步骤的引入是一种有效的策略,以显著提高依赖于双链体去稳定化的测定的特异性。使用dpfret方法或任何其他依赖于探针和样品之间物理距离的方法,解链峰相对较宽,因为在退火过程中已经产生了信号。相反,核酸内切酶依赖于dna的正确碱基配对。因此,仅在双链体形成后才会产生信号,从而产生更窄和更独特的峰。
9、用于基因分型分析的适当的酶是rnase h2酶,其识别rna:dna双链体并切割rna链。us20160130673a1[19]描述了组合使用核酸内切酶活性(例如源自rnase h)和核酸外切酶(例如源自聚合酶)用于检测靶序列。所述系统与探针非常相似,但使用了嵌合dna-rna-dna探针。探针靶向目标小区域,例如snp或indel,并且依赖于探针的rna区域是否与目标靶区域杂交。探针被进一步设计,使错配位于双链体的中心,这是最不稳定的位置。这种分析产生二元答案(即rna部分是否杂交),这是非常适合分析双等位基因座例如snp基因座的特征。然而,这种策略不能应用于str探针,因为这些dna区域的特征在于多个可能的等位基因,这些等位基因的长度不同,而不仅仅是序列不同。实际上,这种探针的传感部分不能定位在探针的中心,而是更靠近末端。因此,对该探针的一些结构调整(例如锚区域和rna碱基的定位)是必不可少的。由于目标基因座比snp基因座长,所以错配的不稳定效应降低。这意味着即使发生错配,rna部分也会杂交,从而使评估和数据分析的方法复杂化。
10、总之,显然仍然非常需要设计一种str基因分型探针和方法,该探针和方法产生高信噪比、没有设计限制并且可以在便携式设备中实施。
技术实现思路
1、本发明涉及组合物,其包含:
2、a)寡核苷酸探针阵列,其中所述探针中的每一个包含从5′到3′或从3′到5的以下3个区域:
3、i.第一侧翼区域,其包含至少一个核苷酸,其与直接紧邻目标特定dna序列的区域退火并且具有比第二侧翼区域更高的解链温度,
4、ii.区域,其包含特定dna序列,所述dna序列与样品内的目标短串联重复区域退火并且其包含至少一个荧光团,和
5、iii.第二侧翼区域,其包含至少2个核苷酸,并且其包含至少一个核糖核苷酸和能够有效猝灭所述荧光团的至少一个猝灭物部分,其中所述荧光团和所述猝灭物部分通过至少一个核糖核苷酸彼此分开,和
6、b)rnasc h2酶,其能够通过在所述探针与所述样品杂交时识别rna:dna双链体来消化所述探针。
7、本发明还涉及如上所述的包含寡核苷酸探针阵列的组合物,其中所述猝灭物附接到所述探针中的每一个的3′或5′末端。
8、本发明还涉及如上所述的包含寡核苷酸探针阵列的组合物,其中所述荧光团附接到所述探针中的每一个的第二侧翼区域的核苷酸上,并且其中所述猝灭物附接到所述探针中的每一个的目标特定dna序列的核苷酸上。
9、在本发明的具体实施方案中,所述荧光团是荧光素衍生物。
10、在本发明的具体实施方案中,所述猝灭物是iowa black fq猝灭物。
11、本发明还涉及如上所述的包含寡核苷酸探针阵列的组合物,所述探针阵列包含多于一个核糖核苷酸。
12、本发明还涉及如上所述的包含寡核苷酸探针阵列的组合物,其中所述核苷酸是核酸类似物。
13、本发明还涉及如上所述的包含寡核苷酸探针阵列的组合物,其中所述探针中的每一个固定在支持物上。
14、本发明还涉及对样品内的短串联重复序列进行基因分型的方法,其包括以下步骤:
15、-提供包含dna的样品,
16、-扩增所述样品中包含目标特定dna序列的dna以获得扩增的单链dna序列,
17、-将如上所述的探针阵列添加到所述dna序列以获得与所述探针退火的单链dna序列的双链体,
18、-添加rnase h2酶,
19、-将样品、探针和rnase h2酶的混合物加热到rnase h2酶被激活的温度,
20、-在激活rnase h2酶后冷却所述混合物时测量荧光,其中荧光强度的增加提供关于在所述样品中是否存在特定的、完全互补的短串联重复序列的信息。
21、本发明还涉及如上所述的基因分型方法,其中所述样品内的所述扩增通过不对称pcr进行,以获得扩增的单链dna序列。
22、本发明还涉及如上所述的基因分型方法,其中所述样品内的所述扩增通过使用生物素标记的引物的对称pcr或随后的λ外切核酸酶消化来进行,以获得扩增的单链dna序列。
23、本发明还涉及如上所述的基因分型方法,其中将所述探针阵列添加到溶液中,或固定在支持物上。
24、发明描述
25、本发明涉及组合物,所述组合物包含寡核苷酸探针阵列和rnase h2酶。探针在本文中被定义为合成制造的寡核苷酸,由彼此共价连接的2个或更多个核苷酸和/或核糖核苷酸组成,其中一些核苷酸和/或核糖核苷酸可以被修饰。这种修饰被定义为附接到寡核苷酸上的分子,该分子不一定存在于天然dna或rna中。修饰的实例是例如荧光部分的存在、猝灭物的存在、用于附接目的的分子的存在、解链温度的调节剂(modifier)等。探针可以合成制造,但寡核苷酸探针的定义在本文中不限于专门合成制造的寡核苷酸。探针通常以它们将与被研究分子相互作用的方式设计,并且将观察和使用探针对这种相互作用的响应以获得所述被研究分子的信息。
26、查格夫规则可以解释dna的互补性,指出腺嘌呤总是与胸腺嘧啶或尿嘧啶形成氢键,而胞嘧啶与鸟嘌呤形成氢键,这一过程也被称为沃森-克里克或hoogsteen碱基配对,从而产生双链dna。杂交或退火在本文中被定义为形成双链体或异源双链体结构,由互补碱基配对后的两条核酸链组成。双链体结构被定义为2条完全互补的碱基配对核酸链的复合物。异源双链体结构被定义为2条部分互补的核酸链的复合物,例如2条dna链被一个或多个4-核苷酸重复延迟(deferring)。
27、本发明公开的探针阵列的功能是短串联重复基因座(str-基因座)的基因分型。str基因座的特征是短的(通常为4个核苷酸)重复序列,这些重复序列在关于重复数量的特定群体中是多态性的。与单核苷酸多态性(snp)相比,这些基因座是多等位基因的,表明群体中存在相当广泛的重复数。通过确定足够基因座的重复数,获得个体的统计学上独特的概况。措辞“探针阵列”是指对于所研究的str基因座的每个等位基因,设计了专用探针。探针阵列由针对某个基因座的所有不同探针组成。应分别分析特定探针和样品之间的相互作用,这意味着所有不同的探针应在物理上分开,例如通过多孔板上的不同孔,或通过将它们固定在表面上的不同点上。
28、如图1所示,本发明公开的寡核苷酸探针从5′至3′或从3′至5′包含第一侧翼区域、特定的目标dna序列和第二侧翼区域。
29、第一侧翼区域是核苷酸序列并且包含至少一个核苷酸。在本发明的更方便的实施方案中,侧翼区域包含20至40个核苷酸。第一侧翼区域与紧邻str区域的区域互补并退火,并确保样品和探针的适当退火,因此用作锚。由于该第一侧翼区域与第二侧翼区域相比具有明显更高的解链温度,所述杂交的起始被特许(privileged)在第一侧翼区域。为了获得正确的基因分型,样品的第一重复与探针的第一重复退火并防止样品滑动至关重要。
30、目标特定dna序列包含至少1个短串联重复序列,并包含至少一个荧光团,并与样品内的短串联重复区域退火。在本发明的一个实施方案中,该样品是dna,其中靶str区域通过例如聚合酶链式反应的方式扩增。
31、第二侧翼区域包含至少1个核苷酸并包含至少一个核糖核苷酸,例如atp、ctp、gtp和utp,并含有能够有效猝灭所述荧光团的至少一个猝灭物部分。
32、荧光团在本文中被定义为以约350nm和900nm之间的最大荧光发射为特征的化合物。常用的荧光素衍生物是5-fam(5-羧基荧光素)。其他常用的荧光团是5-六氯-荧光素、6-六氯-荧光素、5-四氯-荧光素5-tamra(5-羧基四甲基罗丹明)、6-tamra(6-羧基四甲基罗丹明)、cy5(吲哚二羰菁基-5);cy3(吲哚二羰菁基-3)和bodipy fl(2,6-二溴-4,4-二氟-5,7-二甲基-4-硼-3a,4a-二氮杂-s-引达省-3-丙酸)(2,6-dibromo-4,4-difluoro-5,7-dimethyl-4-bora-3a,4a-diaza-s-indacene-3-
33、proprionic acid)。
34、猝灭物被定义为当靠近所述荧光团时抑制荧光团部分发光的部分。一种常见的猝灭机制是荧光共振能量转移(fret),但本文中猝灭物的定义不限于此机制。其他机制是例如光致电子转移。市售的淬火基团有:dabcyl、iowafq和rq、zentm和black hole猝灭物,例如
35、在本发明的具体实施方案中,荧光素衍生物与iowa black fq猝灭物部分组合使用。本领域技术人员将认识到荧光部分和猝灭物的其他组合适用于该目标。荧光团的发射波长与猝灭物的最佳吸收波长相对应是至关重要的。荧光团和猝灭物的可能组合的实例是cy3和black hole猝灭物2。
36、荧光团和猝灭物部分通过至少一个核糖核苷酸彼此分开。如果猝灭物和荧光部分与相同的核苷酸或核糖核苷酸连接,则在探针被合适的酶消化时不会出现信号,因为两个所述部分不会彼此分离。在本发明的更具体的实施方案中,荧光团和猝灭物被15至30个核苷酸分开。
37、在本发明的具体实施方案中,猝灭物附接到探针的3′或5′末端。
38、本发明还涉及如上所述的寡核苷酸探针,其中所述荧光团附接到第二侧翼区域的核苷酸上,并且其中所述猝灭物附接到目标特定dna序列的核苷酸上。本发明进一步涉及如上所述的寡核苷酸探针,其包含多于一个核糖核苷酸。
39、本发明还涉及如上所述的寡核苷酸探针,其中所述核苷酸是核酸类似物,例如lna、pna、gna、tna、吗啉代(pmo)。
40、本发明中描述的所述探针在溶液中和固定在支持物上都具有功能。
41、本发明还涉及对样品内的短串联重复序列进行基因分型的方法,其包括以下步骤:
42、1.提供包含dna的样品。在本发明的更具体的实施方案中,样品包含具有至少一个str基因座的dna。这种dna的来源可以是人类、动物甚至植物。本领域技术人员将认识到这不是限制性列表。
43、2.扩增所述样品中包含目标特定dna序列的dna以获得扩增的dna序列。有多种扩增dna的策略,然而,聚合酶链式反应(pcr)是最常用的方法,以扩增特定目标序列例如str基因座。在pcr反应中,扩增的基因座由专门设计的引物确定。使用dna聚合酶进行扩增。本领域技术人员将认识到存在许多其他扩增dna的策略,包括靶向的或非靶向的。实例为等温dna扩增、全基因组扩增和滚环扩增。
44、3.将如上所述的探针添加到所述扩增的dna序列中以获得与所述探针退火的单链dna序列的双链体。
45、4.添加rnase h2酶或任何其他能够在rna位置与互补核苷酸杂交时切割该位置处探针的酶。在本发明的具体实施方案中,添加25mu的酶。
46、5.将样品、探针和rnase h2酶的混合物加热到rnase h2酶被激活的温度,通常为95℃,使用例如实时pcr仪。
47、6.在激活rnase h2酶后冷却所述混合物时测量荧光,使用例如实时pcr仪。
48、在高温下激活rnase酶后,将混合物缓慢冷却。在本发明的具体实施方案中,混合物以每分钟0.5℃的速率冷却。然而,应该注意的是,更快和更慢的冷却都是可行的。混合物冷却后,探针将与样品中扩增的dna链杂交。由于探针中存在锚区域,杂交被特许在探针的锚侧进行。这确保了探针的第一重复从锚侧开始,将与样品的第一重复杂交。
49、如果所述探针和扩增的dna链具有完全相同的重复数,则完整的探针将与样品杂交。当探针和扩增的dna链不具有相同的重复数时,就会形成异源双链体(图2)。在后一种情况下,与完全互补的情况相比,杂交将在较低的温度下发生。由于rnase h2酶在很宽的温度范围内都具有活性,因此探针与样品杂交后立即被切割(图3)。因此,与错配探针相比,具有与样品相同数量重复的探针的荧光信号在更高的温度下会增加(图4)。
50、因此,本发明描述了通过酶促消化确定探针杂交温度的str测定。对于str基因座,探针和样品之间部分错配的不稳定效应很难评估,因为这些基因座被定义为长基因座。众所周知,探针越长,错配的不稳定效应越低。通过引入依赖于探针中rna单元的特异性杂交的酶促切割步骤,获得了极其尖锐和明显的峰,从而最佳地突出了双链体稳定性的差异。只有在这种核糖核苷酸特异性杂交后,暗示在异源双链体中形成开环结构(如图2所示),才会产生信号。荧光分子与猝灭物部分的组合使用产生了高信噪比。
51、本发明还涉及如上所述的基因分型方法,其中所述样品内的所述扩增通过不对称pcr进行,以获得扩增的单链dna序列。获得单链dna至关重要,因为双链扩增子的重新退火比探针杂交更有利。不对称pcr是一种常用的获得单链dna的技术。为了达到这个目的,将引物以不同的浓度添加到pcr反应混合物中。将掺入与探针互补的链中的引物将被过量添加。在第一个pcr循环中,两个引物都将被消耗,pcr将呈指数增长。在以较低浓度添加的引物耗尽后,pcr将线性发生,因为仅产生所需的链。
52、不对称pcr的替代方法是使用生物素标记的引物的对称pcr。pcr后,将链霉亲和素珠添加到扩增的dna中。生物素标记的引物将与链霉抗生物素共价反应,在双链扩增子变性后,可以分离所需的链。另一种选择是对称pcr和随后的λ核酸外切酶消化。只有源自5′磷酸标记的引物的链会被消化。
53、本发明还涉及如上所述的方法,其中将所述探针添加到溶液中,或固定在支持物上。
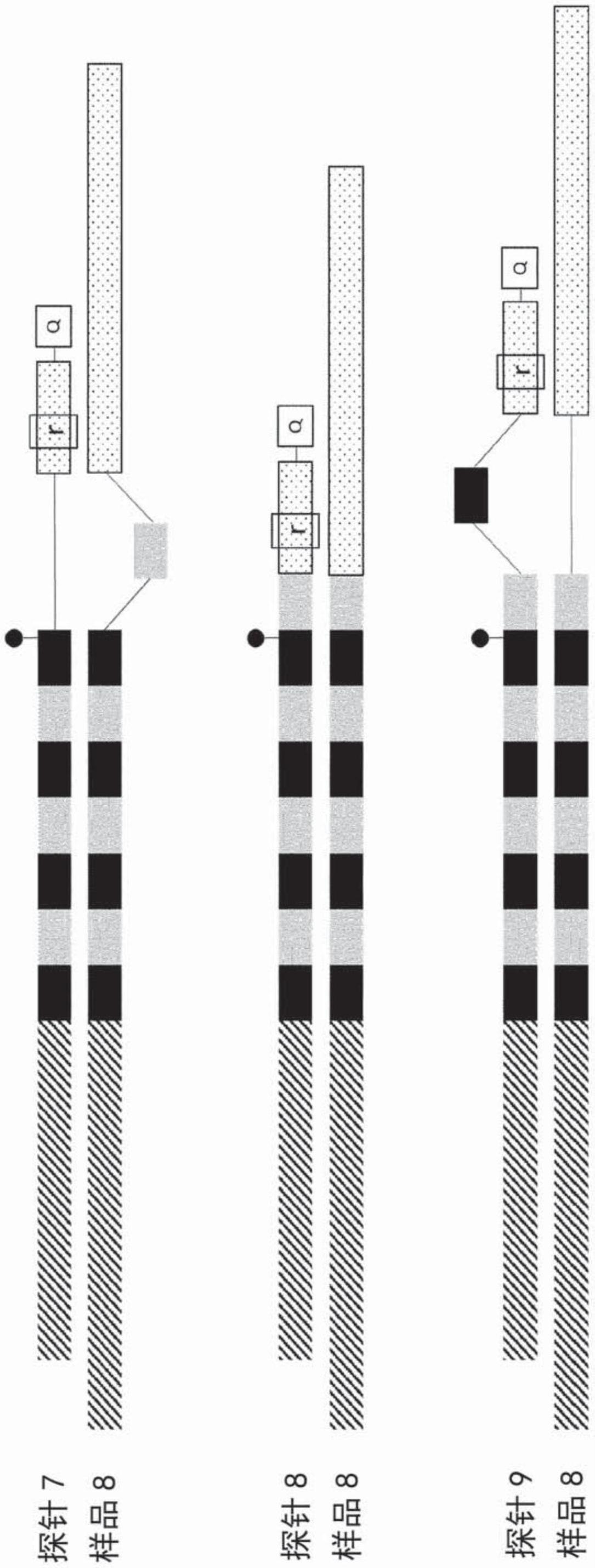
54、实施例
55、实施例1:
56、为th01基因座设计的3种不同探针(具有6、7和8个重复)与具有7个重复的合成制造的互补物混合。探针浓度为0.1μm,合成互补物浓度为1μm。探针序列见表1。添加rnase h2酶后,将混合物加热至95℃10分钟。此后,将混合物缓慢冷却以确保探针和合成互补物的适当杂交。在该杂交阶段,监测荧光。计算荧光相对于温度的一阶导数。
57、
58、表1:用于th01实验的寡核苷酸序列。核糖核苷酸前面有“r”。带下划线的t-核苷酸表示荧光素dt。iowa black fq用作猝灭物。
59、所有3个孔中的荧光均下降,表明所有不同的探针都被酶消化了。然而,与错配探针相比,匹配探针在更高的温度下被消化。这表明错配探针和样品形成了异源双链体。
60、实施例2:
61、将口腔拭子浸入200μl无菌hplc-水中。经过30”的涡旋步骤后,去除拭子并将水用作pcr的输入。使用30μl输入样品进行单重不对称pcr。引物浓度为0.1μm正向引物和1.5μm反向引物。pcr混合物的体积为50μl,含有浓度为0.5mm的mgcl2+、各200μm的dntp、1×qiagenpcr缓冲液和1.3u hotstartaq酶。通过在95℃加热pcr混合物15分钟,然后在95℃1分钟、59℃1分钟和72℃80秒的60个循环来完成聚合酶的活化。引物序列见表1。
62、不对称pcr后,将8.5μl扩增产物的等分试样分装在96孔板中。在每个单独的孔中,以1μm的起始浓度添加1.5μl的一种特定探针。这些混合物在95℃下变性10分钟,然后以0.04℃/s的变温速率(ramp rate)缓慢冷却,同时使用lightcycler(罗氏)连续测量荧光。计算杂交曲线的一阶导数,得到解链峰。探针序列见表2。使用ce分析对样品进行基因分型,并具有等位基因6和7。
63、
64、表2:用于th01实验的寡核苷酸序列。“r”表示以下单位是核糖核苷酸。带下划线的t-核苷酸表示荧光素dt。iowa black fq用作猝灭物。
65、所有杂交曲线的一阶导数如图7所示。可以观察到等位基因6、7和8的明显信号。尽管探针8是显示信号的3个探针中最长的探针,但它显示出明显较低的杂交温度,表明异源双链体形成。其他探针几乎没有显示任何信号,表明错配太不稳定而无法发生杂交。
66、实施例3:
67、如实施例2所述制备、扩增和分析口腔拭子。如实施例2所述使用相同的引物和探针。使用具有等位基因9.3的ce对检查的样品进行基因分型。具有等位基因9.3的样品有10个重复,但其特征是在其第3个重复中缺失了1个核苷酸。这些是具有挑战性的等位基因,因为该样品与探针10的杂交导致异源双链体仅因单核苷酸插入缺失(插入/缺失)而不稳定。
68、不对称pcr后,将8.5μl扩增产物的等分试样分装在96孔板中。向每个单独的孔中添加1.5μl探针(1μm)。这些混合物在95℃下变性10分钟,然后以0.04℃/s的变温速率缓慢冷却,同时使用lightcycler(罗氏)连续测量荧光。计算杂交曲线的一阶导数,得到解链峰。
69、所有杂交曲线的一阶导数如图8所示。可以观察到等位基因8和9.3的明显信号。探针10与阳性等位基因9.3仅有1个核苷酸错配,几乎没有显示信号。探针9是两个阳性等位基因的相邻探针,探针7也是阳性等位基因的相邻探针,与阳性探针相比,在较低温度下显示出解链峰。阳性探针在较低温度下显示出第二个峰,这可用于在探针8和样品9.3之间形成异源双链体,反之亦然。
70、参考文献
71、1.butler,j.m.,forensic dna typing:biology,technology,and genetics ofstr markers.2005:elsevier.
72、2.schneider,p.m.,expansion of the europian standard set of dnadatabase loci-the current situation.profiles in dna,2009.12:p.6-7.
73、3.hares,d.r.,selection and implementation of expanded codis core lociin the united states.forensic science international:genetics,2015.17:p.33-34.
74、4.hennessy,l.k.,et al.,developmental validation studies on therapidhitrm human dna identification system.forensic science international:genetics supplement series,2013.4(1):p.e7-e8.
75、5.bruijns,b.,et al.,microfluidic devices for forensic dna analysis:areview.biosensors,2016.6(3):p.41.
76、6.gale,n.,et al.,oligonucleotides and uses thereof.2016,googlepatents.
77、7.gale,n.,et al.,rapid typing of strs in the human genome bymelting.organic&biomolecular chemistry,2008.6(24):p.4553-4559.
78、8.blackman,s.,et al.,developmental validation of theintelligence system-a novel approach to dna profiling.forensic scienceinternational:genetics,2015.17:p.137-148.
79、9.halpern,m.and p.m.ellis,dye probe fluorescence resonance energytransfer genotyping.2010,google patents.
80、10.halpern,m.d.and j.ballantyne,an str melt curve genotyping assayfor forensic analysis employing an intercalating dye probe fret.journal offorensic sciences,2011.56(1):p.36-45.
81、11.mamone,j.,m.n.feiglin,and h.gamper,str genotyping by differentialhybridization.2017,googlepatents.
82、12.pourmand,n.,r.w.davis,and s.x.wang,dna fingerprinting using abranch migration assay.2009,google patents.
83、13.sosnowski,r.g.and e.tu,methods and apparatus for detectingvariants utilizing base stacking.2004,googlepatents.
84、14.moghaddam,p.p.and f.j.herrmann,randomized full-waveform inversion:a dimenstionality-reduction approach,inseg technical program expandedabstracts 2010.2010,society of exploration geophysicists.p.977-982.
85、15.tsuchiya,t.,method of detecting mismatching regions.2007,googlepatents.
86、16.kemp,j.,et al.,dna profiling and snp detection utilizingmicroarrays.2006,google patents.
87、17.walder,j.a.,et al.,rnase h-based assays utilizing modified rnamonomers.2014,google patents.
88、18.walder,j.a.,j.dobosy,and m.a.behlke,cleavable hairpinprimers.2018,google patents.
89、19.li,j.,et al.,nucleic acid detection by oligonucleotide probescleaved by both exonuclease and endonuclease.2016,google patents.
90、序列表
91、<110> 根特大学
92、<120> 基于水解的探针和str基因分型的方法
93、<130> p2019/085 pct序列表
94、<160> 14
95、<170> patentin version 3.5
96、<210> 1
97、<211> 52
98、<212> dna
99、<213> 人工序列(artificial sequence)
100、<220>
101、<223> 探针
102、<400> 1
103、ctgttcctcc cttatttccc tcattcattc attcattcat tcattcacca tg 52
104、<210> 2
105、<211> 56
106、<212> dna
107、<213> 人工序列(artificial sequence)
108、<220>
109、<223> 探针
110、<400> 2
111、ctgttcctcc cttatttccc tcattcattc attcattcat tcattcattc accatg 56
112、<210> 3
113、<211> 60
114、<212> dna
115、<213> 人工序列(artificial sequence)
116、<220>
117、<223> 探针
118、<400> 3
119、ctgttcctcc cttatttccc tcattcattc attcattcat tcattcattc attcaccatg 60
120、<210> 4
121、<211> 75
122、<212> dna
123、<213> 人工序列(artificial sequence)
124、<220>
125、<223> 与探针互补
126、<400> 4
127、acagactcca tggtgaatga atgaatgaat gaatgaatga gggaaataag ggaggaacag 60
128、gccaatggga atcac 75
129、<210> 5
130、<211> 79
131、<212> dna
132、<213> 人工序列(artificial sequence)
133、<220>
134、<223> 与探针互补
135、<400> 5
136、acagactcca tggtgaatga atgaatgaat gaatgaatga atgagggaaa taagggagga 60
137、acaggccaat gggaatcac 79
138、<210> 6
139、<211> 83
140、<212> dna
141、<213> 人工序列(artificial sequence)
142、<220>
143、<223> 与探针互补
144、<400> 6
145、acagactcca tggtgaatga atgaatgaat gaatgaatga atgaatgagg gaaataaggg 60
146、aggaacaggc caatgggaat cac 83
147、<210> 7
148、<211> 21
149、<212> dna
150、<213> 人工序列(artificial sequence)
151、<220>
152、<223> 引物
153、<400> 7
154、gtgattccca ttggcctgtt c 21
155、<210> 8
156、<211> 22
157、<212> dna
158、<213> 人工序列(artificial sequence)
159、<220>
160、<223> 引物
161、<400> 8
162、attcctgtgg gctgaaaagc tc 22
163、<210> 9
164、<211> 52
165、<212> dna
166、<213> 人工序列(artificial sequence)
167、<220>
168、<223> 探针
169、<400> 9
170、ctgttcctcc cttatttccc tcattcattc attcattcat tcattcacca tg 52
171、<210> 10
172、<211> 56
173、<212> dna
174、<213> 人工序列(artificial sequence)
175、<220>
176、<223> 探针
177、<400> 10
178、ctgttcctcc cttatttccc tcattcattc attcattcat tcattcattc accatg 56
179、<210> 11
180、<211> 60
181、<212> dna
182、<213> 人工序列(artificial sequence)
183、<220>
184、<223> 探针
185、<400> 11
186、ctgttcctcc cttatttccc tcattcattc attcattcat tcattcattc attcaccatg 60
187、<210> 12
188、<211> 60
189、<212> dna
190、<213> 人工序列(artificial sequence)
191、<220>
192、<223> 探针
193、<400> 12
194、ctgttcctcc cttatttccc tcattcattc attcattcat tcattcattc attcaccatg 60
195、<210> 13
196、<211> 67
197、<212> dna
198、<213> 人工序列(artificial sequence)
199、<220>
200、<223> 探针
201、<400> 13
202、ctgttcctcc cttatttccc tcattcattc atcattcatt cattcattca ttcattcatt 60
203、caccatg 67
204、<210> 14
205、<211> 64
206、<212> dna
207、<213> 人工序列(artificial sequence)
208、<220>
209、<223> 探针
210、<400> 14
211、ctgttcctcc cttatttccc tcattcattc attcattcat tcattcattc attcattcac 60
212、catg 64
- 还没有人留言评论。精彩留言会获得点赞!