帕金森氏病的检测方法与流程
1.本发明涉及一种使用帕金森氏病标记物检测帕金森氏病的方法。
背景技术:
2.从病理学上来看,帕金森氏病是一种以lewy小体的形成和中脑黑质的多巴胺神经细胞的变性和细胞死亡为主的进行性神经退行性疾病,其中,lewy小体的形成以α-突触核蛋白聚集体为主,临床上来看,则是一种以肌肉强直、震颤、少动、行走障碍等运动障碍为主的疾病。
3.在神经退行性疾病中,帕金森氏病的数量仅次于阿尔兹海默症,患病率达10万人中120~130人,推测日本约有14万患者。
4.目前,没有能够根治帕金森氏病的疗法,通过l-dopa补充等对症疗法控制症状对保持qol被认为是重要的。
5.但是,出现运动障碍的自觉症状时,已经处于中晚期,所以需要早期诊断疾病并实现早期介入。
6.作为用于检测帕金森氏病的生物标记物,除检测α-突触核蛋白积累之外,还提出了检测源自循环血清的微rna(专利文献1);测定血液中酪氨酸与苯基丙氨酸的浓度比率(专利文献2)等。另外,据报道称,在帕金森氏病患者的皮肤中,与大脑一样可见形成α-突触核蛋白聚集体(非专利文献1);帕金森氏病患者会出现脂溢性皮炎、黑色素瘤、水疱性天疱疮、酒糟鼻等皮肤疾病或症状(非专利文献2),考虑帕金森氏病与皮肤的状态存在某种关系,但科学关联性尚不明确。
7.另一方面,正在开发了通过分析生物试样中的dna和rna等核酸来考察人体内当前和将来的生理状态的技术。利用核酸的分析具有如下优点:已建立了全面的分析方法,一次分析即能得到丰富的信息;及容易基于与单碱基多态性和rna功能等相关的诸多研究报告实现分析结果的功能关联。源自生物的核酸可以从血液等体液、分泌物、组织等中提取,最近有些报道中使用皮肤表层脂质(skin surface lipids;ssl)中所含的rna作为用于分析生物的样本;从ssl能够检测到表皮、汗腺、毛囊和皮脂腺的标记基因(专利文献3)。
8.专利文献1:日本特表2019-506183号公报
9.专利文献2:日本特开2016-75644号公报
10.专利文献3:国际公开公报第2018/008319号
11.非专利文献1:rodriguez-leyva i et al.ann clin transl neurol.2014(modified)
12.非专利文献2:ravn ah et al.clin cosmet investig dermatol.2017
技术实现要素:
13.本发明涉及下面的1)~3)。
14.1)一种被检验者的帕金森氏病的检测方法,包括:对于从被检验者采集的生物试
样,测定选自snora16a、snora24、snora50和rexo1l2p这四种基因群中的至少一种基因或其表达产物的表达水平的工序。
15.2)一种1)的方法中使用的用于检测帕金森氏病的检查用试剂盒,其中,
16.含有与所述基因特异性杂交的寡核苷酸或识别所述基因的表达产物的抗体。
17.3)一种帕金森氏病的检测标记物,其由选自表3-1~3-4和表6-1~6-2所示的基因群的至少一种基因或其表达产物构成。
附图说明
18.图1是由测试数据中的最佳预测模型中的预测值和实测值制成的混淆矩阵。
具体实施方式
19.本发明涉及一种用于检测帕金森氏病的标记物和使用该标记物检测帕金森氏病的方法。
20.本发明人等从帕金森氏病患者和健康者的皮肤上采集ssl,将ssl中所含的rna的表达状态作为序列信息进行全面分析,结果发现,在两者之间,特定的基因的表达水平存在显著差异,可以以此为指标来检测帕金森氏病。
21.根据本发明,可以简单无创地在早期以高精度、灵敏度和特异度检测帕金森氏病。
22.本文中引用的全部专利文献、非专利文献和其它刊物整体作为参考引用在本文中。
23.在本说明书中,术语“核酸”或“多核苷酸”表示dna或rna。dna中包括cdna、基因组dna和合成dna的全部,“rna”中包括total rna、mrna、rrna、trna、non-coding rna和合成rna的全部。
24.在本说明书中,“基因”除表示包含人基因组dna的双链dna之外,还表示包含cdna的单链dna(正链)、具有与该正链互补的序列的单链dna(互补链)和包含这些片段的物质,并且,在构成dna的碱基的序列信息中包含一些生物学信息。
25.另外,该“基因”不仅包含特定的碱基序列所表示的“基因”,还包含它们的同源物(即,同系物或者直系同源物)、基因多型等变异体和编码衍生物的核酸。
26.本说明书中所公开的基因名称基于ncbi([www.ncbi.nlm.nih.gov/])所记载的官方代号(official symbol)。另一方面,关于基因本体(gene ontology、go),基于string([string-db.org/])所记载的pathway id.。
[0027]
在本说明书中,基因的“表达产物”是包含基因的转录产物和翻译产物的概念。“转录产物”为从基因(dna)转录生成的rna,“翻译产物”是指基于rna翻译合成的被基因编码的蛋白质。
[0028]
在本说明书中,“帕金森氏病”表示以黑质致密部多巴胺神经细胞的变性为主要主病变,缓慢进行性地表现出三种运动症状(静息性震颤、强直、运动缓慢和运动不能)的特发性且进行性的疾病。
[0029]
在本说明书中,帕金森氏病的“检测”是指明确帕金森氏病存在或不存在,也可以有检查、测定、判定、评价或辅助评价这样的术语替换。其中,在本说明书中,“判定”或“评价”这样的术语不包括医生进行的这些行为。
[0030]
本发明的由snora16a、snora24、snora50和rexo1l2p构成的四种基因是后述实施例所示的选自下述表a中记载的33种基因的基因,其是在帕金森氏病患者中,源自ssl的rna的表达量相比健康者显著上升(up)或下降(down)的基因,并且是至今都没有被知晓为与帕金森氏病有关联的基因(表中由粗体表示)。
[0031]
[表a]
[0032][0033]
[0034]
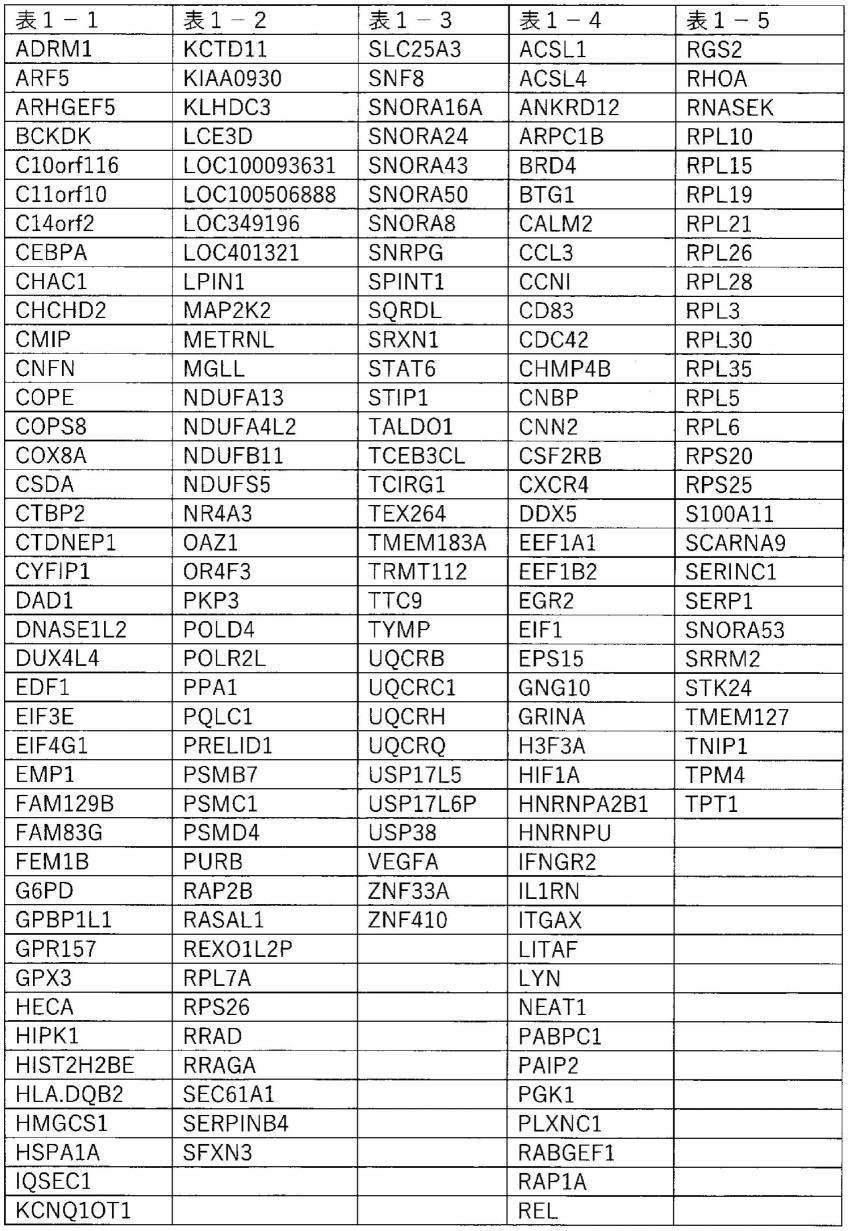
将从两个试验(试验1:健康者/帕金森氏病患者各15名,试验2:健康者/帕金森氏病患者各50名)的被检验者的ssl中所提取的rna的表达量的数据(读取计数值)转换为经样品间的总读取数(总read数)的差异校正后的rpm值,将该rpm值转换成以2为底的对数值,基于由此得到的值(log2rpm值),鉴定与健康者进相比在帕金森氏病患者中student’s t-test的p值为0.05以下的rna(试验1:111种表达上升基因、68种表达下降基因(共计179种基因、表1-1~1-5);试验2:565种表达上升基因、294种表达下降基因(共计859种基因、表1-6~1-27),选择在试验1和试验2中均上升的基因(18种)和均下降的基因(15种),这些基因即是表a所示的33种基因。
[0035]
因此,选自由该179种基因和859种基因(除去重复后共计1005种基因)构成的组的基因或其表达产物可以作为用于检测帕金森氏病的帕金森氏病标记物,其中,选自由表a所示的33种基因构成的组的基因或其表达产物是优选的帕金森氏病标记物。
[0036]
表a和下述表1中,“p值(p value)”表示在统计学检验中观测到比在归无假说下实际上根据数据算得的统计量更加极端的统计量的概率。因此,“p值”越小,则可以视为比较对象间存在显著差异。
[0037]“up”所示的基因是帕金森氏病患者中表达水平升高的基因,而“down”所示的基因是帕金森氏病患者中表达水平降低的基因。
[0038]
经过利用了基因本体(go)富集分析的生物过程(biological process(bp))的探索和kegg pathway探索表明,上述表达变动的基因群中包括与帕金森氏病(hsa05012)关联的基因(参见下述表2)。另一方面,在上述表达变动的基因群中,下述表3-1~3-4所示的基因是过去完全没有被报道过与帕金森氏病的关系相关的基因。因此,选自这些基因群的至少一种基因或其表达产物是用于检测帕金森氏病的新型帕金森氏病标记物,特别优选将试验1和试验2中相同的选自由snora16a、snora24、snora50和rexo1l2p构成的组的至少一种基因或其表达产物用作新型帕金森氏病标记物。更优选选自该组的两种以上,进一步优选三种以上,更进一步优选全部四种。另外,优选至少包含上述表a和下述表b中均包含的snora24。
[0039]
另外,作为rna的表达量的数据(读取计数值),可以使用例如通过deseq2(love mi et al.genome biol.2014)校正的计数值(normalized count值、标准化计数值)或者加上整数1的以2为底的对数值(log2(count+1)值)来进行表达变动rna的鉴定。
[0040]
例如,从上述两个试验的被检验者的ssl中提取的rna的表达量的数据使用标准化计数值,鉴定与健康者相比在帕金森氏病患者中似然比检验的p值的校正值(fdr)为0.25以下的rna时,试验1中得到74种表达上升的基因,209种表达下降的基因,共计283种基因(表4-1~4-8),试验2中得到151种表达上升的基因,308种表达下降的基因,共计459种基因(表4-9~4-20)。并且,试验1和试验2中均上升的基因有7种(anxa1、aqp3、emp1、krt16、polr2l、serpinb4、snora24),均下降的基因有10种(atp6v0c、bhlhe40、ccl3、ccni、cxcr4、egr2、gabarapl1、rhoa、rnasek、serinc1),共计17种(表b)。
[0041]
因此,选自由该283种基因和459种基因(除去重复后共计725种基因)构成的组的基因或其表达产物可以作为用于检测帕金森氏病的帕金森氏病标记物,其中,选自由表b所示的17种基因构成的组的基因或其表达产物是优选的帕金森氏病标记物,其中,与上述表a所示的基因相同的选自由下述表c所示的11种基因群构成的组的基因或其表达产物是更优
选的帕金森氏病标记物。
[0042]
另外,在上述表达变动的基因群中,下述表6-1~6-2所示的基因是目前完全没有被报道过与帕金森氏病存在相关关系的基因。因此,选自这些基因群的至少一种基因或其表达产物是用于检测帕金森氏病的新型帕金森氏病标记物,特别是试验1和试验2中共有的snora24(表中由粗体表示)或其表达产物是优选的新型帕金森氏病标记物。
[0043]
[表b]
[0044][0045]
需要说明的是,只要可以作为用于检测帕金森氏病的生物标记物,则上述可以作为帕金森氏病标记物的基因(下面也称为“靶标基因”)也包括具有与构成该基因的dna的碱基序列基本相同的碱基序列的基因。其中,基本相同的碱基序列表示,在使用例如同源性计算算法ncbi blast,在期待值=10;允许缺失;过滤=on;匹配得分=1;错配得分=-3的条件下进行检索时,与构成该基因的dna的碱基序列具有90%以上、优选为95%以上、更优选为98%以上、进一步优选为99%以上的同一性。
[0046]
本发明的帕金森氏病的检测方法包括下述工序:对于从被检验者采集的生物试样,测定靶标基因的表达水平,在一种方案中,测定选自由snora16a、snora24、snora50和rexo1l2p构成的组的至少一种基因或其表达产物的表达水平。
[0047]
在本发明的帕金森氏病的检测方法中,作为供采集生物试样的被检验者,可列举
包括人类和非人类哺乳动物的哺乳动物,优选为人类。在被检验者为人类的情况下,其性別、年龄和人种等不受特别限定,可以包括从婴儿到老人。优选该被检验者为需要或希望进行帕金森氏病检测的人。例如,该被检验者是怀疑帕金森氏病发病的人或具有帕金森氏病遗传因素的人。
[0048]
本发明中使用的生物试样只要为本发明的基因的表达随着帕金森氏病的发生和发展产生变化的组织和生物材料即可。具体而言,可列举:脏器、皮肤、血液、尿液、唾液、汗液、角质层、皮肤表层脂质(ssl)、组织渗出液等体液、由血液制备的血清、血浆、以及粪便、毛发等,优选列举皮肤、角质层或皮肤表层脂质(ssl),更优选列举皮肤表层脂质(ssl)。供ssl采集的皮肤的部位不受特别限定,可列举:头部、面部、颈部、躯干、四肢等身体的任意部位的皮肤,优选皮脂分泌较多的部位,例如头部或面部的皮肤,更优选面部的皮肤。
[0049]
其中,“皮肤表层脂质(ssl)”是指存在于皮肤表面上的脂溶性组分,有时也称为皮脂。通常,ssl主要包含由位于皮肤的皮脂腺等外分泌腺分泌的分泌物,以覆盖皮肤表面的薄层的形式存在于皮肤表面上。ssl包含皮肤细胞中表达的rna(参见所述专利文献3)。另外,在本文中,只要没有特别限定,则“皮肤”是包括角质层、表皮、真皮、毛囊、以及汗腺、皮脂腺和其它腺体等组织在内的区域的总称。
[0050]
可以采用用于从皮肤回收或除去ssl的任意手段来从被检验者的皮肤采集ssl。优选能够使用后述的ssl吸收性素材、ssl粘附性素材或从皮肤刮取ssl的器具。ssl吸收性素材或ssl粘附性素材只要为对ssl具有亲和性的素材,则不受特别限定,可列举例如:聚丙烯、纸浆等。作为从皮肤采集ssl的流程的更详细示例,可列举:用吸油纸、吸油膜等片材吸收ssl的方法、使ssl粘附于玻璃板、胶带等的方法、通过抹刀、刮刀等刮取并回收ssl的方法等。为了提高ssl的吸附性,也可以使用预先包含有高脂溶性溶剂的ssl吸收性素材。另一方面,ssl吸收性素材若包含水溶性高的溶剂或水分,则会抑制ssl的吸附,因此优选水溶性高的溶剂和水分的含量较少。ssl吸收性素材优选在干燥状态下使用。供采集ssl的皮肤的部位不受特别限定,可列举头部、面部、颈部、躯干、四肢等身体的任意部位的皮肤,优选面部等皮脂分泌较多部位的皮肤。
[0051]
从被检验者采集的含rna的ssl可以保存一定时间。为了尽可能抑制所含有的rna分解,采集到的ssl优选在采集后尽可能快速保存在低温条件下。本发明中,该含rna的ssl的保存温度条件只要为0℃以下即可,优选为-20
±
20℃~-80
±
20℃,更优选为-20
±
10℃~-80
±
10℃,进一步优选为-20
±
20℃~-40
±
20℃,进一步优选为-20
±
10℃~-40
±
10℃,进一步优选为-20
±
10℃,进一步优选为-20
±
5℃。该含rna的ssl在该低温条件下保存的时间不受特别限定,优选为12个月以下,例如6小时以上且12个月以下,更优选为6个月以下,例如1天以上且6个月以下,进一步优选为3个月以下,例如3天以上且3个月以下。
[0052]
在本说明书中,作为靶标基因或其表达产物的表达水平的测定对象,可列举:由rna人工合成的cdna、编码该rna的dna、被该rna编码的蛋白质、与该蛋白质相互作用的分子、与该rna相互作用的分子、或与该dna相互作用的分子等。其中,作为与rna、dna或蛋白质相互作用的分子,可列举:dna、rna、蛋白质、多糖、低聚糖、单糖、脂质、脂肪酸、和它们的磷酸化物、烷基化物、糖加成物等、以及上述任意的复合物。另外,表达水平全面地表示该基因或表达产物的表达量和活性。
[0053]
在本发明的方法中,作为优选方案,作为生物试样使用ssl,在该情况下,分析ssl
中所含的rna的表达水平,具体而言,将rna通过逆转录转换为cdna后,测定该cdna或其扩增产物。
[0054]
从ssl中提取rna时,可以使用通常用于从生物试样提取或精制rna的方法,例如,苯酚/氯仿法、agpc(acid guanidinium thiocyanate-phenol-chloroform extraction,酸性硫氰酸胍-酚-氯仿提取)法或利用trizol(注册商标)、rneasy(注册商标)、qiazol(注册商标)等色谱柱的方法、使用包被有硅胶的特殊磁性体粒子的方法、使用固相载体可逆化固定(solid phase reversible immobilization)磁性体粒子的方法、利用isogen等市售rna提取试剂进行提取的方法等。
[0055]
该逆转录可以使用靶向待分析的特定rna的引物,为了进行更全面的核酸保存和分析,更优选使用随机引物。该逆转录能够使用通常的逆转录酶或逆转录试剂盒。优选使用准确性和效率较高的逆转录酶或逆转录试剂盒,作为其的示例,优选使用:m-mlv逆转录酶(reverse transcriptase)和其变体、或者市售的逆转录酶或逆转录试剂盒,例如primescript(注册商标)逆转录酶系列(takara bio公司)、superscript(注册商标)逆转录酶系列(thermo scientific社)等。优选使用superscript(注册商标)iii逆转录酶、superscript(注册商标)vilo cdna synthesis kit(均由thermo scientific公司制)等。
[0056]
优选地,该逆转录中的延伸反应将温度调节为优选42℃
±
1℃、更优选42℃
±
0.5℃、进一步优选42℃
±
0.25℃,同时,将反应时间调节为优选60分钟以上、更优选80~120分钟。
[0057]
作为测定表达水平的方法,在以rna、cdna或dna为对象的情况下,可以选自下述方法:以与它们杂交的dna为引物的pcr法、实时rt-pcr法、多重pcr、smartamp法、lamp法等所代表的核酸扩增法、将与它们杂交的核酸用作探针的杂交法(dna芯片、dna微阵列、斑点印迹杂交、狭线印迹杂交、northern印迹杂交等)、确定碱基序列的方法(测序)、或以上的组合方法。
[0058]
在pcr中,可以使用以要分析的特定的dna为靶标的引物对,只扩增该特定的1种dna,也可以使用多个引物对,同时扩增多个特定的dna。优选该pcr为多重pcr。多重pcr是通过在pcr反应系统中同时使用多个引物对而同时扩增多个基因区域的方法。多重pcr可以使用市售的试剂盒(例如ion ampliseq转录组人基因表达试剂盒(transcriptome human gene expression kit);life technologies japan株式会社等)实施。
[0059]
该pcr的退火和延伸反应的温度因依赖于所使用的引物而不能一概而论,在使用上述的多重pcr试剂盒的情况下,优选为62℃
±
1℃,更优选为62℃
±
0.5℃,进一步优选为62℃
±
0.25℃。因此,在该pcr中,优选退火和延伸反应在1个步骤中进行。该退火和延伸反应的步骤的时间可以根据所扩增的dna的大小等来调整,优选为14~18分钟。该pcr中的变性反应的条件可以根据所扩增的dna来调整,优选以95~99℃进行10~60秒。上述那样的温度和时间条件下的逆转录和pcr可以使用常用于pcr的热循环仪实施。
[0060]
利用该pcr得到的反应产物的精制优选通过反应产物的粒析(sizeseparation)进行。利用粒析,能够将目标的pcr反应产物与pcr反应液中所含的引物和其它的杂质分离。dna的粒析例如可以利用粒析柱、粒析芯片、能够用于粒析的磁珠等进行。作为能够用于粒析的磁珠的优选的例子,可以列举ampurexp等的固相可逆化固定(solid phase reversible immobilization,spri)磁珠。
[0061]
对于精制后的pcr反应产物,为了进行之后的定量分析,也可以进行必要的更进一步的处理。例如为了进行dna的测序,可以将精制后的pcr反应产物在适当的缓冲液溶液中进行制备,或切断pcr扩增得到的dna所含的pcr引物区域,或向扩增得到的dna中进一步添加接头序列。例如可以将精制后的pcr反应产物在缓冲液溶液中进行制备,对扩增dna进行pcr引物序列的除去和接头连接,使所得到的反应产物根据需要扩增,制作用于定量分析的文库(library)。例如可以使用superscript(注册商标)vilo cdna合成试剂盒(life technologies japan株式会社)所附带的5
×
vilo rt reaction mix和ion ampliseq转录组人基因表达试剂盒(life technologies japan株式会社)所附带的的5
×
ion ampliseq hifi mix和ion ampliseq transcriptome human gene expression core panel,基于各试剂盒所带的操作规程进行这些操作。
[0062]
在利用northern印迹杂交法测定靶标基因或来自其的核酸的表达量的情况下,例如,首先利用放射性同位素、荧光物质等对探针dna进行标记,接着,使所得到的标记dna与按照通常方法转移至尼龙膜等的来自生物体试样的rna杂交。之后,检测来自标记物的信号,由此能够对所形成的标记dna与rna的双链进行测定。
[0063]
在利用rt-pcr法测定靶标基因或来自其的核酸的表达量的情况下,例如,首先按照通常方法由来自生物体试样的rna制备cdna,以其为模板,使以本发明的靶标基因能够扩增的方式制备的一对引物(与上述cdna(-链)结合的正链,与+链结合的反链)与其杂交。之后,按照通常方法进行pcr法,对所得到的扩增双链dna进行检测。在扩增得到的双链dna的检测中,可以采用对通过使用预先用ri、荧光物质等标记过的引物进行上述pcr而产生的标记双链dna进行检测的方法等。
[0064]
在利用dna微阵列测定靶标基因或来自其的核酸的表达量的情况下,例如将本发明的来自靶标基因的核酸(cdna或dna)的至少1种固定在支承体上,使用所得到的阵列,使由mrna制备的标记化cdna或crna结合在微阵列上,检测微阵列上的标记,由此测定mrna的表达量。
[0065]
作为上述阵列所固定的核酸,只要是在严格的条件下特异性(即实质上只是目标核酸)地进行杂交的核酸即可,例如可以为具有本发明的靶标基因的全部序列的核酸,也可以为由部分序列构成的核酸。其中,“部分序列”可以列举至少由15~25碱基构成的核酸。其中,严格的条件通常可以列举“1
×
ssc、0.1%sds、37℃”左右的清洗条件,作为更严格的杂交条件,可以列举“0.5
×
ssc、0.1%sds、42℃”左右,作为进一步严格的杂交条件,可以列举“0.1
×
ssc、0.1%sds、65℃”左右的条件。杂交条件记载于j.sambrook et al.,molecular cloning:a laboratory manual,thrd edition,cold spring harbor laboratory press(2001)等中。
[0066]
在利用测序测定靶标基因或来自其的核酸的表达量的情况下,例如可以列举使用下一代测序仪(例如ion s5/xl系统、life technologies japan株式会社)进行分析。基于测序中制作的读取数(read count),能够对rna表达进行定量。
[0067]
上述测定所使用的探针或引物、即本发明的用于特异性地识别并扩增靶标基因或来自其的核酸的引物、或用于特异性地检测该rna或来自其的核酸的探针与其相当,它们可以基于构成该靶标基因的碱基序列进行设计。其中,“特异性识别”是指,例如在northern印迹法中,实质上只能够检测本发明的靶标基因或来自其的核酸,另外,例如在rt-pcr法中,
如实质上只扩增该核酸那样,能够判断该检测物或产物为该基因或来自其的核酸。
[0068]
具体而言,能够利用含有与本发明的由构成靶标基因的碱基序列构成的dna或其互补链互补的一定数量的核苷酸的寡核苷酸。其中,“互补链”是指与由a:t(rna时为u)、g:c的碱基对构成的双链dna的一条链相对应的另一条链。另外,“互补”并不限于在该一定数量的连续的核苷酸区域内完全是互补序列的情况,只要具有优选80%以上、更优选90%以上、进一步优选95%以上的碱基序列上的同一性即可。碱基序列的同一性可以利用上述blast等算法进行确定。
[0069]
在这样的寡核苷酸作为引物使用的情况下,只要是特异性退火和链增长即可,通常可以列举具有例如10个碱基以上、优选15个碱基以上、更优选20个碱基以上并且例如100个碱基以下、优选50个碱基以下、更优选35个碱基以下的链长。
[0070]
另外,在作为探针使用的情况下,只要是特异性杂交即可,可以使用具有本发明的由构成靶标基因的碱基序列构成的dna(或其互补链)的至少部分或全部的序列、例如10个碱基以上、优选15个碱基以上并且例如100个碱基以下、优选50个碱基以下、更优选25个碱基以下的链长的寡核苷酸。
[0071]
另外,其中,“寡核苷酸”可以为dna或rna,既可以是合成的,也可以是天然的。或者,杂交所使用的探针通常使用标记过的寡核苷酸。
[0072]
另外,在测定本发明的靶标基因的翻译产物(蛋白质)、与该蛋白质发生相互作用的分子、与rna发生相互作用的分子、或与dna发生相互作用的分子的情况下,可以使用蛋白芯片分析、免疫测定法(例如elisa等)、质谱分析(例如lc-ms/ms、maldi-tof/ms)、单杂交法(pnas 100,12271-12276(2003))和双杂交法(biol.reprod.58,302-311(1998))这样的方法,并且可以根据对象适当选择。
[0073]
例如在使用蛋白质作为测定对象的情况下,通过以下的方式实施:使与本发明的表达产物相对应的抗体与生物体试样接触,对与该抗体结合的试样中的蛋白质进行检测,测定其水平。例如利用蛋白质印迹法时,可以如下所述地进行:作为一次抗体,使用上述的抗体后,作为二次抗体,使用用放射性同位素、荧光物质或酶等标记了的与一次抗体结合的抗体,对其一次抗体进行标记,利用放射线测定器、荧光检测器等测定来自这些标记物质的信号。
[0074]
另外,与上述翻译产物相对的抗体可以为多克隆抗体,也可以为单克隆抗体。这些抗体可以按照公知的方法进行制造。具体而言,使用按照通常方法在大肠杆菌等中表达并精制得到的蛋白质,或者按照通常方法合成该蛋白质的部分多肽,使家兔等非人动物免疫后,能够从该免疫动物的血清中按照通常方法得到多克隆抗体。
[0075]
另一方面,利用按照通常方法在大肠杆菌等中表达并精制得到的蛋白质或该蛋白质的部分多肽,使小鼠等非人动物免疫后,将所得到的脾细胞和骨髓瘤细胞进行细胞融合,能够从所制备的杂交瘤细胞中得到单克隆抗体。另外,单克隆抗体也可以通过噬菌体展示来制作(griffiths,a.d.;duncan,a.r.,current opinion in biotechnology,volume 9,number 1,february 1998,pp.102-108(7))。
[0076]
这样,能够测定从被检验者采集的生物体试样中的本发明的靶标基因或其表达产物的表达水平,并且基于该表达水平能够检测帕金森氏病。在一个实施方式中,检测具体通过将所测得的本发明的靶标基因或其表达产物的表达水平与对照水平进行比较而进行。
[0077]
在通过测序对多种靶标基因的表达水平进行分析的情况下,如上所述,作为指标,优选使用作为表达量数据的读取计数值(read count值)、对于该读取计数值校正了样品间的总读取数的差异而得到的rpm值、将该rpm值转换成以2为底的对数值的值(log2rpm值)或利用deseq2校正得到的计数值(normalized count值,标准化计数值)或加上整数1后以2为底的对数值(log2(count+1)值)。
[0078]
另外,作为rna-seq的定量值,也可以是一般的利用fragments per kilobase of exon per million reads mapped(fpkm)、reads per kilobase of exon per million reads mapped(rpkm)、transcripts per million(tpm)等算出的值。还可以为利用微阵列法得到的信号值和其校正值。另外,在通过rt-pcr等只对特定的靶标基因进行表达水平的分析的情况下,优选将对象基因的表达量转换成以管家基因的表达量为基准的相对表达量(相对定量)后再进行分析的方法、或使用包含靶标基因区域的质粒对绝对复制数进行定量(绝对定量)并分析的方法。也可以为利用数字pcr法得到的复制数。
[0079]
其中,作为“对照水平”,例如可以列举健康者的该靶标基因或其表达产物的表达水平。健康者的表达水平也可以为从健康者群体中测得的该基因或其表达产物的表达水平的统计值(例如平均值等)。在靶标基因为多种的情况下,优选对于各基因或其表达产物,求出基准表达水平。
[0080]
另外,本发明中,帕金森氏病也可以通过本发明的靶标基因或其表达产物的表达水平的上升/减少来检测。在这种情况下,来自被检验者的生物体试样中的靶标基因或其表达产物的表达水平与各基因或其表达产物的截止值(参照值)进行比较。关于参照值,只要预先取得健康者的该靶标基因或其表达产物的表达水平作为基准数据,基于以其为基础的表达水平的平均值或标准偏差等统计数值,适当确定即可。
[0081]
并且,可以利用源自帕金森氏病患者的靶标基因或其表达产物的表达水平和源自健康者的靶标基因或其表达产物的表达水平的测定值,构建区分帕金森氏病患者和健康者的判别公式(预测模型),并利用该判别公式来检测帕金森氏病。即,将来自帕金森氏病患者的靶标基因或其表达产物的表达水平和源自健康者的靶标基因或其表达产物的表达水平的测定值作为训练样品,构建区分帕金森氏病患者和健康者的判别公式(预测模型),并基于该判别公式求得判别帕金森氏病患者和健康者的截止值(参照值)。需要说明的是,建立判别公式时,可以通过主成分分析(pca)进行降维,并将主要成分作为说明变量。
[0082]
接着,同样由从被检验者采集的生物试样测定靶标基因或其表达产物的水平,将得到的测定值代入该判别公式,将根据该判别公式得到的结果与截止值比较,由此能够评价被检验者存在或不存在帕金森氏病。
[0083]
其中,作为构建判别式的算法,可以利用机械学习所使用的算法等公知的算法。作为机械学习算法的例子,可以列举随机森林(random forest)、线性核的支持向量机(svm linear)、rbf核的支持向量机(svm rbf)、神经网络(nerural net)、一般线性模型(generalized linear model)、正则化线性判别分析(regularized linear discriminant analysis)、正则化逻辑回归(regularized logistic regression)等。可以向所构建的预测模型输入检验用的数据,算出预测值,选取该预测值与实测值最适合的模型、例如准确率(accuracy)最大的模型作为最佳预测模型。另外,可以根据预测值和实测值计算检测率(recall)、精度(precision)和作为它们的调和平均的f值,选取其f值最大的模型作为最佳
预测模型。
[0084]
截止值(参照值)的确定方法没有特别限制,可以按照公知的方法进行确定。例如,可以利用使用判别式所制作的roc(receiver operating characteristic curve、受试者操作特征曲线)曲线求出。在roc曲线中,在纵轴绘制阳性患者中出现阳性结果的概率(灵敏度),在横轴绘制1减去阴性患者中出现阴性结果的概率(特异度)而得到的值(假阳性率)。关于roc曲线所示的“真阳性(灵敏度)”和“假阳性(1-特异度)”,可以将“真阳性(灵敏度)
”‑“
假阳性(1-特异度)”最大的值(约登指数,youden index)作为截止值(参照值)。
[0085]
如后述的实施例所示,将根据表a所示的靶标基因(33种基因或选自其中的四种基因)的表达量的数据(log2rpm值)得到的各主成分的值作为说明变量,以健康者和帕金森氏病患者作为目标变量,使用机器学习算法构建预测模型之后,可以通过使用snora16a、snora24、snora50、rexo1l2p这四种的模型来预测帕金森氏病。另外还表明,也可以通过使用33种的模型来高精度地预测帕金森氏病。
[0086]
因此,在建立区分上述帕金森氏病患者群和健康者群的判别公式的情况下,作为靶标基因,除snora16a、snora24、snora50和rexo1l2p这四种靶标基因之外,适当添加选自表a所示的除以上四种靶标基因之外的29种基因的至少一种基因或其表达产物的表达数据,优选基于下面所示的表8所示的变量重要度,添加适当数量变量重要度较高的基因,能够建立显示较高检测率和精度的判别公式,从而可以更高精度地检测帕金森氏病。具体而言,优选egr2、rhoa、ccni、rnasek、csf2rb、serp1、ankrd12、slc25a3这8种,优选再加入cd83、cxcr4、itgax、uqcrh这4种后的12种,优选再加入kcnq1ot1、ccl3、c10orf116、serpinb4、lce3d、cnfn这6种后的18种,优选再加入全部29种。
[0087]
或者,作为靶标基因,除snora16a、snora24、snora50和rexo1l2p这四种靶标基因之外,还可以适当地加入至少一个除snora24之外的基因或其表达产物的表达数据,这些基因或表达产物选自下表c所示的11种基因,这些基因在前面的表a和表b中都显示为表达变动基因。
[0088]
[表c]
[0089][0090]
并且,在建立区分帕金森氏病患者群和健康者群的判别公式的情况下,所用的靶标基因也可以使用选自表b所示的基因的至少一种基因或其表达产物的表达数据,优选包含snora24,并优选使用选自此外的至少一种基因,更优选使用表c所示的基因或其表达产物的表达数据,进一步优选使用表b所示的全部基因或其表达产物的表达数据。
[0091]
本发明的用于检测帕金森氏病的检查用试剂盒包括用于测定从患者分离出的生物试样中本发明的靶标基因或其表达产物的表达水平的检查试剂。具体而言,可列举:包含与本发明的靶标基因或源自其的核酸特异性结合(杂交)的寡核苷酸(例如,用于pcr的引物)的、用于核酸扩增、杂交的试剂;或者包含识别本发明的靶标基因的表达产物(蛋白质)的抗体的、用于免疫学测定的试剂等。如上所述,该试剂盒中所包含的寡核苷酸、抗体等可以通过公知的方法获得。
[0092]
另外,该检查用试剂盒除了含有上述抗体以外,还可以含有标记试剂、缓冲液、显色底物、二次抗体、阻滞剂、试验所需的器具以及对照、用于采集生物试样的用具(例如,用于采集ssl的吸油膜等)等。
[0093]
下面示出本发明的方案和优选实施方案。
[0094]
《1》一种被检验者的帕金森氏病的检测方法,其包括:
[0095]
对于从被检验者采集的生物试样,测定选自snora16a、snora24、snora50和rexo1l2p这四种基因群中的至少一种基因或其表达产物的表达水平的工序。
[0096]
《2》根据《1》的帕金森氏病的检测方法,其中,至少包括测定snora24基因或其表达产物的表达水平。
[0097]
《3》根据《1》或《2》的方法,其中,基因或其表达产物的表达水平的测定为mrna的表达量的测定。
[0098]
《4》根据《1》~《3》中任一项的方法,其中,基因或其表达产物为所述被检验者的皮肤表层脂质中所含的rna。
[0099]
《5》根据《1》~《4》中任一项的方法,其中,将表达水平的测定值与所述各基因或其表达产物的参照值比较,评价存在或不存在帕金森氏病。
[0100]
《6》根据《1》~《4》中任一项的方法,其中,将来自帕金森氏病患者的所述基因或其表达产物的表达水平和源自健康者的所述基因或其表达产物的表达水平的测定值作为训练样品,制作能够区分帕金森氏病患者和健康者的判别式,
[0101]
将由从被检验者采集的生物试样得到的所述基因或其表达产物的表达水平的测定值代入该判别公式,将得到的结果与参照值比较,以评价被检验者存在或不存在帕金森氏病。
[0102]
《7》根据《6》的方法,其中,测定所述四种基因群中的所有基因或其表达产物的表达水平。
[0103]
《8》根据《6》或《7》的方法,其中,除了测定选自所述四种基因群中的至少一种基因之外,还测定选自下述29种基因群中的至少一种基因或其表达产物的表达水平:
[0104]
ankrd12、c10orf116、ccl3、ccni、cd83、cnfn、cnn2、csf2rb、cxcr4、egr2、emp1、itgax、kcnq1ot1、lce3d、litaf、ndufa4l2、ndufs5、polr2l、rhoa、rnasek、rpl7a、rps26、serinc1、serp1、serpinb4、slc25a3、snrpg、srrm2、uqcrh。
[0105]
《9》根据《8》的方法,其中,除了测定选自所述四种基因群中的至少一种基因之外,还测定选自下述10种基因群中的至少一种基因或其表达产物的表达水平:
[0106]
ccl3、ccni、cxcr4、egr2、emp1、polr2l、rhoa、rnasek、serinc1、serpinb4。
[0107]
《10》根据《6》或《7》的方法,其中,除了测定选自所述四种基因群中的至少一种基因之外,还测定选自下述16种基因群中的至少一种基因或其表达产物的表达水平:
[0108]
anxa1、aqp3、atp6v0c、bhlhe40、ccl3、ccni、cxcr4、egr2、emp1、gabarapl1、krt16、polr2l、rhoa、rnasek、serinc1、serpinb4。
[0109]
《11》根据《6》或《7》的方法,其中,除了测定选自所述四种基因群中的至少一种基因之外,还测定选自下述表3-1~3-4和下述表6-1~6-2所示的基因群的至少一种基因(其中,所述四种基因除外。)或其表达产物的表达水平。
[0110]
《12》根据《6》或《7》的方法,其中,除了测定选自所述四种基因群中的至少一种基因之外,还测定选自下述表1-1~1-27所示的1005种和下述表4-1~4-20所示的725种中除所述四种基因之外的基因群的至少一种基因或其表达产物的表达水平。
[0111]
《13》一种《1》~《10》中任一项的方法中使用的用于检测帕金森氏病的检查用试剂盒,其中,含有与所述基因或源自其的核酸特异性杂交的寡核苷酸或识别所述基因的表达产物的抗体。
[0112]
《14》一种选自下述表3-1~3-4和下述表6-1~6-2所示的基因群的至少一种基因或其表达产物作为帕金森氏病的检测标记物的用途。
[0113]
《15》一种选自snora16a、snora24、snora50和rexo1l2p这四种基因群中的至少一种基因或其表达产物作为帕金森氏病的检测标记物的用途。
[0114]
《16》一种帕金森氏病的检测标记物,其由选自下述表3-1~3-4和下述表6-1~6-2所示的基因群的至少一种基因或其表达产物构成。
[0115]
《17》根据《16》的帕金森氏病的检测标记物,其中,所述检测标记物由选自snora16a、snora24、snora50和rexo1l2p这四种基因群中的至少一种基因或其表达产物构
成。
[0116]
实施例
[0117]
下面,基于实施例对本发明进行更详细说明,但本发明并不限定于此。
[0118]
实施例1使用从ssl提取的rna检测帕金森氏病
[0119]
1)ssl采集
[0120]
进行以下的试验1和试验2各两次。
[0121]
试验1:将15名健康者(40~89岁男女)和15名帕金森氏病患者(pd)(40~89岁男女)作为被检验者。
[0122]
试验2:将50名健康者(40~89岁男)和50名pd(40~89岁男)作为被检验者。
[0123]
pd提前由脑神经内科医诊断为帕金森氏病(hoehn&yahri期或ii期)。使用吸油膜(5
×
8cm,聚丙烯制,3m公司)从各被检验者的全脸回收皮脂之后,将该吸油膜转移至小瓶中,以-80℃保存至用于rna提取,期间约1个月。
[0124]
2)rna制备和测序
[0125]
将上述1)的吸油膜切成适当的大小,使用qiazol裂解试剂(lysis reagent)(qiagen),按照附带的操作规程提取rna。基于所提取的rna,使用superscript vilo cdna合成试剂盒(synthesis kit)(life technologies japan株式会社),以42℃进行90分钟逆转录,进行cdna的合成。逆转录反应的引物使用试剂盒所附带的随机引物。利用多重pcr,由所得到的cdna制作包含来自20802种基因的dna的文库。使用ion ampliseq转录组人基因表达试剂盒(life technologies japan株式会社),在[99℃、2分钟
→
(99℃、15秒
→
62℃、16分钟)
×
20次循环
→
4℃、保持(hold)]的条件下进行多重pcr。所得到的pcr产物用ampure xp(beckman coulter株式会社)精制后,进行缓冲液的重构、引物序列的消化、接头连接和精制、扩增,制作文库。将所制作的文库载入ion 540chip中,使用ion s5/xl系统(life technologies japan株式会社)进行测序。
[0126]
3)数据分析
[0127]
i)rna表达分析1
[0128]
在上述2)中测得的源自被检验者的rna的表达量的数据(读取计数值)中,将读取计数小于10的数据作为缺失值处理,将表达量的数据转换为经样品间的总读取数的差异校正后的rpm值,然后,使用singular value decomposition(svd)imputation方式对缺失值进行填充。其中,只将在全部样品的80%以上的样品中得到了不是缺失值的表达量数据的基因用于以下的分析。分析时,为了使符合负二项分布的rpm值近似正态分布,使用将读取计数的rpm值转换成以2为底的对数值的rpm值(log2rpm值)。
[0129]
基于上述得到的来自健康者和pd的ssl的rna表达量(log2rpm值),鉴定与健康者相比pd中student’s t-test的p值为0.05以下的表达变动rna。试验1中,与健康者相比pd中111种rna表达上升(表1-1~1-3),68种表达下降(表1-4~1-5)。另一方面,试验2中,565种rna表达上升(表1-6~1-19),294种表达下降(表1-20~1-27)。以上试验1和试验2中均表达上升的rna有18种,均表达下降的rna有15种(表中粗体表示的基因)。
[0130]
[表1-1]
[0131][0132]
[表1-2]
[0133][0134]
[表1-3]
[0135][0136]
[表1-4]
[0137][0138]
[表1-5]
[0139][0140]
[表1-6]
[0141][0142]
[表1-7]
[0143][0144]
[表1-8]
[0145]
试验2creld20.6850454830.009010899up试验2cript0.6230886610.000802774up试验2crnn1.4018841120.001214875up
试验2cst60.5899665310.016466862up试验2csta0.7842551160.002502729up试验2cul4a0.4895584050.013487958up试验2cuta0.5859871270.001016471up试验2cyb5a0.6419394070.009668198up试验2cyb5b0.5446801180.005232354up试验2dancr0.431263360.041709971up试验2dcaf120.5454546480.011615314up试验2ddrgk10.3723477480.044309125up试验2ddt0.4727600040.010925051up试验2degs10.5459846890.037107489up试验2dennd2c0.5022887920.047224257up试验2dhps0.5188456830.012866877up试验2dhx290.6821061050.006066935up试验2dhx320.5065092590.033864568up试验2dhx400.3965736040.015540946up试验2dnaja10.2525523720.019713754up试验2dnaja40.4830453510.044641278up试验2dnajc130.4703629360.029093404up试验2dnajc150.4692115630.013979287up试验2dnajc210.4527090720.022814459up试验2dnajc70.3196763870.022220543up试验2dnajc90.5751269540.012161694up试验2dock60.5457197830.03676478up试验2dock90.6217579860.012261459up试验2dph11.1580398186.72e-05up试验2dpy300.3983177570.02828779up试验2drg10.5812536410.004984247up试验2dsg10.5673992180.037732972up试验2dusp110.4733256180.006136292up试验2dym0.8161785130.00274631up试验2dync1li10.5832428580.0067388up试验2dynll10.3746364060.035010075up试验2dynlrb10.3300536740.027194242up试验2echs10.3742192630.043974114up试验2efnb20.6610196930.019887237up试验2eif1ax0.6005238640.00135969up试验2eif2s20.6669625340.008564954up
[0146]
[表1-9]
[0147][0148]
[表1-10]
[0149]
试验2gtf2a20.3303726470.040423351up试验2gtf2e20.5348550780.004830931up试验2gtf2h50.6118792080.000741758up
试验2gtf3c50.3889856630.032287492up试验2gtf3c60.5628512940.008550842up试验2h1fx0.382898240.039887459up试验2hadh0.5968863840.031698277up试验2hbegf0.358247570.029353225up试验2hdac10.4129968260.019794177up试验2hddc20.4810288650.038549638up试验2heatr5a0.5416757690.004141004up试验2hexb0.483266380.016112958up试验2hibadh0.4914099110.028149152up试验2hibch0.5889438010.025448145up试验2hist1h1e0.4363344760.040849694up试验2hist1h2ae0.4720221850.032486571up试验2hist1h2ag0.5549521960.026912916up试验2hist1h2ai0.537486170.034833553up试验2hist1h2am0.5054659220.015356542up试验2hist1h2bn0.5471503640.019828836up试验2hist1h3b1.0614769480.000400823up试验2hist1h3i0.6013093930.011107396up试验2hist1h4b0.8395444680.00079634up试验2hist1h4e0.7783350850.00020329up试验2hist1h4f0.5511757910.032237462up试验2hist1h4h0.7150817020.000190121up试验2hmox20.3751245920.032277484up试验2hnrnpa00.430122240.023849018up试验2homer10.5720561220.027336542up试验2hook10.6896474120.000609804up试验2hpgd0.5314616620.034525209up试验2hrsp120.7481638860.00429738up试验2hsd17b100.5255804310.005390788up试验2hsp90aa10.4506715140.012853222up试验2hspd10.3535242680.038337878up试验2hypk0.4955087320.000812946up试验2ide0.5614864040.030266606up试验2idh3a0.7157414830.000740982up试验2ifi271.0887182710.000166105up试验2il320.6354646480.022927371up试验2il36a1.1935571690.000147742up
[0150]
[表1-11]
[0151][0152]
[表1-12]
[0153]
试验2lce3e0.8365639720.000810209up试验2lcmt10.6018428690.025859542up试验2lcn20.733257720.003321up
试验2lemd30.4409940150.016865308up试验2leprotl10.3778795150.038317178up试验2linc006750.5735233060.034073972up试验2llph0.4849982990.007188702up试验2lmbr10.6650833530.00191151up试验2lnx10.9527134430.000293549up试验2loc1005057380.4770537450.024409up试验2loc5506430.6346724370.002533558up试验2loc6468620.7475721650.025405318up试验2lrba0.5292803510.038783597up试验2lrrc150.9064566720.002321669up试验2lsm100.5085072420.013416263up试验2lsm20.6758782850.004242908up试验2lsm70.5706177640.005193872up试验2ltf0.7170426620.012011178up试验2ly6d0.6389092470.038220601up试验2lynx11.0060122620.002091327up试验2mafa0.6465098390.018661569up试验2mal1.1573936950.00203966up试验2mall1.0828535460.000258882up试验2maoa0.4894522890.017793881up试验2map4k30.5674995350.022681249up试验2map70.5977830190.034525239up试验2mccc10.6777830160.008565533up试验2mcts10.4994486750.013734219up试验2micalcl0.5191282130.00748038up试验2mnf10.4488900250.045325106up试验2mphosph60.4314639620.044290704up试验2mpv170.4620107920.022209637up试验2mrpl110.4441699530.041198724up试验2mrpl120.4592607380.023664309up试验2mrpl240.494230420.034251269up试验2mrpl320.5705275450.004685957up试验2mrpl470.5222501560.004855696up试验2mrps110.7235722330.000171238up试验2mrps18b0.6066422850.003402311up试验2mrps240.4246101030.027109976up试验2mt1x0.9131478160.000517578up
[0154]
[表1-13]
[0155][0156]
[表1-14]
[0157][0158]
[表1-15]
[0159][0160]
[表1-16]
[0161][0162]
[表1-17]
[0163][0164]
[表1-18]
[0165][0166]
[表1-19]
[0167][0168]
[表1-20]
[0169][0170]
[表1-21]
[0171][0172]
[表1-22]
[0173]
试验2fam53c-0.4146466520.043215387down试验2fbxo11-0.5875676860.033095048down试验2fcgrt-0.5931040230.019455764down
试验2fgr-0.5736045180.025328892down试验2flna-0.5039784570.020310777down试验2fnip1-0.5592599470.024530856down试验2fosb-1.0916223630.000150044down试验2fosl2-0.7415466330.000377548down试验2foxn3-0.3546371740.046745405down试验2foxo4-0.46672230.043783003down试验2furin-0.4591057150.001341881down试验2fzr1-0.3641476220.028337243down试验2gabarapl1-0.555975230.006898537down试验2gadd45b-0.4704815270.001471104down试验2gapvd1-0.4103698440.017202036down试验2gatad2a-0.4270737710.023639602down试验2gga1-0.3964271180.011108895down试验2gla-0.4323861630.046129953down试验2gmip-0.4392554430.025650159down试验2gnb1-0.318475510.015581144down试验2gnb2-0.3197211490.049773636down试验2gpr108-0.4419033220.042000281down试验2gpx1-0.4190154760.012872784down试验2gramd1a-0.9322636431.44e-05down试验2grk6-0.6546154240.008370268down试验2grn-0.5515009850.014350263down试验2gtpbp1-0.4035032040.015278622down试验2hexim1-0.3659865020.049415504down试验2hipk3-0.4632026590.018014847down试验2hla.a-1.2367924060.020861464down试验2hlx-0.6243230890.020911612down试验2hspa4-0.596232710.022837699down试验2ids-0.2408814110.028746962down试验2ier3-0.2875208380.017217201down试验2impdh1-0.6104056950.010620152down试验2ino80d-0.375473780.007394184down试验2inpp5k-0.4233725010.028673174down试验2iqsec1-0.3904271360.017062257down试验2irak2-0.6581698820.010698571down试验2irs2-0.4204968940.042158917down试验2iscu-0.2878691250.027433296down
[0174]
[表1-23]
[0175][0176]
[表1-24]
[0177]
试验2nab1-0.416318670.034705696down试验2nagk-0.4009389580.039418511down试验2ncf1b-0.6329881610.032521317down
试验2ncf1c-0.5649584340.023648103down试验2ncoa1-0.352687490.025504935down试验2nfkb2-0.6862259060.006490871down试验2nfkbib-0.4173753310.020054211down试验2nfkbid-0.5790202160.039512351down试验2ninj1-0.6665213990.007758421down试验2nlrc5-0.5182899680.04615466down试验2notch2nl-0.3805269310.002073988down试验2nrip1-1.3229583780.002632999down试验2numb-0.4948707670.00364152down试验2ogfr-0.4576660830.021935407down试验2os9-0.4726493910.045293803down试验2pan3-0.4907597140.037403044down试验2patl1-0.4254941610.039431793down试验2pcbp1-0.1768490950.0308842down试验2pdpk1-0.3517648480.030720043down试验2per1-0.5202149270.038720114down试验2pfkfb3-0.3719379970.012048698down试验2phf1-0.5094904180.018640047down试验2pik3ap1-0.6304453340.004184868down试验2pik3r5-0.6124464750.004720621down试验2pim3-0.4675771740.002878904down试验2pitpna-0.4744704220.00241514down试验2plau-0.650310110.029875395down试验2plekhb2-0.3055830540.044277802down试验2plekhm3-0.3687944160.029647876down试验2plin5-0.6769601810.015080446down试验2ppp1r15a-0.4180723370.005793369down试验2ppp1r18-0.5062619320.019385963down试验2ppp2r5c-0.4715076430.029204209down试验2ppp4r1-0.5786493710.006631286down试验2prr14-0.460517950.0377872down试验2prr24-0.394698830.038397986down试验2prrc2c-0.3832432670.047022553down试验2ptger4-0.4674315270.024894507down试验2ptk2b-0.4294048020.005990901down试验2pttg1ip-0.4815909330.044232468down试验2rab11fip1-0.224579180.041085596down
[0178]
[表1-25]
[0179][0180]
[表1-26]
[0181][0182]
[表1-27]
[0183]
试验2xpo6-0.5977093480.046102314down试验2ypel5-0.2875063770.038298209down试验2zc3h12a-0.5112174610.009065486down
试验2zfp36-0.4685061720.020859393down试验2zmiz1-0.6513370520.00341487down试验2znfx1-0.4477276120.044337198down试验2zzef1-0.3562474350.015261504down
[0184]
使用作为公共数据库的string,进行了利用基因本体(go)富集分析的生物过程(bp)的探索。其结果显示了,与pd患者中表达上升或者下降的基因群关联的kegg pathway在试验1中得到30个,试验2中得到39个,两个试验中均包含显示帕金森氏病的项目hsa05012(parkinson’s disease)(表2-1~2-2)。
[0185]
[表2-1]
[0186][0187]
[表2-2]
[0188][0189]
针对所述表1-1~表1-27所示的试验1或试验2中至少任意一项试验中发现表达变动的基因,就其与帕金森氏病的关联性对现有文献进行了确认,结果发现,试验1中发现表达变动的基因中表3-1中所示的21种和试验2中发现表达变动的基因中表3-2~3-4所示的92种目前尚未被报告有关它们与帕金森氏病的关联性,表明它们可以用作帕金森氏病的新型检测标记物。需要说明的是,表中粗体所表示的基因是试验1和试验2中共有的基因。
[0190]
[表3-1]
[0191][0192]
[表3-2]
[0193][0194]
[表3-3]
[0195][0196]
[表3-4]
[0197]
试验2ino80ddown试验2kiaa0232down试验2map7d1down试验2mllt6down
试验2ncf1bdown试验2prr24down试验2sde2down试验2sled1down试验2smg1p1down试验2tmem167bdown
[0198]
ii)rna表达分析2
[0199]
在上述2)中测得的源自被检验者的rna的表达量的数据(读取计数值)中,使用deseq2方法进行校正。其中,排除未检测到4161个以上基因的样品,仅将排除后的全部样品中被检验者的表达量的数据中90%以上的样品被检验者不是缺失值的表达量的数据的基因用于以下分析。分析中使用通过deseq2方法校正后的计数值(标准化计数值)。
[0200]
基于上述得到的来自健康者和pd的ssl的rna表达量(标准化计数值),鉴定与健康者相比pd中似然比检验的p值的校正值(fdr)为0.25以下的表达变动rna。试验1中,与健康者相比pd中74种rna表达上升(表4-1~4-2),209种表达下降(表4-3~4-8)。另一方面,试验2中,151种rna表达上升(表4-9~4-12),308种表达下降(表4-13~4-20)。以上试验1和试验2中均表达上升的rna有7种,均表达下降的rna有10种(表中粗体表示的基因)。
[0201]
[表4-1]
[0202][0203]
[表4-2]
[0204][0205]
[表4-3]
[0206][0207]
[表4-4]
[0208][0209]
[表4-5]
[0210]
试验1hbp1-1.6914818950.02450771down试验1helz-2.6898663660.003678702down试验1hif1a-1.2248277690.238174085down试验1hint1-1.4537626920.04751536down
试验1hint3-1.9471520320.140445468down试验1hist1h1e-1.6701406060.103600407down试验1hmgn1-2.0631656820.073126006down试验1hnrnpa2b1-1.2742699150.142104577down试验1hnrnpk-1.966404370.000238125down试验1hnrnpu-1.7037156060.009237092down试验1iars2-2.5025780810.04751536down试验1icam1-2.3831303110.162465282down试验1ide-1.8622232740.11590529down试验1ier3ip1-2.1290408870.191902648down试验1jak1-2.4784296770.04751536down试验1jmy-2.2634968730.155218477down试验1kat2b-1.5502569040.157035049down试验1kiaa1551-1.3676282590.228182221down试验1kif16b-1.7120883160.170079315down试验1klf10-2.5079208550.01186505down试验1klf3-2.6712240650.011682374down试验1lgalsl-1.8682460090.165136091down试验1march7-1.3581428670.242073043down试验1mbd2-1.9663851720.008794332down试验1mbd6-2.2431040330.157035049down试验1mdm2-2.1749801920.067390396down试验1med13l-1.639028930.104520242down试验1med19-3.5810059560.007535427down试验1mrpl15-2.3867658750.094888262down试验1napa-1.4201334420.067390396down试验1nr4a2-1.2567726970.231685231down试验1nrbf2-0.8719163250.124019241down试验1nrbp1-1.1225308810.242957593down试验1nsfp1-1.1670247180.104520242down试验1ogfrl1-1.4596131620.06499322down试验1p4hb-0.9388007960.184113444down试验1paip2-1.4844161160.04751536down试验1pdxk-1.2652832010.246917684down试验1pgk1-0.9211051780.135858235down试验1pgrmc2-2.3090100580.142104577down试验1phf20l1-2.0988093690.18841032down
[0211]
[表4-6]
[0212][0213]
[表4-7]
[0214]
试验1sertad2-2.0298305020.192395571down试验1set-1.9374447120.007533471down试验1sh3bgrl3-0.6633057350.067390396down试验1slmo2-1.813290580.137966894down
试验1sms-2.7045178020.045626018down试验1snap29-1.5900855810.16114262down试验1snora53-1.5865126850.15598759down试验1snx13-3.0044749610.000999599down试验1snx9-1.7799580510.028713164down试验1srek1ip1-1.8398555190.154290596down试验1srsf5-0.9841136660.13064644down试验1ssr2-1.7493372530.146923279down试验1ssu72-1.2453251920.027093818down试验1stk24-2.7349185840.000596053down试验1stt3b-1.9518002280.150544954down试验1taf10-1.324526470.094888262down试验1taok1-1.9273490240.030576015down试验1terf2ip-1.7928636030.084366841down试验1tlk2-2.6059067070.170106979down试验1tma7-1.3678951950.028713164down试验1tmem106b-1.868359980.247784004down试验1tmem127-1.1907037940.044486644down试验1tmem167b-1.7967139660.116792089down试验1tnfsf13b-1.788146540.131657412down试验1tpgs2-1.8098746690.11590529down试验1tram1-1.8395460050.092035865down试验1trip12-1.7254353130.025949689down试验1trpm7-2.0383577710.182595713down试验1tsg101-1.0052379890.209643258down试验1txnl1-1.5498712730.032012546down试验1ube2a-1.5928299160.088783965down试验1ube2b-1.4365133640.078520181down试验1ube2h-3.4058186370.004532329down试验1usmg5-1.0463901490.136226208down试验1usp22-1.1815074830.174760541down试验1usp53-3.7614886130.006574907down试验1usp6nl-1.791260360.192642777down试验1usp7-1.9937086290.079281961down试验1wipf1-2.7424650490.000134039down试验1wtap-1.4461703370.200942058down试验1xbp1-1.3261238650.14231193down
[0215]
[表4-8]
[0216]
试验1ywhaq-3.2301537290.000308536down
试验1zcrb1-2.4551395760.104520242down试验1zmat2-1.6355394880.104520242down试验1znf148-2.2375739810.088783965down
[0217]
[表4-9]
[0218][0219]
[表4-10]
[0220][0221]
[表4-11]
[0222]
试验2rpl110.2720493570.188973375up试验2rpl120.2722314470.203917134up试验2rpl13a0.3328828360.087496195up试验2rpl180.236319010.203917134up
试验2rpl210.2340572220.228358936up试验2rpl260.2751784880.203917134up试验2rpl270.253619320.203917134up试验2rpl27a0.2860640330.203917134up试验2rpl290.2273232010.202573111up试验2rpl30.2679374050.185751097up试验2rpl300.223031170.208405568up试验2rpl320.3666980610.05120094up试验2rpl350.3531611750.075426886up试验2rpl360.3218428620.084037369up试验2rpl36a0.3384768570.089104057up试验2rpl37a0.4296271490.035341619up试验2rpl380.3615051450.069516286up试验2rpl70.4077221450.013807457up试验2rpl7a0.38652740.033308231up试验2rplp00.4758366970.00985997up试验2rplp10.4662274570.019217132up试验2rplp20.3516413720.151691535up试验2rps100.2781009190.129200547up试验2rps120.5572117650.001079959up试验2rps150.3437735690.061406696up试验2rps15a0.2413595390.204723138up试验2rps180.5454563580.003707257up试验2rps190.3048564290.188426153up试验2rps210.2992950520.230255192up试验2rps260.4953755450.056702789up试验2rps280.3469427630.045753193up试验2rps30.4679017370.121272078up试验2rps4x0.4039821390.053047353up试验2rps50.3604494080.079217394up试验2rps60.3238630580.083321514up试验2rps80.2761548050.156533343up试验2s100a140.776723440.035341619up试验2s100a70.493515680.203917134up试验2s100a7a0.821074650.057323067up试验2s100a90.4152038980.204723138up试验2sbds0.4336291330.094970832up
[0223]
[表4-12]
[0224][0225]
[表4-13]
[0226][0227]
[表4-14]
[0228][0229]
[表4-15]
[0230][0231]
[表4-16]
[0232]
试验2grn-0.5357152530.191525939down试验2gtpbp1-0.463081940.148123279down试验2hdac7-0.4908420460.248266371down试验2hla-a-1.1048334250.203917134down
试验2hpcal1-0.4477926450.216748647down试验2hs3st6-0.5016631960.207332199down试验2hspa4-0.9065817830.092289404down试验2ids-0.2556741670.191525939down试验2ier3-0.4417385960.034346406down试验2impdh1-0.6187508530.202573111down试验2inpp5k-0.391525190.179929185down试验2irak2-0.9413100190.041102123down试验2irf1-0.6333287230.219872648down试验2itga5-0.6141005210.148123279down试验2itgax-0.5843823780.191525939down试验2itpk1-0.6816008430.167922188down试验2junb-0.4056331070.174126215down试验2kiaa0247-0.433540890.185751097down试验2kiaa0368-0.3868519050.149347833down试验2kiaa0494-0.3759613130.203917134down试验2kiaa1191-0.7595706470.07612678down试验2klf2-0.8296226720.095805056down试验2klf6-0.6152370690.032647959down试验2larp1-0.3716919280.191525939down试验2lgals3-0.336607040.228358936down试验2lilrb2-0.7132178290.141991209down试验2lilrb3-0.5862520860.203917134down试验2limk2-0.6938711450.19331656down试验2litaf-0.4725820530.129200547down试验2loc146880-0.5969533760.248133155down试验2loc729737-0.6418144220.204723138down试验2lpcat1-1.0108757420.013807457down试验2lpin1-0.5208062570.196547493down试验2lsp1-0.6695553470.066340963down试验2ltbr-0.7151044480.155367959down试验2maf1-0.4752688420.232304966down试验2map4k4-0.5171398430.204723138down试验2map7d1-0.4495599950.189823772down试验2mapkapk2-0.4759838180.191525939down试验2marcks-0.5644912320.18041519down试验2mboat7-0.7511602660.145937048down
[0233]
[表4-17]
[0234]
试验2mef2d-0.6264281060.045753193down
试验2megf9-0.3629997140.203917134down试验2mepce-0.8646152290.073987946down试验2metrnl-0.2815068630.183513502down试验2mgea5-0.3456550870.180032429down试验2mknk2-0.4103647190.126316838down试验2mlf2-0.3873930690.075659228down试验2mllt6-0.8824912180.041252415down试验2mmp25-0.7437211490.204723138down试验2msrb1-0.3847338340.160228921down试验2mthfs-0.578388180.191525939down试验2mtmr14-0.6903690510.156533343down试验2myo9b-0.6075026150.191525939down试验2naa50-0.4447309060.019217132down试验2nbeal2-0.5359654130.2421585down试验2ncf1b-0.9295174740.094970832down试验2nfkb2-0.9265777720.023042585down试验2nfkbia-0.4946042510.189823772down试验2nfkbib-0.5802666890.221139649down试验2nfkbid-0.8613159020.050427226down试验2nfkbie-0.7309711950.083321514down试验2ninj1-0.820508450.035341619down试验2nipbl-0.3908396960.188426153down试验2nlrc5-0.7943667430.185751097down试验2notch2nl-0.3343549650.094970832down试验2nr4a3-0.5891587210.249272492down试验2ntan1-0.6208257770.126316838down试验2ogdh-0.4090530890.156533343down试验2osm-0.6095225220.240103288down试验2p2ry4-0.8300416870.204723138down试验2pacsin2-0.4303592530.196505966down试验2pdhx-0.6814064830.24513842down试验2pdlim7-0.6462657740.236147723down试验2per1-0.7150315620.129200547down试验2pfkl-0.4729249490.189087808down试验2phf1-0.6160909550.203917134down试验2pik3ap1-0.7549381390.131526721down试验2pik3r5-0.6998132380.141991209down试验2pilra-0.5782936450.221139649down试验2pim2-0.5438632090.248133155down
试验2pim3-0.6029223870.032647959down
[0235]
[表4-18]
[0236][0237]
[表4-19]
[0238]
试验2shisa5-0.703130860.200987694down试验2shkbp1-0.6100472610.203917134down
试验2sirpa-0.5120225060.03912317down试验2slc11a1-0.6563086160.199786172down试验2slc15a3-0.6372806480.205001407down试验2slc15a4-0.7162058450.130446509down试验2slc31a1-0.4516798470.159048563down试验2slc3a2-0.8285535430.079852349down试验2slc41a1-1.0107837730.079217394down试验2slc43a2-0.9401369090.032647959down试验2slc43a3-0.6906134120.225295536down试验2slc45a4-0.7490479690.093863146down试验2slc6a6-0.7584008640.073352416down试验2smg1p1-0.6932269770.094970832down试验2snora8-0.5198458060.211019733down试验2sort1-0.6230315990.050123349down试验2sphk1-1.1084245010.016355202down试验2spint2-0.3396701230.203917134down试验2sqstm1-0.2768872280.240103288down试验2srebf2-1.2274415480.007293838down试验2srp54-0.3197710580.248266371down试验2srrm2-0.4183038810.180898511down试验2srxn1-0.5829797760.038524973down试验2stk40-0.4880017790.0381434down试验2stx11-0.6587240540.205349428down试验2stx6-0.5136191310.230255192down试验2stxbp2-0.5085856930.130446509down试验2tagap-0.8014152080.118903158down试验2tap1-0.6541260470.218710004down试验2tcf25-0.4610704980.230255192down试验2tcirg1-0.8041261360.079217394down试验2tecpr2-0.8251393880.170236917down试验2tex264-0.5054906450.227224075down试验2tle3-0.4661449840.203917134down试验2tmbim6-0.2912609170.207332199down试验2tmem123-0.3952426630.05837947down试验2tmem134-0.4724399080.204723138down试验2tmem189-0.5636154640.204723138down试验2tnfaip2-0.8915745630.035341619down试验2tnfrsf14-0.7226119490.19331656down试验2tnip1-0.4257196330.122393306down
[0239]
[表4-20]
[0240][0241][0242]
使用公共数据库string进行基于基因本体(go)富集分析的生物过程(biological process(bp))和kegg pathway搜索。结果表明,与pd患者中表达上升或者下降的基因群关联的kegg pathway在试验1中得到了30个、试验2中得到了28个,两个试验中均包含显示帕金森氏病的项目hsa05012(parkinson’s disease)(表5-1~5-2)。
[0243]
[表5-1]
[0244]
[0245][0246]
[表5-2]
[0247]
[0248][0249]
针对所述表4-1~表4-20所示的试验1或试验2中至少任意一项中发现表达变动的基因,就其与帕金森氏病的关联性对现有文献进行了确认,结果发现,试验1中发现表达变动的基因中表6-1所示的19种和试验2中发现表达变动的基因中表6-2所示的30种目前尚未被报告有关它们与帕金森氏病的关联性,表明它们可以用作帕金森氏病的新型检测标记物。需要说明的是,表中粗体所表示的基因是试验1和试验2中共有的基因。
[0250]
[表6-1]
[0251][0252]
[表6-2]
[0253][0254]
实施例2判别模型的制作和验证1
[0255]
1)使用数据
[0256]
与实施例1的rna表达分析1相同地,在源自被检验者的ssl的rna的表达量的数据(读取计数值)中,将读取计数小于10的数据作为缺失值处理,将表达量的数据转换为经样品间的总读取数的差异校正后的rpm值,然后,使用singular value decomposition(svd)imputation方法对缺失值进行填充。其中,只将在全部样品的80%以上的样品中得到了不是缺失值的表达量数据的基因用于以下的分析。构建机器学习模型时,为了使符合负二项分布的rpm值近似正态分布,使用转换成以2为底的对数值的rpm值(log2rpm值)。
[0257]
2)数据集分割
[0258]
在由试验1的被检验者得到的rna谱数据集中,将10名健康者和10名pd共计20名被检验者的rna谱数据作为pd预测模型的训练数据,将剩余10名被检验者的rna谱数据作为用
于评价模型精度的测试数据。在由试验2的被检验者得到的rna谱数据集中,将40名健康者和40名pd共计80名被检验者的rna谱数据作为pd预测模型的训练数据,将剩余20名被检验者的rna谱数据作为用于评价模型精度的测试数据。
[0259]
3)特征量基因的选择
[0260]
在实施例1的rna表达分析1中,将与健康者相比试验1和试验2中pd患者均表达上升的18种rna和均表达下降的15种rna(表1-1~1-27中粗体表示的基因)选作特征量基因,通过主成分分析将这些表达量的数据转换为主成分之后,将第1主成分~第10主成分作为说明变量。另外,在试验1和试验2中pd患者均表达上升的18种rna和均表达下降的15种rna中,将snora16a、snora24、snora50、rexo1l2p这四种选作特征量基因,通过主成分分析将这些表达量的数据转换为主成分之后,将第1主成分~第4主成分作为说明变量。
[0261]
4)模型构建
[0262]
将根据选自源自ssl的rna的所述特征量基因的表达量的数据(log2rpm值)得到的各主成分的值用作说明变量,其中,上述表达量的数据为训练数据;将健康者(hl)和pd用作目标变量,构建预测模型。针对每个待预测项目,使用随机森林(random forest)、线性核的支持矢量机(svm linear)、rbf核的支持矢量机(svm rbf)、神经网络(nerural net)、一般线性模型(generalized linear model)、正则化线性判别分析(regularized linear discriminant analysis)、正则化逻辑回归(regularized logistic regression)这7种算法,进行十折交叉验证,使预测模型进行学习。在每种算法中,向学习后的模型中输入根据测试数据的所述特征量基因表达量(log2rpm值)得到的各主成分的值,并计算各预测项目的目标预测值。根据预测值和实测值计算检测率(recall)、精度(precision)和作为它们的调和平均的f值,并选择该f值最大的模型作为最佳预测模型。
[0263]
5)结果
[0264]
表7中示出待预测项目的使用算法、检测率、精度、f值。另外,测试数据中由最佳预测模型中的预测值和实测值制成的混淆矩阵示于图1。需要说明的是,图中的数值表示各象限的样品数。
[0265]
表8中示出使用随机森林构建模型时各特征量基因的变量重要度的计算结果。
[0266]
使用snora16a、snora24、snora50、rexo1l2p这四种基因的模型的f1在试验1中为0.67,试验2中为0.75,综合试验1+试验2后达到0.76,表明其可以预测pd。使用pd患者中上升的18种rna和下降的15种rna这共计33种rna的模型的f1在试验1中为0.91,试验2中为0.80,综合试验1+试验2后达到0.82,表明其可以更高精度地预测pd。
[0267]
[表7]
[0268][0269]
rf:随机森林;svm linear:线性核的支持矢量机;svm rbf:rbf核的支持矢量机;nnet:神经网络;glm:一般线性模型;rlda:正则化线性判别分析;rlogistic:正则化逻辑回
归
[0270]
[表8]
[0271][0272]
实施例3判别模型的制作和验证2
[0273]
1)使用数据
[0274]
与实施例1的rna表达分析2相同地,在源自被检验者的ssl的rna的表达量的数据(读取计数值)中,使用deseq2方法进行校正。其中,排除未检测到4161个以上基因的样品,仅将排除后的全部样品中被检验者的表达量的数据中90%以上的样品被检验者不是缺失值的表达量的数据的基因用于以下分析。分析中使用通过deseq2方法校正后的计数值(标准化计数值)。
[0275]
2)数据集分割
[0276]
在由试验1的被检验者得到的rna谱数据集中,将9名健康者和6名pd共计15名被检验者的rna谱数据作为pd预测模型的训练数据,将剩余4名健康者和1名pd共计5名被检验者
的rna谱数据作为用于评价模型精度的测试数据。在由试验2的被检验者得到的rna谱数据集中,将37名健康者和35名pd共计72名被检验者的rna谱数据作为pd预测模型的训练数据,将剩余13名健康者和11名pd共计24名被检验者的rna谱数据作为用于评价模型精度的测试数据。
[0277]
3)特征量基因的选择
[0278]
在实施例1的rna表达分析2中,将与健康者相比试验1和试验2中pd患者均表达上升或下降的17种rna(表4-1~4-20的粗体表示的基因)选作特征量基因,通过主成分分析将这些表达量的数据转换为主成分之后,将第1主成分~第4主成分作为说明变量。
[0279]
4)模型构建
[0280]
根据选自源自ssl的rna的所述特征量基因的表达量的数据(标准化计数值加1后制成以2为底的对数值后的值)得到的各主成分的值用作说明变量,其中,上述表达量的数据为训练数据;将健康者(hl)和pd用作目标变量,构建预测模型。针对每个待预测项目,使用随机森林(random forest)、线性核的支持矢量机(svm linear)、rbf核的支持矢量机(svm rbf)、神经网络(nerural net)、一般线性模型(generalized linear model)、正则化线性判别分析(regularized linear discriminant analysis)、正则化逻辑回归(regularized logistic regression)这7种算法,进行十折交叉验证,使预测模型进行学习。在每种算法中,向学习后的模型输入根据测试数据的所述特征量基因表达量(标准化计数值加1并制成以2为底的对数值后的值)得到的各主成分的值,并计算各预测项目的目标预测值。根据预测值和实测值计算检测率(recall)、精度(precision)和作为它们的调和平均的f值,并选择该f值最大的模型作为最佳预测模型。
[0281]
5)结果
[0282]
表9中示出待预测项目的使用算法、检测率、精度、f值。
[0283]
使用在试验1和试验2中利用deseq2校正后的似然比检验的结果中均表达上升或下降的17种rna的模型的f值在试验1中为1,在试验2中达到0.87,表明其可以预测pd。
[0284]
[表9]
[0285][0286]
rf:随机森林;svm linear:线性核的支持矢量机;svm rbf:rbf核的支持矢量机;nnet:神经网络;glm:一般线性模型;rlda:正则化线性判别分析;rlogistic:正则化逻辑回归
[0287]
实施例4判别模型的制作和验证3
[0288]
1)使用数据
[0289]
与实施例1的rna表达分析2相同地,在源自被检验者的ssl的rna的表达量的数据(读取计数值)中,使用deseq2方法进行校正。其中,排除未检测到4161个以上基因的样品,仅将排除后的全部样品中被检验者的表达量的数据中90%以上的样品被检验者不是缺失值的表达量的数据的基因用于以下分析。分析中使用通过deseq2方法校正后的计数值(标准化计数值)。
[0290]
2)数据集分割
[0291]
在由试验1的被检验者得到的rna谱数据集中,将9名健康者和6名pd共计15名被检验者的rna谱数据作为pd预测模型的训练数据,剩余4名健康者和1名pd共计5名被检验者的rna谱数据作为用于评价模型精度的测试数据。在由试验2的被检验者得到的rna谱数据集中,将37名健康者和35名pd共计72名被检验者的rna谱数据作为pd预测模型的训练数据,剩余13名健康者和11名pd共计24名被检验者的rna谱数据作为用于评价模型精度的测试数据。
[0292]
3)特征量基因的选择
[0293]
在实施例1的rna表达分析2中,将与健康者相比在试验1中pd患者表达上升或者下降的19种rna(表6-1所示的基因)、或与健康者相比在试验2中pd患者表达上或者下降的30种rna(表6-2所示的基因)选作特征量基因,通过主成分分析将这些表达量的数据转换为主成分之后,将第1主成分~第4主成分作为说明变量。
[0294]
4)模型构建
[0295]
将根据选自源自ssl的rna的所述特征量基因的表达量的数据(标准化计数值加1并制成以2为底的对数值后的值)得到的各主成分的值用作说明变量,上述表达量的数据为训练数据;将健康者(hl)和pd用作目标变量,构建预测模型。针对每个待预测项目,使用随机森林(random forest)、线性核的支持矢量机(svm linear)、rbf核的支持矢量机(svm rbf)、神经网络(nerural net)、一般线性模型(generalized linear model)、正则化线性判别分析(regularized linear discriminant analysis)、正则化逻辑回归(regularized logistic regression)这7种算法,进行十折交叉验证,使预测模型进行学习。在每种算法中,向学习后的模型输入根据测试数据的所述特征量基因表达量(标准化计数值加1并制成以2为底的对数值后的值)得到的各主成分的值,并计算各预测项目的目标预测值。根据预测值和实测值计算检测率(recall)、精度(precision)和作为它们的调和平均的f值,并选择该f值最大的模型作为最佳预测模型。
[0296]
5)结果
[0297]
表10、表11中示出待预测项目的使用算法、检测率、精度、f值。
[0298]
使用试验1中通过deseq2校正后的似然比检验的结果中,表达上升或下降的rna中目前未报道与帕金森氏病的关联性的19种rna的模型的f值为1,表明可以预测pd。使用在试验2中通过deseq2校正后的似然比检验的结果中,表达上升或下降的rna中目前尚未报道与帕金森氏病的关联性的30种rna的模型的f值为0.87,表明可以预测pd。
[0299]
[表10]
[0300][0301]
rf:随机森林;svm linear:线性核的支持矢量机;svm rbf:rbf核的支持矢量机;nnet:神经网络;glm:一般线性模型;rlda:正则化线性判别分析;rlogistic:正则化逻辑回归
[0302]
[表11]
[0303][0304]
rf:随机森林;svm linear:线性核的支持矢量机;svm rbf:rbf核的支持矢量机;nnet:神经网络;glm:一般线性模型;rlda:正则化线性判别分析;rlogistic:正则化逻辑回归
[0305]
实施例5判别模型的制作和验证4
[0306]
1)使用数据
[0307]
与实施例1的rna表达分析1相同地,在源自被检验者的ssl的rna的表达量的数据(读取计数值)中,将读取计数小于10的数据作为缺失值处理,将表达量的数据转换为经样品间的总读取数的差异校正后的rpm值,然后,使用singular value decomposition(svd)imputation方法对缺失值进行填充。其中,只将在全部样品的80%以上的样品中得到了不是缺失值的表达量数据的基因用于以下的分析。构建机器学习模型时,为了使符合负二项分布的rpm值近似正态分布,使用转换为以2为底的对数值的rpm值(log2rpm值)。
[0308]
2)数据集分割
[0309]
在由试验1的被检验者得到的rna谱数据集中,将10名健康者和10名pd共计20名被检验者的rna谱数据作为pd预测模型的训练数据,将剩余10名被检验者的rna谱数据作为用于评价模型精度的测试数据。在由试验2的被检验者得到的rna谱数据集中,将40名健康者和40名pd共计80名被检验者的rna谱数据作为pd预测模型的训练数据,将剩余20名被检验者的rna谱数据作为用于评价模型精度的测试数据。
[0310]
3)特征量基因的选择
[0311]
在实施例1的rna表达分析1中,将与健康者相比在试验1中pd患者表达上升或者下降的21种rna(表3-1所示的基因)、或与健康者相比在试验2中pd患者表达上升或者下降的92种rna(表3-2~3-4所示的基因)选作特征量基因,通过主成分分析将这些表达量的数据转换为主成分之后,将第1主成分~第4主成分作为说明变量。
[0312]
4)模型构建
[0313]
将根据选自源自ssl的rna的所述特征量基因的表达量的数据(log2rpm值)得到的各主成分的值用作说明变量,其中,上述表达量的数据为训练数据;将健康者(hl)和pd用作目标变量,构建预测模型。针对每个待预测项目,使用随机森林(random forest)、线性核的支持矢量机(svm linear)、rbf核的支持矢量机(svm rbf)、神经网络(nerural net)、一般线性模型(generalized linear model)、正则化线性判别分析(regularized linear discriminant analysis)、正则化逻辑回归(regularized logistic regression)这7种算法,进行十折交叉验证,使预测模型进行学习。在每种算法中,向学习后的模型输入根据测试数据的所述特征量基因表达量(log2rpm值)得到的各主成分的值,并计算各预测项目的目标预测值。根据预测值和实测值计算检测率(recall)、精度(precision)和作为它们的调和平均的f值,并选择该f值最大的模型作为最佳预测模型。
[0314]
5)结果
[0315]
表12、表13中示出待预测项目的使用算法、检测率、精度、f值。
[0316]
使用试验1中通过log2rpm校正后的检验结果中,表达上升或者下降的rna中目前尚未报道与帕金森氏病的关联性的21种rna的模型的f值为0.91,表明其可以预测pd。使用试验2中通过log2rpm校正后的检验结果中,表达上升或者下降的rna中目前尚未报道与帕金森氏病的关联性的92种rna的模型的f值为0.9,表明其可以预测pd。
[0317]
[表12]
[0318][0319]
rf:随机森林;svm linear:线性核的支持矢量机;svm rbf:rbf核的支持矢量机;nnet:神经网络;glm:一般线性模型;rlda:正则化线性判别分析;rlogistic:正则化逻辑回归
[0320]
[表13]
[0321][0322]
rf:随机森林;svm linear:线性核的支持矢量机;svm rbf:rbf核的支持矢量机;nnet:神经网络;glm:一般线性模型;rlda:正则化线性判别分析;rlogistic:正则化逻辑回归
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1