编码氨基酸序列、编码氧化还原酶的多核苷酸的制作方法
编码氨基酸序列、编码氧化还原酶的多核苷酸
1.本发明属于用于生产精细化学品的发酵方法领域,并且本发明涵盖了多核苷酸,其编码氨基酸序列、编码氧化还原酶,以及使用氧化还原酶的发酵方法。
2.患有帕金森病的患者会出现震颤、僵硬或运动迟缓等症状,这是由于大脑基底神经节中的细胞死亡引起的低水平多巴胺导致的。由于多巴胺不能通过血脑屏障,其前体分子l-dopa(l-二羟基苯丙氨酸)被用作减轻患者症状的药物。自其1977年首次推出以来,l-dopa(左旋多巴)已发展成为排在第一个的帕金森治疗药物,全球市场量为600mt。
3.现有的生产方法基于化学合成,特别是不对称氢化或氢化和手性拆分。这是一个由来已久的制造方法,但转化率和整体效率都很差。或者,l-dopa通过酶促转化产生,即丙酮酸和儿茶酚的酶促偶联。然而,这种酶促转化方法具有原料成本高的缺点。
4.文献中记载了使用来自大肠杆菌(escherichia coli)(e.coli)的4-羟基苯乙酸3-单加氧酶hpab和同源的4-羟基苯乙酸3-单加氧酶还原酶hpac的发酵方法,并且由于更高的转化率、对映选择性和更经济的方法而可能是有利的。
5.为了实现具有经济上有吸引力的方法,至关重要的是,一方面优化酪氨酸生产力,另一方面通过增加hpab活性将l-酪氨酸转化为l-dopa。
6.prieto等人,1993中记载了4-羟基苯乙酸单加氧酶的广泛的底物谱,并指出,与非天然底物l-酪氨酸的酶活性仅为与天然底物4-hpa(4-羟基苯乙酸)的酶活性的5%。因此,酶建模被认为是增加酶活性的有希望的选择。
7.shen等人,scientific reports(2019)9:7087/https://doi.org/10.1038/s41598-019-43577-w,p.1-11记载了对来自大肠杆菌的黄素依赖性羟化酶(hpab)的催化多功能性的结构见解。shen等人提到来自大肠杆菌的4-羟基苯乙酸3-羟化酶(echpab)能够有效地对多种酚类化合物进行邻羟基化,并显示出广泛的化学酶学应用的巨大潜力。为了了解其催化多功能性的结构和机制基础,shen等人通过x射线晶体学阐明了echpab的晶体结构,其揭示了覆盖活性位点的独特环状结构。shen等人进一步对该环进行了诱变研究,以探究其在底物特异性和催化活性中的作用。他们的结果不仅表明该环具有很强的可塑性和对充分诱变的强抗性,而且还提出了一种柔性环,所述柔性环使底物能够进入并稳定结合活性位点,其可能是酶催化多功能性的关键因素。报告了不同echpab突变体的环序列和结构。然而,没有记载对于l-dopa的选择性得到增强。
8.fordjour等人,microbial cell factories(2019)18:74/https//doi.org/10.1186/s12934-019-1122-0,p.1-10记载了用于从d-葡萄糖从头生产l-dopa的e.coli bl21(de3)的代谢工程改造。通过缺失tyrr、ptsg、crr、phea和pykf同时通过过表达galp和glk来引导碳流来工程改造e.coli bl21(de3)。还过表达tkta和ppsa以增强e4p和pep的积累。对hpab应用位点定向诱变以优化其活性。鉴定了三个突变体g883r、g883a和l1231m,它们相对于野生型hpab的l-dopa产量分别显示出3.03倍、2.9倍和2.56倍增加的活性改善。使用菌株lp-8导致摇瓶和5l生物反应器中l-dopa的产量分别为691.24mg/l和25.53g/l。
9.ep3150712a1(symrise)记载了用于提供3,4-二羟基苯基化合物及其甲基化变体的生物技术方法。ep3150712a1因此涉及通过合理设计原型酶4-羟基苯乙酸3-羟化酶
(4hpa3h)的活性位点结合袋获得的基因修饰的酶,用于羟基化4-羟基苯基化合物以产生3,4-二羟基苯基化合物,并涉及包括使用所述酶或其催化活性片段的体内和体外方法的生物技术方法。
10.cn107541483a(tianjin)记载了用于重组生产左旋多巴的大肠杆菌菌株、其构建方法及应用。cn107541483a因此提供了一种采用大肠杆菌重组菌株t002生产l-dopa的方法,其中实施aroe基因的上调以增强3-脱氢酶莽草酸脱氢酶的表达,其能够进而生产l-dopa。
11.本发明的一个目的是提供一种经济上有吸引力的用于生产l-dopa的发酵方法,具有优化的酪氨酸产量和l-酪氨酸到l-dopa的高转化率。需要具有改进的产量或更高的细胞内和/或培养基中产物的最终浓度的l-dopa的生产方法。该方法需要可扩展,以确保l-dopa的高效生产。
12.本发明的又一目的是提供一种细胞,其以使得它能够产生大量的l-dopa的方式被修饰。
13.本发明的发明人已经出乎意料地确定,氧化还原酶hpab的突变导致l-dopa的产量增加和l-酪氨酸到l-dopa的转化率更高。
14.本发明涉及一种多核苷酸,其编码氨基酸序列、编码氧化还原酶,,所编码的氨基酸序列或氧化还原酶与seq id no:1(地芽孢杆菌属的一个种(geobacillus sp.)pa9)、seq id no:3(嗜热栖热菌(thermus thermophilus))、seq id no:4(球孢链霉菌(streptomyces globisporus))、seq id no:5(氨基丁酸梭状芽孢杆菌(clostridium aminobutyricum))、seq id no:6(洋葱伯克霍尔德菌(burkholderai cepacia))、seq id no:8(颤藻属的一个种(oscillatoria sp.)pcc 6506)、seq id no:9(瘤状副伯克霍尔德菌(paraburkholderia phymatum))的氨基酸序列至少≥50%相同,
15.其特征在于在seq id no:1的位置202、203、204、205、206、207、208、209、210、211、212、213、214中的一个或多个位置中或者在seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:8、seq id no:9的氨基酸序列的对应位置处的氨基酸交换,其中氨基酸交换不是a210s或s212a。
16.根据本发明,在seq id no:1的位置210处或在seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:8、seq id no:9的氨基酸序列的对应位置处从丙氨酸到丝氨酸的氨基酸交换和在seq id no:1的位置212处或在seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:8、seq id no:9的氨基酸序列的对应位置处从丝氨酸到丙氨酸的氨基酸交换被排除并且不应成为本发明的一部分。
17.在备选配置中,多核苷酸具有在seq id no:1的位置202、203、204、205、206、207、208、209、211、213、214中的一个或多个位置处或者在seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:8、seq id no:9的氨基酸序列的对应位置处的氨基酸交换。
18.在优选实施方案中,氧化还原酶不是seq id no:2的4-羟基苯乙酸3-单加氧酶。
19.在优选实施方案中,氨基酸交换导致l-dopa的产量增加和/或l-酪氨酸到l-dopa的更高的转化率。
20.在优选实施方案中,由核苷酸序列编码的氧化还原酶是4-羟基苯乙酸3-单加氧酶。
21.根据本发明由seq id no:1、seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:7、seq id no:8、seq id no:9和seq id no:10编码的酶是4-羟基苯乙酸3-单加氧酶。
22.选择来自地芽孢杆菌属的一个种pa-9的同源物作为合理酶设计的起点,并在标准化的全细胞筛选系统中测试了一组突变。将筛选系统获得的有希望的结果转移到1l发酵规模上证实了这些结果,并且显示与表达大肠杆菌和地芽孢杆菌属的一个种pa-9野生型基因的参考菌株相比,突变酶的性能显著提高。
23.表述“seq id no:1的对应位置”或“与氨基酸序列的位置202、203、204、205、206、207、208、209、210、211、213、214可比较的位置”是指这样的事实,通过在编码多肽的n端区域(基于seq id no:1的位置202-214)插入或缺失一个编码氨基酸的密码子,位置说明和长度说明在插入的情况下正式增加一个单位或者在缺失的情况下减少一个单位。以相同的方式,通过在编码多肽的c端区域(基于位置202-214)插入或缺失一个编码氨基酸的密码子,长度说明在插入的情况下正式增加一个单位或者在缺失的情况下减少一个单位。这种可比较的位置可以很容易地通过以“比对”的形式通过比较氨基酸序列来进行鉴定,例如使用clustal w programme(thompson等人,nucleic acids research 22,4637-4680(1994))或mafft programme(katoh等人,genome information 2005;16(1),22-33)。当提到来自不同物种的不同seq id no:(例如seq id no:3
–
seq id no:10),表述“seq id no:1的对应位置”是指待比较的晶体结构内的同源位置。这样的同源位置也可以通过如上所述的以“比对”的形式比较氨基酸序列或基于结构预测来进行鉴定。
24.在本发明的情况下,seq id no:1的位置202、203、204、205、206、207、208、209、210、211、212、213、214对应于各种序列的以下位置:
25.seq id no:3的位置194、195、196、197、198、199、200、201、202、203、204、205;
26.seq id no:4的位置212、213、214、215、216、217、218、219、220、221、222;
27.seq id no:5的位置202、203、204、205、206、207、208、209、210;
28.seq id no:6的位置202、203、204、205、206、207、208、209、210、211、212;
29.seq id no:7的位置203、204、205、206、207、208、209、210、211、212、213;
30.seq id no:8的位置209、210、211、212、213、214、215、216、217;
31.seq id no:9的位置192、193、194、195、196、197、198、199、200、201;
32.seq id no:10的位置203、204、205、206、207、208、209、210、211、212、213、214、215、216、217。
33.关于本发明的氧化还原酶,用clustal omega比对了同源物hpab酶的序列并且在第二步,预测了hpab序列的二级结构并且使用二级结构预测重新比对了序列。
34.这样的插入和缺失基本上没有影响酶促活性。“基本上没有影响”意味着所述变体的酶促活性与具有seq id no:1的氨基酸序列的多肽的活性相差最多10%、最多7.5%、最多5%、最多2.5%或最多1%。
35.优选地,编码氨基酸序列的多核苷酸编码氧化还原酶,其与seq id no:1(地芽孢杆菌属的一个种pa9)、seq id no:3(嗜热栖热菌)、seq id no:4(球孢链霉菌)、seq id no:5(氨基丁酸梭状芽孢杆菌)、seq id no:6(洋葱伯克霍尔德菌)、seq id no:7(钩虫贪铜菌(cupriavidus necator))、seq id no:8(颤藻属的一个种pcc 6506)、seq id no:9(瘤状副
伯克霍尔德菌)的氨基酸序列至少≥65%相同,
36.其特征在于在seq id no:1的位置202、203、204、205、206、207、208、209、210、211、212、213、214中的一个或多个位置中或者在seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:7、seq id no:8、seq id no:9的氨基酸序列的对应位置处的氨基酸交换。
37.更优选地,编码氨基酸序列的多核苷酸编码氧化还原酶,其与seq id no:1(地芽孢杆菌属的一个种pa9)、seq id no:3(嗜热栖热菌)、seq id no:4(球孢链霉菌)、seq id no:5(氨基丁酸梭状芽孢杆菌)、seq id no:6(洋葱伯克霍尔德菌)、seq id no:7(钩虫贪铜菌)、seq id no:8(颤藻属的一个种pcc 6506)、seq id no:9(瘤状副伯克霍尔德菌)、seq id no:10(皮氏罗尔斯顿菌(ralstonia pickettii))的氨基酸序列至少≥90%相同,
38.其特征在于在seq id no:1的位置202、203、204、205、206、207、208、209、210、211、212、213、214中的一个或多个位置中或者在seq id no:3、seq id no:4、seq id no:5、seq id no:6、seq id no:7、seq id no:8、seq id no:9、seq id no:10的氨基酸序列的对应位置处的氨基酸交换。
39.优选地,编码氨基酸序列的多核苷酸,其编码氧化还原酶,所编码的氧化还原酶选自:
40.·
seq id no:1(地芽孢杆菌属的一个种pa9),其中在位置202、203、204、205、206、207、208、209、210、211、212、213、214中的一个或多个位置处具有氨基酸交换;
41.·
seq id no:3(嗜热栖热菌),其中在位置194、195、196、197、198、199、200、201、202、203、204、205中的一个或多个位置处具有氨基酸交换;或者seq id no:21;
42.·
seq id no:4(球孢链霉菌),其中在位置212、213、214、215、216、217、218、219、220、221、222中的一个或多个位置处具有氨基酸交换;或者seq id no:22;
43.·
seq id no:5(氨基丁酸梭状芽孢杆菌),其中在位置202、203、204、205、206、207、208、209、210中的一个或多个位置处具有氨基酸交换;或者seq id no:23;
44.·
seq id no:6(洋葱伯克霍尔德菌),其中在位置202、203、204、205、206、207、208、209、210、211、212中的一个或多个位置处具有氨基酸交换;或者seq id no:24;
45.·
seq id no:7(钩虫贪铜菌),其中在位置203、204、205、206、207、208、209、210、211、212、213中的一个或多个位置处具有氨基酸交换;或者seq id no:25;
46.·
seq id no:8(颤藻属的一个种pcc 6506),其中在位置209、210、211、212、213、214、215、216、217中的一个或多个位置处具有氨基酸交换;或者seq id no:26;
47.·
seq id no:9(瘤状副伯克霍尔德菌),其中在位置192、193、194、195、196、197、198、199、200、201中的一个或多个位置处具有氨基酸交换;或者seq id no:27;
48.·
seq id no:10(皮氏罗尔斯顿菌),其中在位置203、204、205、206、207、208、209、210、211、212、213、214、215、216、217中的一个或多个位置处具有氨基酸交换;或者seq id no:28。
49.这些不同序列中的那些位置应当是意味着根据本发明的“氨基酸序列的对应位置”。
50.编码氨基酸序列的多核苷酸编码氧化还原酶,所编码的氧化还原酶与seq id no:11、seq id no:12、seq id no:13或seq id no:14的氨基酸序列至少≥90%、≥92%、≥
94%、≥96%、≥97%、≥98%、≥99%或100%、优选≥97%、特别优选≥98%、非常特别优选≥99%并且极度优选100%相同,其中seq id no:11、seq id no:12、seq id no:13和seq id no:14在位置207或氨基酸序列的对应位置处具有除l-缬氨酸以外的蛋白质氨基酸。
51.多核苷酸优选是这样的序列,其中该多核苷酸是编码来自地芽孢杆菌属微生物的4-羟基苯乙酸3-单加氧酶的可复制核苷酸序列,其中由此编码的蛋白质序列在对应于seq id no:1的位置207的位置处含有除l-缬氨酸以外的蛋白质氨基酸。
52.在本发明的有利配置中,由该多核苷酸编码的氨基酸序列在位置207或对应位置处具有选自苏氨酸、亮氨酸、谷氨酰胺和甘氨酸的氨基酸。
53.特别优选的是,当由该多核苷酸编码的蛋白质序列
[0054]-在位置206或氨基酸序列的对应位置处含有除l-苏氨酸以外的蛋白质氨基酸,优选l-甲硫氨酸或l-丙氨酸;或者
[0055]-在位置208或氨基酸序列的对应位置处含有除l-赖氨酸以外的蛋白质氨基酸,优选l-精氨酸。
[0056]
在优选配置中,编码氨基酸序列的多核苷酸编码氧化还原酶,所编码的氧化还原酶与seq id no:15、seq id no:16.、seq id no:17、seq id no:18、seq id no:19、seq id no:20、seq id no:29、seq id no:30、seq id no:31、seq id no:32的氨基酸序列至少≥90%、≥92%、≥94%、≥96%、≥97%、≥98%、≥99%或100%、优选≥97%、特别优选≥98%、非常特别优选≥99%并且极度优选100%相同。
[0057]
本发明相应地还涉及这样的多核苷酸和核酸分子,其包含这样的序列并编码seq id no:1至20和seq id no:29至32的多肽变体,其含有一个或多个插入或缺失。优选地,多肽含有最多5个、最多4个、最多3个或最多2个氨基酸的插入或缺失。
[0058]
本发明还涉及包含由根据本发明的核苷酸序列编码的氨基酸序列的多肽。
[0059]
本发明还涉及这样的埃希菌(escherichia)、假单胞菌(pseudomonas)或棒状杆菌(corynebacterium)属的微生物,其包含根据本发明的多核苷酸、载体和/或多肽,并且在所述微生物中,编码4-羟基苯乙酸单加氧酶的酶的核苷酸序列优选以过表达的形式存在。
[0060]
优选地,由多核苷酸编码的多肽可以在位置207处或对应位置处显示这样的氨基酸,其选自丝氨酸、亮氨酸、谷氨酰胺和甘氨酸。
[0061]
由此编码的蛋白质序列可以在位置206或氨基酸序列的相应位置处含有除l-苏氨酸以外的蛋白质氨基酸,优选l-甲硫氨酸。
[0062]
由此编码的蛋白质序列还可以在位置208或氨基酸序列的相应位置处含有除l-赖氨酸以外的蛋白质氨基酸,优选l-精氨酸。
[0063]
本发明还涉及这样的质粒和载体,其包含根据本发明的多核苷酸并且任选地在棒状杆菌、假单胞菌或埃希菌属的微生物中复制或适用于其中。
[0064]
本发明还涉及这样的棒状杆菌、假单胞菌或埃希菌属的微生物,其包含根据本发明的多核苷酸、载体和多肽。
[0065]
本发明还涉及根据本发明的微生物,其特征在于根据本发明的多核苷酸整合在染色体中。同源重组允许使用根据本发明的载体将由载体转运进入细胞的根据本发明的多核苷酸交换染色体上的dna部分(section)。为了在载体的环型dna分子和染色体上的靶dna之间进行有效的重组,提供了在末端具有与靶位点同源的核苷酸序列的含有根据本发明的多
核苷酸的待交换dna区域;这些决定了载体的整合和dna交换的位点。
[0066]
例如,根据本发明的多核苷酸可以在染色体中的天然基因位点处交换天然hpab基因或在进一步的基因位点处整合。
[0067]
本发明提供了大肠杆菌、恶臭假单胞菌(p.putida)或谷氨酸棒状杆菌(c.glutamicum)种的微生物,其包含要求保护的任意多核苷酸或要求保护的任意多肽或要求保护的任意载体。
[0068]
微生物可以是这样的微生物,其中多核苷酸以过表达的形式存在。
[0069]
微生物的特征可以在于微生物具有生产精细化学品的能力。精细化学品优选为l-二羟基苯丙氨酸(l-dopa)。
[0070]
过表达通常意味着,与起始菌株(亲本菌株)或野生型菌株(如果这是起始菌株)相比,核糖核酸、蛋白质(多肽)或酶的细胞内浓度或活性增加。起始菌株(亲本菌株)是指这样的菌株,对所述菌株进行导致过表达的措施。
[0071]
在过表达中,重组过表达的方法是优选的。这些包括使用体外提供的dna分子生产微生物的所有方法。这样的dna分子包括,例如,启动子、表达盒、基因、等位基因、编码区等。这些通过转化、接合、转导或类似方法转化为所需的微生物。
[0072]
可以通过测量基因转录的mrna的量、测定多肽的量以及测定酶活性来确定表达或过表达的程度。
[0073]
本发明公开了生产精细化学品的发酵方法,其包括以下步骤:
[0074]
a)在培养基中发酵包含编码氨基酸序列的多核苷酸的微生物,所述多核苷酸编码氧化还原酶,所编码的氧化还原酶与seq id no:1或seq id no:3至seq id no:32的氨基酸序列至少≥90%、≥92%、≥94%、≥96%、≥97%、≥98%、≥99%或100%、优选≥97%、特别优选≥98%、非常特别优选≥99%并且极度优选100%相同,
[0075]
b)在培养基中积累精细化学品,其中获得发酵液。
[0076]
如实施例3所示,根据本发明的这种方法的使用导致与相应的起始菌株相比,产品浓度和l-dopa:l-酪氨酸的比率显著增加。
[0077]
待使用的培养基或发酵培养基必须适当地满足相应菌株的需求。在american society for bacteriology的手册"manual of methods for general bacteriology"(washington d.c.,usa,1981)中含有各种微生物的培养基的说明。术语培养基和发酵培养基或培养基是可以互换的。
[0078]
可以使用以下作为碳源:糖和碳水化合物,例如葡萄糖、蔗糖、乳糖、果糖、麦芽糖、糖蜜、来自甜菜糖或甘蔗加工的含蔗糖的溶液、淀粉、淀粉水解物和纤维素,油和脂肪,例如大豆油、葵花籽油、花生油和椰子脂,脂肪酸,例如棕榈酸、硬脂酸和亚油酸,醇类例如甘油、甲醇和乙醇,以及有机酸,例如乙酸或乳酸。
[0079]
可以使用以下作为氮源:有机氮化合物,例如蛋白胨、酵母提取物、肉提取物、麦芽提取物、玉米浆、豆粕和尿素或无机化合物,例如硫酸铵、氯化铵、磷酸铵、碳酸铵和硝酸铵。氮源可以单独使用,也可以作为混合物使用。
[0080]
可以使用磷酸、磷酸二氢钾或磷酸氢二钾或相应的含钠盐作为磷源。
[0081]
此外,培养基必须含有盐,例如金属的氯化物或硫酸盐形式,所述金属例如钠、钾、镁、钙和铁,例如硫酸镁或硫酸铁,这是生长所必需的。最后,除上述物质外,还可以使用,必
需的生长物质如氨基酸,例如高丝氨酸和维生素,例如硫胺素、生物素或泛酸。
[0082]
所述起始原料可以以单批的形式加入到培养物中或在培养期间以适当的方式提供。
[0083]
碱性化合物如氢氧化钠、氢氧化钾、氨或氨水,或酸化合物如磷酸或硫酸,以合适的方式用于培养物的ph控制。ph值一般调节至6.0至8.5,优选6.5至8。为了控制泡沫的形成,可以使用消泡剂,例如脂肪酸的聚乙二醇酯。为了维持质粒的稳定性,可以将合适的选择性作用物质例如抗生素添加到培养基中。优选在好氧条件下进行发酵。为了维持所述好氧条件,将氧气或含氧气体混合物例如空气引入培养物中。同样可以使用富含过氧化氢的液体。任选地,发酵在低于大气压的压力下进行,例如在0.03至0.2mpa的低于大气压的压力下进行。培养的温度通常为20℃至45℃,优选25℃至40℃,特别优选30℃至37℃。在分批或补料分批方法的情况下,优选继续培养,直到形成足以用于测量获得所需有机化合物的量。此目标通常在10小时到160小时内达到。在连续方法中,可以延长培养时间。由于微生物的活性,发酵培养基和/或微生物细胞中精细化学品的富集(积累)发生。
[0084]
除其他外,合适的发酵培养基的实例可以在专利文献us 5,770,409、us 5,990,350、us 5,275,940、wo 2007/012078、us 5,827,698、wo 2009/043803、us 5,756,345或us 7,138,266中找到;可以任选地根据所用菌株的要求进行适当的修改。
[0085]
该方法的特征可以在于它是这样的方法,其选自分批方法、分批补料方法、重复分批补料方法和连续方法。
[0086]
该方法的特征还可以在于精细化学品或含有精细化学品的液体或固体产品是从含有精细化学品的发酵液中获得的。
[0087]
在优选配置中,精细化学品是l-二羟基苯丙氨酸(l-dopa)。
[0088]
基于其中存在根据本发明的启动子变体的微生物的方法或发酵方法,根据本发明的方法或发酵方法的性能就选自以下的参数中的一个或多个增加了至少0.5%、至少1%、至少1.5%或至少2%:浓度(每体积形成的化合物)、产率(每碳源消耗形成的化合物)、体积生产率(每体积和时间形成的化合物)和生物质特异性生产率(每细胞干质量(dry mass)或生物干质量和时间形成的化合物或每细胞蛋白质和时间形成的化合物)或其他方法参数及其组合。
[0089]
由于发酵措施,获得含有所需精细化学品、优选氨基酸或有机酸的发酵液。
[0090]
然后,提供或生产或获得含有精细化学品的液体或固体形式的产品。
[0091]
在优选实施方案中,发酵液是指微生物在其中在一定时间和一定温度下培养的发酵培养基或营养培养基。发酵培养基或发酵过程中使用的培养基包含确保生产所需化合物的所有物质或成分,并且通常确保生长和/或活力。
[0092]
发酵完成后,所得发酵液相应地含有
[0093]
a)由微生物细胞的生长产生的微生物的生物质(细胞团块(cell mass)),
[0094]
b)在发酵过程中形成的所需精细化学品,
[0095]
c)在发酵过程中可能形成的有机副产品,以及
[0096]
d)发酵未消耗的所使用的发酵培养基或起始原料的成分,例如,例如,维生素例如生物素,或盐,例如硫酸镁。
[0097]
有机副产品包括除了发酵中使用的微生物所需的相应化合物之外产生的物质,并
no:1(地芽孢杆菌属的一个种pa9)、seq id no:3(嗜热栖热菌)、seq id no:4(球孢链霉菌)、seq id no:5(氨基丁酸梭状芽孢杆菌)、seq id no:6(洋葱伯克霍尔德菌)、seq id no:8(颤藻属的一个种pcc 6506)、seq id no:9(瘤状副伯克霍尔德菌)的氨基酸序列至少≥50%相同,
[0114]
其特征在于在seq id no:1的位置202、203、204、205、206、207、208、209、210、211、212、213、214中的一个或多个位置中,或在seq id no:3的位置194、195、196、197、198、199、200、201、202、203、204、205处,或在seq id no:4的位置212、213、214、215、216、217、218、219、220、221、222处,或在seq id no:5的位置202、203、204、205、206、207、208、209、210处,或在seq id no:6的位置202、203、204、205、206、207、208、209、210、211、212处,或在seq id no:8的位置209、210、211、212、213、214、215、216、217处,或在seq id no:9的位置192、193、194、195、196、197、198、199、200、201处的氨基酸交换,其中氨基酸交换不是a210s或s212a。
[0115]
在进一步的优选实施方案中,排除seq id no:1的位置210和212处的氨基交换。
[0116]
在优选实施方案中,编码氨基酸序列的多核苷酸编码氧化还原酶,其与seq id no:1(地芽孢杆菌属的一个种pa9)、seq id no:3(嗜热栖热菌)、seq id no:4(球孢链霉菌)、seq id no:5(氨基丁酸梭状芽孢杆菌)、seq id no:6(洋葱伯克霍尔德菌)、seq id no:7(钩虫贪铜菌)、seq id no:8(颤藻属的一个种pcc 6506)、seq id no:9(瘤状副伯克霍尔德菌)的氨基酸序列至少≥65%相同,
[0117]
其特征在于在seq id no:1的位置202、203、204、205、206、207、208、209、210、211、212、213、214中的一个或多个位置中,或在seq id no:3的位置194、195、196、197、198、199、200、201、202、203、204、205处,或在seq id no:4的位置212、213、214、215、216、217、218、219、220、221、222处,或在seq id no:5的位置202、203、204、205、206、207、208、209、210处,或在seq id no:6的位置202、203、204、205、206、207、208、209、210、211、212处,或在seq id no:7的位置203、204、205、206、207、208、209、210、211、212、213处,或在seq id no:8的位置209、210、211、212、213、214、215、216、217处,或在seq id no:9的位置192、193、194、195、196、197、198、199、200、201处的氨基酸交换,其中氨基酸交换不是a210s或s212a。
[0118]
在进一步的优选实施方案中,排除seq id no:1的位置210和212处的氨基交换。
[0119]
在优选实施方案中,编码氨基酸序列的多核苷酸编码氧化还原酶,其与seq id no:1(地芽孢杆菌属的一个种pa9)、seq id no:3(嗜热栖热菌)、seq id no:4(球孢链霉菌)、seq id no:5(氨基丁酸梭状芽孢杆菌)、seq id no:6(洋葱伯克霍尔德菌)、seq id no:7(钩虫贪铜菌)、seq id no:8(颤藻属的一个种pcc 6506)、seq id no:9(瘤状副伯克霍尔德菌)、seq id no:10(皮氏罗尔斯顿菌)的氨基酸序列至少≥90%相同,
[0120]
其特征在于在seq id no:1的位置202、203、204、205、206、207、208、209、210、211、212、213、214中的一个或多个位置中,或在seq id no:3的位置194、195、196、197、198、199、200、201、202、203、204、205处,或在seq id no:4的位置212、213、214、215、216、217、218、219、220、221、222处,或在seq id no:5的位置202、203、204、205、206、207、208、209、210处,或在seq id no:6的位置202、203、204、205、206、207、208、209、210、211、212处,或在seq id no:7的位置203、204、205、206、207、208、209、210、211、212、213处,或在seq id no:8的位置209、210、211、212、213、214、215、216、217处,或在seq id no:9的位置192、193、194、195、196、197、198、199、200、201处或在seq id no:10的位置203、204、205、206、207、208、209、
210、211、212、213、214、215、216、217处的氨基酸交换,其中氨基酸交换不是a210s或s212a。
[0121]
在进一步的优选实施方案中,排除seq id no:1的位置210和212处的氨基交换。
[0122]
优选实施方案涉及编码氨基酸序列的多核苷酸,其编码氧化还原酶,所编码的氧化还原酶与以下的氨基酸序列至少≥30%相同:
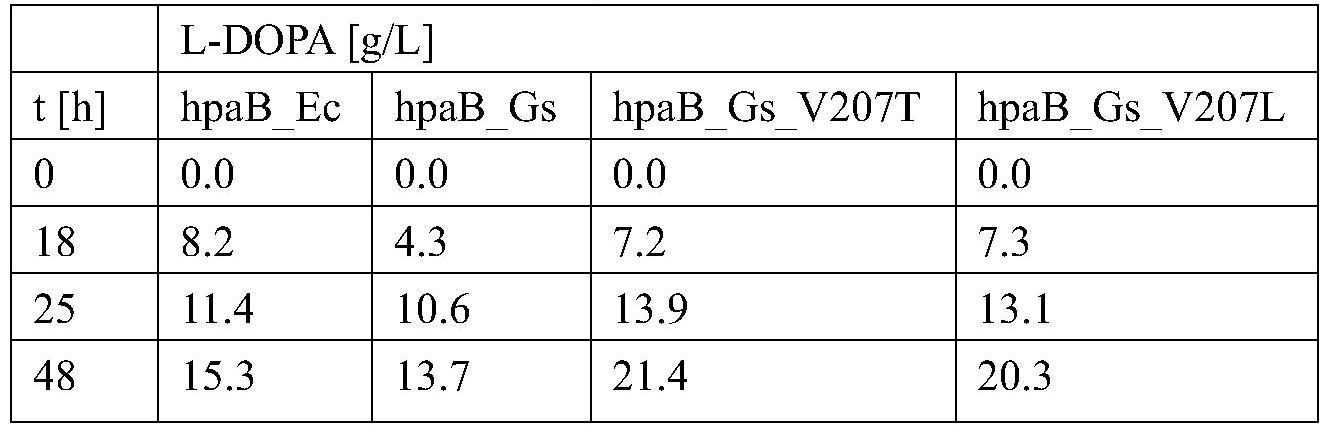
[0123]
·
seq id no:1(地芽孢杆菌属的一个种pa9),其中在位置202、203、204、205、206、207、208、209、210、211、213、214中的一个或多个位置处具有氨基酸交换;
[0124]
·
seq id no:3(嗜热栖热菌),其中在位置194、195、196、197、198、199、200、201、202、203、204、205中的一个或多个位置处具有氨基酸交换;或者seq id no:21;
[0125]
·
seq id no:4(球孢链霉菌),其中在位置212、213、214、215、216、217、218、219、220、221、222中的一个或多个位置处具有氨基酸交换;或者seq id no:22;
[0126]
·
seq id no:5(氨基丁酸梭状芽孢杆菌),其中在位置202、203、204、205、206、207、208、209、210中的一个或多个位置处具有氨基酸交换;或者seq id no:23;
[0127]
·
seq id no:6(洋葱伯克霍尔德菌),其中在位置202、203、204、205、206、207、208、209、210、211、212中的一个或多个位置处具有氨基酸交换;或者seq id no:24;
[0128]
·
seq id no:7(钩虫贪铜菌),其中在位置203、204、205、206、207、208、209、210、211、212、213中的一个或多个位置处具有氨基酸交换;或者seq id no:25;
[0129]
·
seq id no:8(颤藻属的一个种pcc 6506),其中在位置209、210、211、212、213、214、215、216、217中的一个或多个位置处具有氨基酸交换;或者seq id no:26;
[0130]
·
seq id no:9(瘤状副伯克霍尔德菌),其中在位置192、193、194、195、196、197、198、199、200、201中的一个或多个位置处具有氨基酸交换;或者seq id no:27;
[0131]
·
seq id no:10(皮氏罗尔斯顿菌),其中在位置203、204、205、206、207、208、209、210、211、212、213、214、215、216、217中的一个或多个位置处具有氨基酸交换;或者seq id no:28,其中氨基酸交换不是a210s或s212a。
[0132]
优选实施方案涉及编码氨基酸序列的多核苷酸,其编码氧化还原酶,所编码的氧化还原酶与以下的氨基酸序列至少≥40%或至少≥50%、或至少≥60%、或至少≥70%、或至少≥80%、或至少≥90%、或至少≥95%、或至少≥97%、或至少≥98%、或至少≥99%相同:
[0133]
·
seq id no:1(地芽孢杆菌属的一个种pa9),其中在位置202、203、204、205、206、207、208、209、210、211、212、213、214中的一个或多个位置处具有氨基酸交换;
[0134]
·
seq id no:3(嗜热栖热菌),其中在位置194、195、196、197、198、199、200、201、202、203、204、205中的一个或多个位置处具有氨基酸交换;或者seq id no:21;
[0135]
·
seq id no:4(球孢链霉菌),其中在位置212、213、214、215、216、217、218、219、220、221、222中的一个或多个位置处具有氨基酸交换;或者seq id no:22;
[0136]
·
seq id no:5(氨基丁酸梭状芽孢杆菌),其中在位置202、203、204、205、206、207、208、209、210中的一个或多个位置处具有氨基酸交换;或者seq id no:23;
[0137]
·
seq id no:6(洋葱伯克霍尔德菌),其中在位置202、203、204、205、206、207、208、209、210、211、212中的一个或多个位置处具有氨基酸交换;或者seq id no:24;
[0138]
·
seq id no:7(钩虫贪铜菌),其中在位置203、204、205、206、207、208、209、210、211、212、213中的一个或多个位置处具有氨基酸交换;或者seq id no:25;
[0139]
·
seq id no:8(颤藻属的一个种pcc 6506),其中在位置209、210、211、212、213、214、215、216、217中的一个或多个位置处具有氨基酸交换;或者seq id no:26;
[0140]
·
seq id no:9(瘤状副伯克霍尔德菌),其中在位置192、193、194、195、196、197、198、199、200、201中的一个或多个位置处具有氨基酸交换;或者seq id no:27;
[0141]
·
seq id no:10(皮氏罗尔斯顿菌),其中在位置203、204、205、206、207、208、209、210、211、212、213、214、215、216、217中的一个或多个位置处具有氨基酸交换;或者seq id no:28,其中氨基酸交换不是a210s或s212a。
[0142]
优选实施方案涉及编码氨基酸序列的多核苷酸,其编码氧化还原酶,所编码的氧化还原酶与seq id no:11、seq id no:12、seq id no:13或seq id no:14的氨基酸序列至少≥30%、≥40%、≥50%、≥60%、≥70%、≥80%、≥90%相同,其中seq id no:11、seq id no:12、seq id no:13和seq id no:14在位置207或氨基酸序列的对应位置处具有除l-缬氨酸以外的蛋白质氨基酸。
[0143]
优选实施方案是编码氨基酸序列的多核苷酸,其编码氧化还原酶,所编码的氧化还原酶与seq id no:11、seq id no:12、seq id no:13或seq id no:14的氨基酸序列至少≥90%、≥92%、≥94%、≥96%、≥97%、≥98%、≥99%或100%、优选≥97%、特别优选≥98%、非常特别优选≥99%并且极度优选100%相同,其中seq id no:11、seq id no:12、seq id no:13和seq id no:14在位置207或氨基酸序列的对应位置处具有除l-缬氨酸以外的蛋白质氨基酸。
[0144]
在优选实施方案中,编码氨基酸序列的多核苷酸编码氧化还原酶,其与seq id no:11、seq id no:12、seq id no:13or seq id no:14、seq id no:15、seq id no:16.、seq id no:17、seq id no:18、seq id no:19、seq id no:20、seq id no:29、seq id no:30、seq id no:31、seq id no:32的氨基酸序列至少≥30%、≥40%、≥50%、≥60%、≥70%、≥80%、≥90%相同。
[0145]
在优选实施方案中,编码氨基酸序列的多核苷酸编码氧化还原酶,其与seq id no:11、seq id no:12、seq id no:13or seq id no:14、seq id no:15、seq id no:16.、seq id no:17、seq id no:18、seq id no:19、seq id no:20、seq id no:29、seq id no:30、seq id no:31、seq id no:32至少≥90%、≥92%、≥94%、≥96%、≥97%、≥98%、≥99%或100%、优选≥97%、特别优选≥98%、非常特别优选≥99%并且极度优选100%相同。
实施例
[0146]
下面将参考示例性实施方案更详细地描述本发明。
[0147]
实施例1:生产具有hpab(地芽孢杆菌)突变体的大肠杆菌菌株
[0148]
先前在de102004043748a1中描述的pom17c-质粒被用作起点,其中公开了质粒的完整序列(含有来自斯氏假单胞菌(pseudomonas stutzeri)ak61的cyanidase基因的序列)。保留该质粒上复制起点和氨苄青霉素抗性基因的序列,添加野生型地芽孢杆菌属的一个种pa-9hpab基因、野生型大肠杆菌hpac基因、食油假单胞菌(pseudomonas oleovorans)palkb启动子和大肠杆菌rrnb终止序列以及alks转录调节基因(yuste等人,j bacteriol.1998oct;180(19):5218-26)。使用地芽孢杆菌属的一个种野生型单切限制性内切酶agei和pmli[new england biolabs gmbh,br
ü
ningstraβe 50,geb.b852,
65926frankfurt am main]消化该质粒。使用hifi dna assembly master mix[new england biolabs gmbh br
ü
ningstrasse50,geb.b852 65926frankfurt am main]根据制造商的说明书用质粒主链克隆合成的dna片段[eurofins genomics germany gmbh anzinger str.7a de-85560ebersberg]。因此,该质粒含有大肠杆菌hpac和不同hpab变体。转化5-alpha electrocompetent e.coli[new england biolabs gmbh br
ü
ningstrasse 50,geb.b852 65926frankfurt am main]之后,将细胞铺板于含有100μg/ml的氨苄青霉素的lb-agar上。进行限制性分析和测序以正确选择克隆的质粒。如果提及,将质粒稳定性毒素-抗毒素序列hok/sok(thisted,embo j.1994apr 15;13(8):1950-9)和cer决定因子(summers&sherratt,1984,doi:10.1016/0092-8674(84)90060-6)添加到质粒以增加质粒稳定性。
[0149]
产l-酪氨酸菌株dpd6021-s用于转化所选质粒。dpd6021-s是dpd4145的s期变体,其记载于us 7700328 b2并且其中另外去除了pmt100质粒。为了进行质粒转化,将菌株接种到od
600
为0.05的来自lb过夜培养物,并培养至od
600
为0.7。冰上孵育30min之后,收获细胞(10min,5500g;4℃),用50ml的h2o
demin
洗涤两次。在用1ml预冷的10%(v/v)甘油进行额外的洗涤步骤后,将细胞重悬于200μl预冷的10%(v/v)甘油中,并使用40μl等分试样进行转化。因此,将100ng质粒转移到电穿孔比色皿中,与40μl细胞溶液混合,并在gene pulser xcell electorporation system[bio-rad laboratoriesgmbh,kapellenstraβe 12,d-85622feldkirchen]中在2500v,200ω&25μf进行负载。在加入1ml soc培养基并在37℃下再生45分钟后,将100μl悬浮液铺板至含有100μg/ml氨苄青霉素的lb-琼脂。从抗性菌落中分离出质粒,并通过限制性分析证实了质粒的真实性。
[0150]
产生了以下菌株:
[0151]
hpabc_ec:大肠杆菌hpab(野生型;参考);
[0152]
hpab_gs:地芽孢杆菌属的一个种pa-9hpab(野生型);
[0153]
hpab_v207l_gs:地芽孢杆菌属的一个种pa-9hpab(突变v207l);
[0154]
hpab_v207t_gs:地芽孢杆菌属的一个种pa-9hpab(突变v207t);
[0155]
hpab_v207q_gs:地芽孢杆菌属的一个种pa-9hpab(突变v207q);
[0156]
hpab_v207g_gs:地芽孢杆菌属的一个种pa-9hpab(突变v207g);
[0157]
hpab_v207t_k208r_gs:地芽孢杆菌属的一个种pa-9hpab(突变v207t_k208r);
[0158]
hpab_t206m_v207t_gs:地芽孢杆菌属的一个种pa-9hpab(突变t206m_v207t);
[0159]
hpab_t206a_v207t_gs:地芽孢杆菌属的一个种pa-9hpab(突变t206a_v207t).
[0160]
hpab_v207t_a210s_t211n_s212t:地芽孢杆菌属的一个种pa-9hpab(突变v207t_a210s_t211n_s212t)
[0161]
hpab_v207t_a210v_t211m_s212n:地芽孢杆菌属的一个种pa-9hpab(突变v207t_a210v_t211m_s212n)
[0162]
hpab_v207t_g213a_e214q_d215n:地芽孢杆菌属的一个种pa-9hpab(突变v207t_g213a_e214q_d215n)
[0163]
hpab_i152l_v207t:地芽孢杆菌属的一个种pa-9hpab(突变i152l_v207t)
[0164]
实施例2:生产l-dopa和l-酪氨酸(使用biolector筛选)
[0165]
在biolector[m2p-labs;arnold-sommerfeld-ring 2,52499baesweiler]小规模
测试系统中测试了具有表达地芽孢杆菌属的一个种pa-9基因的野生型和突变的变体的质粒的dpd6021-s菌株以及表达野生型基因的参考菌株。
[0166]
因此,将菌株在振荡摇瓶中在10ml lb培养基(100μg/ml氨苄青霉素)中在37℃、200rpm培养18小时。将培养物接种到含有1mllb培养基、ph 5.5(补充有7.5mm的l-酪氨酸;100μg/ml氨苄青霉素;0.25%(v/v)dcpk)的biolector flowerplates中,以得到0.1的起始od
600
。在37℃、1200rpm和85%的相对湿度下进行培养24小时,直到该过程停止,使用高效液相色谱(hplc)测量l-dopa和l-酪氨酸浓度。
[0167]
在agilent 1200(agilent technologies,palo alto,calif.)上进行hplc。使用inertsil ods-3,5μm,4.6x150 mm的柱子(agilent technologies)。所使用的方法要求柱流速为1.00ml/min,停止时间为18分钟。流动相由如表1所示的溶剂a(2.72g/l的kh2po4、2.5ml/l浓磷酸、40ml/l的乙腈)和溶剂b(乙腈)的比例组成。
[0168]
表1:hplc分析的流动相组成
[0169]
时间[min]溶剂a[%]溶剂b[%]010006.5100010752513752513.011000181000
[0170]
光谱从100nm扫描到380nm,在290nm处记录了l-dopa的信号,保留时间为2.8min,在278nm处记录了l-酪氨酸的信号,保留时间为3.9分钟。
[0171]
表2:使用biolector筛选用单个突变体生产l-dopa和l-酪氨酸
[0172] l-dopa[mm]l-酪氨酸[mm]ld-ec-12[hpabc_ec]2.75.4hpab_gs3.64.5hpab_v207q_gs4.04.0hpab_v207t_gs4.33.6hpab_v207l_gs4.43.8
[0173]
表3:使用biolector筛选用单个和多个突变体生产l-dopa和l-酪氨酸(质粒含有质粒稳定性元件)
[0174] l-dopa[mm]l-酪氨酸[mm]hpab_v207t_gs5.23.5hpab_t206m_v207t_gs6.42.2hpab_v207t_k208r_gs5.43.6hpab_v207g_gs4.44.4hpab_t206a_v207t_gs4.73.8hpab_v207t_a210s_t211n_s212t4.04.5hpab_v207t_a210v_t211m_s212n4.04.6
hpab_v207t_g213a_e214q_d215n3.84.7hpab_i152l_v207t4.05.0
[0175]
筛选结果总结在表2和表3中,并可视化于图1和图2中。图1显示了使用biolector筛选用单个突变体生产l-dopa和l-酪氨酸,图2显示了使用biolector筛选用单个和多个突变体生产l-dopa和l-酪氨酸(质粒含有质粒稳定性元件)。
[0176]
实施例3:生产l-dopa和l-酪氨酸(发酵方法)
[0177]
如us 7700328 b2的实施例8所述进行菌株dpd4145的发酵,并评估菌株的l-酪氨酸和l-dopa的产量。与us 7700328 b2的实施例8不同,没有用iptg而是用二环丙基酮(dcpk)诱导发酵,并且在氨苄青霉素(100mg/l)存在下在ph 6.8进行发酵。定期从发酵器中取出样品,并分析l-酪氨酸、l-dopa和生物质浓度。结果总结在表4-6中,表4显示了48h内l-dopa的产量,表5显示了相同发酵过程48h内的l-酪氨酸的产量,表6显示了l-dopa/l-酪氨酸的比例。结果可视化于相应的图3-4中。
[0178]
图3显示了使用发酵方法用大肠杆菌hpab(上)和地芽孢杆菌属的具体种hpab(下)48h内生产l-dopa和l-酪氨酸,图4显示了使用发酵方法用地芽孢杆菌属的具体种hpab v207t突变体(上)和地芽孢杆菌属的具体种hpab v207l突变体(下)48h内生产l-dopa和l-酪氨酸。
[0179]
表4:用发酵方法生产l-dopa的产量
[0180][0181]
表5:用发酵方法生产l-酪氨酸的产量
[0182][0183]
表6:用发酵方法生产的l-dopa/l-酪氨酸的比例
[0184][0185]
通过使用来自地芽孢杆菌属的hpab作为野生型酶,并且用不同的突变以发酵生产l-dopa 48h,可以清楚地表明,l-dopa/l-酪氨酸的比例高于使用大肠杆菌野生型酶时(表
6)。地芽孢杆菌属hpab酶的突变体甚至具有更高的l-dopa/l-酪氨酸的比例值。
[0186]
还在发酵方法中测试了其他突变体gs_v207t-t206m和gs_v207t-k208r,并获得了l-dopa/l-酪氨酸的比例值较高的相似结果。
[0187]
实施例5:结构同源性分析
[0188]
关于本发明的氧化还原酶,地芽孢杆菌属的一个种hpab序列用于在protein data bank(pdb)中搜索结构同源物。使用matt(menke,m.,berger,b.&cowen,l.matt:local flexibility aids protein multiple structure alignment.plos comput.biol.4,e10(2008))构建结构比对。
[0189]
本发明的氧化还原酶的这样的比对显示在图5中,其显示了地芽孢杆菌属的一个种hpab和2yyj(来自嗜热栖热菌的氧化还原酶)的比对。
[0190]
图6:地芽孢杆菌属的一个种hpab和4oo2(来自球孢链霉菌的氧化还原酶)的比对
[0191]
图7:地芽孢杆菌属的一个种hpab和1uv8(来自氨基丁酸梭状芽孢杆菌的氧化还原酶)的比对
[0192]
图8:地芽孢杆菌属的一个种hpab和3hwc(来自洋葱伯克霍尔德菌的氧化还原酶)的比对
[0193]
图9:地芽孢杆菌属的一个种hpab和4g5e(来自钩虫贪铜菌的氧化还原酶)的比对
[0194]
图10:地芽孢杆菌属的一个种hpab和4irn(来自颤藻属的一个种pcc 6506的氧化还原酶)的比对
[0195]
图11:地芽孢杆菌属的一个种hpab和5idu(来自瘤状副伯克霍尔德菌的氧化还原酶)的比对
[0196]
图12:地芽孢杆菌属的一个种hpab和6jhm(来自皮氏罗尔斯顿菌的氧化还原酶)的比对
[0197]
建模hpab结构的重合显示这些蛋白比对得很好。
[0198]
实施例6:生产具有不同的氧化还原酶突变体的大肠杆菌菌株
[0199]
用enyzme eari[new england biolabs gmbh br
ü
ningstrasse 50,geb.b852 65926frankfurt am main]对带有野生型地芽孢杆菌属的一个种pa-9hpab基因和野生型大肠杆菌hpac基因的实施例1中所述的pom17c质粒进行限制性酶切以去除野生型地芽孢杆菌属的一个种pa-9hpab基因以及palkb,alks序列和一部分大肠杆菌hpac序列。将所得质粒主链用合成的dna片段进行克隆,所述合成的dna片段含有先前去除的palkb和alks序列和一部分大肠杆菌hpac序列以及编码具有以下的氨基酸序列的酶的基因的野生型序列:seq id no:3(嗜热栖热菌)、seq id no:4(球孢链霉菌)、seq id no:5(氨基丁酸梭状芽孢杆菌)、seq id no:6(洋葱伯克霍尔德菌)、seq id no:7(钩虫贪铜菌)、seq id no:8(颤藻属的一个种pcc 6506)、seq id no:9(瘤状副伯克霍尔德菌)、seq id no:10(皮氏罗尔斯顿菌)。
[0200]
另外,将所述质粒主链用合成的dna片段进行克隆,所述合成的dna片段含有先前去除的palkb和alks序列和一部分大肠杆菌hpac序列以及得到活性增加的酶变体的基因的突变体序列,所述酶变体具有seq id no:21、seq id no:22、seq id no:23、seq id no:24、seq id no:25、seq id no:26、seq id no:27、seq id no:28的氨基酸序列。
[0201]
所得质粒用于转化如实施例1中所述的产l-酪氨酸菌株dpd6021-s。
[0202]
所得菌株在如实施例2所述的小规模筛选系统中培养或如实施例3所述发酵,以评
估表达突变酶变体的菌株的转化率。如实施例1所述定量l-dopa的形成。检测到所有变体的l-dopa产生。
[0203]
蛋白序列
[0204]
seq id no:1地芽孢杆菌属的一个种pa9 hpab
[0205]
seq id no:2大肠杆菌(hpab)4-羟基苯乙酸3-单加氧酶加氧酶成分
[0206]
seq id no:3嗜热栖热菌
[0207]
seq id no:4球孢链霉菌
[0208]
seq id no:5氨基丁酸梭状芽孢杆菌
[0209]
seq id no:6洋葱伯克霍尔德菌
[0210]
seq id no:7钩虫贪铜菌
[0211]
seq id no:8颤藻属的一个种pcc 6506
[0212]
seq id no:9瘤状副伯克霍尔德菌
[0213]
seq id no:10皮氏罗尔斯顿菌
[0214]
seq id no:11地芽孢杆菌属的一个种pa9 hpab v207l
[0215]
seq id no:12地芽孢杆菌属的一个种pa9 hpab v207t
[0216]
seq id no:13地芽孢杆菌属的一个种pa9 hpab v207q
[0217]
seq id no:14地芽孢杆菌属的一个种pa9 hpab v207g
[0218]
seq id no:15地芽孢杆菌属的一个种pa9 hpab t206m
[0219]
seq id no:16地芽孢杆菌属的一个种pa9 hpab t206a
[0220]
seq id no:17地芽孢杆菌属的一个种pa9 hpab k208r
[0221]
seq id no:18地芽孢杆菌属的一个种pa9 hpab v207t_k208r
[0222]
seq id no:19地芽孢杆菌属的一个种pa9 hpab t206m_v207t
[0223]
seq id no:20地芽孢杆菌属的一个种pa9 hpab t206a_v207t
[0224]
seq id no:21嗜热栖热菌(已突变)
[0225]
seq id no:22球孢链霉菌(已突变)
[0226]
seq id no:23氨基丁酸梭状芽孢杆菌(已突变)
[0227]
seq id no:24洋葱伯克霍尔德菌(已突变)
[0228]
seq id no:25钩虫贪铜菌(已突变)
[0229]
seq id no:26颤藻属的一个种pcc 6506(已突变)
[0230]
seq id no:27瘤状副伯克霍尔德菌(已突变)
[0231]
seq id no:28皮氏罗尔斯顿菌(已突变)
[0232]
seq id no:29地芽孢杆菌属的一个种pa9 hpab v207t_a210s_t211n_s212t
[0233]
seq id no:30地芽孢杆菌属的一个种pa9 hpab v207t_a210v_t211m_s212n
[0234]
seq id no:31地芽孢杆菌属的一个种pa9 hpab v207t_g213a_e214q_d215n
[0235]
seq id no:32地芽孢杆菌属的一个种pa9 hpab i152l_v207t
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1