一组预测肝癌预后联合标志物及其应用的制作方法
一组预测肝癌预后联合标志物及其应用
发明领域
1.本发明属于生物医药领域,具体涉及一组预测肝癌预后的联合标志物及其应用,具体涉及一组新的转录因子家族e2f相关的基因集,该基因集可以作为肝癌的预后标志物。
背景技术:
2.肝癌是临床上最常见的恶性肿瘤之一,死亡人数在所有癌症中位居第三。早期难以发现,70%以上的肝癌患者在晚期确诊,因此,肝癌患者的预后极差。此外,传统的可识别的临床和病理症状在预测肝癌预后方面存在很大缺陷,为了延长肝癌患者的总体存活率,需要寻找更好发预测预后的新方法。
3.肝癌作为一种异质性疾病,并非由单个基因或其产物所决定,越来越多的文献报道,来自患者肿瘤组织的多基因预后特征比单基因更能准确地预测癌症患者的预后,特别是mrna的多基因预后特征比非编码预后基因具有更好的预后准确性,可以提供更有效的个体化治疗。然而,目前在肝癌中尚缺乏mrna联合生物标志物对肝癌预后的研究。因此,寻找有效的联合生物标志物对于评估肝癌的预后具有重要的意义。
4.e2f是编码一系列转录因子、具有多功能的转录因子家族。目前已报道e2f家族可通过结合共识dna结合序列参与调控肿瘤细胞周期、dna损伤反应、细胞分化和细胞死亡,从而影响肿瘤细胞的生长和侵袭。大量的证据表明,在多种癌症类型中,e2f通过控制其下游的靶标因子参与肿瘤的发生发展。本发明公开的联合生物标志物可用于肝癌患者的预后判断,对我国肝癌的治疗与预后判断现状具有显著意义。
技术实现要素:
5.鉴于在预测肝癌预后的现有技术中缺乏足够的生物标志物,本发明提供一组用于预测肝癌预后的联合生物标记物及其确立方法和应用。为实现该目的,将进行如下说明:
6.第一方面,本发明提供了一组用于预测肝癌预后的联合生物标记物,所述联合生物标记物包括cdca3,cdca8,hn1,kif4a以及ssrp1;所述联合标志物由risk score表征:risk score=0.3915*expression of gene hn1-0.3864*expression ofgene kif4a-0.2886*expression ofgene cdca3+0.4415*expression ofgene cdca8+0.8842*expression ofgene ssrp1。
7.第二方面,本发明提供了将上述联合生物标记物用于预测肝癌预后的方法,所述方法包括如下步骤:
8.(1)从tcga数据库,检索肝癌患者的癌组织及癌旁组织的rna-seq测序数据,并下载患者的临床病理资料;
9.(2)利用gsea功能富集分析,筛选肝癌组织和癌旁正常组织中存在差异的基因集:采用gsea功能富集分析,以|nes|》1并且nom p-val《0.05为标准,选取具有显著统计学差异的基因集,用于确定肝癌治疗中有价值的标志性联合生物标志物;nes代表归一化后的富集分析评分,nom p-val代表校正后的p value,表征富集结果的可信度;其中,转录因子e2f基
因集|nes|=2.071552,nom p-val=0.001961,是在肝癌组织和癌旁正常组织中差异最大的基因集,并对其进行了进一步的分析;
10.(3)单因素cox筛选差异基因集中影响预后的基因:利用单因素cox回归分析,筛选出差异基因集中,影响肝癌患者预后的基因,以p《0.05为标准;
11.(4)多因素cox构建肝癌预后的风险模型:从单因素cox分析结果中筛选p《0.001的预后基因,采用多因素cox回归分析模拟并建立肝癌的预后模型,最终筛选出cdca3、cdca8、ssrp1、hn1、kif4a构建预测肝癌患者预后的风险模型;对所选基因的表达水平进行加权,与多因素cox回归分析得到的回归系数进行线性积分,风险评分=0.3915*expression ofgene hn1-0.3864*expression of gene kif4a-0.2886*expression of gene cdca3+0.4415*expression of gene cdca8+0.8842*expression ofgene ssrp1,风险评分公式可用于计算每个肝癌患者的风险值,根据风险值的大小可以预测肝癌患者的预后;
12.(5)预后模型中基因的异常表达:利用tcga数据库和geo数据库中肝癌组织、正常肝组织的rna-seq数据,采用配对以及非配对t检验,对比cdca3、cdca8、ssrp1、hn1和kif4a在肝癌组织及正常肝组织中的表达量的差异;
13.(6)风险模型准确性的验证:使用roc曲线和kaplan
–
meier(k-m)曲线评估模型的准确性;roc曲线下面积反映该预后模型的准确性和特异性,k-m曲线反映高风险组和低风险组患者预后的差异,以p《0.05为标准确定是否具有统计学意义;
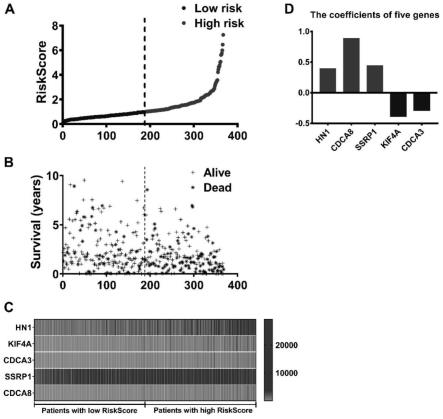
14.(7)收集肝癌组织和癌旁正常肝组织,通过实时荧光定量pcr检测预后模型中基因cdca3、cdca8、ssrp1、hn1和kif4a在肝癌组织和癌旁正常肝组织中的表达差异;
15.(8)统计分析:数据显示为平均值
±
sd/sem,p值小于0.05认为是统计学有差异;
16.其中,所述步骤(1)中,检索数据并处理rna序列数据,具体为:从tcga下载了422例肝癌组织和88例癌旁正常肝组织的rna-seq数据和临床数据,网址如下:https://portal.gdc.cancer.gov/。
17.第三方面,本发明提供一组用于预测肝癌预后的标记物,该标记物含有上述的一组用于预测肝癌预后的生物标记物,该标记物在制备辅助判断肝癌预后试剂盒中的应用。
18.最后,本发明还提供一种辅助判断肝癌预后的试剂盒,该试剂盒含有上述一组用于预测肝癌预后的联合生物标记物。
19.有益效果
20.本发明提供一种联合生物标志物及其作为肝癌预后预测的方法,区别于单基因生物标志物,有更准确、有效的优点,将大大提高肝癌预后判断的准确性。总体生存分析显示,联合生物标志物中cdca3,cdca8,hn1,kif4a以及ssrp1的基因表达水平高,患者的总生存时间缩短,roc曲线下面积为0.755,表明上述联合生物标志物具有较高的灵敏度和准确性,因此,由这5个基因组成的联合生物标志物可作为优异的肝癌预后生物标志物。
21.附图1为实施例3中肝癌预后模型的构建。其中,图1a是患者由低到高的风险评分;图1b横坐标是患者评分由低到高,纵坐标是患者的生存时间,星号*和加号+分别代表患者的生存状态为死亡和存活;图1c的横坐标是按照患者的风险评分依次升高,该图代表随着患者风险评分的升高模型中5个基因的表达情况;图1d是模型公式中5个基因表达量的系数值。
22.附图2为实施例4中非配对以及配对t检验检测这5个基因在tcga数据库,肝癌组织
和癌旁正常肝组织中的表达量差异。其中,图2a-2e是模型中5个基因在肝癌组织和癌旁正常肝组织表达量的非配对t检验的结果,图2f-2j是模型中5个基因在肝癌组织和癌旁正常肝组织表达量的配对t检验的结果;非配对t检验和配对t检验结果均表明模型中5个基因在肝癌组织中存在异常高表达,且具有统计学意义。
23.附图3为实施例4中非配对以及配对t检验检测这5个基因在geo数据库,肝癌组织和癌旁正常肝组织中的表达量差异。其中,图3a-3e是模型中5个基因在肝癌组织和癌旁正常肝组织表达量的非配对t检验的结果,图3f-3j是模型中5个基因在肝癌组织和癌旁正常肝组织表达量的配对t检验的结果;非配对t检验和配对t检验结果均表明模型中5个基因在肝癌组织中存在异常高表达,除ssrp1的配对t检验无统计学意义外,其它结果均有统计学意义;分析原因是可能由于样本量少导致的。
24.附图4为实施例5中roc曲线和kaplan-meier生存曲线验证预后模型的准确性和特异性。其中,4a是高风险评分患者和低风险评分患者的kaplan-meier生存曲线,高风险评分患者的预后明显差于低风险评分患者;模型的roc曲线下面积为0.755,证明该模型在预测患者预后方面具有很好的敏感性和特异性。
25.附图5为实施例6中,肝癌预后模型中的基因在临床样本中表达量的验证。通过提取临床肝癌组织和癌旁正常组织中的总rna,分别进行逆转录和qrt-pcr检测模型中5个基因在临床肝癌组织和癌旁正常肝组织中的表达量,实验结果和生物信息学分析的结果一致,模型中的5个基因在肝癌组织中均存在显著高表达。
具体实施方式
26.为了使本发明的目的、技术方案及优点更加清楚明白,以下结合实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅用以解释本发明,并不用于限定本发明。
27.本发明适用的数据包括转录组数据和临床数据,适用标本包括冻存在-80℃的组织标本。
28.实施例1tcga数据库rna-seq测序数据和患者临床病理资料的下载
29.从tcga数据库下载肝癌患者的肝癌组织、癌旁正常组织的rna-seq测序数据,并下载肝癌患者的临床病理资料,下载网址为:https://portal.gdc.cancer.gov/。tcga数据库中,422例肝癌患者具有临床病理资料;373例肝癌患者有肝癌组织的rna-seq测序数据。既有临床病理资料又有肝癌组织测序数据的肝癌患者总计369例,其中,50例肝癌患者具有肝癌组织和癌旁正常肝组织的配对的rna-seq测序数据。由于mrna的表达谱数据已经通过tcga标准化,因此不对这些数据进行进一步的标准化,肝癌患者病理参数如表1所示:
30.表1.肝癌患者的临床病理参数
[0031][0032]
实施例2肝癌患者中差异表达基因集的筛选
[0033]
采用gsea 4.1.0版,利用肝癌组织和癌旁正常肝组织的rna-seq测序数据,分析肝癌组织和癌旁正常肝组织中差异表达的基因集。以|nes|》1.5,nom p-val《0.05为标准筛选在肝癌组织中异常表达的基因集,用于确定肝癌治疗中对预后有预测价值的基因集;|nes|代表归一化后的富集分析评分,nom p-val代表校正后的p value,表征富集结果的可信度;其中,转录因子e2f基因集中含有197个基因,其|nes|=2.071552,nom p-val=0.001961,是在肝癌组织和癌旁正常组织中差异最大的基因集(表2)。
[0034]
表2.肝癌患者中异常表达的基因集
[0035][0036]
实施例3肝癌预后模型的构建
[0037]
利用单因素cox回归分析,从gsea筛选出的差异基因集中筛选影响肝癌患者预后,且p《0.001的基因,共筛选出20个基因;采用多因素cox回归分析建立预后相关模型,最终筛选出hn1、kif4a、cdca3、cdca8及ssrp1构建预测肝癌患者预后的风险模型,所选预后基因的多变量cox生存分析的详细结果如表3所示。风险评分=0.3915*expression of gene hn1-0.3864*expression of gene kif4a-0.2886*expression of gene cdca3+0.4415*expression of gene cdca8+0.8842*expression of gene ssrp1。在构建的风险模型中,b(cox)是相应基因表达量的系数,p value是基因多因素cox生存分析影响预后的p值,hr是基因的风险系数(表3)。根据风险评分中位数将肝癌患者分为低危组和高危组(图1a),本发明发现高危组总生存时间较低危组短,死亡人数较高(图1b)。
[0038]
表3.肝癌预后模型中基因的多因素cox分析结果
[0039][0040][0041]
实施例4肝癌预后模型中基因在肝癌组织和癌旁正常组织中的表达差异
[0042]
采用tcga以及geo数据库,使用非配对以及配对t检验在肝癌组织以及正常组织中分析预后模型中5个基因表达量的差异,结果显示筛选的5个基因在肝癌组织中的表达均高于相邻正常组织(图2、图3)。
[0043]
实施例5roc曲线和kaplan-meier生存曲线验证预后模型的准确性和特异性
[0044]
将肝癌患者按风险评分中位数分为高低风险组,构建roc曲线,roc曲线下面积为0.755,表明风险评分在预测肝癌患者预后方面具有较高的特异性和敏感性(图4b)。该结果证明预后模型在预测肝癌患者预后方面具有较好的准确性和特异性。
[0045]
利用kaplan-meier生存曲线,分析高低风险组患者的预后,低危组患者的生存时间明显优于高危组(图4a)。对肝癌患者的预后进行分层分析,以确定风险评分的有效性。该结果表明风险值高的患者的预后较差,预后模型可以很好的预测肝癌患者的预后。
[0046]
实施例6肝癌预后模型中的基因在临床样本中表达量的验证(图5)
[0047]
(1)肝癌肿瘤组织及配对正常组织样本的获得及总rna提取
[0048]
获得经手术分离的肝癌肿瘤组织样本21个,癌旁正常组织样本21个,用聚合美生物科技公司的trigent试剂提取总的rna,对提取的总rna进行定量。
[0049]
(2)实时荧光定量rt-pcr检测cdca3、cdca8、hn1、kif4a以及ssrp1在肝癌肿瘤样本中的表达。
[0050]
利用实时荧光定量rt-pcr检测步骤(1)中获得的21个肝癌肿瘤样本及21个癌旁正常组织样本中5个基因的表达情况,具体步骤如下:
[0051]
1)rna反转录:使用艾科瑞生物工程有限公司的反转录试剂盒(evo m-mlv mix kit with gdna clean for qpcr,货号:ag11706-s)进行rna样本的反转录反应,具体按照试剂盒说明书的方法进行,步骤如下:
①
去除基因组dna,取1微克提取的总rna样本,加入2μl 5
×
gdna clean reaction mix,3μl rnase free water,混匀后置42℃,反应2分钟;
②
反转录反应,在
①
的反应液中加入4μl 5
×
evo m-mlv rt reaction mix,6μl rnase free water,总体积为20μl;混匀后短暂离心,置于bio-rad t100 thermal cycler仪中进行反转录反应,反应参数为37℃,15分钟;85℃,5秒;随后置于4℃保存。
[0052]
2)实时荧光定量pcr:模型中基因的引物设计来源于生工生物工程有限公式,使用艾科瑞生物工程有限公司的green premix pro taq hs qpcr kit(货号:ag11701)对样本中的cdca3,cdca8,hn1,kif4a以及ssrp1的表达进行定量检测,具体方法按试剂盒的说明书进行,步骤如下:取2μl逆转录产物,加入5μl 2
×
sybr green pro taq hs premix,0.4μl(10μm)上游引物,0.4μl(10μm)下游引物,最后加入2.2μl rnase free water,反应总体积为10μl。混匀后短暂离心,置于bio-rad cfx96 optics module实时定量pcr仪中进行pcr扩增反应,反应参数为95℃预变性30秒,95℃变性5秒,60℃退火延伸30秒;循环次数为40个循环。每个反应设置3个重复;
[0053]
表4.肝癌预后模型中基因的引物序列如下:
[0054][0055]
3)数据分析:对同一样本分别检测其中目标rna及内参rna的表达;以内参rna的表达量为基准,对目标rna的表达进行归一化处理;随后采用通常使用的delta delta ct法对目标rna的表达量进行定量,本发明的内参为gapdh。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1