一种单细胞全序列转录组文库构建的方法与流程
1.本发明属于基因测序领域,具体涉及一种单细胞全序列转录组文库构建的方法。
背景技术:
2.高通量单细胞rna检测主要有基于油包水的液滴区隔技术、基于微孔板的beads标记技术以及微流控三种实现方式。基于油包水的液滴区隔技术以10x genomics、drop-seq平台及indrop平台为代表。该类技术通过微流控技术将barcode标记的微珠和单个细胞包裹在单个油滴中并裂解释放含有polya尾巴的rna;每一个凝胶微珠偶联了含有细胞标签和分子标签的oligo dt核酸序列;mrna结合到含有细胞标签和分子标签的oligo dt核酸分子后通过逆转录给不同细胞来源的cdna标记上不同的细胞标签并用于以后的混合建库并测序分析。基于微孔板的beads标记技术以bd cytoseq、seqwell及microwell-seq为代表。该技术将细胞自然沉降至细胞数量十倍以上的微孔阵列中保证单细胞入孔率,然后在微孔中加入细胞标签标记的微珠用以捕获细胞裂解后的mrna;mrna结合到含有细胞标签和分子标签的逆转录引物后通过逆转录给不同细胞来源的cdna标记上不同的细胞标签并用于以后的混合建库并测序分析。
3.以上提到的高通量单细胞rna建库技术都只能实现靠近rna特定3’尾巴端或者5’起始端的碱基序列检测,而不能有效的在高通量单细胞层面实现对rna全长序列的分析。
技术实现要素:
4.有鉴于此,本发明提出一种单细胞全序列转录组文库构建的方法,以得到在cdna任一位置标记的cdna,构建cdna全序列文库。
5.为达到上述目的,本发明提供一种单细胞全序列转录组文库构建的方法,所述单细胞全序列转录组文库构建的方法包括:
6.提供多个细胞的目标mrna;
7.采用逆转录引物对各细胞的所述目标mrna进行逆转录,得到各细胞的cdna第一链;
8.采用cdna第二链引物对对以各细胞的所述cdna第一链为模板合成各细胞的多个cdna第二链片段,其中,所述cdna第二链引物对包括正向引物和反向引物,所述正向引物包括第一序列和随机序列,所述反向引物包括第二序列和固定序列,所述第二序列与所述第一序列互补;
9.将多个细胞的多个所述cdna第二链片段与多个标签体一一对应连接,得到多个标记cdna片段,其中,各所述标签体包括细胞标签、分子标签和连接序列,所述连接序列与所述固定序列互补,任意两个所述标签体的所述分子标签不同,同个细胞的多个所述cdna第二链片段连接的多个所述标签体的所述细胞标签相同,不同细胞的多个所述cdna第二链片段连接的多个所述标签体的所述细胞标签不同;
10.将多个所述标记cdna片段进行富集后,并引入文库标签,得到所述单细胞全序列
转录组文库。
11.可选地,所述提供多个细胞的目标mrna的步骤包括:
12.提供多个检测区域和多个待检测细胞;
13.使多个所述待检测细胞在多个所述检测区域一一对应沉积;
14.将沉积后的所述待检测细胞采用捕获体进行目标mrna富集。
15.可选地,多个所述检测区域设置在一个检测区域集成体上,且在所述检测区域集成体间隔排布,各所述检测区域的内径为10~200um的检测孔。
16.可选地,所述采用逆转录引物对目标mrna进行逆转录,得到与目标mrna互补的cdna第一链的步骤包括:
17.采用连接有所述逆转录引物的捕获体富集目标mrna,并对富集的目标mrna进行逆转录。
18.可选地,所述逆转录引物为oligo-(dt)n,n的数量在10~100之间。
19.可选地,所述将多个细胞的多个所述cdna第二链片段与多个标签体一一对应连接,得到多个标记cdna片段的步骤包括:
20.提供含有所述标签体的多种功能微珠,每种所述功能微珠包括标签本体,与所述标签本体连接的多个断点,各所述标签体的所述细胞标签与所述分子标签共同构成各标记段,所述连接序列连接在所述标记段的一端,多个含有相同细胞标签的所述标记段的另一端与多个所述断点一一对应连接,任意两种所述功能微珠的所述标签体的所述细胞标签不同,且所述标签本体与所述标记段可在所述断点条件性断开;
21.将同一细胞的多个所述cdna第二链片段与一种所述功能微珠混合,不同细胞的多个所述cdna第二链片段与不同所述功能微珠混合;
22.断开所述标签本体与所述标记段;
23.将所述连接序列与所述cdna第二链片段连接,得到多个所述标记cdna片段。
24.可选地,所述断点为可在紫外光照条件下断裂的pc linker。
25.可选地,所述标签体还包括引物结合序列;所述将多个所述标记cdna片段进行富集后,并引入文库标签,得到所述单细胞全序列转录组文库的步骤包括:
26.将各所述标记cdna片段经过单端接头连接后,加入与所述引物结合序列结合的扩增引物,进行富集及建库,得到所述单细胞全序列转录组文库。
27.可选地,所述标签体还包括引物结合序列;所述将多个所述标记cdna片段进行富集后,并引入文库标签,得到所述单细胞全序列转录组文库的步骤包括:
28.将各所述标记cdna片段依次经过杂交、延伸后,加入与所述引物结合序列结合的扩增引物,进行index扩增,得到所述单细胞全序列转录组文库。
29.本发明中,采用带有随机序列的正向引物合成多个cdna第二链片段,通过粘性末端在每个cdna第二链片段连接分子标签,同时在不同细胞来源的cdna片段上连接不同的细胞标签,以此将不同的细胞进行分辨,实现了高通量单细胞全序列测序。
附图说明
30.为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅为本
发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
31.图1为本发明单细胞全序列转录组文库的构建流程图;
32.图2为本发明单个cdna片段结构图;
33.图3为本发明一实施例的捕获体的结构示意图;
34.图4为本发明一实施例的标签体的结构示意图;
35.图5为本发明实施例1单细胞全序列转录组文库的构建流程图;
36.图6为本发明实施例1获得文库片段大小测试图;
37.图7为本发明实施例1核酸序列测试图。
38.本发明目的的实现、功能特点及优点将结合实施例,参照附图做进一步说明。
具体实施方式
39.为使本发明实施例的目的、技术方案和优点更加清楚,下面将对本发明实施例中的技术方案进行清楚、完整地描述。显然,所描述的实施例仅仅是本发明的一部分实施例,而不是全部的实施例。
40.需要说明的是,实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。另外,全文中出现的“和/或”的含义,包括三个并列的方案,以“a和/或b”为例,包括a方案、或b方案、或a和b同时满足的方案。此外,各个实施例之间的技术方案可以相互结合,但是必须是以本领域普通技术人员能够实现为基础,当技术方案的结合出现相互矛盾或无法实现时应当认为这种技术方案的结合不存在,也不在本发明要求的保护范围之内。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
41.鉴于现有技术中,无法在高通量层面进行cdna全长序列的测序,而这个问题的主要原因在于目前的细胞标签都只能标记在cdna全长的3’或者5’端。
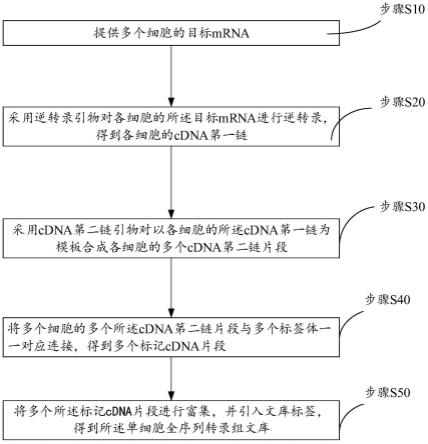
42.本发明提出一种单细胞全序列转录组文库的构建方法,如图1所示,包括以下步骤:
43.步骤s10:提供多个细胞的目标mrna;
44.步骤s20:采用逆转录引物对各细胞的所述目标mrna进行逆转录,得到各细胞的cdna第一链;
45.步骤s30:采用cdna第二链引物对对以各细胞的所述cdna第一链为模板合成各细胞的多个cdna第二链片段,其中,所述cdna第二链引物对包括正向引物和反向引物,所述正向引物包括第一序列和随机序列,所述反向引物包括第二序列和固定序列,所述第二序列与所述第一序列互补;
46.步骤s40:将多个细胞的多个所述cdna第二链片段与多个标签体一一对应连接,得到多个标记cdna片段,其中,各所述标签体包括细胞标签、分子标签和连接序列,所述连接序列与所述固定序列互补,任意两个所述标签体的所述分子标签不同,同个细胞的多个所述cdna第二链片段连接的多个所述标签体的所述细胞标签相同,不同细胞的多个所述cdna第二链片段连接的多个所述标签体的所述细胞标签不同;
47.步骤s50:将多个所述标记cdna片段进行接头连接和富集,得到所述单细胞全序列转录组文库。
48.本发明中,采用带有随机序列的正向引物合成如图2所示的多个cdna第二链片段,图2中,1为第一序列,2为随机序列,3为逆转录产物,4为第二序列,5为固定序列形成的粘性末端,通过粘性末端在cdna第二链片段连接细胞标签和分子标签,以此,在高通量层面,其构建的单细胞全序列转录组文库可在cdna的任意基因的位置进行测序。
49.具体地,在本发明中,第一序列的3’端连接随机序列后,可以形成所述正向引物,其随机序列可以是6-20个atgc四种碱基核苷酸的随机组合dn(6-20),如nnnnnn(n代表atcg的任一碱基核苷酸),当然,也可以在随机序列中固定几个碱基,而除固定部分其余也为atcg的任一碱基核苷酸的随机组合;例如random primer-f:gcttgtgtcttttnnnnnn,序列中,nnnnnn用于起始逆转录反应的随机引物,gcttgtgtctttt为互补配对序列,第二序列的3’端连接固定序列,使其最终形成的cdna第二链其5’端具有粘性末端,因此在每个cdna片段的5’端连接有细胞标签和分子标签,例如:
50.random primer-r:
51.aaaagacacaagcaaaaaaaaaaaaaaaaaa,序列中aaaagacacaagc用于与random primer-f中的gcttgtgtctttt互补配对;序列:aaaaaaaaaaaaaaaaaa作为粘性末端与cell barcode中的ployt互补配对,介导连接反应,对应的连接序列为ployt。
52.第一序列和所述第二序列是四种碱基中任意三种碱基的随机组合。所述第一序列和所述第二序列的长度范围17~50个核苷酸,优选地,其长度可以是17-25个核苷酸之间。所述第一序列和所述第二序列之间互补形成的双链体的解链温度(tm)在40~80℃,进一步优选为55~70℃。
53.在一些实施例中,所述提供多个细胞的目标mrna步骤包括:
54.步骤s101:提供多个检测区域和多个待检测细胞;
55.步骤s102:使多个所述待检测细胞在多个所述检测区域一一对应沉积;
56.步骤s103:将沉积后的所述待检测细胞采用捕获体进行目标mrna富集。
57.通过将在划分区域进行不同的细胞的逆转录文库构建,在提高效率的同时,避免不同细胞的污染。具体地,多个所述检测区域设置在一个检测区域集成体上,且在所述检测区域集成体间隔排布,各所述检测区域的内径为10~200um的检测孔。在本发明中,所述不同的检测区域集成在微孔芯片,该微孔芯片可以含有100个到10000000个微孔,同一张芯片的每个微孔的直径是相同的,其直径可以在10-200um之间。投入微孔芯片的细胞数不应超过芯片微孔数的1/5,可以通过重力使细胞自然沉降到孔内。在使用时,其单个细胞均在单一的微孔中进行,避免的不同细胞rna的污染。
58.在一些实施例中,采用连接有所述逆转录引物的捕获体富集目标mrna,并对富集的目标mrna进行逆转录。如图3所示,所述捕获体包括捕获本体及连接在所述捕获本体的捕获序列,图3中,1为捕获本体,2为逆转录引物。
59.本发明中,所述逆转录引物为oligo-(dt)n,n的数量在10~100之间,采用oligo-(dt)可以与真核细胞的特有的polya进行互补,以此实现对真核细胞的mrna进行富集的同时并可对其逆转录。
60.在一些实施例中,所述采用捕获体将目标mrna进行富集的步骤包括:采用细胞裂
解液将细胞裂解后,捕获体进行目标mrna富集。具体地,在微孔芯片中依次加入细胞和捕获体,后采用细胞裂解液裂解细胞释放mrna,捕获体对目标mrna进行富集。通过采用细胞裂解液进行细胞裂解,可以使细胞中的mrna完全释放。
61.需要说明的是,在可连接捕获序列的前提下,捕获本体其材质选择不受限制,具体至本发明中,捕获本体的材质为磁珠,可采用偶联反应与捕获序列进行偶联。
62.在一些实施例中,所述步骤s30包括:
63.步骤s301:将所述cdna第二链引物对与杂交液混合,进行杂交反应,得到待延伸体系;
64.步骤s302:往所述待延伸体系中加入延伸液,进行延伸反应后,得到多个cdna第二链片段。
65.在本步骤中,由于采用的随机引物,因此与cdna互补碱基较少,所以先在高盐条件下进行,提高引物与模板的结合率;结合后去除高盐buffer,加入延伸反应液,进行二链合成。具体地,在本发明中的杂交和延伸方法均是给基于现有杂交和延伸操作,具体可参考专利cn 114096678 a。进一步,在本发明中,采用的是北京寻因科技公司的mm试剂盒中的试剂进行的杂交和延伸。
66.在一些实施例中,所述步骤s40包括:
67.步骤s401:提供含有所述标签体的多种功能微珠,每种所述功能微珠包括标签本体,与所述标签本体连接的多个断点,各所述标签体的所述细胞标签与所述分子标签共同构成各标记段,所述连接序列连接在所述标记段的一端,多个含有相同细胞标签的所述标记段的另一端与多个所述断点一一对应连接,任意两种所述功能微珠的所述标签体的所述细胞标签不同,且所述标签本体与所述标记段可在所述断点条件性断开;
68.步骤s402:将同一细胞的多个所述cdna第二链片段对应与一种所述功能微珠混合,不同的多个所述cdna第二链片段对应与不同所述功能微珠混合;
69.步骤s403:断开所述标签本体与所述标记段;
70.步骤s404:将所述连接序列与所述cdna第二链片段连接,得到多个所述标记cdna片段。
71.通过引入标签本体可以负载大量的细胞标签,且可以提高结合率。具体地,在本发明中,一个标签本体负载105~10
10
个标记段。进一步,其条件性断开是指在施加一定的条件下,标记段与标记本体在断点处进行断裂,具体地双硫修饰、du修饰、rna碱基修饰、di修饰、dspacer修饰、ap位点修饰、光断裂pc linker以及限制性内切酶识别序列。进一步,在本发明中,所述断点为可在紫外光照条件下断裂的pclinker。
72.在一些实施例中,所述标签体还包括引物结合序列,具体地,所述引物结合序列在标记段与断点之间或者在连接头201与标记段之间;
73.需要说明的是,本发明采用富集以及文库标签的引入其方法不限,具体地,在本发明中,所述标签体还包括引物结合序列;步骤s50包括:
74.将各所述标记cdna片段经过单端接头连接后,加入与所述引物结合序列结合的扩增引物,进行富集及建库,得到所述单细胞全序列转录组文库,具体地,在cdna第二链片段的3’端进行单端接头连接,为后续扩增提供另一端引物结合位点。或,
75.将各所述标记cdna片段依次经过杂交、延伸后,加入与所述引物结合序列结合的
扩增引物,进行index扩增,得到所述单细胞全序列转录组文库。
76.在一些实施例中,所述功能微珠的具体结构如图4所示,包括标签本体1,其标签本体负载的功能序列2,其中,每条功能序列依次包括连接的连接头201、断点202、引物结合序列203、细胞标签204、分子标签205和连接序列206,其中,连接头与所述标签体连接,一个功能磁珠上的分子标签205不同,但细胞标签204相同。通过在一条功能序列中集成不同作用的序列,可以得到反应集成体,以此,更加提高文库的构建效率。例如:功能序列设计为:
77.acactctttccc/pclinker/tacacgacgctcttccgatctnnnnnnnnactggtgannnnnnnnggtagtgacannnnnnnnnnnnnnnntttttttttttttttttt,序列中,acactctttccc为接头序列增加序列与beads间间隔;tacacgacgctcttccgatct用于连接后cdna扩增的引物结合序列,nnnnnnnnactggtgannnnnnnnggtagtgacannnnnnnn细胞标签,nnnnnnn分子标签,tttttttttttttttttt与cdna中的polya互补配对,介导连接,其中,每个n和n均选自atcg中的任一碱基。
78.需要说明的是,本发明的所述的标签本体在可以进行连接序列的前提下,其材质不限,可以是任何硬质材料或软质材料,如为固体珠子、半固态水凝胶珠等等。
79.以下结合具体实施例和附图对本发明的技术方案作进一步详细说明,应当理解,以下实施例仅仅用以解释本发明,并不用于限定本发明。
80.实施例1
81.本实施例提供一种pbmc的单细胞全序列转录组文库的构建及测序分析,具体操作如图5所示:
82.1.抽取外周血并获得新鲜的pbmc细胞,重悬于pbs中;
83.2.提供微孔芯片,该微孔芯片可以含有100个到10000000个微孔,同一张芯片的每个微孔的直径是相同的,其直径可以在10-200um之间。
84.3.按照mm 3’单细胞转录组文库构建试剂盒中提供的微孔芯片说明书处理芯片,并将3000个孵育好的pbmc细胞投入到微孔芯片中;
85.4.加入350μl经pbs洗涤的偶联有oligo(dt)25的3um直径磁珠至微孔板中,磁吸入孔后清洗掉多余磁珠;
86.5.加入mm 3’试剂盒中自带的裂解液裂解2min;
87.6.按照mm 3’单细胞文库构建试剂盒中说明配置逆转录体系400ul;
88.试剂400μl体系buffera-t80μldntp16μlenzyme a-t40μl0.1m dtt20μlrnase-free water补充至400μl
89.并取350μl配置好的逆转录反应体系加入微孔板中,封口后置于以下温度条件反应:
90.步骤温度时间137℃30min
91.7.逆转录反应完成后使用pbs冲洗3次,加入mm 3’试剂盒中的buffer c,与试剂盒不同的是将bufferc中的引物更换为自制随机引物,随机引物为以下两个序列的退火产物。
[0092][0093][0094]
置于恒温反应器中,运行如下程序:
[0095]
温度时间95度5min37度5min25度15min
[0096]
8.反应完成后使用pbs冲洗3次,按照mm 3’单细胞文库构建试剂盒说明书配置400μl如下反应体系:
[0097]
试剂400μl体系bufferd376μlenzymec16μlenzymed8μl
[0098]
取350μl配制好mix加入到步骤11的产物中,于恒温反应器上进行反应,设置如下:
[0099]
温度时间37度10min53度30min
[0100]
9.二链延伸反应完成后使用pbs冲洗3次,加入cell barcoded beads。此处加入cell barcoded beads,在cell barcoded beads偶联有具体序列如下所示的序列:
[0101][0102]
10.按以下体系配置连接反应体系并加入到微孔板中,使用紫外光裂解1min后置于22℃反应15min;
[0103]
试剂400μl体系2x rapid ligation buffer200μlt4 dna ligase12μlrnase-free water补充至400μl
[0104]
11.从微孔板中取出磁珠,高温变性后取上清使用1.6
×
dna clean beads纯化,15
μl low-edta te洗脱,以得到细胞标签标记的cdna二链产物。
[0105]
12.按照abclonal(rk20228)试剂盒说明书配置如下反应体系:
[0106]
试剂25μl体系t7 buffer4μlt7 adapter2.5μlt7 enzyme mix ii3μllow-edta te补充至25μl
[0107]
将配制好的mix加入到步骤11的产物中,于pcr仪中(热盖105度)进行反应,程序如下:
[0108]
37度15min95度2min4度hold
[0109]
13.按照abclonal(rk20228)试剂盒说明书配置如下反应体系:
[0110]
试剂46μl体系2xsynthesis mix43μlsynthesis reagent3μl
[0111]
将配制好的mix加入到步骤12的产物中,置于pcr仪(提前预热到98度,热盖105度)上进行反应,设置如下:
[0112]
温度时间98度1min60度2min68度5min4度hold
[0113]
14、反应结束后,1.2
×
dna clean beads纯化,使用北京寻因科技公司的mm试剂盒中引物universal oligo进行产物富集及dd 3’单细胞文库构建试剂盒中的建库试剂进行文库构建,得到的文库片段大小如图6中所示,其长度主要集中在200bp~1500bp范围内。
[0114]
对比例1
[0115]
根据北京寻因科技公司的mm试剂盒中记载的操作方法对pbmc细胞(外周血单核细胞)进行文库构建。
[0116]
测试实施例
[0117]
illumina novaseq 6000测序,并分析测到的核酸序列在rna的位置。如图7所示,该方法测得的核酸序列(a)比seekone mm 3’转录组文库(b)更均匀分布在rna全长上。
[0118]
以上仅为本发明的优选实施例,并非因此限制本发明的专利范围,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包括在本发明的专利保护范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1