生物标志物CD83及其应用的制作方法
生物标志物cd83及其应用
技术领域
1.本技术涉及医学领域,特别是疾病筛查、诊断、和/或监测领域;具体而言,本技术提供了用于冠心病的筛查、诊断、和/或监测的生物标志物及相关产品和用途。
2.资助信息
3.本专利申请得到了中国科技部的资助,涉及国家重点研发计划:新生儿/儿童危重症体外生命支持应用评价和质量改善研究(批准号:2021yfc2701700;2021yfc2701703)。
4.发明背景
5.冠心病具有较高致死率和致残率,已经成为全球重大公共健康问题。冠心病发病正趋向年轻化,严重威胁青年人群体身体健康。早发冠心病被认为是45岁之前的急性心肌梗死或冠状动脉造影中观察到的70%以上的冠状动脉狭窄。传统冠心病危险因素如代谢性疾病、吸烟和不健康的生活方式在冠心病发病中发挥关键作用。但是,经典的传统冠心病危险因素作为疾病预测和危险分层的指标存在明显不足。流行病学调查研究发现,约40%的冠心病患者具有遗传易感性特征,ldl-c≧190mg/dl的个体更可能携带家族性高胆固醇血症基因,其冠心病患病概率是不携带这些突变个体的3倍。遗传易感性与冠心病的发病机制密切相关。多种基因变异可以直接或间接作用通过参与多种生物学途径发挥致病作用,包括血压、血脂血糖代谢、血管稳态、抗炎促炎失衡等。没有明确传统冠心病危险因素的早发冠心病群体发病可能更容易受到遗传危险因素的影响。因此,在较少合并传统冠心病危险因素的早发冠心病群体中,遗传易感性被认为是不可忽略的危险因素。
6.因此,阐明早发冠心病遗传易感性特征和鉴别新的生物标志物对于早发冠心病的早期预警、诊疗和/或改善临床预后至关重要。
技术实现要素:
7.第一方面,本技术提供了用于检测cd83特异性核酸序列的试剂或包含所述试剂的组合物,其中所述试剂包含引物或探针,优选地,所述特异性核酸序列的长度为至少约20个核苷酸,更优选30-441个核苷酸,最优选为42-132个核苷酸。
8.第二方面,本技术提供了用于筛查、诊断和/或监测个体中冠心病的试剂盒,其包含如上述第一方面所述的试剂或组合物、或者检测由上述第一方面所述的特异性核酸序列编码的多肽的试剂。
9.第三方面,本技术提供了用于检测来自个体的样本中cd83特异性核酸序列表达水平的试剂在制备用于筛查、诊断和/或监测所述个体中冠心病的试剂盒或药物中的用途,其中如果cd83特异性序列的表达水平高于在对照样本中的水平,则所述个体患有或可能患有冠心病。
10.在一些实施方案中,所述试剂检测cd83特异性核酸序列。在一些实施方案中,所述特异性核酸序列包含如seq id nos:1-11和45中任一项所示的序列或由如seq id nos:1-11和45中任一项所示的序列组成。
11.在一些实施方案中,所述引物包含如seq id nos:23-44中任一项所示的序列。
12.在一些实施方案中,所述多肽包含如seq.id nos:12-22和46中任一项所示的序列或由如seq id nos:12-22和46中任一项所示的序列组成。
13.在具体实施方案中,上述述冠心病为早发冠心病。
14.在一些实施方案中,上述用于测量由cd83基因表达的蛋白的量的试剂包括特异性结合由cd83基因表达的蛋白的抗体或适配体。
15.在一些实施方案中,cd83 mrna的量通过选自以下的至少一种方法测量:原位杂交、聚合酶链式反应(pcr)、逆转录(rt)-pcr、实时pcr、rna酶保护测定(rpa)、northern印迹、微阵列和高通量测序等。
16.在一些实施方案中,cd83蛋白的量通过选自以下的至少一种方法测量:蛋白质印迹、放射免疫测定(ria)、放射免疫扩散法、酶联免疫吸附测定(elisa)、免疫沉淀、流式细胞术、免疫荧光、ouchterlony双向免疫扩散法、补体固定测定和蛋白质芯片等。
17.在一些实施方案中,上述样本是外周血、全血、血清、或血浆样品。在优选的实施方案中,所述样本为外周血样本。
18.在本技术中,鉴定了与冠心病相关的表达特异性增加的生物标志物,因此可以通过测量所述生物标志物基因的特异性序列的表达水平(例如mrna或蛋白质水平)来筛查、诊断和/或监测冠心病特别是早发性冠心病的发生。然而,本技术的效果不限于上述效果,本发明所属技术领域的技术人员将从以下描述中清楚地理解未提及的其它效果。
19.附图简述
20.图1显示了差异表达基因(differentially expressed genes,degs)的识别。图1a显示gse66360数据集的冠心病患者样本和健康对象样本之间的degs;图1b显示高通量测序数据集的早发冠心病患者样本和健康对象样本之间的degs,其中蓝色点代表下调基因,灰色点代表非显著表达基因,红色点代表上调基因。图1c显示gse66360数据集的冠心病患者样本和健康对象样本之间的degs;图1d显示高通量测序数据集的早发冠心病患者样本和健康对象样本之间degs,其中蓝色矩形代表低表达基因,红色矩形代表高表达基因。图1e和f分别显示两个数据集重叠共有的上调及下调基因数。
21.图2显示了degs的富集分析。图2a和d显示degs的显著富集通路。图2b和c显示bp和mf两个层面中degs的富集显著项。图2的气泡图中,y轴代表富集显著项,x轴代表基因占比率;和弦图中,degs显示在图的左半侧,显著富集通路显示在图的右半侧。
22.图3显示了早发冠心病所有degs的富集分析。图3a显示degs的显著富集通路。图3b分别从bp、cc及mf三个功能显示degs显著富集项。
23.图4显示了高通量测序数据集的gsea分析。
24.图5的a显示了高通量测序数据集的免疫浸润分析,示出了免疫细胞亚群的比例。
25.图6a显示了一个由17个节点和28条边的ppi交互网络;图6b显示了将所有高通量测序数据集的degs放入string中,并使用cytohubba算法筛选出的前100个核心基因。
26.图7显示了核心基因的外部验证结果。
27.图8显示了目标ncrnas预测和网络构建,其中图8a为来自gse31568数据集和在线mirna数据库的重叠mirna的维恩图;图8b为来自gse160717数据集和在线circrnas数据库的重叠circrnas的维恩图。图8为cd83的cerna网络,其中红色节点代表核心基因,蓝色节点代表靶向mirna,橙色节点代表靶向circrna。
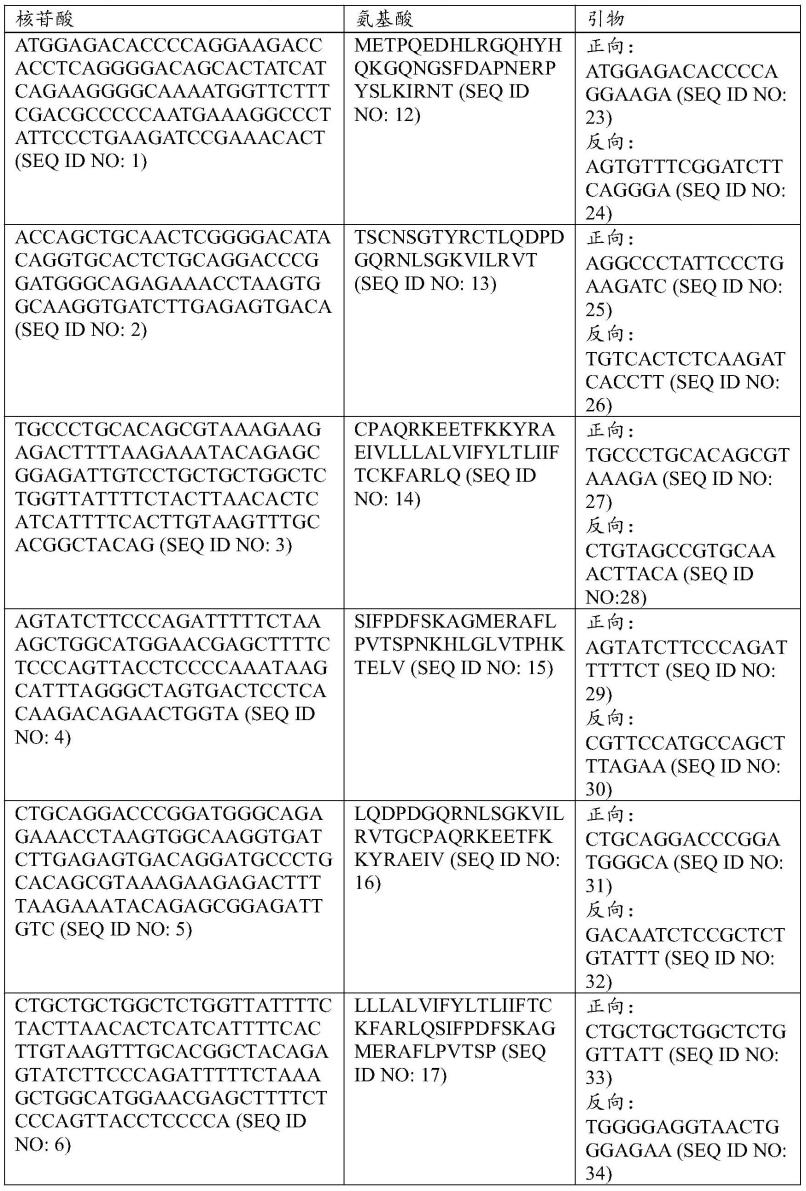
具体实施方式
28.在下文中,将更详细地描述本技术。除非本文另有定义,否则本文使用的所有技术和科学术语具有与本技术所属领域的技术人员通常理解的相同的含义。通常,本文使用的命名法和下文将描述的实验方法是本领域熟知和常用的那些。
29.本技术提供了用于筛查、诊断和/或监测个体中冠心病的组合物或试剂盒,其包含用于检测来自所述个体的样本中cd83基因表达水平的试剂。发明人鉴定了与冠心病相关的表达特异性增加的生物标志物cd83,通过测量cd83基因的特异性序列的表达水平(例如mrna或蛋白质水平)可以筛查、诊断和/或监测冠心病特别是早发性冠心病的发生。如果测试个体的样本中cd83特异性序列表达水平高于在对照样本中的水平,则所述个体患有或可能患有冠心病。
30.cd83是免疫球蛋白超家族的成员,是树突状细胞表面的一种特征性标志物,持续高表达的cd83代表着丰富的树突状细胞参与抗原呈递。发明人发现上调的cd83基因是免疫紊乱及早发冠心病发病的重要危险因素。在本技术中,发明人鉴定出了cd83基因的特异性核苷酸序列以及相应的氨基酸序列。在一些实施方案中,所述特异性核酸序列的长度为至少约20个核苷酸。在具体的实施方案中,所述特异性核酸序列的长度为30-441个核苷酸。在更具体的实施方案中,所述特异性核酸序列的长度为42-132个核苷酸。
31.以下表中列出了示例性的特异性核苷酸序列、相应的氨基酸序列和引物序列。经验证,这些序列能够成功地用于本技术的相关目的。
32.33.[0034][0035]
在一些实施方案中,cd83的全长序列如seq id no:45所示,其编码的多肽如seq id no:46所示。
[0036]
本文所用术语“个体”或“对象”可互换地用于指被分类为哺乳动物的所有动物,并且包括但不限于家畜和农场动物、灵长类动物和人类,例如人、非人类灵长类动物、牛、马、猪、绵羊、山羊、狗、猫或啮齿动物。优选地,个体是任何年龄或种族的男性或女性。
[0037]
来源于个体的生物样本可以包括外周血、全血、血清、或血浆样品等,但不限于此。在具体的实施方案中,所用的生物样本是外周全血样本。
[0038]
本文所用的术语“诊断”是指试图确定和/或鉴定对象中可能疾病的过程,即诊断程序,以及通过该过程所达成的意见,即诊断意见。
[0039]
术语“筛查”在本文中被理解为检查或测试属于一般人群的一组无症状个体,或者具有一个或多个风险因素的一组个体(即,疑似发生疾病或处于发生疾病的风险中的对象),目的是将健康个体与患有或疑似患有疾病的那些个体区分开来。筛查的方法通常用于疾病的早期检测。早期检测是指在存在临床征象之前进行检测。
[0040]
本文所用的术语“监测”是指确定疾病的演变和/或疗法的功效,例如确定是否存在疾病的缓解;或者相反,是否存在疾病进展或复发。
[0041]
本文所用的术语“生物标志物”是指疾病的标志物,其通常是在个体样品中发现的可以容易地被测量的物质。测量的量可以与潜在的疾病病理生理学,如是否存在冠心病,例如早发性冠心病,或者与其预后相关。
[0042]
在一些实施方案中,用于检测cd83基因表达水平的试剂是用于测量cd83基因的mrna的量的试剂。用于测量基因的mrna量的试剂是指能够特异性结合并识别基因的mrna或者扩增所述基因的mrna的量的试剂。作为具体实例,它可以是但不限于特异性结合mrna或者通过mrna的逆转录制备的cdna的核苷酸序列的引物或探针。
[0043]
本文所用的术语“引物”是指具有游离的3
’‑
末端羟基的短核酸序列,互补模板链与其形成碱基对,并因此当核酸聚合酶复制并扩增模板链时,其用于提供起始点。引物可以通常被合成为15-30个碱基对的长度,但是可以根据使用目的而变化,并且可以通过已知的方法通过甲基化、加帽等进行修饰。
[0044]
本文所用的术语“探针”是指长度为几个到几百个碱基的核酸片段,其由能够特异性结合mrna或cdna的序列组成,可以通过酶促化学分离纯化或合成产生。探针可以被标记上放射性同位素、酶或磷光体以鉴定是否存在mrna,并且可通过已知的方法进行设计和修饰。
[0045]
在一些实施方案中,基因的mrna的量可以使用基因序列的有义和反义引物通过诸如pcr、rt-pcr、竞争性rt-pcr和实时rt-pcr的方法进行测量,可以使用具有能够特异性结合基因的mrna或通过逆转录制备的cdna的序列的探针通过诸如northern印迹和微阵列的方法进行测量,以及此外,可以通过诸如rna酶保护测定和测序的方法进行测量,但本技术不限于此,可以使用本领域技术人员已知的任何方法来测量基因的mrna表达水平。
[0046]
在一些实施方案中,用于检测cd83基因表达水平的试剂是用于测量由cd83基因表达的蛋白的量的试剂。用于测量由基因表达的蛋白的量的试剂是指能够特异性结合并识别
蛋白的试剂。作为具体的实例,它可以是但不限于特异性结合蛋白质的抗体或适配体。
[0047]
本文所用的术语“抗体”是指免疫学上特异性结合蛋白质表位并具有反应性的免疫球蛋白分子,其包括但不限于单克隆抗体、多克隆抗体、具有全长链结构的抗体、具有至少抗原结合功能的功能片段的抗体和重组抗体。术语“适配体”是指具有能够靶向并特异性结合蛋白质的特性的稳定的三维结构的单链核酸分子,可以通过指数富集(selex)技术等使用配体的系统进化来合成对蛋白质具有特异性的适配体。
[0048]
在一些实施方案中,可以通过诸如蛋白质印迹、蛋白质微阵列(蛋白质芯片)、酶联免疫吸附测定(elisa)、二维电泳、免疫组织化学(ihc)、免疫荧光、流式细胞术、共免疫沉淀测定、荧光激活细胞分类器(facs)、放射免疫测定(ria)、放射免疫扩散、基质辅助激光解吸/电离飞行时间质谱(maldi-tof)、ouchterlony双向免疫扩散法、补体固定测定等的至少一种方法来测量由基因表达的蛋白质的量,但本技术不限于此,可以使用本领域技术人员已知的任何方法来测量蛋白质的表达水平。
[0049]
高通量测序、微阵列分析和临床生物信息学技术方法的广泛应用,在发掘参与多种疾病的差异表达基因(differentially expressed genes,degs)方面取得了突破性进展。这些degs与冠心病的多种生物学功能密切相关,因此,这些技术将从全基因组维度深入了解冠心病的致病机制。基于degs构建的竞争性内源性rna(competing endogenous rna,cerna)网络能够阐明疾病进展和恢复过程中的转录调控机制。此外,血液循环中的degs因具有相对稳定、易于检测和疾病特异相关的属性,已经成为有希望的候选生物标志物。在具体的实施方案中,发明人通过高通量测序和生物信息学分析的方法,比较早发冠心病患者和年轻健康个体的全血转录组差异,鉴定了可以用于早期预警、风险分层和/或精准干预的潜在靶点,例如cd83基因。
[0050]
在一些实施方案中,高通量测序通过新一代测序技术直接获得所有捕获片段的序列和数量。然而,微阵列测序需要提前合成感兴趣的基因序列,并确定这些基因是否通过核酸杂交引入的荧光信号表达。高通量测序的直接性必然使其整体准确性和测序深度优于微阵列测序。
[0051]
在一些实施方案中,利用基因本体(gene ontology,go)富集分析方法将所有degs的生物学特征归纳总结为分子功能(molecular function,mf)、生物学过程(biological process,bp)和细胞成分(cell components,cc)三个层次。此外,还可以例如通过京都基因和基因组百科全书(kyoto encyclopedia of genes and genomes,kegg)通路富集分析,明确各个degs参与的分子通路,并且较为全面地了解基因间相互作用。
[0052]
在具体的实施方案中,利用本文公开的组合物或试剂盒来检测个体样本中cd83基因的表达水平,如果cd83基因的表达水平显著高于在对照样本中的水平,则指示个体患有冠心病或具有患冠心病的风险。
[0053]
对照样本可以是来自健康个体的样本、来自未患冠心病的个体的样本、来自多个未患冠心病的个体的混合样本,或者是已知为未患冠心病的个体的平均表达水平的对照或基线表达水平。
[0054]
在某些实施方案中,本技术的方法和试剂盒还可以用于监测化学疗法治疗的有效性。在可选的实施方案中,在某一个体中,如果治疗方案有效,cd83生物标志物的水平可以随时间降低;如果治疗方案无效,所述生物标志物的水平将不改变或者可以随时间增加。
[0055]
在某些实施方案中,本技术的方法和试剂盒还可以用于筛选有望治疗冠心病的潜在药物。例如,如果对个体施用某一化合物后,cd83基因表达被抑制或降低,则该化合物可以作为治疗冠心病的候选物被进一步研究或开发。
[0056]
本说明书和权利要求书中,词语“包括”、“包含”和“含有”等词意指“包括但不限于”,且并非意图排除其他部分、添加物、组分或步骤。
[0057]
应该理解,在本发明的特定方面、实施方案或实施例中描述的特征、特性、组分或步骤,可适用于本文所描述的任何其他的方面、实施方案或实施例,除非与之矛盾。
[0058]
上述公开内容总体上描述了本技术,通过下面的实施例进一步示例本技术。描述这些实施例仅为说明本技术,而不是限制本技术的范围。尽管本文中使用了特殊的术语和值,这些术语和值同样被理解为示例性的,并不限定本技术的范围。除非特别指明,本说明书中的实验方法和技术为本领域技术人员所公知的方法和技术。
[0059]
实施例
[0060]
提供以下实施例仅是对本技术的一些实施方案进行举例说明,没有任何限制的目的。
[0061]
下述实施例中所使用的方法如无特殊说明,均为常规方法。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
[0062]
材料与方法
[0063]
纳入对象和样本采集
[0064]
本研究经中国人民解放军总医院伦理委员会批准,45名早发冠心病患者被纳入此研究。纳入标准:
①
年龄≤45岁;
②
有明确的胸闷或胸痛等临床表现;
③
有完整的入院或外院实验室化验及检查;
④
在本院经过冠状动脉造影证实冠心病的诊断。排除标准:
①
已接受冠状动脉搭桥治疗或心脏移植患者;
②
无冠状动脉造影结果;
③
明确的凝血异常或活动性出血患者;
④
合并大动脉炎、风湿性疾病或癌症患者;
⑤
怀疑主动脉夹层、肺栓塞、心脏瓣膜病等胸痛患者;
⑥
当前感染或患有自身免疫性疾病等患者。同时,将8名45岁以下的青年健康个体纳入对照组。所有参与者都对本次研究充分了解,并获得知情同意,入院后均采集了外周血样本,进行高通量测序。
[0065]
外周血rna提取、测序及数据处理
[0066]
按照产品说明书,使用试剂(wuhan servicebio technology,wuhan,china)从全血样品中分离提取总rna量。使用1%琼脂糖凝胶电泳来评估rna是否降解超标或存在污染。通过nanodrop 2000分光光度计(thermo scientific,ma,usa)计算rna的纯度和浓度。使用agilent 2100生物分析仪(agilent technologies,santa clara,ca,usa)帮助准确评估rna完整性。然后,使用illumina公司的专用试剂盒nebnext ultra directional rna library prep kit(neb,ispawich,usa)构建rna测序所需的cdna文库。然后通过agilent 2100生物分析仪测试文库的质量,使用qpcr(kapa biosystems,woburn,ma,usa)进行量化,并按照制造商的方案在illumina hiseq
tm 2000测序平台(illumina,san diego,ca,usa)上测序。我们通过以下三个标准过滤原始测序读数以确保信息分析的质量:(1)如果单端读数中未知碱基的百分比超过10%,则丢弃成对的读数;(2)丢弃带有测序接头或接头的成对的读数;(3)如果单端读数中低质量碱基的百分比超过50%,则丢弃成对的读数。此外,还检测了所有测序读数的q20、q30和gc碱基含量,选择了高质量、清洁的读数进
retrieval of interacting genes/proteins database(string;http://www.string-db.org/)构建了此次研究中degs的ppi网络。使用cytoscape软件优化ppi网络的可视化展现形式。同时,该软件可以通过minimal common oncology data elements(mcode)模块发现重要的互作基因簇,并通过cytohubba插件识别核心基因。
[0079]
cernas网络的构建
[0080]
将获得的核心基因输入到三个在线mirna数据库,即mirdb、mirwalk和targetscan数据库,可预测核心基因对应靶向的mirna,并选择在三个数据库中共同存在的mirna进行后续分析。应用starbase数据库(http://starbase.sysu.edu.cn/contact.php)可识别上述筛选出的mirna对应靶向的circrna。最后,整理从线数据库筛选出的mirna和circrna,并与从geo数据库获得的冠心病相关mirna和circrna相交,便可得到既与核心基因相关、又与冠心病发病相关的mirna和circrna,利用cytoscape软件对结果进行可视化展示。
[0081]
统计学分析
[0082]
所有统计数据处理和分析均通过r语言软件和spss statistics26.0。通过使用t检验进行组间定量资料的比较。pearson相关分析用于展现基因之间相关性。p值<0.05被认为具有统计学意义。
[0083]
结果
[0084]
degs的鉴定
[0085]
根据预先设定的log2(fc)值及q值,分别从高通量测序数据集和gse66360数据集鉴定出1692个和885个degs,均使用火山图和热图进行可视化(图1a-d)。其中,高通量测序数据集包含235个上调基因和1457个下调基因,gse66360数据集包含666个上调基因和219个下调基因。使用维恩图中分别展示了两个数据集重叠共有35个degs,包括31个上调基因和4个下调基因(图1e-f)。
[0086]
富集分析结果
[0087]
首先对两个数据集共有的35个重叠的degs进行了kegg通路分析,这些degs主要富集于旺盛的免疫反应,包括il-17信号通路、核因子κβ(nuclear factor kappa beta,nf-κβ)信号通路、肿瘤坏死因子(tumor necrosis factor,tnf)信号通路、核苷酸结合寡聚化结构域(nucleotide-binding oligomerization domain,nod)样受体信号通路(图2a和d)。在这35个degs的go富集分析中,bp层面显著富集在对外部刺激反应的正向调节、造血调节、中性粒细胞活化和炎症反应的正向调节,并且mf层面包含细胞因子活性和toll样受体(toll-like receptor,tlr)结合(图2b和c)。
[0088]
为全面评估早发冠心病的遗传属性及其可能发病机制,我们对高通量测序数据集进行了单独的富集分析。kegg通路富集分析表明,所有degs主要富集于粘着斑、紧密连接和细胞外基质(extracellular matrix,ecm)受体相互作用(图3a)。go富集分析的bp层面包含体液免疫反应、吞噬作用、补体激活、免疫球蛋白介导的免疫反应和b细胞介导的免疫反应;cc层面主要包含免疫球蛋白复合物和质膜外侧;mf层面主要包含通道活性和被动跨膜转运蛋白活性(图3b)。gsea分析发现,显着富集的基因集包含心肌收缩、先天免疫反应激活信号转导、il-1介导的信号通路和对il-12的反应(图4)。
[0089]
高通量测序数据集的免疫浸润特征
[0090]
为了明确早发冠心病的免疫浸润环境,通过cibersort算法分析可知早发冠心病
患者体内静息记忆cd4+t淋巴细胞、静息肥大细胞、嗜酸性粒细胞、中性粒细胞含量较为丰富,而静息自然杀伤细胞(natural killer cell,nk)细胞、m2(macrophages2)巨噬细胞、记忆b淋巴细胞、cd8 t+淋巴细胞、幼稚cd4 t+淋巴细胞含量相对较少。中性粒细胞的比例与cd8 t+淋巴细胞和调节性t(regulatory t,treg)淋巴细胞的比例呈负相关。参见图5。
[0091]
ppi网络分析结果
[0092]
将共有的35个共表达degs输入在线工具string,删除分散节点后可构建一个由17个节点和28条边的ppi交互网络(图6a)。通过cytohubba算法识别出cxcl8、jun、bcl2a1、cxcl2、nfkbia、cd83(例如seq id no:45所示)、nfkbiz、fosb、nr4a2、s100a9这10个功能相对重要的核心基因(使用红色及黄色标出)。此外,将所有高通量测序数据集的degs放入string中,并使用cytohubba算法筛选前100个核心基因(图6b)。
[0093]
核心基因的外部验证
[0094]
在外部gse19339数据集中,验证10个核心基因的表达情况。结果发现cd83(例如seq id no:45所示)、cxcl2、cxcl8、jun和nr4a2水平仍呈显著性升高表达(p<0.05)(图7)。
[0095]
cerna网络构建
[0096]
基于3个mirna数据库共发现201个与核心基因相关的mirna,同时在gse31568数据集中通过差异基因表达分析筛选出397个与冠心病相关mirna。最后,通过维恩图重叠相交确定共有19个靶向mirna,并使用互相作用网络图可视化展示(图8a)。使用与上述相同的方法预测靶向circrna(图8b)。我们依次搜索starbase数据库、分析gse160717数据集,并将交集作为靶向circrna。最后,根据相互作用关系,构建了cd83的cerna网络,该cerna网络可能是早发冠心病的潜在致病机制(图8c)。
[0097]
讨论
[0098]
本研究以45名早发冠心病患者作为主要研究对象,并抽取外周血样本进行高通量测序。发明人在对数据处理和差异基因表达分析后发现,与健康个体相比,早发冠心病患者有明确的差异性基因表达,提示早发冠心病与基因表达情况明确相关。发明人从高通量测序基因集及公共数据库冠心病数据集中确定了共有35个degs重叠。对其进行go和kegg通路富集分析,发现与健康人相比,冠心病患者的中性粒细胞激活、il-17信号通路、nf-κβ信号通路、tnf信号通路和nod样受体信号通路等免疫反应更加显著。
[0099]
为更好地揭示早发冠心病的遗传特征,发明人进一步对所有与早发冠心病相关的高通量测序数据集中degs额外进行了上述两种富集分析,结果同样表明这些degs也大多富集在过度的免疫激活途径和丰富的信号转导通路上。gsea分析发现早发冠心病患者的大部分基因主要集中表达在心肌收缩、先天免疫反应激活信号转导、对il-12反应及il-1介导的信号通路上,提示免疫功能异常是早发冠心病的重要致病机制。通过cibersort算法计算高通量测序数据集的免疫浸润情况,结果发现较高比例的中性粒细胞和嗜酸性粒细胞与早发冠心病相关,这与本研究中go和kegg富集分析的结果较为一致,同时m2巨噬细胞、cd8 t+淋巴细胞和treg淋巴细胞相对较少。本研究通过综合生物信息学方法,在转录组和细胞水平上确定了早发冠心病患者发病过程中重要的炎症通路和免疫细胞亚群,为更加深入了解疾病的发病机制,提供了详细的理论基础。
[0100]
发明人通过构建ppi网络、使用cytoscape对网络进行分析,结果发现cxcl8、jun、bcl2a1、cxcl2、nfkbia、cd83、nfkbiz、fosb、nr4a2、s100a9这10个核心基因在此网络中发挥
重要作用。为了降低实验的偏移及误差,本研究选择另外一组冠心病相关数据集进行核心基因表达水平的外部验证,结果提示仅有cd83(例如seq id no:45所示)、cxcl2、jun、cxcl8和nr4a2在内的五个基因仍在冠心病及健康群体间表现出显著差异性。mirna可与靶向基因结合,使基因表达下调、甚至沉默,而其上游的circrna可干扰mirna响应元件调节基因表达。为了更加明确核心基因的分子调控机制,本研究通过geo、mirna相关数据库和starbase数据库构建出cd83的cerna网络。
[0101]
本研究表明,上调的cd83基因与早发冠心病的发生发展密切相关,可以作为早发冠心病早期预警、精准治疗和/或预后评估的生物标志物。
[0102]
可以理解,尽管本技术以某种形式被说明,但本技术并不局限于本说明书中所显示和描述的内容。对本领域的技术人员显而易见的是,在不偏离本技术的范围的前提下还可做出各种变化。这些变化都在本技术要求保护的范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1