一种苏紫猪产仔性状相关全基因组SNPs筛选方法
本发明属于畜牧业中的生物育种,具体涉及一种苏紫猪产仔性状相关全基因组snps筛选方法,能筛选到影响猪产仔数的全基因组snps,为猪产仔数性状的育种提供帮助。
背景技术:
1、产仔数是养猪生产中一个重要的育种性状、繁殖性状和经济性状,而总产仔数和产活仔数一直是种猪选育中最常记录的两个产仔数指标。
2、由于产仔数遗传力较低且又是伴性性状,利用常规的育种方法选育难度较大,且进展缓慢。随着分子生物学的发展,全基因组snps分子标记逐渐应用于猪产仔数的选择育种中。利用全基因组snps分子标记,可以进行全基因组关联分析(genome-wideassociation study,gwas)和全基因组选择育种(genomic selection)。gwas是利用全基因组范围内筛选出的高密度分子标记(现大多为snp)进行试验群体的全基因组扫描,再通过统计学方法将扫描所得的分子标记基因型与表型性状关联分析,最后鉴定出影响表型性状的相关分子标记和候选基因。近年来已在奶牛和生猪育种生产上得到广泛的应用,其中在猪上,研究人员利用全基因组关联分析技术在嵊县花猪、金华猪群体分别筛选了160个和124个达到显著性水平的snp。
3、本发明拟解决实验猪高产仔数关键位点和基因的筛选,研究结果将进一步揭示猪高产仔性状的遗传基础,提高猪育种效率和经济效益有较大的借鉴意义。
技术实现思路
1、本发明要解决的技术问题是提供一种苏紫猪产仔性状相关全基因组snps筛选方法,筛选到影响猪产仔数的全基因组snps,为猪产仔数性状的育种提供帮助。
2、为解决上述技术问题,本发明的实施例提供一种苏紫猪产仔性状相关全基因组snps筛选方法,包括如下步骤:
3、s1、确定试验样品:苏紫猪能繁母猪;
4、s2、组织样采集:对试验样品统一采集耳组织样得到猪组织样,置于-20℃冰箱保存、备用;
5、s3、dna提取:将步骤s2采集的猪组织样剪碎,加入组织裂解液中,上下颠倒至混匀,然后加入蛋白酶k至浓度200μg/ml,50℃-55℃水浴锅内消化过夜,得到消化好的耳组织样溶液;
6、s4、在消化好的耳组织样溶液中加入与之等体积苯酚抽提,12000rpm离心10min,第一次取上清;在第一次取得上清液中加入与之等体积苯酚和氯仿抽提,苯酚和氯仿的体积比为1:1,12000rpm离心10min,第二次取上清;在第二次取得上清液中加入与之等体积氯仿抽提,12000rpm离心10min,第三次取上清;在第三次取得上清液中加入其两倍体积的无水乙醇沉淀dna,12000rpm离心10min,弃上清,留沉淀物;
7、s5、用70%乙醇快速洗涤步骤s4得到的沉淀物后常温晾干,然后用30μl灭菌双蒸水溶解dna;0.7%的琼脂糖电泳初步检测dna的浓度和纯度,紫外分光光度仪测定dna含量,至此,dna提取完成;
8、s6、dna质控检测合格后,进行illumina全基因组猪50k snps芯片检测,删除没有检测结果的snp位点后得到的有效数据用于后期的全基因组snps基因型质控检测;
9、s7、snps基因型质控:snps基因型质控过程删除达到以下标准的个体或位点:分型缺失数>10%的位点、最小等位基因频率低于0.01的位点、哈代-温伯格检验p值<0.000001的位点,符合以上三条件中的任何一个,数据均会被删除;
10、s8、猪产仔性状表型统计:记录每头苏紫猪能繁母猪的耳号、样品号、出生日期、出生体重、乳头数、配种日期、产仔日期、胎次、总产仔数、产活仔数、产健仔数、活仔公猪数、活仔母猪数、弱仔数、死胎数、出生窝重、出生个体均重、断乳窝重和断乳头数;
11、s9、全基因组关联分析:进行猪全基因组snps基因型与产仔性状表型之间的gwas分析;
12、建立gwas分析模型:y=μ+xb+u+e;
13、其中,y表示苏紫猪产仔性状表型值;μ表示产羔数平均值;x表示固定效应矩阵,b为固定效应向量;u表示剩余多基因效应;e表示产仔表型值的随机残差,u和e均服从正态分布;
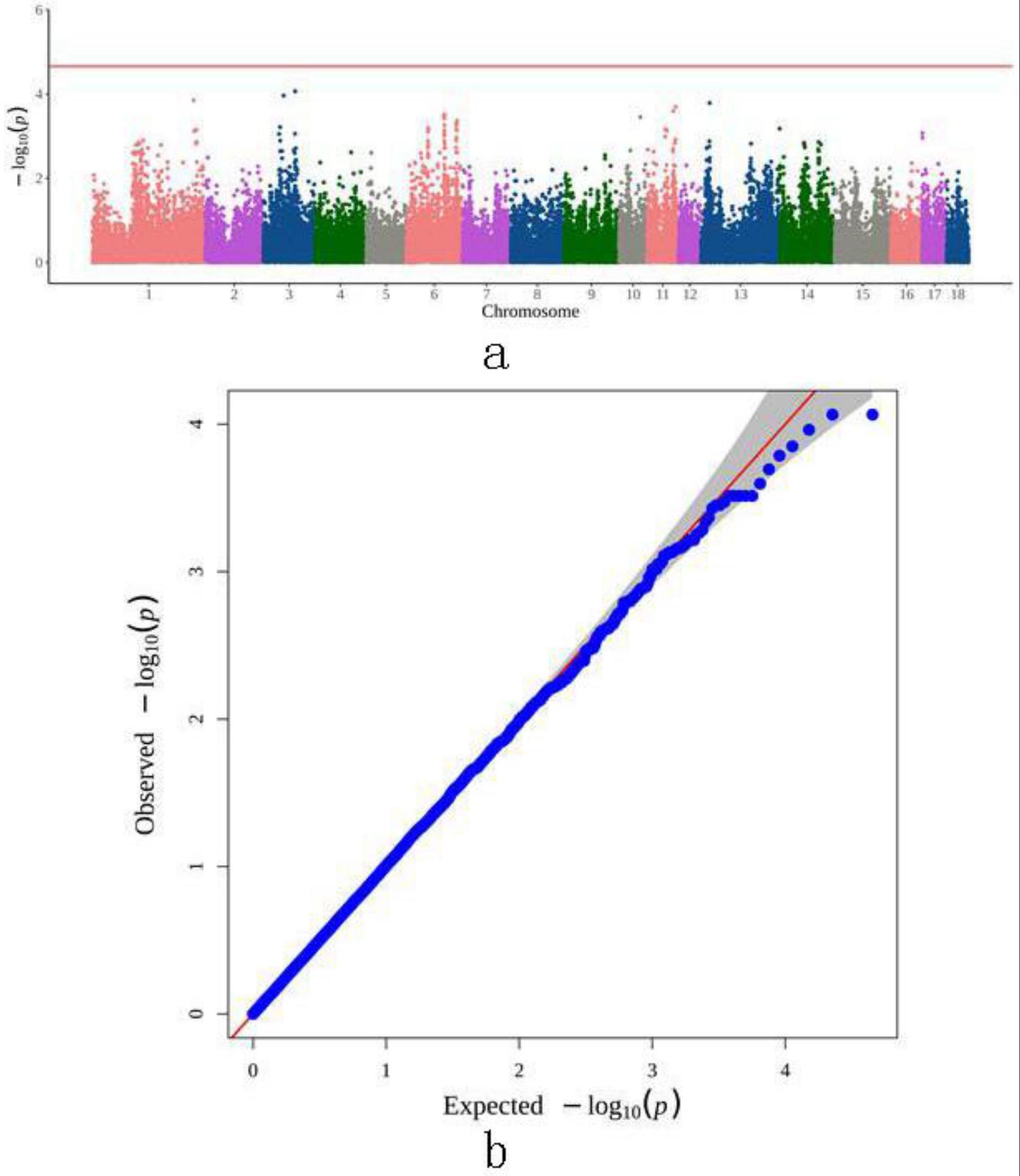
14、s10、显著snps鉴定:对苏紫猪gwas分析结果中的差异显著性水平p值采用bonferroni校正,并使用r软件中的qqman绘制曼哈顿图和q-q图,曼哈顿图上高于阈值线的snps即为苏紫猪产仔性状显著关联snps;
15、s11、候选基因注释:利用ensembl网站的猪基因组序列sscrofa11.1,对苏紫猪产仔性状显著关联snps前后40kb范围内定位的候选基因进行筛选,再利用ensembl网站基因注释工具和pubmed文献数据库进行基因的功能注释,完成苏紫猪产仔性状相关全基因组snps筛选。
16、其中,步骤s9中,使用gemma软件中的混合线性模型,进行猪产仔表型性状与全基因组snps之间的gwas分析。
17、其中,与苏紫猪产仔性状显著关联的全基因组snps共有6个,分别为:cnc10013348、cnc10071931、cnc10110924、cnc10082712、cnc10082643和cnc10082645。
18、其中,全基因组范围内苏紫猪产仔性状的8个候选基因,包括:enssscg00000004980、enssscg00000002260、enssscg00000026251、enssscg00000009457、rap1gds1、enssscg00000009188、enssscg00000009188、bmpr1b和pdlim5。
19、本发明的上述技术方案的有益效果如下:
20、本发明利用illumina全基因组猪50k snps芯片检测了苏紫猪的基因组,并与这些猪的总产仔数、产活仔数性状进行了关联分析。进一步对获得的显著关联snps染色体物理位置100kb范围的基因进行筛选挖掘,最后对这些候选基因进行go功能和kegg通路注释。解决试验猪高产仔数关键位点和基因的筛选,研究结果将进一步揭示猪高产仔性状的遗传基础,提高猪育种效率和经济效益有较大的借鉴意义。
技术特征:
1.一种苏紫猪产仔性状相关全基因组snps筛选方法,其特征在于,包括如下步骤:
2.根据权利要求1所述的苏紫猪产仔性状相关全基因组snps筛选方法,其特征在于,步骤s9中,使用gemma软件中的混合线性模型,进行猪产仔表型性状与全基因组snps之间的gwas分析。
3.根据权利要求1所述的苏紫猪产仔性状相关全基因组snps筛选方法,其特征在于,与苏紫猪产仔性状显著关联的全基因组snps共有6个,分别为:cnc10013348、cnc10071931、cnc10110924、cnc10082712、cnc10082643和cnc10082645。
4.根据权利要求1所述的苏紫猪产仔性状相关全基因组snps筛选方法,其特征在于,全基因组范围内苏紫猪产仔性状的8个候选基因,包括:enssscg00000004980、enssscg00000002260、enssscg00000026251、enssscg00000009457、rap1gds1、enssscg00000009188、enssscg00000009188、bmpr1b和pdlim5。
技术总结
本发明提供一种苏紫猪产仔性状相关全基因组SNPs筛选方法,组织样采集后提取DNA,DNA质控检测合格后,进行Illumina全基因组猪50K SNPs芯片检测,质控后得到有效数据用于后期的变异检测;然后进行全基因组关联分析:进行猪产仔表型性状与全基因组SNPs之间的GWAS分析;建立GWAS分析模型:y=μ+Xb+u+e;对苏紫猪GWAS分析结果中的差异显著性水平P值采用Bonferroni校正,并使用R软件中的qqman绘制曼哈顿图和Q‑Q图。本发明利用Illumina全基因组猪50K SNPs芯片检测了苏紫猪的基因组,并与这些猪的总产仔数、产活仔数性状进行了关联分析。解决试验猪高产仔数关键位点和基因的筛选,研究结果将进一步揭示猪高产仔性状的遗传基础,提高猪育种效率和经济效益有较大的借鉴意义。
技术研发人员:付言峰,程金花,廖超,李辉,赵为民,涂枫,戴超辉,王学敏,李碧侠
受保护的技术使用者:江苏省农业科学院
技术研发日:
技术公布日:2024/1/15
- 还没有人留言评论。精彩留言会获得点赞!