基于SNP标记的菊花DNA指纹图谱及其构建方法和应用
本发明属于生物,具体涉及菊花dna指纹图谱及其构建方法和应用。
背景技术:
1、菊花(chrysanthemum morifolium ramat.)为菊科(asteraceae)菊属的多年生草本花卉,是我国栽培历史悠久的传统名花和世界四大切花之一。其品种繁多,色彩艳丽,栽培应用十分广泛,具有很高的观赏价值和商业价值。品种鉴定是菊花种质资源评价的重要环节。花色作为菊花主要观赏特征是其开展品种鉴定的重要性状之一,然而传统的形态学鉴定方法如花色受外界栽培环境(光照、温度等)影响较大,在同一色系品种鉴定中有一定局限性。因此,基于不同色系构建dna指纹图谱,对同一色系内品种的准确区分和鉴定具有重要意义。
2、现有技术中,基于ssr、srap、rapd、aflp和rflp等分子标记的dna指纹图谱已广泛用于植物品种鉴定。近年来,随着测序技术的快速发展,snp标记作为第三代分子标记技术,因其具有稳定性高、多态性好、数量多等优点,被认为是极具有应用前景的分子标记技术,并在国际植物品种权保护联盟(upov)的bmt测试指南中被确定为进行dna指纹数据库构建的推荐标记。目前获取snp数据的方法主要包括snp芯片和基因组测序,其中snp芯片的准确度和可信度更高,且具备较为成熟的数据分析流程,但其定制价格昂贵且主要针对已知的变异位点,随着测序成本的不断降低,基因组测序能在足够的深度下检测到所有类型的变异。dna指纹图谱在菊花品种鉴定中已有零星报道,主要利用issr、ssr、srap等多种分子标记对菊花进行品种鉴定与dna指纹图谱构建,与snp标记相比以上标记难以实现高通量检测,目前基于snp标记技术的菊花品种dna指纹图谱构建还未见报道。因此,通过分析snp位点信息,筛选多态性信息含量较高的snp位点为核心snp标记,构建不同色系菊花品种的指纹图谱,可为菊花品种准确性和特异性鉴定提供科学依据。
技术实现思路
1、发明目的:针对现有技术中存在的不足,本发明提供了一种可用于同一花色或不同花色菊花品种准确性和特异性鉴定的基于snp标记的菊花dna指纹图谱。
2、本发明提供一种基于snp标记的菊花dna指纹图谱的构建方法。
3、本发明还提供基于snp标记的菊花dna指纹图谱或菊花dna指纹图谱构建方法在鉴别菊花品种中的应用。
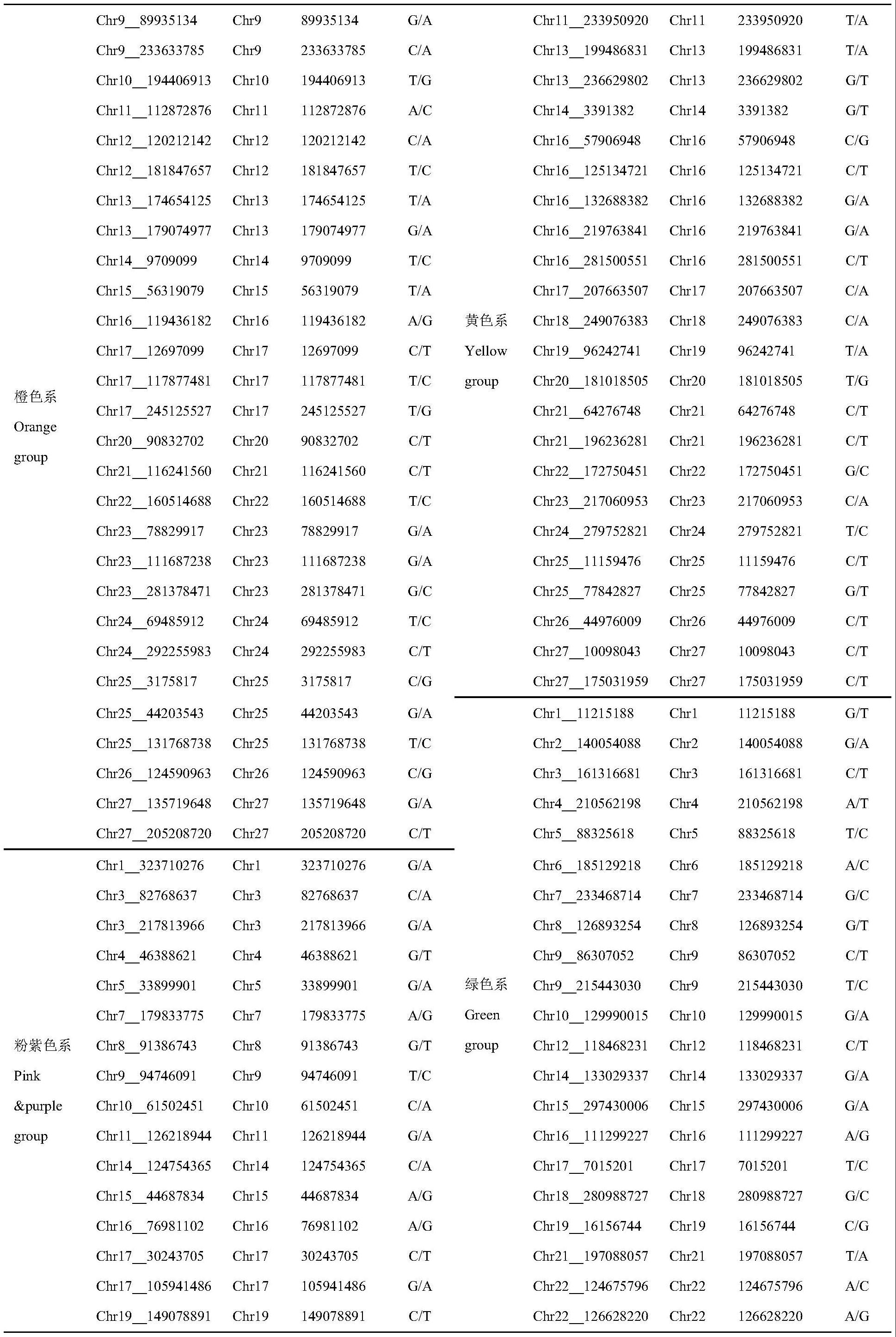
4、技术方案:为了实现上述目的,本发明所述的一种基于snp标记的菊花dna指纹图谱,所述dna指纹图谱包括179个snp位点,所述位点及其特异性核苷酸如下所示:
5、
6、
7、
8、本发明所述的一种基于snp标记的菊花dna指纹图谱的构建方法,包括如下步骤:
9、(1)利用ctab法提取141份不同色系菊花材料的基因组dna,对dna样品质量进行检测后,用限制性内切酶(msei)对基因组dna进行酶切;
10、(2)将步骤(1)得到的酶切片段与测序接头连接,然后pcr扩增、样品混池、片段回收构建gbs文库,gbs文库质检合格后,利用illumina hiseq测序平台,进行双末端(paired-end)150测序;
11、(3)将步骤(2)gbs基因组测序所得的高质量clean data,采用bwa软件(参数:mem-t 4-k 32-m)将高质量clean reads比对到菊花参考基因组,分别利用samtools软件和vcftools软件对snp检测和过滤,筛选出菊花不同色系群体的高质量snp位点370,562个;
12、(4)将步骤(3)筛选出的snp位点根据缺失率、maf值、杂合度和pic值对不同色系群体的snp位点再分别进行筛选,去除ld值中较高两个位点中的一个,筛选出菊花不同色系群体的snp位点共293个,最后通过python程序,筛选能区分所有品种的最少标记集,各色系最终得到核心snp位点共179个;
13、(5)基于snp标记的不同色系菊花的dna指纹图谱构建成功。
14、其中,步骤(3)snp分型数据处理为基于gbs(genotyping-by-sequencing)基因组测序数据所过滤得到的高质量clean data,采用bwa软件(参数:mem-t 4-k 32-m)将高质量clean reads比对到菊花参考基因组,利用samtools软件进行snp calling,gbs基因组测序与snp标记开发由北京诺禾致源生物信息科技有限公司完成。利用vcftools软件按条件“--mindp 3--min-alleles 2--max-alleles2--max-missing 0.80--maf 0.05”过滤获得370,562个高质量snp位点用于后续分析。
15、作为优选,所述步骤(4)中对不同色系群体的snp位点再分别进行筛选的标准为缺失率为0、maf值>0.1、杂合度<0.2和和白色系pic值>0.20,橙色系pic值>0.35,粉紫色系pic值>0.20,红色系pic值>0.35,黄色系pic值>0.20,绿色系pic值>0.25。
16、进一步地,步骤(4)核心snp位点筛选为根据基因分型结果,利用vcftools软件和plink1.9软件计算不同色系品种群体内最小等位基因频率(minor allele frequency,maf)、观测杂合度(observed heterozygosity,ho)、多态性信息含量(polymorphisminformation content,pic)和连锁不平衡水平(linkage disequilibrium,ld)。并按照以下标准对不同色系群体的snp位点分别进行筛选:(1)数据缺失率为0;(2)maf值>0.1;(3)杂合度<0.2;(4)白色系pic值>0.20,橙色系pic值>0.35,粉紫色系pic值>0.20,红色系pic值>0.35,黄色系pic值>0.20,绿色系pic值>0.25。对于筛选所获得的snp位点,按条件--indep-pairwise 50 10 0.3利用plink1.9软件去除ld值较高的两个位点中的一个,并通过python(https://github.com/wan230114/get-least-diff-feature)程序进一步筛选能够区分出全部品种的最少标记构成核心snp标记集。
17、其中,所述基于snp标记的菊花dna指纹图谱在鉴别同一色系或不同色系菊花品种中的应用。
18、进一步地,所述基于snp标记的菊花dna指纹图谱在鉴别同一色系菊花品种中的应用。
19、进一步地,所述基于snp标记的菊花dna指纹图谱在鉴别不同色系菊花品种中的应用。
20、其中,所述基于snp标记的菊花dna指纹图谱构建方法在鉴别同一色系或不同色系菊花品种中的应用。
21、进一步地,基于snp标记的菊花dna指纹图谱构建方法在鉴别同一色系菊花品种中的应用。
22、进一步地,基于snp标记的菊花dna指纹图谱构建方法在鉴别不同色系菊花品种中的应用。
23、其中,所述基于snp标记的菊花dna指纹图谱在鉴别同一色系或不同色系菊花品种中的应用的具体过程为:
24、(1)对需鉴定品种利用ctab法提取其dna。
25、(2)对dna样品质量进行检测后,用限制性内切酶(msei)对dna进行酶切,将得到的酶切片段与测序接头连接,然后pcr扩增、样品混池、片段回收构建gbs文库,gbs文库质检合格后,利用illumina hiseq测序平台,进行双末端(paired-end)150测序。
26、(3)获得该品种对应色系核心snp位点的基因分型,与指纹图谱进行比较,若和对应的指纹图谱一致,则为该品种,不一致则不是。
27、其中,所述步骤(4)中各色系最终得到核心snp位点分别为白色23、橙色43、粉紫色24、红色21、黄色40和绿色28个。
28、选择适合于不同色系菊花品种群体鉴定的核心snp标记是构建dna指纹图谱的关键,标记筛选标准因群体大小和基因型的不同而不同,筛选得到的核心snp标记数量也不同。本发明根据不同色系品种群体中snp位点的pic值分布范围不同而设定不同标准,最终获得各色系核心snp位点构成标记集。核心snp标记集与370,562个snp标记集的群体结构分析表明,核心snp标记集具有较强的代表性和鉴别能力,因此利用更少标记来区分尽可能多的品种,更便于品种鉴定、降低应用成本、节约鉴定时间。本发明利用简化基因组测序鉴定到的370,562个高质量snp位点,根据特定的最小等位基因频率(maf)、观测杂合度(ho)、多态性信息含量(pic)和连锁不平衡水平(ld)等参数,分别从6个色系菊花品种群体中筛选出核心snp位点,构建了不同色系菊花品种的dna指纹图谱。为推动snp分子标记在菊花种质资源精准高效鉴定中的应用提供科学依据。
29、有益效果:与现有技术相比,本发明具有如下显著优点:
30、(1)本发明构建dna指纹图谱的方法以同一色系或不同色系菊花dna的特异性为依据,为菊花品种指纹图谱构建提供了通用策略,并结合群体结构分析构建了同一色系或不同色系菊花品种的dna指纹图谱,在鉴别同一色系以及不同花色的菊花品种中具有应用成本低、鉴定时间短、准确率高和效果更佳的优点。
31、(2)本发明的菊花dna指纹图谱在鉴别同一色系或不同花色的菊花品种中具有应用成本低、鉴定时间短、准确率高和效果更佳的优点。
32、(3)本发明菊花dna指纹图谱可用于同一色系或不同花色菊花品种准确性和特异性鉴定,可以准确区分出全部参试的同色不同品种菊花资源。同时,在新品种保护,品种初步鉴定,判定品种的真实性与特异性方面起到指导作用。
33、(4)本发明指纹图谱构建基于snp标记实现高通量检测,且数据分析简单准确,选择适合于同一色系或不同色系菊花品种群体的稳定性好、鉴别能力强和分布较为均匀的核心snp标记是指纹图谱构建的关键。
- 还没有人留言评论。精彩留言会获得点赞!