靶向PBP2a的非天然核酸适配体获取方法
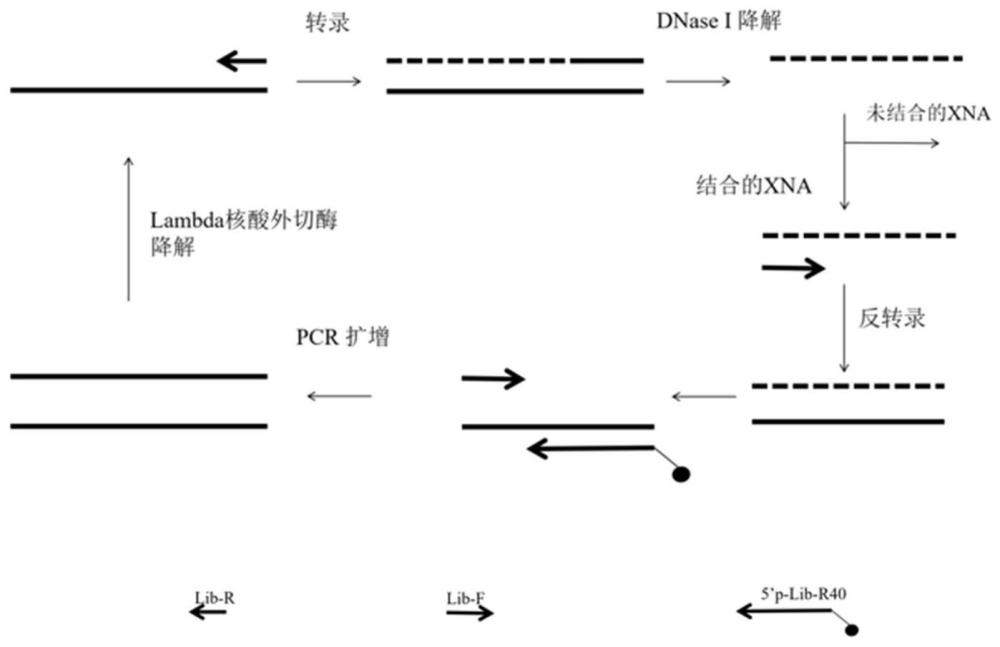
本发明涉及生物,具体为利用定向进化后的sfm4-6聚合酶在selex每一轮筛选当中转录dna文库得到2’-全修饰的非天然核酸适配体文库并用该文库筛选靶向pbp2a的2’-全修饰非天然核酸适配体,最终得到一条69nt的靶向pbp2a的2’-全修饰非天然核酸适配体。
背景技术:
1、抗生素是现代医学的基础,以青霉素、头孢类药物为代表的β-内酰胺类抗生素是目前临床抗感染最普遍应用且有效治疗的一类抗生素。抗生素的使用给世界范围内的传染病治疗带来了革命性的变化。研究发现,虽然用于治疗感染的大部分抗生素和人类病原体获得的抗生素耐药性基因都有环境来源,但是大部分归因于这类药物的滥用误用,使病原菌对抗菌药物的耐药性问题日趋严重。
2、青霉素结合蛋白(pbps)是细菌细胞壁合成过程中所必需的转肽酶。pbps是抗生素治疗的有效靶位,各种β-内酰胺类抗生素通过与pbps结合,抑制它的转肽酶活性,从而阻止正常的转肽反应,使细胞壁不能合成,从而最终导致细菌死亡。
3、而耐甲氧西林金黄色葡萄球菌(mrsa)通过meca编码产生的特殊蛋白-青霉素结合蛋白2a(pbp2a)对β-内酰胺类抗生素的亲和力低,导致多种抗菌药物不能与之结合,当高亲和力的pbps被抑制时,pbp2a可以继续执行转肽酶功能,完成细胞壁功能,维持细菌的生长,所以pbp2a对所有的β-内酰胺类抗生素均产生抗药性。由于科学家对pbp2a的分子生物学特性的探索,通过快速准确筛查出pbp2a,将极大的推进mrsa这一难题的攻克。
4、目前已有报道的pbp2a的核酸适配体,但都是天然核酸适配体,其生物稳定性差,易被核酸酶降解,有一定的局限性,而非天然核酸适配体生物稳定性更好,能很好的改善这个问题,同时为核酸药物的开发提供一个新的选择。
5、寡核苷酸适配体是具有靶向选择性高亲和特性的单链dna或rna,被认为是取代单克隆抗体的核酸基亲和配体。与抗体相比,适配体具有较小的复杂性和免疫原性,适配体除了对多种靶标(蛋白质、细胞或小分子)具有较高的亲和力和特异性外,它们有更大的灵活性,因此,可以附着在内部不可接近的表位上,抗体不容易锁定这些表位。利用适配体与靶标蛋白具有较高的亲和力和特异性的特性可以建立快速检测pbp2a的方法。同时,适配体与靶标蛋白的特异性结合可能会抑制其活性,从而达到杀菌、抑菌的目的。
6、非天然核酸(xnas)是通过对核酸分子上的碱基或骨架修饰而获得,它是天然dna和rna的合成替代物。与天然的dna或rna适配体相比,xna适配体具有更强的稳定性以及耐核酸酶降解能力,这使得xna核酸适配体替代天然的dna或rna适配体成为可能。
7、由于聚合酶对底物的特异性,天然的聚合酶无法高效的识别和合成非天然核酸,但是随着科学家对来自不同家族的dna和rna聚合酶进化定向进化,使聚合酶识别非天然核苷酸底物变得可能,实验室酶法合成非天然核酸序列变得更加容易。在不改变适配体结合活性的情况下,如何获取2’-全修饰的非天然核酸适配体,并提高其对各种核酸酶的稳定性是一个急需解决的问题。本专利通过使用定向进化的taq dna聚合酶stoffel片段(stoffelfragment(sf)突变株——sfm4-6转录出糖环2’-c,f-c、u,ome-a、g的xna文库,利用selex技术对制备得到的文库与固定好的靶标蛋白pbp2a进行孵育结合,通过多轮的洗涤,将未结合的xna序列除掉,最后用变性条件洗脱得到结合靶标蛋白的xna,然后再通过反转录,pcr扩增,转录构建下一轮用于筛选的文库,经过多轮筛选,最终得到靶向pbp2a的xna适配体。最后通过emsa测试xna适配体对的pbp2a的结合活性,证明筛选到的xna适配体具有较高的结合pbp2a的活性,为未来用于mrsa检测和诊断提供一种思路。
技术实现思路
1、本发明包括2’-全修饰非天然核酸适配体文库制备方法,其具体操作是采用2’-f修饰的尿嘧啶核苷酸,2’-f修饰的胞嘧啶核苷酸,2’-ome修饰的腺嘌呤核苷酸以及2’-ome修饰的鸟嘌呤核苷酸的非天然核苷酸为元件,在sfm4-6聚合酶的作用下,以(n20)-lib文库为模板,直接转录合成dna/xna杂合体产物,经dnase i酶降解后,制备得到2’-全修饰的xna文库。
2、本发明包括筛选靶向pbp2a全修饰非天然核酸适配体的技术。筛选靶向pbp2a非天然核酸适配体的过程中,第二轮以后的转录文库制备方法主要采用5’-磷酸化扩增引物对筛选到的xna序列反转录产物做pcr扩增,该引物与模板3’-端互补40nt,包含与转录产物5’-端互补的20nt以及转录引物部分。pcr扩增产物用lambda核酸外切酶降解,通过控制降解时间,能最大效率降解掉磷酸化的序列以获取单链dna文库,然后再使用sfm4-6聚合酶转录获取下一轮的xna文库(如图1所示为筛选流程)。在筛选靶向pbp2a非天然核酸适配体的过程中,第一轮洗脱次数为10次,大多数未结合的xna文库被除去,随着筛选轮数的增加,与靶标蛋白有结合活性的xna逐渐富集。
3、与现有技术相比,本发明的优势在于:
4、相较于固相合成来说,固相合成全修饰xna难度非常大,通常超过30nt的xna序列成本就非常高,且成功率低。本发明利用定向进化后的sfm4-6聚合酶转录酶法合成xna适配体文库,合成效率高,合成的文库序列更长,并且生产成本显著降低。与现今常用的利用t7聚合酶突变株转录得到xna序列相比定向进化后的sfm4-6聚合酶能够耐高温,通过高温转录得到的文库多样性更大,并且转录效率更高,在适宜的条件下转录得到的全长产物更多,操作流程更加简便。
技术特征:
1.靶向pbp2a的非天然核酸适配体获取方法,其特征在于,筛选靶向pbp2a非天然核酸适配体的过程中,第二轮以后的转录文库制备方法主要采用5’-磷酸化扩增引物和反转录引物对筛选到的xna序列反转录产物做pcr扩增,该引物与模板3’-端互补40nt,包含与转录产物5’-端互补的20nt以及转录引物5’-aatacgactcactattaggg-3’部分;pcr扩增产物用lambda核酸外切酶降解,通过控制降解时间,能最大效率降解掉磷酸化的序列以获取单链dna文库,然后再使用sfm4-6聚合酶转录获取下一轮的xna文库;在筛选靶向pbp2a非天然核酸适配体的过程中,第一轮洗脱次数为10次,大多数未结合的xna文库被除去,随着筛选轮数的增加,与靶标蛋白有结合活性的xna逐渐富集。
2.根据权利要求1所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,所述xna文库的制备方法为:采用一种组合的非天然核苷酸为元件在sfm4-6聚合酶的作用下,以(n20)-lib文库为模板,用转录引物直接转录合成dna/xna杂合体产物,经dnase i酶降解后,制备得到2’-全修饰的xna文库。
3.根据权利要求2所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,所述采用一种组合的非天然核苷酸为元件具体为:采用2’-f修饰的尿嘧啶核苷酸,2’-f修饰的胞嘧啶核苷酸,2’-ome修饰的腺嘌呤核苷酸以及2’-ome修饰的鸟嘌呤核苷酸的非天然核苷酸为元件。
4.根据权利要求2所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,用于高通量测序引物seq-f和seq-r进行测序,最终得到适配体序列,其特征在于,靶向pbp2a的2’-f-c,u,2’-ome-a,g修饰的xna适配体的序列为:
5.根据权利要求1所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,所述5’-磷酸化扩增引物的序列为:
6.根据权利要求1所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,所述反转录引物序列为:5’-atacgactcactattagggc-3’。
7.根据权利要求2所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,所述(n20)-lib文库序列为:
8.根据权利要求2所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,所述2’-全修饰的xna文库的序列为:
9.根据权利要求4所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,所述用于高通量测序引物seq-f的序列为:
10.根据权利要求4所述靶向pbp2a的非天然核酸适配体获取方法,其特征在于,seq-r的序列为:5’-ctagcataaccccttggggcaatacgactcactattaggg-3’。
技术总结
本发明公开靶向PBP2a的非天然核酸适配体获取方法。该方法为筛选靶向PBP2a非天然核酸适配体的过程中,第二轮以后的转录文库制备方法主要采用5’‑磷酸化扩增引物对筛选到的XNA序列反转录产物做PCR扩增,该引物与模板3’‑端互补40nt,包含与转录产物5’‑端互补的20nt以及转录引物部分。PCR扩增产物用Lambda核酸外切酶降解,通过控制降解时间,能最大效率降解掉磷酸化的序列以获取单链DNA文库,然后再使用SFM4‑6聚合酶转录获取下一轮的XNA文库。在筛选靶向PBP2a非天然核酸适配体的过程中,大多数未结合的XNA文库被除去,随着筛选轮数的增加,与靶标蛋白有结合活性的XNA逐渐富集。
技术研发人员:陈庭坚,刘巧玲,彭乐丽,张瑞敏,杜宇辉
受保护的技术使用者:华南理工大学
技术研发日:
技术公布日:2024/3/11
- 还没有人留言评论。精彩留言会获得点赞!