使用一堆散焦全息图像来识别生物颗粒的方法与流程
本发明大体涉及生物颗粒的光学分析的领域,特别适用于微生物诊断,更特别地,适用于微生物和/或它们应激反应条件的识别。生物颗粒的光学分析还可以应用于细胞培养的监测。
背景技术:
数字全息显微术,dhm,是一种已知的成像技术,使传统的光学显微术的景深限制被克服。示意性地,数字全息显微术包括:记录由所观测的物体所衍射的光波和具有空间相干性的参考波之间的干涉所形成的全息图。在myungk.kim于2010年1月在spiereviews第1卷第1号发表的题为《principlesandtechniquesofdigitalholographicmicroscopy(数字全息显微术的原理和技术)》的文章中可以找到对数字全息显微术的概述。
近来提出了将数字全息显微术用于微生物的自动识别。例如,n.wu等人在computationalandmathematicalmethodsinmedicine第2013卷、文章号id162105中发表的题为《three-dimensionalidentificationofmicroorganismsusingadigitalholographicmicroscope(使用数字全息显微镜的微生物三维识别)》的文章描述了一种方法,该方法用于通过朝着与颗粒上的聚焦对应的平面的数值传播来识别待分析的体积中细菌的不同类型。在不同深度聚焦的图像被用于对微生物的三维表示进行重构。然后,使用非线性3d滤波来对在它们进行分类。
类似地,ahmedelmallahi于2013年1月在appliedoptics第52卷第1号中发表的题为《automatedthree-dimensionaldetectionandclassificationoflivingorganismsusingdigitalholographymicroscopywithpartialspatialcoherentsource:applicationtomonitoringofdrinkingwaterresources(使用具有局部空间相干源的数字全息显微术的活有机体的自动三维检测和分类:应用于饮用水资源的监测)》的文章描述了一种方法,该方法包括:用于检测待分析的体积中的细菌的位置的第一步骤;使用数值传播而在体积中的不同深度处聚焦的聚焦步骤;随后根据细菌的形态特征来对细菌进行分类。
然而,前述识别方法是复杂的,因为它们需要在相继的焦平面中聚焦。相反地,对于以低误检率识别微生物类型来说,在单个焦平面中(换言之,在单个分析深度处)的聚焦一般而言是不够的。
因此,本发明的目的是提出一种用于通过数字全息显微术来识别有机颗粒的方法,该方法允许获得低误检率而同时该方法是简单又鲁棒的。
技术实现要素:
本发明是由一种根据使用光学系统所获得的一堆全息图像来识别生物颗粒的方法来限定的,其中:
–针对相对于焦平面的多个散焦偏差来获得所述全息图像,沿着所述光学系统的光轴取得所述散焦偏差;
-在所述一堆全息图像中选择与焦平面最接近的图像作为参考全息图像;
-针对感兴趣生物颗粒,从所述一堆全息图像中提取包括所述感兴趣生物颗粒的一堆图像块;
-从所提取的一堆图像块中识别感兴趣颗粒的类型。
最小散焦偏差优选地大于感兴趣生物颗粒的最大厚度,使得图像块包括感兴趣生物颗粒的不多于单个聚焦图像。
根据实施例的第一变形例,由所述光学系统针对沿着所述光轴的多个位置来获取所述全息图像。
根据实施例的第二变形例,由所述光学系统来获取第一全息图像,并且使用数值传播模型根据所述第一全息图像来计算所述一堆全息图像中的其他全息图像。在该情况下,按照相对于所述焦平面的非零散焦偏差来取得所述第一全息图像。
可以从所述一堆全息图像中选择使预定对比度准则最大化的图像作为所述参考图像。
接下来,在所述一堆图像块中选择属于所述参考图像的且以所述感兴趣颗粒为中心的图像块作为参考块,然后选择所述散焦偏差的原点作为所述参考块在所述光轴上的位置。
可以通过在以所述感兴趣颗粒为中心的且属于所述一堆全息图像中所述参考图像的相邻图像的图像块中搜索使预定对比度准则最大化的块来更新参考块。
根据第一实施例,针对所述一堆图像块中的每个块,计算该块上的至少一个特征量的值,并且根据由此计算出的特征量的值沿着所述光轴来获得所述特征量的轮廓。
然后,将所述特征量的轮廓与阈值进行比较,并且当该轮廓超过该阈值时推断所述感兴趣生物颗粒是第一类型的,而当该轮廓不超过该阈值时推断所述感兴趣生物颗粒是第二类型的。
或者,使用相似度准则,将所述特征量的轮廓与针对不同的已知生物颗粒类型而获得的多个标准轮廓进行比较,并且根据与所述特征量的轮廓有最大相似度的标准轮廓来推断生物颗粒的类型。
所述相似度准则可以选自相关性、皮尔森相关系数、二次偏差。
又或者,使用监督学习方法而在多个轮廓种类之中对所述特征量的轮廓进行分类,每个种类与给定的生物颗粒类型对应。
根据第二实施例,在所述一堆图像块中选择与预定散焦偏差对应的多个块。
然后,使用相似度准则在所述选择的多个块与同一批多个标准块之间进行比较,每批多个标准块与给定的生物颗粒类型对应,根据与所述选择的多个块有最大相似度的多个标准块来推断感兴趣生物颗粒的类型。
这里,所述相似度准则也可以是相关性、二次偏差、空间傅里叶变换后的二次偏差、基于主成分分析的准则。
附图说明
在参照附图来阅读优选实施例时,本发明的其他特征和优点将变得明显,在附图中:
图1示意性地示出了可在用于识别生物颗粒的本发明方法中使用的、用于记录全息图像的装置;
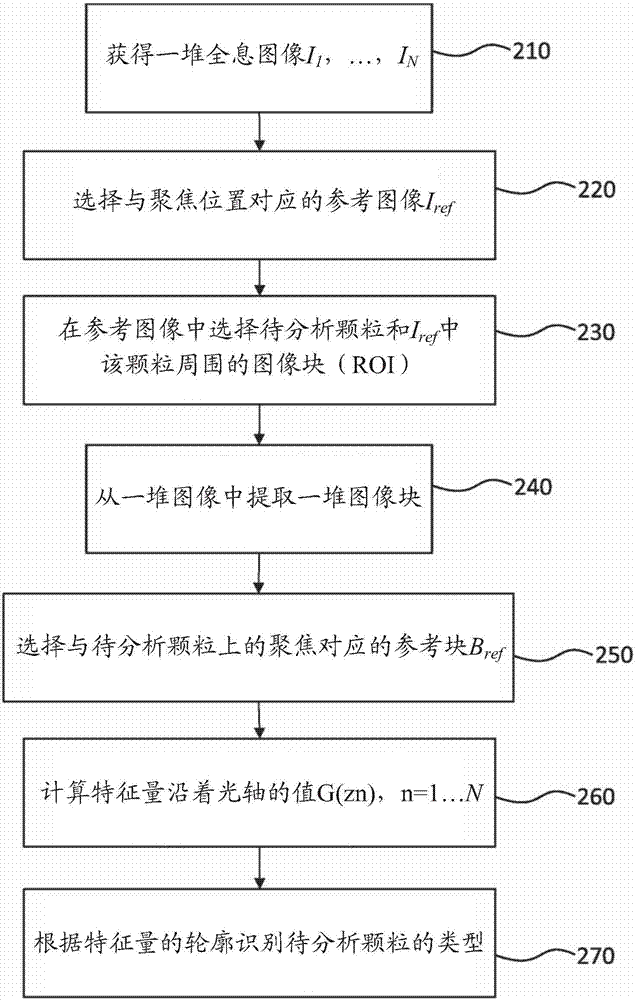
图2示意性地示出了根据本发明第一实施例的用于识别生物颗粒的方法的流程图;
图3示意性地示出了根据一堆图像提取一堆图像块;
图4示意性地示出了在一堆块中搜索参考块;
图5a和图5b分别示出了两个已知生物颗粒类型的特征量的轴向轮廓;
图6示意性地示出了根据本发明第二实施例的用于识别生物颗粒的方法的流程图;
图7给出了用于不同的已知生物颗粒类型的标准块的m元组的示例。
具体实施方式
本发明的用于识别生物颗粒的方法依赖于用于记录全息图像的设备,例如,结合图1描述的用于记录全息图像的设备。
该设备100包括优选地时间上相干的、窄谱宽的光源110,谱宽例如小于200nm或者甚至小于100nm,或者甚至小于25nm。该光源尤其可以是激光二极管、或发光二极管。优选地,光源是空间上相干的。光源发射的光束由待分析的样品下方的光纤120来传递。该样品是含有待识别生物颗粒p的液体,诸如水、缓冲溶液、培养基或反应介质。或者,样品可以是固体介质的形式,优选地,透明的固体介质,诸如包含所考虑的颗粒的琼脂。样品还可以是气体介质。生物颗粒可以被包含于样品的表面上或介质内部。
待识别的生物颗粒可以是微生物,例如,细菌或酵母。待识别的生物颗粒还可以是细胞、多细胞生物或任何其他污染类型颗粒或尘埃颗粒。
所观察的颗粒的尺寸介于100nm与几百微米(甚至几毫米)之间。
样品被容纳在分析室130中,分析室130是由下载玻片131(例如,传统的显微镜载玻片)和上载玻片132来竖直地界定的。分析室是由粘合剂135或任何其他防渗材料来横向地界定的。对于光源的波长来说,下载玻片和上载玻片是透明的。生物颗粒被固定于分析室中,这是因为含有生物颗粒的介质是固体的(琼脂)或因为介质是流体但是颗粒附着于上载玻片的内表面132i。如果颗粒的运动速度足够慢使得在测量时间期间能够认为颗粒是固定的,则颗粒可以是移动的。
装置100还包括光学系统140,光学系统140例如由显微镜物镜145和镜筒透镜141形成。光学系统在光学上配备有滤波器143,滤波器143可以位于物镜的前方或位于物镜和镜筒透镜之间。
光学系统140尤其由其光轴δ、物平面或焦平面、以及其像平面πi表征,焦平面在距物镜的距离d处,像平面通过光学系统与物平面共轭。换言之,对于位于物平面πo中的物体,在像平面πi中存在该物体的对应清晰图像。物平面和像平面与光轴δ正交。
图像传感器150(例如,ccd或cmos传感器)位于像平面πi中,或在像平面附近。因此,传感器150通过焦平面的一部分的透射来获取图像。
光学系统140相对于分析室130的位置可以被竖直地调整。例如,物镜被固定于能够沿着竖直轨道移动的透镜支架。因此,可以聚焦于一个或多个感兴趣的生物颗粒上。
在图像传感器上形成的图像是全息图像,这是由于该图像是由生物颗粒所衍射的波与穿过样品而不与样品相互作用的参考波之间的干涉得到的。
或者,可以将光束分成两个分量,例如使用半透明板(未示出)来将光束分成两个分量。然后,第一分量用作参考波,而第二分量被样品衍射,光学系统的像平面中的图像是由衍射波和参考波之间的干涉得到的。
然后,使用本发明的识别方法来处理由此获取的样品图像。
图2示意性地示出了根据本发明第一实施例的用于识别生物颗粒的方法的流程图。
在步骤210,获得样品中的感兴趣生物颗粒的多个(或一堆)全息图像。
根据第一变形例,在实验中通过成像系统来获取这些图像,这些图像中的每个图像对应于沿着光轴δ取得的距分析室的不同距离。
根据第二变形例,在距分析室的第一距离处获取第一图像,该距离不一定对应于这些颗粒上的聚焦条件。尤其是要注意到,当对于光源的波长而言待分析生物颗粒为透明的时,该第一图像不会是在聚焦条件下取得的,而是在距聚焦位置预定距离处得到的。该距离优选地短于2mm,更优选地短于1mm,甚至短于500μm。那么,由此获取的该第一图像被说成是散焦的。
使用如下面阐述的数值传播模型来根据初始的散焦图像来计算额外的图像。计算出的额外图像是会在光学系统与分析室之间的不同距离处(即,在成像系统的不同轴位置处)观察到的图像。
在sang-hyuklee等人于2007年2月19日在opticsexpress第15卷第4号第1505-1512页中发表的题为《holographicmicroscopyofholographicallytrappedthree-dimensionalstructures(全息捕获的三维结构的全息显微术)》的文章中阐述了通过数值传播进行的图像计算方法。
更具体地,如果瑞利-索末菲(rayleigh-sommerfeld)的传播函数被记为hz(r),即:
其中,z是散焦高度,换言之,距焦平面的偏差,r=(x,y)是像平面中的位置,r2=r2+z2和k=2πn/λ是相对于传播介质的波数,那么坐标平面z中的波可以被表达为如下形式:
其中b(q)是焦平面中的衍射波的强度b(r)的傅里叶变换(这里,参考波的强度被假设为恒定),h-z(q)是h-z(r)的傅里叶变换,以及q是傅里叶变换中的r的对耦变量。
因此,将理解到,可以针对沿着光轴的坐标z1,...,zn构建一堆图像i1,...,in,坐标的原点取在轴聚焦位置处,每个图像in均由复数幅度a(r,zn)来限定。
在步骤220,在前述步骤处获得的一堆图像i1,...,in中选择参考图像iref。该参考图像是与感兴趣生物颗粒上的理想聚焦条件最对应的图像。在理想实验条件下,颗粒位于上载玻片132的内表面132i上,并且该表面垂直于光轴δ。然后,理想聚焦条件是其中焦平面与前述内表面合并的条件。
在实践中,当生物颗粒不透明时,可以根据应用于含有生物颗粒的区域的最大对比度准则来选择参考图像。该最大对比度准则可以是例如最大标准差,或该区域中的最大梯度的均值。
当生物颗粒透明时,有利地,使用具有球面像差的光学系统以避免在聚焦位置处的信号的完全消失。
在所有情况下,参考图像的轴位置则会被取为参考位置(z=0),相对于该参考位置来计算该一堆图像中的其他图像的位置。
在步骤230,在参考图像中选择至少一个待分析颗粒。该选择可以是自动的,并且例如基于形态和/或光度准则来执行。
对于每个待分析颗粒,在参考图像中确定待分析颗粒的位置(x,y),按照以该位置为中心的图像块bref的形式来确定感兴趣区域。将注意到,该图像块可以比待分析颗粒的尺寸更大或更小。该图像块bref提取自参考图像iref。
在步骤240,根据该一堆图像中的其他图像,提取与提取自参考图像的图像块的同一位置对应的图像块。这给出了一堆图像块b1,...,bn,即,将该一堆图像限制于以待分析颗粒为中心的感兴趣区域。将理解到,该一堆图像中的每个图像块均与不同轴位置对应,并且因此与相对于参考块bref的不同散焦条件对应。
图3示意性地示出了一堆图像i1,...,in、以及以具有坐标(x,y)的感兴趣颗粒p为中心的一堆图像块b1,...,bn。这里,图像块是方形的,但是将理解到,在不背离发明的范围的情况下可以构想其他块形状。
可选地,在步骤250,精细调节参考图像的选择,并且因此还针对待分析颗粒来精细调节参考图像块的选择。如果不是所有颗粒都位于与光轴正交的同一平面中(当上载玻片的内表面不与光轴完全正交时尤其是这种情况),则一个颗粒与另一个颗粒的焦平面可能不同。然后,在该一堆图像块中进行搜索,并且有利地,在步骤220处选择的参考图像iref的两侧的图像块中搜索与聚焦条件最对应的图像块。如在步骤220处那样,可以使用最大对比度准则来进行该选择,但是这时在该一堆图像块上进行。这里,最大对比度准则也可以是最大标准差,或者图像块上的最大梯度的均值。然后,所选择的图像块成为该一堆图像块中的新参考图像块bref。
图4示出了在生物颗粒的一堆图像块中搜索参考块的示例。沿着x轴给出图像块的下标,每个图像块bn对应于不同的轴位置zn,并且沿着y轴给出图像块中的像素强度的标准差。
步骤220处的参考图像iref的搜索、以及步骤230处的以感兴趣颗粒为中心的图像块的提取给出了第一参考块,第一参考块在该图中被命名为
在步骤260,针对该一堆图像块的每个图像块bn,计算至少一个特征量g(zn),沿着光轴来推导该特征量的轴向轮廓。更具体地,如果图像块b1,...,bn对应于轴位置z1,...,zn,则图像块的轴向轮廓包括序列g(z1),g(z2),...,g(zn)。
该特征量尤其可以是与图像块相关的统计量,例如,图像块中的像素强度的均值、中值、标准差。此外,如果该一堆图像是通过数值传播获得的而不是实验获取的,则复数值可以与图像块的每个像素相关联。那么,特征量可以与这些复数值的实部或虚部相关联。例如,作为特征量,可以取图像块中的复数量的虚部的均值的平方,即,对于图像块bn:。
其中,e(.)表示与r∈bn相关的均值。
可选地,可插入离散值g(z1),g(z2),...,g(zn)以获得特征量的轮廓的更加精细调节的分辨率。
在步骤270,根据由此获得的轴向轮廓来识别生物颗粒的类型。
根据第一特别简单的变形例,轴向轮廓g(z)可以与阈值进行比较,从而区分两类颗粒。
图5a和图5b示出了针对两类生物颗粒(即,表皮葡萄球菌(图5a)和金黄色葡萄球菌)的特征量(这里,图像块中的量的虚部的均值的平方)的轴向轮廓。这里可以看到,阈值th(诸如0.4)能够有效地区分这两类生物颗粒(这里,两类葡萄球菌)。如果轴向轮廓超过该阈值,则该生物颗粒将被识别为表皮葡萄球菌,而如果轴向轮廓不超过该阈值,则该生物颗粒将被识别为金黄色葡萄球菌。
将注意到,该第一变形例不要求精确确定参考块bref。然而,必须在步骤230在参考图像iref中进行搜索以选择待分析的颗粒。
在第二变形例中,使用相似度准则来对特征量的轴向轮廓与例如存储在数据库中的之前针对已知类型的生物颗粒获得的该同一特征量的轴向轮廓g1(z),...,gk(z)进行比较。轮廓g1(z),...,gk(z)被记为标准轮廓。使用共同原点(参考块)的事实便于进行比较。
相似度准则可以是例如相关性值,或皮尔森相关系数。与轮廓g(z)最接近的标准轮廓gk(z)的下标kmax∈{1,...,k}给出了生物颗粒的类型。
在第三识别变形例中,在步骤270,针对该一堆图像中的每个图像块bn来计算几个特征量
在第四变形例中,颗粒识别使用了监督学习方法。该变形例假设针对多个k类生物颗粒事先获取轴向轮廓。然后,使用轴向轮廓的值或者优选地该轮廓的特征作为描述符来将生物颗粒分类为k类,能够由l维空间中的一组点来表示每类。
根据待分析的生物颗粒的轴向轮廓,确定l维空间中的对应点,并搜索该组点,即,该组点所属于的类。
例如,如果表达式(3)所限定的量被取为图像块的特征量,并且其最大值被取为轮廓特征,则能够显示出可以获得细菌:约氏不动杆菌、产气肠杆菌、大肠杆菌、表皮葡萄球菌的有效分类。因此,在已知种群的样品上,获得了以下混合矩阵:
其中,不同行对应于不同类型的种群,不同列对应于识别方法所预测的类。将注意到,混合矩阵是对角占优的并且因此识别错误率相对低。
本领域技术人员将理解到,通常的监督学习方法,诸如贝叶斯分类技术或支持向量机,可用于根据生物颗粒的轴向轮廓来对生物颗粒进行分类。
图6示例性地示出了根据本发明第二实施例的用于识别生物颗粒的方法的流程图。
本发明的第二识别方法还基于以感兴趣生物颗粒为中心的一堆图像块。从如之前所描述的所获取的或所计算的一堆图像中提取这些图像块。更具体地,步骤610至650与根据本发明第一实施例的识别方法的步骤610至650分别相同。因此,将不进一步描述这些步骤。
然而,在步骤660,不计算特征量,而是在一堆图像块中选择位于相对于参考块bref预定偏差(enz)处(z=0)的子多个m个图像块。在实践中,以有规律的间隔获取或计算(通过数值传播)该一堆图像,从一堆图像块中进行选择将涉及相对于参考块下标的预定下标。
在步骤670,使用相似度准则,将所选的图像块的m元组与标准块的m元组进行比较,标准块的每个m元组涉及一个已知类型的生物颗粒,每个m元组自身中的图像块是以前述预定距离来获得的。
相似度准则可以是待分析颗粒的图像块的傅里叶变换与标准图像块的变换之间的空间相关性、皮尔森相关系数、二次偏差,甚至可以基于主成分分析(pca)。在基于主成分分析的情况下,对于待分析颗粒的图像块的m元组中的每个图像块和标准图像块的m元组中的每个图像块,可以确定像素分布的主轴,并且可以在待分析颗粒的图像块的主轴与标准块的主轴的对准之间进行比较(例如,利用方向向量的标量积)。
不论所选择的相似度准则如何,在步骤680,与待分析颗粒的图像块的m元组最接近的标准块的m元组给出了生物颗粒的类型。
图7给出了针对不同生物颗粒的标准块的m元组的示例。
在所示出的情况下,m=3并且预定散焦偏差分别为15μm(第一列中的图像块)、6μm(第二列中的图像块)和0μm(焦平面中的第三列中的图像块),在光束的传播方向上正向地计算该偏差。
第一行中的标准块涉及种类大肠杆菌,第二行中的标准块涉及种类约氏不动杆菌,第三行中的标准块涉及种类表皮葡萄球菌。
可以看出,种类大肠杆菌与种类约氏不动杆菌的标准块在15μm处具有非常相似的结构,而表皮葡萄球菌的标准块具有很不同的结构。类似地,种类约氏不动杆菌和种类表皮葡萄球菌的标准块在0μm处具有非常类似的结构,而大肠杆菌具有很不同的结构。因此,使用三个一组的图像块能够消除识别模糊。
- 还没有人留言评论。精彩留言会获得点赞!