一种用于鉴别与定量多元细菌的表面增强拉曼光谱方法
1.本发明属于细菌检测领域,尤其是涉及一种用于鉴别与定量多元细菌的表面增强拉曼光谱方法。
背景技术:
2.食源性疾病是我国食品安全面临的主要公共卫生问题,许多由病原菌感染或污染引起的食源性疾病都危及生命,提高病原菌检测水平是预防这类疾病的重要途径。在实际微生物污染中,往往不局限于单一病原菌,而是呈现多种病原菌混合污染的复杂局面。因此,亟需开发快速无损的多种病原菌的检测方法以有效控制细菌污染。然而,现有的微生物检测大多依赖于培养法,这种方法检测与鉴定时间较长,往往需要24-72h。此外,聚合酶链式反应与酶联免疫吸附技术在多元菌的同时检测中取得了一些成果,但是仍存在检测灵敏度与干扰性的问题。近年来,基于光学的检测技术在病原菌的检测应用中取得了良好的成果,并且与多元统计分析以及深度学习算法的结合,可以提高对数据处理的通量和分析的准确性,在多元菌的鉴别和检测分析中有良好的应用前景。
3.表面增强拉曼光谱散射(sers)技术是一种基于金属表面的等离子体共振激发活性基底的局域电场产生“热点”,从而使拉曼信号大大增强的光谱分析技术,因此活性金属基底的选择对sers的产生至关重要。纳米溶胶基底制备方法成熟,使用场景灵活方便,便于大多数场景的快速检测。当病原菌与金属纳米溶胶接触后,产生的热运动使得纳米溶胶发生一定程度的聚集而形成sers“热点”,从而便于病原菌的高灵敏检测。目前,使用多元统计分析方法驱动的sers技术在多元菌的鉴别检测方面有一些应用,但是操作方法还比较繁琐,而在多元混合菌的鉴别分析及同时定量检测方面还鲜有应用。
技术实现要素:
4.有鉴于此,针对混合细菌污染的实际样本中细菌含量多、定量检测难度大等问题,本发明提出一种适用于多元细菌同步鉴别和灵敏检测的表面增强拉曼光谱方法。
5.为达到上述目的,本发明的技术方案是这样实现的:
6.本发明提供了一种用于鉴别与定量多元细菌的表面增强拉曼光谱方法,包括如下步骤和方法:
7.(1)对多元菌纳米溶液进行拉曼光谱测定,对得到的光谱进行预处理后,使用主成分分析、偏最小二乘判别分析或正交偏最小二乘判别分析中的至少一种进行分析,对多元菌纳米溶液进行鉴别;
8.(2)对多元菌纳米溶液进行拉曼光谱测定,对得到的光谱进行预处理后,使用偏最小二乘回归法或人工神经网络分析算法进行分析,对多元菌纳米溶液进行定量。
9.进一步,所述的多元菌纳米溶液包括一种或两种以上的单一菌;所述的多元菌纳米溶液中单一菌的浓度均为10
5-107cfu/ml;所述的多元菌纳米溶液中多元菌的总浓度为10
5-107cfu/ml。(所述的多元菌为两种以上的单一菌)
10.进一步,所述的单一菌为大肠埃希氏菌、金黄色葡萄球菌、鼠伤寒沙门氏菌、志贺氏菌、李斯特氏菌、副溶血性弧菌、铜绿假单胞菌或枯草芽孢杆菌中的至少一种。
11.进一步,所述的步骤(1)中的预处理步骤的方法为多元散射校正(msc)、标准正态变量(snv)、一阶导数(1-d)、savitzky-golay平滑(s-g)或行中心化(rc)中的至少一种;优选的,对得到的光谱进行标准正态变量处理后,使用正交偏最小二乘判别分析进行分析,对多元菌纳米溶液进行鉴别。
12.进一步,所述的步骤(2)中的预处理步骤的方法为多元散射校正、标准正态变量、一阶导数、savitzky-golay平滑或行中心化中的至少一种。
13.r2x和r2y分别为x和y矩阵的模型可解释率,q2代表模型的预测能力。根据这三个参数评价模型的预测能力,从而筛选出最佳判别分析模型。
14.进一步,所述的步骤(2)中的偏最小二乘回归法对光谱进行模型优化;所述的模型优化的方法为变量重要性投影(vip)、连续投影算法(spa)或竞争性自适应重加权采样法(cars)中的一种;所述的偏最小二乘回归法的模型评判参数为交叉验证均方根误差(rmsecv)、均方根误差(rmse)或相关系数r2中的至少一种。误差越小越好,相关系数越高越好。交叉验证是通过k折交叉验证实现的,k=7。但是在预测集中,同样选取均方根误差和相关系数r2作为模型评判参数,这时均方根误差没有做交叉验证。
15.进一步,所述的步骤(2)中的人工神经网络分析算法包括输入层、隐含层和输出层;所述的人工神经网络分析算法对隐藏层、学习率、隐藏节点数或训练回合数进行筛选来建立最优的人工神经网络定量模型;所述的隐藏层的层数为1-4层;所述的学习率为0.1、0.01、0.001或0.0001;所述的隐藏节点数为16、32或48;所述的训练回合数为100、200、300、400或500;所述的筛选的方法为热图分析法,通过绘制隐藏层与学习率和隐藏节点数与训练回合数的四维热图,通过热图颜色变化找到均方根误差最小的超参数值,来建立最优的人工神经网络定量分析模型。不同对应的(rmse)值,值越大,颜色越浅,模型效果越差。反之,颜色越深,值越小,颜色越深,模型效果误差越小,模型效果越好。运用这种方法可以便捷准确的筛选出最佳的人工神经网络算法中的超参数组合,从而找到最佳的人工神经网络模型用于细菌定量分析。需要说明的是,人工神经网络算法得出的定量模型均比偏最小二乘回归法定量模型效果好,说明这种方法在处理细菌的拉曼光谱数据时更有优势。
16.所述的光谱的数量大于200条,选取其中80%的光谱作为测试集样本,剩余光谱作为验证集样本用于建模分析;所述的光谱的分析处理范围为400-1800cm-1
;
17.所述的拉曼光谱测定的激发波长为633nm,功率为2-3mw,光谱采集时间为10-20s,光谱采集范围为50-3500cm-1
。
18.进一步,所述的多元菌纳米溶液由包括如下步骤的方法制成:将纳米胶体溶液与多元菌溶液混合,将得到的混合液滴加到金箔纸芯片上即成;所述的纳米胶体溶液与多元菌溶液的体积比为1:1;所述的纳米胶体溶液的浓度为0.02-1mm。
19.进一步,所述的多元菌溶液由多元菌与无机盐溶液混合而成;所述的无机盐溶液的阳离子浓度为0.1-1mol/l;所述的无机盐溶液由至少一种氯化物溶液与至少一种磷酸盐溶液混合而成;所述的氯化物为氯化钠或氯化钾中的至少一种;所述的磷酸盐为磷酸氢二钠、磷酸二氢钠、磷酸二氢钾或磷酸氢二钾中的至少一种;
20.所述的多元菌溶液由包括如下步骤的方法制成:震荡培养多元菌,37℃条件下
100-250r/min培养16-18小时,使用所述的无机盐溶液将得到的多元菌培养液在3000-5000r/min下洗涤5-15min,将洗涤后的多元菌溶液冷藏即成;
21.所述的纳米胶体溶液为纳米金、纳米银、纳米金核银壳合金、纳米银核金壳合金、纳米金核银壳硅最外壳合金或纳米银核金壳硅最外壳合金中的一种;所述的纳米胶体溶液中的纳米胶体的粒径为50-100nm。
22.相对于现有技术,本发明具有以下优势:
23.本发明所述的用于鉴别与定量多元细菌的表面增强拉曼光谱方法结合多元统计分析可快速实现对五种以上病原菌的判别分析;通过分别与偏最小二乘回归分析、深度神经网络的结合,可同时快速对多元病原菌进行定量分析,具有分析速度快、方便快捷等优势。
24.本发明所述的用于鉴别与定量多元细菌的表面增强拉曼光谱方法所采用的纳米胶体溶液性质稳定,尺寸均一,可长期存储,增强因子高,将病原菌的拉曼信号大大增强,得到病原菌的高质量sers谱图。
25.本发明所述的用于鉴别与定量多元细菌的表面增强拉曼光谱方法中的纳米胶体溶液与病原菌的混合方法简单,在一定ph值范围内通过简单的涡旋混合即可实现病原菌与纳米粒子的结合,制样简单,使用灵活方便。
26.本发明所述的用于鉴别与定量多元细菌的表面增强拉曼光谱方法只需将溶解在无机盐溶液中的细菌与纳米胶体溶液以等体积比例混合,在金箔纸芯片上即可获得增强型的细菌拉曼信号,并通过特定的模型预处理方法,建立可靠的定性判别模型和准确的定量分析模型。
附图说明
27.图1为本发明实施例1所述的复合纳米材料(au@ag@sio2)的透射电镜图。
28.图2为本发明实施例1所述的紫外光谱图:其中曲线a为au纳米粒子,b为au@ag纳米粒子,c为au@ag@sio2纳米粒子。
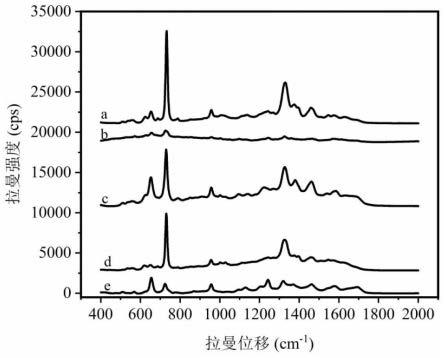
29.图3为本发明实施例3所述的五种细菌的拉曼谱图,其中a为枯草芽孢杆菌,b为铜绿假单胞菌,c为鼠伤寒沙门氏菌,d为金黄色葡萄球菌,e为大肠埃希氏菌。
30.图4为本发明实施例3所述的金黄色葡萄球菌、大肠埃希菌与鼠伤寒沙门氏菌三种混合菌的拉曼谱图。
31.图5为本发明实施例3中不同浓度枯草芽孢杆菌的拉曼谱图对比效果图。
32.图6为本发明实施例4所述的5种细菌定性分析的pca得分散点图。
33.图7为本发明实施例4所述的三类两两混合菌与三种纯菌液的opls-da判别模型效果图。
34.图8为本发明实施例4所述的三种混合菌与三种单一细菌的opls-da判别模型效果图。
35.图9为本发明实施例4中聚类分析(hca)的模型效果图。
36.图10为本发明实施例5和实施例6所述的三种混合细菌最优的偏最小二乘回归法(plsr)定量分析模型的预测指标实际值与预测值相关图:10-a为金黄色葡萄球菌的校正集,10-b为金黄色葡萄球菌的预测集,10-c为大肠埃希氏菌的校正集,10-d为大肠埃希氏菌
的预测集,10-e为鼠伤寒沙门氏菌的校正集,10-f为鼠伤寒沙门氏菌的预测集。
37.图11为本发明实施例7所述的金黄色葡萄球菌的热图分析图。
38.图12为本发明实施例7所述的三种混合菌最优的人工神经网络分析(ann)模型的预测指标实际值与预测值相关图:12-a为金黄色葡萄球菌的校正集,12-b为金黄色葡萄球菌的预测集,12-c为大肠埃希氏菌的校正集,12-d为大肠埃希氏菌的预测集,12-e为鼠伤寒沙门氏菌的校正集,12-f为鼠伤寒沙门氏菌的预测集。
具体实施方式
39.除有定义外,以下实施例中所用的技术术语具有与本发明所属领域技术人员普遍理解的相同含义。以下实施例中所用的试验试剂,如无特殊说明,均为常规生化试剂;所述实验方法,如无特殊说明,均为常规方法。
40.本发明实施例中的金属试纸条购于苏州中通传感科技有限公司。
41.本发明实施例中的肠道致病性大肠埃希氏菌(保藏号为cicc10372)、金黄色葡萄球菌(保藏号为cicc 10473)、鼠伤寒沙门氏菌(保藏号为cicc 21483)、铜绿假单胞菌(保藏号为cicc 21636)购买自中国工业微生物菌种保藏管理中心。枯草芽孢杆菌(保藏号为cmcc(b)63501)购买自中国菌种保藏中心。
42.其余的试剂都为普通化学试剂,实验用水为二次蒸馏水。
43.下面结合实施例来详细说明本发明。
44.实施例1纳米胶体溶液(au@ag@sio2)的制备
45.首先,配制50ml摩尔浓度为1mm的氯金酸溶液与5ml质量百分比为1%的柠檬酸钠溶液,将50ml氯金酸溶液置于圆底烧瓶中加热至微沸,接下来在快速搅拌与回流的状态下,注入5ml配置好的柠檬酸钠溶液,并继续加热10min,停止加热后继续搅拌冷却至室温。之后收集与玻璃瓶中置于4℃保存,得到金纳米粒子。
46.然后,在取得上述金纳米粒子的基础上制备au@ag纳米粒子。以质量分数为1%的柠檬酸钠作为稳定剂,摩尔质量为0.1m的抗坏血酸作为还原剂在金纳米种子溶液表面还原银纳米粒子,具体操作步骤为:将0.7ml金纳米种子溶液与20ml超纯水以及1.0ml柠檬酸钠和3.0ml抗坏血酸在室温下搅拌5min。在剧烈的搅拌下,使用电动注射器以0.15ml/min的速率滴加0.7ml摩尔浓度为10mm的agno3溶液至上述混合溶液中,待反应完成后,制备得到的合金纳米粒子在100℃下老化2h。最终制备得到au@ag纳米粒子,并保存于4℃条件下。
47.最后,在上述au@ag纳米粒子的基础上包覆sio2壳层,具体操作步骤为:将上述步骤制备得到的30mlau@ag纳米粒子置于圆底烧瓶,加入0.4ml摩尔浓度为1mm的3-氨丙基三甲氧基硅烷作为涂层交联剂在室温下快速搅拌15min。与此同时,将硅酸钠稀释至质量分数为0.54%的稀溶液,使用稀盐酸调节硅酸钠的ph值至10.5,然后,向反应物中加入3.2ml酸化后的硅酸钠,继续在室温下搅拌3min。之后,将反应温度升至90℃持续45分钟。最后,将热反应物转移到ep管中并在冰水浴中冷却使反应停止。接着将冷却后的复合纳米材料以5500rmp离心15分钟去除上清液后使用超纯水洗涤一次,再次离心得到纯净的au@ag@sio2纳米颗粒,粒径为50nm,4℃保存备用。图1为所得到纳米粒子的透射电子显微图片。图2为au纳米粒子,au@ag纳米粒子和au@ag@sio2纳米粒子的紫外吸收光谱图。
48.实施例2多元菌纳米溶液的制备
49.以大肠埃希氏菌、金黄色葡萄球菌、鼠伤寒沙门氏菌、铜绿假单胞菌和枯草芽孢杆菌作为判别分析模型菌,使用平板计数法对培养后的五种单一细菌菌液进行计数,然后分别进行梯度稀释,将单种细菌的菌液浓度控制在5
×
107cfu/ml。
50.以大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌的混合菌作为模型混合菌。具体的配置方法为以浓度分别为5
×
107cfu/ml的大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌为起始原料菌液,三种菌的体积按照0-1.0ml范围混合加入,保证混合菌的总体积维持在1ml,使得混合菌液总浓度为仍为5
×
107cfu/ml。
51.将纳米胶体溶液(au@ag@sio2)与混合菌液以1:1的体积比混合,涡旋15min后使其充分混合均匀得到多元菌纳米溶液。
52.实施例3多元菌纳米溶液拉曼信号的获得
53.通过拉曼显微镜在10倍物镜下聚焦观察样品,光谱采集条件为:激光器633nm,激光能量2mw,光谱范围50-3500cm-1
,扫描时间:16s。每个测试基片上随机采集50条拉曼光谱,最少平行测试三个基片。图3为五种细菌的拉曼谱图,其中a为枯草芽孢杆菌,b为铜绿假单胞菌,c为鼠伤寒沙门氏菌,d为金黄色葡萄球菌,e为大肠埃希氏菌。可以看出5种细菌的拉曼谱图重叠比较严重,直接鉴别分析难度较大。图4为金黄色葡萄球菌、大肠埃希菌与鼠伤寒沙门氏菌三种混合菌的拉曼谱图。混合光谱图中兼具每种细菌谱峰信息,但是重叠干扰严重,为混合样本的直接分析带来难度。
54.图5为不同浓度枯草芽孢杆菌的拉曼谱图对比效果图。从图上可以看出,当浓度为104cfu/ml时,细菌基本没有拉曼信号,以730cm-1
峰位为例,细菌浓度从107cfu/ml降至104cfu/ml时,拉曼强度对应的变化为11440,4288,758,0。说明当细菌浓度为104cfu/ml时,此时的数据不能用于鉴别或定量分析。所以,本发明对单一菌种或混合细菌的浓度范围进行了限定,以保证分析方法的准确。
55.实施例4细菌鉴别分析模型的建立:
56.结合不同模型对测试光谱数据进行分析,以x矩阵的累计解释率r2x(cum),y矩阵的累计解释率r2y(cum)和模型的可预测性q2(cum)作为模型评价指标,不同预处理方法与模型的检测性能见表1。对比发现,对光谱进行snv预处理,采用正交偏最小二乘判别分析(opls-da)对五种细菌的判别分析效果最佳,此时r2x(cum)为0.947,r2y(cum)为0.960,q2(cum)为0.939,表明构建的opls-da模型对细菌的判别分析效果较优。
57.图6为所述的大肠埃希氏菌、金黄色葡萄球菌、鼠伤寒沙门氏菌、铜绿假单胞菌和枯草芽孢杆菌5种细菌pca分析的得分散点图,可以看出5种细菌基本能得到区分。
58.表1不同预处理结合分类模型效果对比
59.分析方法r2x(cum)r2y(cum)q2(cum)pca0.919/0.888pls-da0.930.9410.901opls-da0.9030.8360.814msc+pls-da0.9570.9620.926snv+pls-da0.9430.9450.9291-d+pls-da0.6980.9540.929s-g+pls-da0.9570.9330.892
msc+opls-da0.9550.9410.912snv+opls-da0.9470.9600.9391-d+opls-da0.6980.9500.927s-g+opls-da0.9310.8320.812
60.图7为大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌及其两两混合菌的opls-da模型效果图,从图中可以看出不仅大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌三种细菌得到了准确区分,且他们两种菌的混合菌种都分布在对应两种菌的中间,证实了我们的判别模型分析方法不仅可以用于区分单一菌种,也可以用于分析混合菌种。
61.图8为大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌三类细菌及他们三者混合细菌的opls-da模型效果图,从图中可以看出不仅大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌三种细菌得到了准确区分,且他们三种菌的混合菌种样本都分布在对应三种菌的中间,进一步证实了我们的判别模型分析方法不仅可以用于区分单一菌种,也可以用于分析三元混合菌种。
62.图9显示了聚类分析(hca)的模型效果。横坐标为大肠埃希氏菌、金黄色葡萄球菌、鼠伤寒沙门氏菌、铜绿假单胞菌和枯草芽孢杆菌5种细菌样本,编号分别为a,b,c,d,e,每种细菌随机采集45条光谱数据,共得到225条光谱数据(由于横坐标长度有限,不能完全显示225个样本)。运用hca方法对得到的225条光谱数据样本进行聚类分析。在此图中,集群的高度与集群之间的距离成正比。即当垂直线很高时,集群之间的距离很远,而当垂直线很短时,集群之间的距离很近。当hca树状图将样本分为为5类时,组1中包含a与b两种细菌,组2中包含a与d两种细菌,组3中包含a、c、d三种细菌,组4包含d与e两种细菌,组5中包含a、b、e、c四种细菌。可以明显看出,hca方法对单一细菌的分类效果很差,可想而知,对混合菌的分析效果就更无法保证了。而我们本发明中所建立的鉴别模型效果较好,对几类单一细菌和混合细菌都能进行很好的鉴别。
63.实施例5基于plsr的定量分析模型的建立
64.通过随机取样将光谱划分为校正集和预测集,按照4:1的比例划分,得到对应的校正集与预测集。以不同比例的大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌三类细菌的混合细菌作为混合模型样本,通过不同预处理方式对混合菌的原始光谱数据进行处理,并建立了混合样本中三种菌对应的plsr定量模型。以均方根误差(rmse)和相关系数(r2)作为模型评价指标,不同预处理方法与plsr模型结合的检测性能见表2。通过对比发现:采用rc预处理后,金黄色葡萄球菌的定量模型效果最好,plsr的校正集相关系数高达0.9169,且其对应的rmse值为0.0787,预测集相关系数达到了0.9273,rmse为0.0749,同组最低,说明预测值与实际值有良好的相关性,且模型的稳定性较好。而在大肠埃希氏菌的定量模型中,采用snv作为预处理方法的定量模型效果最好,plsr的校正集相关系数为0.8598,rmse为0.1003,预测集相关系数也达到了0.8790,rmse为0.0965,同组最低,表明预测值与实际值有良好的相关性。在鼠伤寒沙门氏菌的定量模型中,采用rc预处理后的plsr定量效果最好,校正集相关系数达0.8963,rmse为0.0899,预测集相关系数为0.8936,rmse为0.0899,说明预测值与实际值有良好的相关性。
65.表2不同预处理方法与plsr结合模型效果对比
[0066][0067][0068]
实施例6基于波段选取的plsr模型的优化
[0069]
以不同比例的大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌三类细菌的混合细菌作为混合模型样本,选取不同菌种最佳光谱预处理方法,考察vip、spa、cars三种波段预处理方法对plsr模型效果的影响,并与全波段数据建立的模型进行对比分析。同样以rmse和r2作为模型评价指标,不同波段筛选方法与plsr模型结合的检测性能见表3。
[0070]
表3不同波段筛选方法与plsr结合模型效果对比
[0071][0072]
对比表2和表3可以看出,对于大肠埃希氏菌和鼠伤寒沙门氏菌,经过cars波段筛选处理后,所得到的plsr模型准确率更高。在大肠埃希氏菌的模型中,预测集中r2和rmse分别为0.8825和0.0951;在鼠伤寒沙门氏菌的模型中,预测集中r2和rmse分别为0.9087和0.0834。但是对于金黄色葡萄球菌,经过这三种波段筛选后,plsr的模型效果都没有表3中全波段数据得到的模型效果好,因此要结合细菌光谱数据合理选择优化方法。图10显示的是由实施例5和6中得出的三种细菌的最优plsr模型关系图。
[0073]
实施例7基于ann的定量分析模型的建立
[0074]
通过随机取样将光谱划分为校正集和预测集,按照4:1的比例划分得到对应的校正集与预测集。以不同比例的大肠埃希氏菌、金黄色葡萄球菌和鼠伤寒沙门氏菌三类细菌的混合细菌作为混合模型样本进行ann定量模型研究。使用python3.9搭建ann神经网络模型。输入节点数为波段数总目1453,输出节点为1,模型选择relu函数激活隐含层,最后一次激活函数为linear,其他超参数设置如下:隐层层数设置1,2,3,4;每个隐层结点数设置16,32,48;学习率设置为0.1,0.01,0.001,0.0001;训练回合数设置为100,200,300,400,500。根据验证集rmse选择最佳网络模型。通过绘制隐藏层与学习率和隐藏节点数与训练回合数的四维热图,通过热图颜色变化找到rmse最小的超参数值,来筛选最优的人工神经网络定量分析模型。图11为金黄色葡萄球菌的热图分析图,通过颜色判断最小的rmse值为0.0352,
此时对应的隐层数为3,每个隐层结点数为48,学习率为0.001,回合数为400时,模型效果最佳。同理,依据此法找到大肠埃希氏菌的最小的rmse值为0.0382,此时对应的隐层数为4,每个隐层结点数为48,学习率为0.001,回合数为400时,模型效果最佳。鼠伤寒沙门氏菌的最小的rmse值为0.0398,此时对应的隐层数为3,每个隐层结点数为32,学习率为0.001,回合数为400时,模型效果最佳。图12显示的是经过热图分析得出的三种细菌的最优ann模型关系图。可以看出与图10相比,在相同浓度样本中,ann模型效果比plsr的定量模型效果要好,说明ann模型处理拉曼光谱数据的优势可能更大。
[0075]
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1