基于半监督学习的脑部CT图像出血区域分割方法及系统与流程
本发明涉及机器学习和图像处理领域,尤其涉及一种基于半监督学习的脑部CT图像出血区域分割方法及系统。
背景技术:
颅内出血(ICH)是最严重的急性脑血管疾病中的一种,同时也是急性神经紊乱疾病,如偏瘫等的重要发病诱因。因此,对于临床治疗来说,颅内出血的早期诊断具有重要意义。同临床表现相比,可以进行无创伤检测的计算机断层(CT)扫描和磁共振成像(MRI)扫描可以更直接、更准确地反映颅内出血的严重程度和演化趋势。同时又因为CT检测的费用要比MRI检测的费用少得多,所以大多数人患者会选择CT检测的方式。在CT图像中新鲜血肿通常显示为边界模糊的高亮度区域。通常情况下,血肿的形状为肾形,圆形或不规则形,并常常被低密度水肿包围。
现在的出血区域检测方法主要集中于模糊C均值聚类(FCM)或以规则为基础的区域分类等算法。然而,这些方法有两个缺点。首先,这些方法中的大多数使用了非常简单的分割算法,诸如聚类和阈值等,虽然这些方法可能在自然图像处理过程中表现良好,但在复杂的情况下,如出血区域与脑组织重叠或出血的边缘没有足够的辨别度时,这些方法的效果并不好。其次,现有的算法大多仅适用于处理二维图像。但CT成像是一个三维的过程,因此会产生一系列的平行扫描图像帧,而2维分割算法会忽略掉一些重要的帧间信息。但使用机器学习以及3D分割的方法可以避免这些问题,增强对复杂情况的处理能力,更好的利用这些在2D方法中被忽视了的帧间信息。
技术实现要素:
本发明的目的在于针对目前医学图像分割领域内现有技术的不足,提供了一种基于半监督学习的脑部CT图像出血区域分割方法及系统。本发明运行效率高,对CT图像中的噪声、伪影有较强的鲁棒性,分割得到的结果准确率高。
为实现上述技术目的,本发明采用的技术方案如下:一种基于半监督学习的脑部CT图像出血区域分割方法,包含步骤1:训练Tri-training模型和步骤2:基于Tri-training模型的出血区域分割;
所述步骤1,Tri-training模型训练阶段包括以下步骤:
(1.1)转换CT图像格式:从计算机断层扫描设备或者数据库中获取包含出血区域的CT图像序列,截取像素值的有效区间,转换成常用的bmp或jpg图像处理格式。
(1.2)标记训练样本:将CT图像序列分为两部分,一部分序列作为有标记样本集,另一部分作为无标记样本集,针对有标记样本,手动标出出血区域,其中出血区域标记为1,其余部分标记为0。
(1.3)图像三维重建:将CT图像序列重建到三维区间,通过三维滤波去除噪声,得到三维矩阵。
(1.4)超体素分割:对重建得到的三维矩阵,应用三维简单线性迭代聚类算法(3DSLIC)对其进行分割,得到规则排列的超体素。该步骤包括以下子步骤:
(1.4.1)计算三维矩阵中的体素总数N,确定要划分的超体素个数K,计算超体素的初始边长以Ns为步长在三个维度上均匀抽样,作为初始的聚类中心Ck=[gk,xk,yk,zk]T,其中,gk为第k个聚类中心的灰度值,xk,yk,zk为第k个聚类中心的位置坐标。
(1.4.2)在以聚类中心点为中心的3×3×3邻域范围内,选取梯度最小点作为新的聚类中心点,梯度G(x,y,z)计算方法如下:
G(x,y,z)=[g(x+1,y,z)-g(x-1,y,z)]2+[g(x,y+1,z)-g(x,y-1,z)]2+[g(x,y,z+1)-g(x,y,z-1)]2其中,g(x+1,y,z)表示坐标(x+1,y,z)处的像素值,g(x-1,y,z)等同理。
(1.4.3)初始化体素标签l(i)=-1,体素到聚类中心的距离d(i)=+∞,相邻的两次聚类中心的差异阈值为threshold;
(1.4.4)以每个聚类中心点Ck为中心,在2Ns×2Ns×2Ns的邻域范围内计算体素i到聚类中心Ck的距离D(i,Ck),其中p,q为调解参数,gi,xi,yi,zi分别为体素i的像素值和三维坐标。
如果D(i,Ck)≤d(i),令体素的标签l(i)=k,体素到聚类中心的距离d(i)=D(i,Ck)。
(1.4.5)对每个聚类中心点的邻域都计算完距离后,根据体素标签计算新的聚类中心点Ck(new):
其中,Nk表示属于第k个聚类中心的体素的总个数。
(1.4.6)计算新聚类中心和原聚类中心之间的差异E:
更新聚类中心Ck=Ck(new),如果差异E≤threshold,结束循环,反之,重复步骤(1.4.4)到步骤(1.4.6),直到差异E≤threshold。
(1.4.7)对于有标签体素集,统计每个超体素内的体素标签,选择体素最多的标签作为整个超体素的标签。
(1.5)提取特征:对每个超体素,提取超体素的灰度直方图作为特征。灰度直方图的统计范围为[Gmedian-40,Gmedian+80],特征总数为40。其中Gmedian表示CT图像中脑部区域的灰度中值,由统计得到。
(1.6)训练半监督模型:用有标签样本和无标签样本一起训练构成tri-training模型的三个不同类型的分类器。该步骤包括以下子步骤:
(1.6.1)对有标记样本集进行可重复取样以获得三个有标记训练样本集,三个训练样本集分别用来训练产生一个分类器,这里的三个分类器分别为人工神经网络(ANN),支持向量机(SVM)和随机森林(RF)。其中ANN分类器是一个标准的三层神经网络,隐层节点数为20,激活函数为sigmoid函数。SVM分类器是通过LIBSVM工具箱实现的,核函数为高斯径向基函数,参数C设定为1。RF分类器的树数量为100。
(1.6.2)三个分类器分别对无标记样本集进行标注,如果两个分类器对同一个未标记样本的标签预测相同,用该标签标记这个样本,然后将其加入第三个分类器的有标记训练样本集;
(1.6.3)用更新过的有标记训练样本集再次训练三个分类器;
(1.6.4)重复步骤(1.6.2)、(1.6.3)直到分类器的参数不再发生变化。
所述基于Tri-training模型的出血区域分割阶段包括以下步骤:
(2.1)转换CT图像格式:对于从计算机断层扫描设备中导出的包含出血区域的CT图像序列,截取像素值的有效区间,转换成常用的计算机图像处理格式。
(2.2)图像三维重建:将CT图像重建到三维区间,通过三维滤波去除噪声,得到三维矩阵。
(2.3)超体素分割:对重建得到的三维矩阵,应用三维简单线性迭代聚类算法(3DSLIC)对其进行分割,得到规则排列的超体素。该步骤具体方法同上一阶段的超体素分割步骤(1.4.1)到步骤(1.4.6)。
(2.4)提取特征:对每个超体素,提取超体素的灰度直方图作为特征。灰度直方图的统计范围为[Gmedian-40,Gmedian+80],特征总数为40。
(2.5)分类样本:用步骤(1.1)到(1.6)中训练好的tri-training分类器分类超体素,即为每一个超体素分配一个标签,其中出血区域标签ti=1,其他区域标签ti=0。
(2.6)三维重建:将所有ti=1的超体素在三维空间进行重建,通过去噪,平滑等处理得到出血区域的三维显示,实现脑部CT图像出血区域的分割。
一种基于半监督学习的脑部CT图像出血区域分割系统,包括图像预处理模块,超体素划分模块,特征提取模块,分类模块和三维重建模块。所述图像预处理模块对二维CT图像序列进行格式转换,简单图像处理并将二维图像存储到三维矩阵。所述超体素划分模块将三维矩阵划分为超体素。所述特征提取模块包括计算灰度直方图模块和计算标签模块。所述分类模块包括训练分类器模块和分类样本模块,在训练过程中用于训练分类器,在实际应用过程中将超体素分为前景部分和背景部分。所述三维重建模块将属于前景的超体素在三维空间进行重建。
进一步地,所述图像预处理模块从计算机断层扫描设备或者数据库中获取包含出血区域的CT图像,截取像素值的有效区间,转换成常用的计算机图像处理格式,并将二维图像序列存储到三维矩阵。
进一步地,所述超体素划分模块应用三维简单线性迭代聚类算法(3DSLIC)对三维矩阵进行分割,得到规则排列的超体素作为样本。
进一步地,所述特征提取模块包括计算灰度直方图模块和计算标签模块:计算灰度直方图模块提取超体素的灰度直方图作为超体素的特征;计算标签模块用于训练过程中计算样本的标签。
进一步地,所述特征分类模块包括训练分类器模块和分类样本模块:训练分类器模块在tri-training模型下,用样本训练人工神经网络(ANN),支持向量机(SVM)和随机森林(RF)三个分类器;分类样本模块根据超体素特征,运用训练好的三个分类器组合而成的tri-training模型分类超体素。
进一步地,所述三维重建模块将分类得到的出血区域超体素重建到三维空间并显示。
本发明的有益效果是:
1、CT图像的前期处理方法简单,不需要像常规算法一样提取颅内结构。
2、灰度直方图作为超体素特征在可区分性的前提下简单易提取。
3、加入超体素划分模块减小了孤立的噪声点对分割的影响,增强了算法的鲁棒性的同时大幅度降低了分类算法的运算数据量。
4、三个不同分类器的引入增强了tri-training分类模型的分类准确率。
5、tri-training分类模型充分利用了少量有标记数据和大量无标记数据。
6、本发明通过将CT图像从二维空间转换到三维空间,充分利用了CT图像之间的帧间信息。
附图说明
图1为本发明方法在一种实施方式中的流程图;
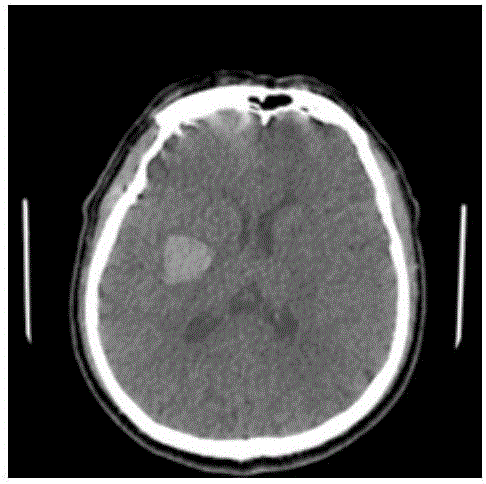
图2为格式转换后得到的二维CT图像;
图3为CT图像划分为超体素后截取的二维图像;
图4为出血区域中某个超体素的灰度直方图;
图5为背景区域中某个超体素的灰度直方图;
图6为分割得到的出血区域的二维截图;
图7为分割得到的出血区域的三维图像;
图8为本发明系统在一种实施方式中的结构示意图;
图9为本发明系统中图像预处理模块的结构示意图;
图10为本发明系统中图像预处理模块的结构示意图。
具体实施方式
本发明适用于医学颅脑CT图像中的出血区域分割,是一种基于半监督学习和三维超体素的脑部CT图像出血区域分割方法。
本发明流程图如图1,主要包括包含Tri-training模型训练阶段和基于Tri-training模型的出血区域分割阶段。
其中Tri-training模型训练阶段包括以下步骤:
(1.1)转换CT图像格式:从计算机断层扫描设备或者数据库中获取包含出血区域的CT图像序列,截取像素值的有效区间,转换成常用的计算机图像处理格式。图2即为CT图像转换格式后得到的图像。
(1.2)标记训练样本:将CT图像序列分为两部分,一部分序列作为有标记样本集,另一部分作为无标记样本集,针对有标记样本,手动标出出血区域,其中出血区域标记为1,其余部分标记为0。
(1.3)图像三维重建:将CT图像序列重建到三维区间,通过三维滤波去除噪声,得到三维矩阵。
(1.4)超体素分割:对重建得到的三维矩阵,应用三维简单线性迭代聚类算法(3DSLIC)对其进行分割,得到规则排列的超体素。该步骤包括以下子步骤:
(1.4.1)计算三维矩阵中的体素总数N,确定要划分的超体素个数K,计算超体素的初始边长以Ns为步长在三个维度上均匀抽样,作为初始的聚类中心Ck=[gk,xk,yk,zk]T,其中,gk为第k个聚类中心的灰度值,xk,yk,zk为第k个聚类中心的位置坐标。
(1.4.2)在以聚类中心点为中心的3×3×3邻域范围内,选取梯度最小点作为新的聚类中心点,梯度G(x,y,z)计算方法如下:
G(x,y,z)=[g(x+1,y,z)-g(x-1,y,z)]2+[g(x,y+1,z)-g(x,y-1,z)]2+[g(x,y,z+1)-g(x,y,z-1)]2其中,g(x+1,y,z)表示坐标(x+1,y,z)处的像素值,g(x-1,y,z)等同理。
(1.4.3)初始化体素标签l(i)=-1,体素到聚类中心的距离d(i)=+∞,相邻的两次聚类中心的差异阈值为threshold;
(1.4.4)以每个聚类中心点Ck为中心,在2Ns×2Ns×2Ns的邻域范围内计算体素i到聚类中心Ck的距离D(i,Ck),其中p,q为调解参数,gi,xi,yi,zi分别为体素i的像素值和三维坐标。
如果D(i,Ck)≤d(i),令体素的标签l(i)=k,体素到聚类中心的距离d(i)=D(i,Ck)。
(1.4.5)对每个聚类中心点的邻域都计算完距离后,根据体素标签计算新的聚类中心点Ck(new):
其中,Nk表示属于第k个聚类中心的体素的总个数。
(1.4.6)计算新聚类中心和原聚类中心之间的差异E:
更新聚类中心Ck=Ck(new),如果差异E≤threshold,结束循环,反之,重复步骤(1.4.4)到步骤(1.4.6),直到差异E≤threshold。图3为划分为超体素后的二维截图。
(1.4.7)对于有标签体素集,统计每个超体素内的体素标签,选择体素最多的标签作为整个超体素的标签。
(1.5)提取特征:对每个超体素,提取超体素的灰度直方图作为特征。灰度直方图的统计范围为[Gmedian-40,Gmedian+80],特征总数为40。其中Gmedian表示CT图像中脑部区域的灰度中值,由统计得到。图4,5为从前景和背景中随机抽取的两个超体素的灰度直方图。
(1.6)训练半监督模型:用有标签样本和无标签样本一起训练构成tri-training模型的三个不同类型的分类器。该步骤包括以下子步骤:
(1.6.1)对有标记样本集进行可重复取样以获得三个有标记训练样本集,三个训练样本集分别用来训练产生一个分类器,这里的三个分类器分别为人工神经网络(ANN),支持向量机(SVM)和随机森林(RF)。其中ANN分类器是一个标准的三层神经网络,隐层节点数为20,激活函数为sigmoid函数。SVM分类器是通过LIBSVM工具箱实现的,核函数为高斯径向基函数,参数C设定为1。RF分类器的树数量为100。
(1.6.2)三个分类器分别对无标记样本集进行标注,如果两个分类器对同一个未标记样本的标签预测相同,用该标签样本这个标记,然后将其加入第三个分类器的有标记训练样本集;(1.6.3)用更新过的有标记训练样本集再次训练三个分类器;
(1.6.4)重复步骤(1.6.2)(1.6.3)直到分类器的参数不再发生变化。
Tri-training模型的出血区域分割阶段包括以下步骤:
(2.1)转换CT图像格式:对于从计算机断层扫描设备中导出的包含出血区域的CT图像序列,截取像素值的有效区间,转换成常用的计算机图像处理格式。
(2.2)图像三维重建:将CT图像重建到三维区间,通过三维滤波去除噪声,得到三维矩阵。
(2.3)超体素分割:对重建得到的三维矩阵,应用三维简单线性迭代聚类算法(3DSLIC)对其进行分割,得到规则排列的超体素。该步骤具体方法同上一阶段的超体素分割步骤(4.1)到(4.6)。
(2.4)提取特征:对每个超体素,提取超体素的灰度直方图作为特征。灰度直方图的统计范围为[Gmedian-40,Gmedian+80],特征总数为40。
(2.5)分类样本:用训练好的tri-training分类器分类超体素,即为每一个超体素分配一个标签,出血区域标签ti=1,其他区域标签ti=0。图6为分割后得到结果的二维截图。
(2.6)三维重建:将所有ti=1的超体素在三维空间进行重建,通过去噪,平滑等处理得到出血区域的三维显示,实现脑部CT图像出血区域的分割。图7为实例1三维的分割结果。
本发明系统模块结构图如图8,包括图像预处理模块,超体素划分模块,特征提取模块,分类模块和三维重建模块。其中图像预处理模块对二维CT图像序列进行格式转换,简单图像处理并将二维图像存储到三维矩阵。超体素划分模块将三维矩阵划分为超体素。所述特征提取模块包括计算灰度直方图模块和计算标签模块。所述分类模块包括训练分类器模块和分类样本模块,在训练过程中用于训练分类器,在实际应用过程中将超体素分为前景部分和背景部分。三维重建模块将属于前景的超体素在三维空间进行重建。
图像预处理模块从计算机断层扫描设备或者数据库中获取包含出血区域的CT图像,截取像素值的有效区间,转换成常用的计算机图像处理格式,并将二维图像序列存储到三维矩阵。
超体素划分模块应用三维简单线性迭代聚类算法(3DSLIC)对三维矩阵进行分割,得到规则排列的超体素作为样本。
如图9所示,特征提取模块包括以下子模块:
计算灰度直方图模块:提取超体素的灰度直方图作为超体素的特征。
计算标签模块:训练过程中用于计算样本的标签。
如图10所示,分类模块包括以下子模块:
训练分类器模块:在tri-training模型下,用样本训练人工神经网络(ANN),支持向量机(SVM)和随机森林(RF)三个分类器
分类样本模块:根据超体素特征,运用训练好的三个分类器组合而成的tri-training模型分类超体素。
三维重建模块将分类得到的出血区域超体素重建到三维空间并显示。
- 还没有人留言评论。精彩留言会获得点赞!