基于句法树和领域特征的生物文本蛋白质指代消解方法与流程
本发明属于文本挖掘
技术领域:
,具体涉及基于句法树和领域特征的生物文本蛋白质指代消解方法。
背景技术:
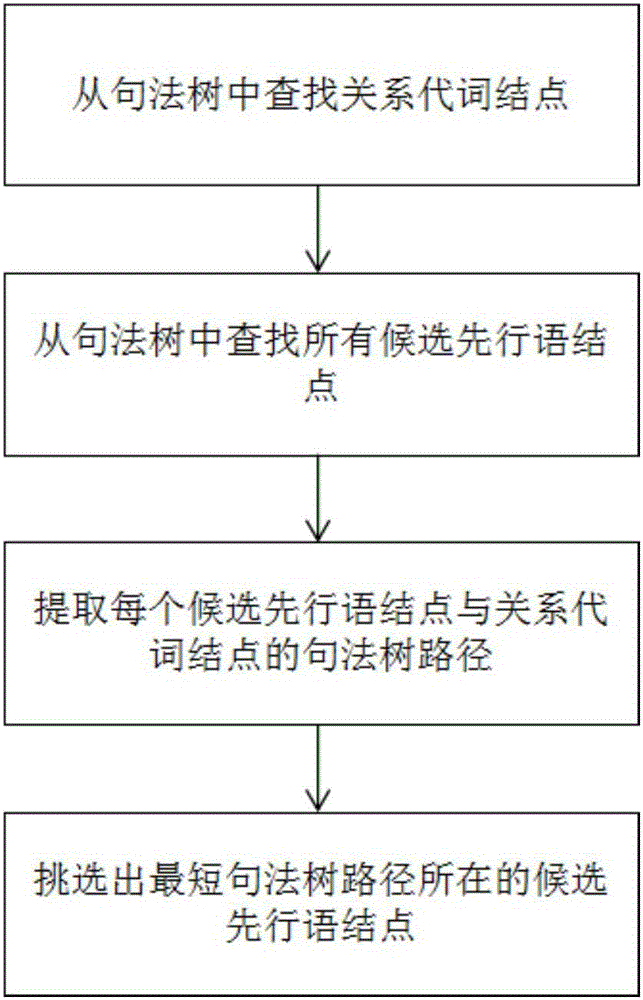
:随着计算机和互联网技术的迅速发展,大量的信息和文献以数字化的形式存在。生物医学领域内的文献已海量存在并且正以指数形式增长,生物医学相关研究学者使用传统的人工阅读模式在庞大的文献面前难以高效的获取有价值的信息,因此自动化的文本信息提取成为一项有意义的工作。作为生物医学领域文本信息提取的一项任务,蛋白质指代消解是找出生物文本中指示着相同蛋白质实体的词和短语,照应语是具有指代作用的对象,例如常见的代词,先行语是被指向的、具有实际内容的对象,例如蛋白质和生物实体,蛋白质指代消解作为一项辅助技术,对生物文本挖掘的许多任务起到了重要的支撑作用,能有效的改善生物信息提取系统的性能。生物自然语言处理任务BioNLPSharedTask2011提供了标准的生物医学领域的蛋白质指代消解语料Coreference,该语料来源于MEDLINE中的摘要,主要解决生物医学文本中的蛋白质实体的指代消解,语料中的蛋白质实体已预先标注出。通用领域指代消解相比于生物医学领域指代消解的研究方法更成熟,但是由于特定领域语料的独特性,这些方法直接移植到生物医学领域中并不能取得很好的效果,开发针对生物文本蛋白质指代消解的方法很有必要。目前在生物文本的蛋白质指代消解中,主要分为基于监督学习的方法、基于规则的方法和融合的方法。基于监督学习的方法通过从蛋白质指代消解训练数据集中提取特征来学习得到一个模型,然后使用这个模型对新数据中的蛋白质指代关系进行消解处理,如YoungjunKim等人2011年在BioNLP任务发表的论文“TheTamingofReconcileasaBiomedicalCoreferenceResolver”中,公开了一种基于特征和分类器模型的监督学习方法用于蛋白质指代消解,该方法提取了词汇特征、句法特征等一系列特征,然后使用一个分类器进行蛋白质指代消解的处理。基于规则的方法通过手工制定的一系列规则处理生物文本中的蛋白质指代消解,如MakotoMiwa等人2012年在杂志《Bioinformatics》第28卷13期发表的论文“Boostingautomaticeventextractionfromtheliteratureusingdomainadaptationandcoreferenceresolution”中,公开了一种基于规则匹配的蛋白质指代消解方法,该方法使用一个照应语候选检测子提取名词短语、代词等作为照应语候选者,然后使用一个先行语候选检测子提取名词短语作为先行语候选者,最后使用一个指代关系连接检测子按照完全匹配、结构匹配、严格的头词匹配、放松的头词匹配、数量匹配等规则的顺序,挑选每个照应语的最优先行语。融合的方法通过同时使用监督学习和规则,对不同类型的蛋白质指代消解采用不同的方法来处理,如JenniferD'Souza和VincentNg于2012年在ACM-BCB会议发表的论文“AnaphoraResolutioninBiomedicalLiterature:AHybridApproach”中,公开了一种基于分类器和规则匹配的蛋白质指代消解方法,该方法使用学习过的分类器处理代词的蛋白质指代消解,使用规则的方法处理名词短语的蛋白质指代消解。蛋白质指代消解方法的性能通常用召回率、准确率和F值来评价,召回率指所提取出正确的蛋白质指代关系和数据中所有蛋白质指代关系的比率,准确率是提取出正确的蛋白质指代关系与所有提取结果的比率,F值是召回率和准确率的调和平均值,作为最终的综合指标。针对生物文本蛋白质指代消解的方法中,基于监督学习的方法需要利用大量的人工标注语料训练才能取得较为理想的结果,但是获取大量人工标注语料需要付出很大的人力物力和很长的时间,当前已有数据并不能满足这样的要求,导致在数量有限的训练数据下监督学习方法得到的模型不够好,模型的召回率很低,从而导致F值很低;基于规则的方法通常只借鉴了通用领域指代消解的一些规则,这些规则只从短语或词的结构和属性方面来考虑,未考虑生物文本独特的句法特性和领域特征,不能有效提取出正确蛋白质指代关系,方法的召回率低从而导致F值低;融合的方法仍然具有监督学习方法中由于训练数据有限导致的模型不够理想,代词类型蛋白质指代消解召回率低的问题,方法整体的召回率虽有改善但仍然不够,使得F值不够理想。当前蛋白质指代消解的结果还需要继续改善,从而才能更有效的为生物文本挖掘其他任务,如生物事件提取,做好前期预处理工作。技术实现要素:本发明的目的在于克服上述现有技术存在的缺陷,提出了一种基于句法树和领域特征的生物文本蛋白质指代消解方法,能够提取出正确蛋白质指代关系,从而提高综合指标F值。为实现上述目的,本发明采取的技术方案为:包括如下步骤:(1)对原始文本进行分句、分词、词性标注、词形还原和句法分析,得到每个句子的句法树Ti,i=1,2,...,N,所有句子的句法树构成句法树集其中i表示句子的序号,N表示所有句子的个数;(2)从句法树Ti中查找关系代词结点和距离该关系代词结点最近的名词短语结点,得到关系代词照应语Mr和关系代词照应语Mr的先行语Ar;(3)从句法树Ti中查找人称代词结点,得到人称代词照应语Mp;并从该人称代词结点所在句法树的并列短语结构、子句句法树或前一句子的句法树中查找该人称代词照应语Mp的先行语Ap;(4)从句法树Ti中查找包含特定生物实体类型关键词的限定性名词短语结点,得到限定性名词短语照应语Md;从句法树集T的子集中查找所有包含生物实体或蛋白质实体的名词短语结点,得到候选先行语集X,基于生物领域特征性质从候选先行语集X中得到限定性名词短语照应语Md的先行语Ad;其中Tj为句法树集T中第j个句子的句法树,k为句子窗口的大小;(5)从步骤(2)至步骤(4)得到的所有指代消解结果中过滤掉先行语不包含蛋白质实体的指代消解,完成基于句法树和领域特征的生物文本蛋白质指代消解。进一步地,步骤(2)中所述的从句法树Ti中查找关系代词结点和距离该关系代词结点最近的名词短语结点,实现步骤为:201、从句法树Ti中查找标记为“WDT”或“WP”的结点,得到关系代词结点Nr和关系代词照应语Mr,从句法树Ti中查找标记为“NP”的所有结点,得到候选先行语集Z;其中WDT代表以wh开头的限定语,WP代表以wh开头的代词,NP表示名词短语;202、从句法树Ti中查找候选先行语集Z的所有候选先行语所在结点,得到候选先行语结点集Nz;203、提取出候选先行语结点集Nz中每个候选先行语结点与关系代词结点Nr的句法树路径;204、从步骤203得到的所有句法树路径中挑选出最短的句法树路径,并以该最短句法树路径所在的名词短语结点作为最近的名词短语结点。进一步地,步骤(3)中先行语Ap的获得具体为:301、以人称代词照应语Mp所在结点为起点,在句法树Ti中自底向上遍历,查找包含并列短语结构的结点Nc,判断结点Nc是否存在,若是,在句法树Ti中提取以结点Nc为根结点的句法子树STc,并在句法子树STc中查找距离人称代词照应语Mp所在结点最远的名词短语结点,得到人称代词照应语Mp的先行语Ap,否则,执行步骤302;302、以人称代词照应语Mp所在结点为起点在句法树Ti中自底向上遍历,查找出子句结点Ns;提取出以子句结点Ns为根结点的句法子树STs,并在句法子树STs中查找距离人称代词照应语Mp所在结点最远的名词短语结点,判断该名词短语结点是否存在,若是,得到人称代词照应语Mp的先行语Ap,否则,执行步骤303;303、从句法树集T中选择出句法树Ti-1,在句法树Ti-1中以最后一个叶结点为起点自底向上遍历,查找出子句结点Nt;提取以子句结点Nt为根结点的句法子树STt,并在该句法子树STt中查找与人称代词照应语Mp单复数相匹配的所有名词短语结点,得到候选先行语集Y;从候选先行语集Y中选择距离人称代词照应语Mp最远的候选先行语,得到人称代词照应语Mp的先行语Ap。进一步地,步骤(3)中并列短语结构是指并列名词短语、并列动词短语或者并列子句结构。进一步地,步骤(4)中先行语Ad的获得具体为:401、判断限定性名词短语照应语Md的头词是否为“proteins”或“genes”,若是,从候选先行语集X中选择所有包含蛋白质实体的候选先行语,得到新的候选先行语集Xs,并从该新的候选先行语集Xs中,按照头词匹配、包含蛋白质实体数量大于1的顺序,挑选距离限定性名词短语照应语Md最近的候选先行语,得到限定性名词短语照应语Md的先行语Ad,否则,执行步骤402;402、判断限定性名词短语照应语Md是否为复数形式,若是,从候选先行语集X中按照头词匹配、包含生物实体数量大于1、包含蛋白质实体数量大于1的顺序,挑选距离限定性名词短语照应语Md最近的候选先行语,得到限定性名词短语照应语Md的先行语Ad,否则,执行步骤403;403、判断限定性名词短语照应语Md的头词是否为“protein”或“gene”,若是,从候选先行语集X中选择所有包含蛋白质实体的候选先行语,得到新的候选先行语集Xs,并从该新的候选先行语集Xs中,按照头词匹配、包含蛋白质实体数量等于1的顺序,挑选距离限定性名词短语照应语Md最近的候选先行语,得到限定性名词短语照应语Md的先行语Ad,否则,执行步骤404;404、从候选先行语集X中按照头词匹配、包含生物实体数量等于1、包含蛋白质实体数量等于1的顺序,挑选距离限定性名词短语照应语Md最近的候选先行语,得到限定性名词短语照应语Md的先行语Ad。进一步地,步骤(4)中的特定生物实体类型关键词,包括“protein”、“gene”、“factor”、“element”、“receptor”、“complex”和“construct”。进一步地,步骤(4)的生物实体,其识别方法包括:由数字开头,并包含字母;由小写字母开头,并包含大写字母或数字或特殊符号;由大写字母开头,并包含数字或特殊符号;或者由大写字母开头,包含小写字母,并包含大写字母或特殊符号。本发明与现有的技术相比,具有以下优点:本发明对原始文本进行预处理后,从句法树中提取关系代词、人称代词和限定性名词短语,基于句法树确定关系代词和人称代词照应语的先行语,基于生物领域特征确定限定性名词短语照应语的先行语,最后过滤非蛋白质实体的指代消解结果。本发明基于句法树对关系代词照应语和人称代词照应语的先行语进行提取,基于领域特征对限定性名词短语照应语的先行语进行提取,能够挖掘出只从短语或词的结构、属性方面得不到的句法结构信息,从而有效提取出更多的正确蛋白质指代关系。由于充分利用了生物医学文本的句法特性和领域特征,相比于目前基于规则的方法能够在保证准确率的同时得到更多正确的蛋白质指代消解结果,提高了召回率,得到了更好的综合性能指标F值,仿真实验结果也表明了这一点。本发明可用于生物医学文本中指向蛋白质实体的关系代词、人称代词和限定性名词短语等指代消解。【附图说明】图1是本发明的实现流程框图;图2是本发明从候选先行语集Z中,挑选出距离关系代词Mr所在结点最近的候选先行语结点的实现流程框图。【具体实施方式】以下结合附图对本发明作进一步详细描述:参照图1:本发明包括如下步骤:步骤1,原始文本预处理。1a)使用GENIA句子分割工具对原始文本进行分句;1b)使用斯坦福大学CoreNLP工具对文本进行分词、词性标注和词形还原;1c)使用Enju句法分析器对每个句子进行句法分析,并将结果转化成PTB格式,得到每个句子的句法树Ti,i=1,2,...,N,所有句子的句法树构成句法树集其中i表示句子的序号,N表示所有句子的个数。步骤2,基于句法树查找关系代词照应语的先行语Ar。2a)从句法树Ti中查找标记为“WDT”或“WP”的结点,得到关系代词照应语Mr,从句法树Ti中查找标记为“NP”的所有结点,得到候选先行语集Z;其中WDT和WP均为通用标注,WDT代表以wh开头的限定语,WP代表以wh开头的代词,NP表示名词短语;;2b)从候选先行语集Z中,挑选出距离关系代词照应语Mr所在结点最近的候选先行语结点,得到关系代词照应语Mr的先行语Ar,其具体实现步骤如图2所示。2011、从句法树Ti中查找关系代词照应语Mr所在的结点,得到关系代词结点Nr;2012、从句法树Ti中查找候选先行语集Z的所有候选先行语所在结点,得到候选先行语结点集Nz;2013、提取出候选先行语结点集Nz中每个候选先行语结点与关系代词结点Nr的句法树路径;2014、从步骤2013得到的所有句法树路径中挑选出最短的句法树路径,并以该最短句法树路径所在的候选先行语结点作为最近的候选先行语结点,得到关系代词照应语Mr的先行语Ar。步骤3,基于句法树查找人称代词照应语的先行语Ap。3a)从句法树Ti中查找由“they”、“them”、“themselves”、“their”、“its”或具有指代作用的“it”构成的人称代词结点,得到人称代词照应语Mp;3b)以人称代词照应语Mp所在结点为起点,在句法树Ti中自底向上遍历,查找包含并列名词短语、并列动词短语或者并列子句结构的结点Nc;3c)判断步骤3b)的查找结果中是否存在结点Nc,若是,在句法树Ti中提取以结点Nc为根结点的句法子树STc,在句法子树STc中查找标记为“NP”的所有结点,并从这些结点中挑选出距离人称代词照应语Mp所在结点最远的结点,得到人称代词照应语Mp的先行语Ap,否则,执行步骤3d);3d)以人称代词照应语Mp所在结点为起点在句法树Ti中自底向上遍历,查找出标记为“S”的子句结点Ns,并提取出以子句结点Ns为根结点的句法子树STs;在句法子树STs中查找标记为“NP”的所有结点,并从这些结点中挑选距离人称代词照应语Mp所在结点最远的名词短语结点,判断该名词短语结点是否存在,若是,得到人称代词照应语Mp的先行语Ap,否则,执行步骤3e);3e)从句法树集T中选择出句法树Ti-1,在句法树Ti-1中以最后一个叶结点为起点自底向上遍历,查找出标记为“S”的子句结点Nt,并提取以子句结点Nt为根结点的句法子树STt;在句法子树STt中查找标记为“NP”的所有结点,并从这些结点中过滤掉与人称代词照应语Mp单复数不匹配的名词短语结点,由剩余的所有名词短语结点得到候选先行语集Y;从候选先行语集Y中选择距离人称代词照应语Mp最远的候选先行语,得到人称代词照应语Mp的先行语Ap;步骤4,基于生物领域特征查找限定性名词短语照应语的先行语Ad。4a)从句法树Ti中查找含有“DT”标记子结点的所有名词短语结点,并从这些名词短语结点中挑选出含有特定生物实体类型关键词“protein”、“gene”、“factor”、“element”、“receptor”、“complex”或“construct”的名词短语结点,得到限定性名词短语照应语Md;4b)从句法树集T的子集如{Ti-2,Ti-1,Ti}中查找标记为“NP”的所有结点,并从这些结点中,过滤掉不包含生物实体和蛋白质实体的名词短语结点,由剩余名词短语结点得到候选先行语集X,其中Tj为句法树集T中第j个句子的句法树,k为句子窗口的大小;所述的生物实体,其识别方法包括:由数字开头,并包含字母;由小写字母开头,并包含大写字母或数字或特殊符号;由大写字母开头,并包含数字或特殊符号;由大写字母开头,包含小写字母,并包含大写字母或特殊符号;4c)判断限定性名词短语照应语Md的头词是否为“proteins”或“genes”,若是,从候选先行语集X中选择所有包含蛋白质实体的候选先行语,得到新的候选先行语集Xs,并从该新的候选先行语集Xs中,按照头词匹配、包含蛋白质实体数量大于1的顺序,挑选距离限定性名词短语照应语Md最近的候选先行语,得到限定性名词短语照应语Md的先行语Ad,否则,执行步骤4d);4d)判断限定性名词短语照应语Md是否为复数形式,若是,从候选先行语集X中按照头词匹配、包含生物实体数量大于1、包含蛋白质实体数量大于1的顺序,挑选距离限定性名词短语照应语Md最近的候选先行语,得到限定性名词短语照应语Md的先行语Ad,否则,执行步骤4e);4e)判断限定性名词短语照应语Md的头词是否为“protein”或“gene”,若是,从候选先行语集X中选择所有包含蛋白质实体的候选先行语,得到新的候选先行语集Xs,并从该新的候选先行语集Xs中,按照头词匹配、包含蛋白质实体数量等于1的顺序,挑选距离限定性名词短语照应语Md最近的候选先行语,得到限定性名词短语照应语Md的先行语Ad,否则,执行步骤4f);4f)从候选先行语集X中按照头词匹配、包含生物实体数量等于1、包含蛋白质实体数量等于1的顺序,挑选距离限定性名词短语照应语Md最近的候选先行语,得到限定性名词短语照应语Md的先行语Ad;步骤5,从所有指代消解结果中过滤掉先行语不包含蛋白质实体的指代消解;完成基于句法树和领域特征的生物文本蛋白质指代消解。其中指代消解结果表示成对关系,包括:关系代词照应语Mr和其先行语Ar、人称代词照应语Mp和其先行语Ap以及限定性名词短语照应语Md和其先行语Ad。以下通过仿真实验,对本发明的技术效果作进一步说明:1、仿真条件:仿真实验采用生物医学自然语言处理共享任务BioNLP2011Coreference数据集,数据集已预先标注出蛋白质实体。仿真实验在CPU为IntelCore(TM)i7-4720HQ、主频2.60GHz,内存为8G的WINDOWS7系统上用JAVA编程语言进行仿真。2、仿真内容及结果分析:使用本发明与现有的基于规则的方法在Coreference数据集上进行生物文本蛋白质指代消解的仿真,实验结果如下:方法召回率(%)准确率(%)F值(%)基于规则的方法50.462.755.9本发明60.263.862.0综上,本发明基于句法树对关系代词照应语和人称代词照应语的先行语进行提取,基于领域特征对限定性名词短语照应语的先行语进行提取,能够挖掘出只从短语或词的结构、属性方面得不到的句法结构信息,从而提取出更多的蛋白质指代关系。由于充分利用了生物医学文本的句法特性和领域特征,相比于目前基于规则的方法能够在保证准确率的同时得到更多正确的蛋白质指代消解结果,提高了召回率,得到了更好的综合性能指标F值,与现有的方法相比具有一定的优势。本发明用于解决现有基于规则的生物文本蛋白质指代消解方法中存在的F值低的技术问题,对原始文本进行预处理;从句法树中查找关系代词和距离该关系代词最近的名词短语,作为该关系代词的先行语;从句法树中查找人称代词,并从句法树的并列短语结构、子句句法树或前一句子的句法树中查找该人称代词的先行语;利用句法树得到限定性名词短语和候选先行语集,并基于生物领域特征如单复数、实体类型、数量等性质从候选先行语集中挑选出最优的作为先行语;非蛋白质指代消解过滤。由于基于句法树处理关系代词和人称代词的蛋白质指代消解,基于生物领域特征规则处理限定性名词短语的蛋白质指代消解,对不同照应语类型的指代消解采用了不同的处理方法,充分利用了生物医学领域文本特定的句法特性和领域特征,实验结果表明,本发明能够有效提取出蛋白质指代关系,实现了生物文本中的蛋白质指代消解,具有更高的综合指标F值。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1